猕猴桃溃疡病病菌新筛选方法与菌种鉴定
2017-12-01田璨熙宋金秋向晓寒崔丽红
田璨熙,宋金秋,向晓寒,崔丽红
(湘西民族职业技术学院 生物工程系, 湖南 吉首 416000)
猕猴桃溃疡病病菌新筛选方法与菌种鉴定
田璨熙,宋金秋,向晓寒,崔丽红*
(湘西民族职业技术学院 生物工程系, 湖南 吉首 416000)
【目的】研究高效筛选猕猴桃溃疡病致病菌的新方法对湘西地区感染溃疡病的猕猴桃植株进行快速病菌分离与鉴定。【方法】以一年生猕猴桃枝条对不同糖浓度和氮浓度基质耐受情况为依据和柯赫氏法则来验证高效筛选病原菌方案的可行性与提取经筛选得到猕猴桃溃疡病病菌的DNA并扩增16SrDNA进行系统发育树分析。【结果】本实验得到一高碳氮比(C∶N=10∶1)的筛选培养基,其对猕猴桃溃疡病致病菌筛选效率优于目前已报道的筛选基质;经筛选获得溃疡病致病菌为丁香假单胞杆菌猕猴桃致病变种,与目前已报道的丁香假单胞杆菌猕猴桃致病变种亲缘关系较远,有区别于以往的文献报道。【结论】以高碳氮比(C/N)的菌种选择压力用于丁香假单胞杆菌猕猴桃致病菌的筛选是高效的。丁香假单胞杆菌猕猴桃致病菌可能出现新的变异。
猕猴桃溃疡病;丁香假单胞杆菌;筛选;猕猴桃,植物病害
【研究意义】湖南湘西地区的猕猴桃种植面积达50 hm2,占总耕地面积比例较高,红阳猕猴桃为湘西州地区主要经济树种,猕猴桃种植及次级产业为该地区地方经济的发展,农民脱贫致富提供了主要的产业支撑。【前人研究进展】猕猴桃溃疡病自20世纪80年代以来已成为该地区乃至全国猕猴桃种植的高发病[1-5],但有效防治猕猴桃溃疡病的技术应用目前仍属于世界性难题,因此解决猕猴桃溃疡病的防治问题已经成为该区域经济发展直接面临的风险和挑战[6-10]。【本研究切入点】本论文基于上述现状,以研究猕猴桃溃疡病病菌自身特性作为研究起点,首先针对湘西地区猕猴桃溃疡病病原菌筛选分离方法进行改进。通过对病菌高效筛选方法的研究,为后期的防治技术研究提供新的思路。【拟解决的关键问题】 本论文旨在研究猕猴桃溃疡病病原菌高效筛选分离方法的可行性。
1 材料与方法
1.1 基质对猕猴桃枝条生长的影响及分离基质配方设计
依据猕猴桃枝条离体培养条件来设计筛选培养及条件:用不同浓度的蔗糖溶液离体培养猕猴桃植株枝条,浓度分别设置为1 %,2 %,3 %,4 %,5 %,6 %;连续培养15 d后,记录枝条上出芽情况;用不同浓度的NH4NO3生理盐水溶液离体培养猕猴桃植株枝条,浓度分别设置为0.1 %,0.2 %,0.3 %,0.4 %,0.5 %,0.6 %,0.7 %,连续培养15 d后,记录枝条上出芽情况。依据培养结果来确定分离培养基的基质组成,并将该分离培养基与目前报道的猕猴桃溃疡病细菌分离培养基[10-11]作比较。
1.2 病原微生物的分离
收集2-4月份不同猕猴桃溃疡病高发地区的红阳猕猴桃溃疡病枝干部病样共15份,再将采集样品分别经无菌水表面冲洗,20 s,75 %乙醇溶液浸泡,无菌水冲洗3次后,用无菌刀片收集枝干上的病变组织,切碎放入100 mL无菌水中,加入无菌玻璃球,24 ℃振荡培养12 h。取0.1 mL稀释菌液涂布于1号固体培养基,24 ℃条件下恒温培养24 h后观察生长情况,挑取在该培养基培养条件下生长较好的菌落进行转接纯化。将纯化后的菌体接种于未染病的猕猴桃枝干培养7 d,选取新染病的枝干再进行细菌分离纯化,与其原始细菌16SrDNA序列作对比,进行柯赫氏法则(Koch's Rule)验证。
1.3 病原菌16SrDNA序列测定及分析
(1)基因组DNA提取。在菌液离心取出置于研钵,液氮研磨,并将研磨好的菌浆转至1.5 mL离心管中,标记菌名称,加入0.6 mL TE(pH 8.0),用枪头吸打均匀,使菌体充分悬浮。加入250 μl 10 % SDS,轻轻倒转混匀。 加入3 μl 蛋白酶K(20 ng/μl),轻轻混匀,37 ℃水浴1 h。加入150 μl 5 mol/L NaCl,轻轻混匀。 加入150 μl 2 % CTAB,轻轻混匀,65 ℃水浴20 min。12 000 r/min常温离心20 min。小心吸取上清至新的1.5 mL离心管,加入等体积异丙醇,充分混匀,室温放置30 min,12 000 r/min,4 ℃离心10 min。吸掉上清,在吸水纸上空干液体,加750 μl 70 %乙醇,轻弹管壁,使沉淀悬浮并反复颠倒几次,12 000 r/min,4 ℃离心2 min。每管加入30 μl纯化水溶解沉淀(水中加Rnase,终浓度10 ng/μl),用手轻弹管壁,4 ℃溶解过夜。
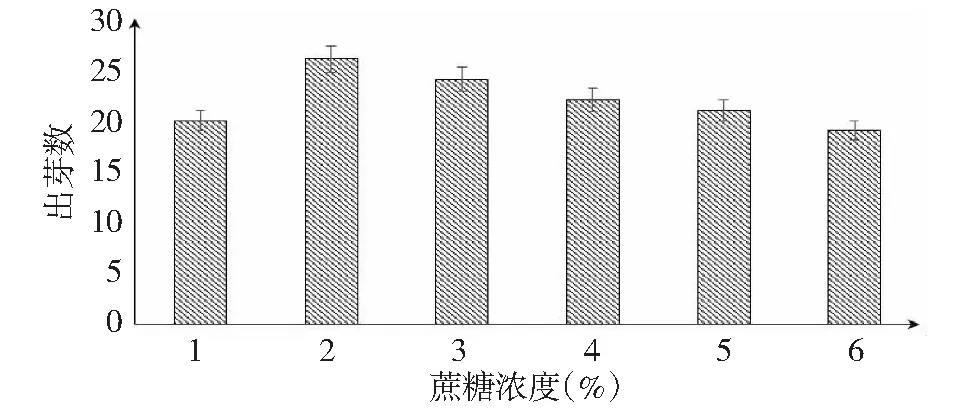
图1 不同蔗糖浓度培养条件下的出芽数Fig.1 The number of cultivate budding in different concentration of sucrose
(2)PCR扩增 16s引物序列。27F: AGAGTTTGATCMTGGCTCAG,1492R:TACGGYTACCTTGTTACGACTT。
反应体系: H2O 17.8 μl ,Buffer 3 μl,d NTP 2 μl,Primer1 3 μl,Primer2 3 μl,DNA模板 1 μl,酶 0.2 μl,总体积 30 μl。
1.4 序列测定与数据分析
PCR 产物直接进行序列测定后用国际基因数据库的比较分析: 将获得的DNA 序列输入Genbank,以blast程序对数据库中的所有序列进行比较分析。将测序结果通过Blast比对,作同源性相似度差异性分析,利用ClustalX1.83软件对序列进行多重比较,采用MEGA7.0软件以N-J法构建系统聚类树,分析其亲缘关系。
2 结果与分析
2.1 猕猴桃枝条离体保存培养效果及分离培养基的选择
如图1~2所示,猕猴桃枝出芽率不会随糖浓度的升高而明显增大,猕猴桃对糖浓度的适应性较好,但猕猴桃枝条的发芽率受铵盐浓度的影响较大。猕猴桃溃疡的发病期大多为初春季,基本符合高碳源的生长环境,可以利用基质碳氮比(C/N)为主要筛选条件来设计分离培养基。经过初步设计,选择以迟效碳源和速效氮源,并使碳氮比达10∶1的高碳氮比(C/N)基质(1号培养基)作为分离条件来对猕猴桃溃疡病病菌进行筛选。表1表明,1号培养基对猕猴桃溃疡病病菌的分离效果优于目前已报道的其它筛选培养基,。
2.2 对不同来源地的红阳溃疡病病菌比较结果
通过对不同来源地的红阳溃疡病病菌按1.2方法进行分离纯化之后,共有2株菌种通过柯赫氏法则的验证且致病力相相同。如图3所示,两菌株经16SrDNA序列测序分析发现其DNA片段大小一致,均为1401 bp。用blast程序对数据库中的所有相似序列进行比较分析,该菌株同丁香假单胞杆菌猕猴桃致病菌(编号AB001439.1)16SrDNA相似率达99 %。因此,结合取样寄主和致病性检测结果,可以确定本试验分离所得菌株为丁香假单胞杆菌猕猴桃致病变种(Pseudomonassyringaepv.actinidae)。
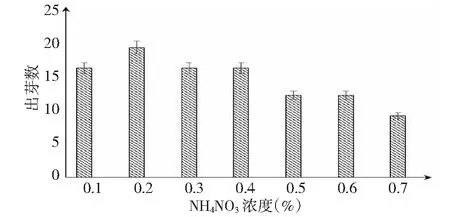
图2 不同NH4NO3浓度培养条件下的出芽数Fig.2 The number of cultivate budding in different concentration of NH4NO3

培养基总菌落数均值(CFU/mL)猕猴桃溃疡病菌落总数均值(CFU/mL)有效菌落数平均比例(%)1号培养基8×1071×10712.5假单胞菌CFC选择培养基4×109未发现未知NA培养基5×1091×1082KB培养基5×1092×1084
2.3 病菌的鉴定与分类
如图3所示,将分离物与丁香假单孢杆菌猕猴桃致病变种相近的物种进行聚类,16SrDNA聚类结果发现分离得到的菌种TC-8与FJ755789.1菌株的亲缘关系最近。而与已报道的丁香假单孢杆菌猕猴桃致病变种(Pseudomonassyringaepv.actinidiae)GQ914994.1、AJ889840.1、JN629035.1、EU906856.1及AB001439.1菌株亲缘关系较远。
3 讨 论
3.1 猕猴桃溃疡病病菌的筛选方法分析
试验采用病理样品多为表面出现褐化脓液的感染后期枝条,杂菌数量较多,一般培养基很难对猕猴桃溃疡病菌进行高效分离,用本试验设计的1号培养基分离目的菌落较为方便。另外,在本试验筛选条件下由于菌落总数较小,不排除病原菌的筛选不完全的情况。

图3 16SrDNA琼脂糖凝胶电泳检测Fig.3 Electrophoesis of PCR reactions of l6SrDNA in agarose gel
目前综合所有已报道猕猴桃溃疡病病菌研究结果可以推论[11-25]:目前猕猴桃溃疡病病菌已存在多菌种致病的情况。
3.2 猕猴桃溃疡病病菌的系统发育树分析
经本试验筛选条件得到的丁香假单胞杆菌猕猴桃致病菌与目前已报道的猕猴桃溃疡病病菌虽同为丁香假单胞杆菌,但菌种在N-J发育树上位于不同分支,可能是由于此丁香假单胞杆菌的传播较快,传播媒介较多,此过程导致丁香假单胞杆菌猕猴桃溃疡病致病菌菌株间出现更多变异,出现进化的迹象,此进化现象的出现应是目前各种防治研究工作制约因素之一。

图4 基于16SrDNA序列的进化树(MEGA 7.0)Fig.4 Phylogenetic tree based on 16SrDNA sequences
4 结 论
本试验采用的筛选培养基为高碳氮比(C/N)的选择性培养基对丁香假单胞杆菌猕猴桃致病菌的筛选效率显著优于目前已报道的相对较低碳氮比(C/N)的筛选培养基(如KB培养基与SPA培养基等)。实验证明合适的高碳氮比筛选条件更有利于的生长,对目标菌种筛选有利,能节省菌种筛选的时间。
[1]Botstein D R, White R L, Skolnick M, et al. Construction of a geneticlinkage map in man using restriction fragment length polymorphism[J].Am J Hum Genet, 1980,32(3): 314-331.
[2]李 淼,李 瑶.猕猴桃溃疡病研究进展[J].安徽农业科学,2002,30(3):91-393.
[3]陈功友,王金生.植物病原细菌致病性决定因子[J].植物病理学报,2002,32(1):1-7.
[4]Tares S, Lemontey J M. Use of species-specific statellite fromBursaphelenchusxylophilusdiagnostic probe[J].Phytopathology, 1994, 84(3):294-298.
[5]Deng S J, Chuji H. Locafion of pathogenic mycoplasma-like organisms in plant tissue in situ hybridization[J]. Proceeding of the Japan Academy, 1991, 67(10): 197-202.
[6]Mas P, Sanchez-Navarro J A, Sanchez-Pina M A, et al.Chemiluminescent and colorigenic detection of Cherry leaf roll virus with digoxigenin-labeled RNA probes[J].Journal of Virological Methods, 1993, 45(1): 93-102.
[7]Opgenorth D C,Lat M,Sorrell M,et al.Pseudomonas canker of kiwifruit [J]. Plant Disease,1983,67(11):1283-1284.
[8]Manulis S, Valinsky L, Liehter A, et al. Sensitive and specific detection ofXanthomonascampestrispv.pelargoniiwith DNA primers and probes identified by random amplified pelymorphie DNA analysis[J].Appl Environ Microbiol, 1994,60(11): 4094-4099.
[9]Koh Y J, Nou I S. DNA markers for identification ofPseudomonassyringaepv.actinidiae[J]. Mol Cells, 2002, 13(2): 309-314.
[10]李 淼,檀根甲,李 瑶,等.不同猕猴桃品种对细菌性溃疡病的抗病性及其聚类分析[J].植物保护,2004,30(5):51-54.
[11]Takikawa Y,Serizawa S,Ichikawa T, et al.Pseudomonassyringaepv.actinidaepv. nov.The causal bacterium of canker of kiwifruit in Japan[J].Ann.Phytopath Soc.Japan, 1989, 55:437-444
[12]Deng S J, Chu H. Location of pathogenic mycoplasma-like organisms in plant tissue in situ hybridization[J]. Proceeding of the Japan Academy, 1991, 67(10): 197-202.
[13]李 瑶,承河元,钱子华.称猴桃溃疡病防治研究[J].安徽农业大学学报,2001,28(2): 139-143.
[14]宋晓斌,王培新,张学武,等.称猴桃细菌性溃疡病生物防治初步研究[J].西北林学院学报,2002, 17(1): 49-50.
[15]盛存波,安德容,鲁燕汉,等.一株抗称猴桃溃疡病的芽抱杆菌分离和筛选研究初报[J].植物保护科学,2005,4(12): 346-348.
[16]Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation [J]. Microbiology Reviews,1995, 59:143-169.
[17]Wise M G, McArther J V, Shimkets L J. Bacterial diversity of a Carolina bay as determined by 16S rRNA gene analysis: Confirmation of novel taxa[J]. Appl. Environ. Microbiol.,1997, 63:1505-1514
[18]Brunel B, Givaudan A, Lanois A, et al. Fast and accurate identification of Xenorhabdus and Photorhabdus species by restriction analysis of PCR-amplified 16S rRNA genes[J]. Appl. Environ. Microbiol.,1997, 63:574-580
[19]Vandamme P,Pot B,Gillis M, et al. MicrobioloAical Reviews, 1996, 60: 407-438.
[20]Harmsen D, Kareh H. 16S rDNA for diagnosing pathogens: aliving tree[J].ASM News,2004,70(19):19-24.
[21]杨 霞,陈 陆,王川庆.16SrRNA基因序列分析技术在细菌分类中应用的研究进展[J].西北农林科技大学学报(自然科学版),2008, 36(2): 55-60.
[22]Kim S B, Yoon J H, Kim H, et al. Int J Svst Bacteriol, 1995, 45:351-356.
[23]Lenoard J, Lascolea J R, Dryyia D. Quantitation of bacteria in cerebrospinal fluid and blood of children and its diagnostic significance[J]. Clin Microbioh, 1984, 19(2): 187.
[24]Paul K, Corkil J E, Hooi P S, et al. Isolation ofWaddliamalaysiensis, a nove lintracellular bacterium, from fruit bat(Eonycterisspelaea)[J]. Emerg Infect Dis,2005,11(2): 271-277.
[25]Tenhaken R,Levine A,Brisson L F,et al.Function of the oxidative burst in hypersensitive disease resistance[J].Proceedings of the National Academy of Sciences of the United States of America,1995,92(10):4158-4163.
(责任编辑 李 洁)
NewScreeningMethodsandIdentificationofKiwifruitCankerPathogen
TIAN Can-xi,SONG Jin-qiu,XIANG Xiao-han,CUI Li-hong*
(Department of Bioengineering, Ethnic Vocational and Technical College, Hunan Jishou 416000, China)
【Objective】A new screening method for kiwifruit pathogenic bacteria was developed and used to isolate and identify pathogenic bacteria of ulcer disease in kiwifruit. 【Method】Based on annual kiwifruit shoots sugar tolerance and nitrogen tolerance results to design pathogen screening program. 【Result】The screening medium with high C/N ratio was obtained, and the screening efficiency of this medium was better than that of other screening media. The pathogenic bacteria were Pseudomonas syringae kiwi pathogens, which have been reported to be distantly related to pathogenic variants ofPseudomonassyringaekiwifruit. The pathogen is a pathogen ofPseudomonassyringaekiwi, which is distantly related to the recently reported pathogen-like variants. 【Conclusion】High-nitrogen ratio (C/N) selection pressure for bacteriaPseudomonassyringaebacteria kiwi screening is efficient.Pseudomonassyringaebacteria kiwi showed possible as a new mutation.
Kiwifruit ulcer disease;Pseudomonassyringae; Screening; Kiwi; Plant diseases
S436.634.1
A
1001-4829(2017)11-2518-04
10.16213/j.cnki.scjas.2017.11.021
2016-12-19
湖南湘西自治州重大科研项目“猕猴桃细菌性溃疡病抗性砧木的选育与研究”(湘西自治州财企指[2016]9号);湖南省教育厅科研项目“猕猴桃溃疡病病菌的拮抗微生物选育研究”(17C1576)
田璨熙(1986-),男,讲师,硕士,主要从事微生物/酶工程与发酵方面研究,*为通讯作者:崔丽红(1974-),女,副教授,硕士, 研究方向:林学。
