蓝鹇源H9N2亚型禽流感病毒HA和NA基因序列分析
2017-11-17王念晨王文清焦培荣李婉萍彭仕明
单 芬 王念晨 王文清 焦培荣 陈 武* 李婉萍 彭仕明
(1.广州动物园,广州,510070;2.华南农业大学,广州,510642)
蓝鹇源H9N2亚型禽流感病毒HA和NA基因序列分析
单 芬1王念晨2王文清2焦培荣2陈 武1*李婉萍1彭仕明1
(1.广州动物园,广州,510070;2.华南农业大学,广州,510642)
为了探查禽流感病毒H9N2在圈养蓝鹇中的流行规律及变异情况,从圈养蓝鹇(Lophuraswinhoii)分离1株H9N2亚型禽流感病毒,对其HA及NA基因进行PCR扩增并进行序列分析。本分离株的HA基因序列与Genbank中广西鸡源2014年分离株A/chicken/Guangxi/CLB02/2014(H9N2)(KT023046)的核苷酸序列相似性最高,为99%;而NA基因与登录号为KM455874,2013人源分离株A/Lengshuitan/11197/2013(H9N2)的NA核苷酸序列相似性最高,为97%。HA蛋白序列分析显示:HA的226位氨基酸残基为亮氨酸(Leu),具有哺乳动物SAα2-6受体结合的特性,裂解位点处的基序PSRSSR↓GL,明显发生1个碱性氨基酸的变异。HA蛋白具有9个潜在N-糖基化位点,分别位于29、82、141、218、298、305、313、492、551位;HA受体结合位点氨基酸变异明显。NA蛋白序列分析显示:NA蛋白颈部63~65位氨基酸缺失,具有7个潜在的糖基化位点,分别位于69、86、146、200、234、264、368位;NA蛋白红细胞结合位点、抗原决定簇区域氨基酸变异活跃。HA、NA基因进化树分析显示该分离株属于欧亚分支中Y280-Like小进化分支,与疫苗株虽属于同一进化分支中,但亲缘关系较远。蓝鹇源H9N2亚型禽流感病毒(LS/GD/P29/16)HA和NA基因发生一定程度的变异,需要加强流行病学监测。
禽流感病毒;H9N2亚型;序列分析;进化树
H9N2亚型禽流感病毒(Avian influenza virus,AIV)最早于1966年从美国威斯康辛州的一个火鸡养殖场中被分离到(A/turkey/Wisconsin/1/1966),虽然表现低致病性,但由于传播广泛、能造成免疫抑制并使宿主容易发生继发感染。自20世纪90年代以后其传播越来越广泛,并在亚洲、中东和北非的许多国家形成地方流行[1-2]。在我国,H9N2亚型AIV从1992年开始在部分省区局部流行,并首次从人源分离到。1998年以来该亚型AIV已在全国大部分地区形成地方流行。经历了多年的持续进化,H9N2亚型AIV的流行特点和生物学特性均产生了一定的变异。同时H9N2亚型AIV还是H5N1/1997、H7N9/2013、H10N8/2013等感染人的流感病毒的内部基因的供体[3-4],因此,对H9N2亚型AlV 的研究工作绝不容忽视。
蓝鹇(Lophuraswinhoii)又名台湾蓝腹鹇,羽毛深蓝,白色羽冠,羽毛艳丽,为台湾特有鸟种,是国家Ⅰ级保护动物,国内外未见蓝鹇感染禽流感的正式报道。本研究从1只罚没死亡的蓝鹇肺脏组织中分离到1株H9N2亚型AIV,并对分离株的HA及NA基因进行序列及进化树分析,对加强 H9N2 亚型禽流感病毒变异株的监测和防控措施研究具有重要的现实意义。
1 材料与方法
1.1 病毒毒株分离鉴定
2015年11月,广东某执法机构罚没1只蓝鹇,这只蓝鹇在救护过程中出现死亡。无菌采集死亡蓝鹇肺脏组织,送华南农业大学禽病研究室完成病料的处理、病毒的分离与鉴定,获得禽流感病毒1株,暂命名为禽流感病毒A/Lophura swinhoii/Guangdong/P29/2016(H9N2),下文简称为LS/GD/P29/16 。
1.2 病毒RNA的提取
使用E.Z.N.A Viral RNA Kit 试剂盒(Omega公司产品)提取病毒RNA,-20℃保存备用。
1.3 cDNA 的制备
取制备的病毒RNA,参照Super-Script III(Invitrogen公司产品)操作说明进行反转录,反应体积为20 μl,在42℃反应1 h,在70℃热灭活15 min,随后在4℃保存备用。
1.4 HA及NA基因片段的扩增
对基因片段进行PCR 扩增,每个体系加入2 μl前述制备的cDNA(含有100 ng RNA 的反转录反应产物),PCR 反应体系为:10×缓冲液5 μl,dNTPs(各2.5 mmol/L)4 μl,PfuUltra II Fusion HS DNA 聚合酶1 μl(Stratagene 公司产品),上游引物和下游引物各25 pmol(引物由华南农业大学禽病实验室提供,引物序列略),用灭菌水补足至总体积50 μl。扩增条件:95℃预变性3 min;95℃变性30 s,53℃退火30 s,72℃延伸1.5 min,30个循环,72℃终延伸8 min。HA片段预期大小为1.7 kb,NA片段预期大小为1.4 kb。
1.5 基因片段的克隆及序列测定
目的片段的PCR扩增产物经凝胶电泳回收后,置于加入10×Taq 缓冲液2 μl,dATP 1 μl,Taq 酶0.5 μl的20 μl 体系内,在72℃反应60 min,随后与pGEM-T载体(Promega 公司产品)连接。将连接产物转化DH 5α感受态细胞,初步鉴定正确的重组质粒由深圳华大基因有限公司测定插入片段的序列,每个片段选择3个阳性克隆质粒测序。
1.6 序列分析
应用Lasergene软件拼接来自LS/GD/P29/16株的HA、NA扩增基因片段的序列,进行核苷酸一致性比较并推导编码的氨基酸序列,应用Clustal W 软件比对得到的序列,并将拼接好的序列登陆NCBI进行Blast分析,下载相关H9N2亚型禽流感病毒NA、HA序列,应用MEGA 5.1 软件对各基因进行系统发生树的绘制。
2 结果
2.1 HA及NA基因片段扩增及测序结果
NA、HA基因片段的测序结果经拼接后,获得LS/GD/P29/16株HA、NA的基因全长序列,其中HA 基因全长1 742 nt,预测编码区为34 ~1 716 nt,编码由560个氨基酸组成的多肽;NA基因全长1 467 nt,预测编码区为21~1 430 nt,编码由469个氨基酸组成的多肽。
2.2 LS/GD/P29/16株NA、HA序列分析
2.2.1 序列Blast分析结果
将拼接好的LS/GD/P29/16株NA、HA序列分别登陆NCBI进行核苷酸序列Blast分析,结果显示:分离株的HA基因序列与登录号为KT 023046,广西鸡源2014年分离株A/chicken/Guangxi/CLB02/2014(H9N2)的核苷酸序列相似性最高,达到99%;而NA基因序列则与登录号为KM 455874,2013年湖南省疾控中心上传的人源分离株A/Lengshuitan/11197/13(H9N2)的NA核苷酸序列相似性最高,为97%。
2.2.2 LS/GD/P29/16分离株HA、NA关键位点分析
参照文献[5]分析LS/GD/P29/16分离株的HA裂解位点序列,发现该毒株在裂解位点处的基序为PSRSSR↓GL,而其他参考序列裂解位点基序较多为PARSSR↓GL,本分离株在裂解位点明显发生一个碱性氨基酸A-S的变异。LS/GD/P29/16分离株HA蛋白的226位氨基酸残基为亮氨酸(Leu),具有哺乳动物SAα2-6受体结合的特性,这与参考毒株G 9/97、Y 280/97等代表毒株特征一致。对LS/GD/P29/16分离株HA蛋白的糖基化位点分析,显示本分离株具有9个潜在N-糖基化位点,分别位于29、82、141、218、298、305、313、492、551位,与其他参考毒株相比,因在315位发生氨基酸残基的变异,而增加了313位的潜在糖基化位点,而其他几个糖基化位点则相对保守。对分离株的HA蛋白受体结合位点进行分析,发现HA受体结合位点109、161、163、202、203位则相对保守,而位点191、198氨基酸变异较为活跃。HA蛋白关键位点氨基酸序列比对情况详见表1。
对LS/GD/P29/16分离株的NA蛋白进行序列分析,发现其他参考株相比本分离株在颈部63~65位氨基酸缺失,没有发生如G1/97代表毒株的38~39位氨基酸的缺失。NA蛋白潜在糖基化位点分析,发现本分离株具有7个潜在的糖基化位点,分别位于69、86、146、200、234、264、368位,与其他参考毒株相比因63~65位氨基酸的缺失造成了61位潜在糖基化位点丢失,而在H264N的突变,与其他毒株相比在此位点增加了1个潜在糖基化位点,其他如86、146、200、234等潜在糖基化位点则相对保守。对NA蛋白红细胞结合位点(HB)分析,发现431~433、366~373、399~404位点均有不同程度的氨基酸变异。其中366~373位氨基酸序列为IKNGSRSG,主要在367~369位发生明显变异;399~404氨基酸序列为DSDDWS,主要在400~402位发生明显变异;而431~433位点则相对保守均为PQE氨基酸基序。研究发现NA蛋白含有7个抗原决定簇均位于其头部区域,分别位于153、197~199、328~336、339~347、367~370、400~403 和431~434位氨基酸。与参考序列进行氨基酸序列比对,分析发现分离毒株NA蛋白抗原决定簇中的199、342、368、369、402、434等位置氨基酸残基变异较为活跃。NA蛋白关键位点氨基酸序列比对情况详见表2。
表1 HA蛋白关键位点氨基酸序列比较
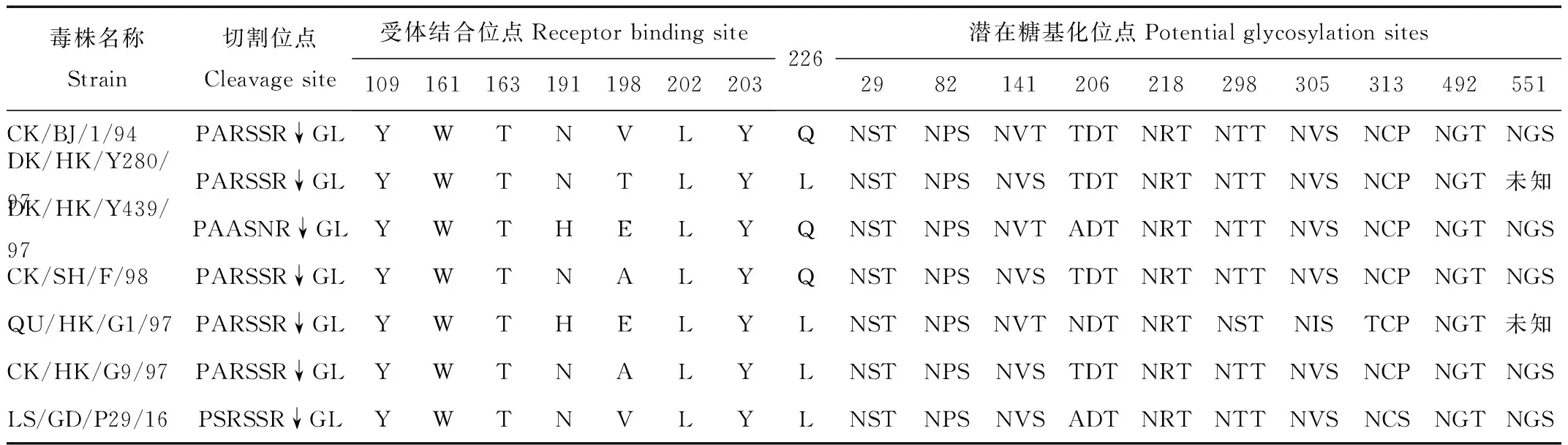
Tab.1 Comparison of the key amino acid residues in the amino acid sequence of HA protein
表2 NA蛋白关键位点氨基酸序列比对
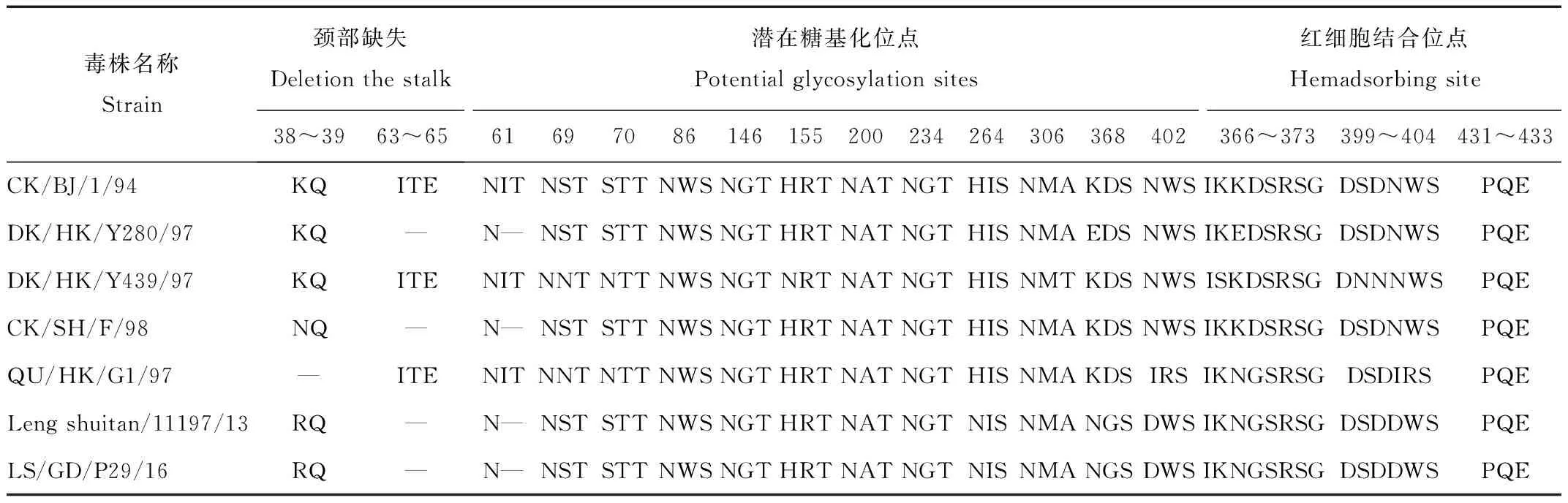
Tab.2 Comparison of the key amino acid residues in the amino acid sequence of NA protein
2.2.3 HA、NA系统进化树分析
从NCBI中下载H9N2亚型AIV的HA,NA序列作为参考,进行基因进化树分析。采用MEGA5.1软件将本分离株的HA、NA基因与国内外部分参考毒株及各个分支的代表毒株进行比对并绘制系统进化树,结果见图1及图2。由图1可知,本研究分离株的HA、NA均位于欧亚分支,属于以Y280/97为代表的Y280亚系中的一个小的进化分支,与其他疫苗株相比在进化关系距离较远。
3 讨论
HA糖蛋白是流感病毒主要表面抗原和保护性抗原,HA糖蛋白的抗原特性直接影响病毒的致病性、宿主特异性及组织嗜性。但HA基因变异频率很高,不同位点,尤其是重要氨基酸位点的变异能导致禽流感病毒的抗原性和致病性变异。HA蛋白潜在的糖基化位点也是影响禽流感病毒抗原性的重要因素,流感病毒可以通过增加糖基化位点的方式来遮蔽抗原表位,从而逃避宿主免疫系统的识别与攻击,对毒力产生一定影响[6-7]。本分离株具有9个潜在N-糖基化位点,与其他参考毒株相比,因在P315S位发生氨基酸残基的变异,而增加了313位的潜在糖基化位点,由于该位点毗邻受体结合区和已知的抗原表位区,该变化是否影响病毒的相关生物学特性还有待进一步研究。对分离株的HA蛋白受体结合位点氨基酸序列进行分析,发现H位点191、198位氨基酸变异较为活跃。
在免疫压力和自然选择下,H9N2 亚型禽流感有可能发生遗传变异而进化出致病力更强的毒株,HA蛋白基因对于病毒的致病性起着决定性的作用,一般高致病力的毒株在裂解位点附近都有连续4个以上碱性氨基酸(R-E-R-R-R-K-K-R),而低致病力的毒株在裂解位点处一般只有单个的碱性氨基酸(Arg),裂解位点处的氨基酸序列和附近糖基化位点的变化决定了致病性的差异[8-10]。本研究毒株在裂解位点处的基序为PSRSSR↓GL,虽然碱性氨基酸的数目未达到4个,而其他参考序列裂解位点基序较多为PARSSR↓GL,本分离株在裂解位点明显发生1个碱性氨基酸A-S的变异,这也符合文献中报道的中国大陆2005年以后此类型切割位点氨基酸序列的毒株逐渐增多的特点。
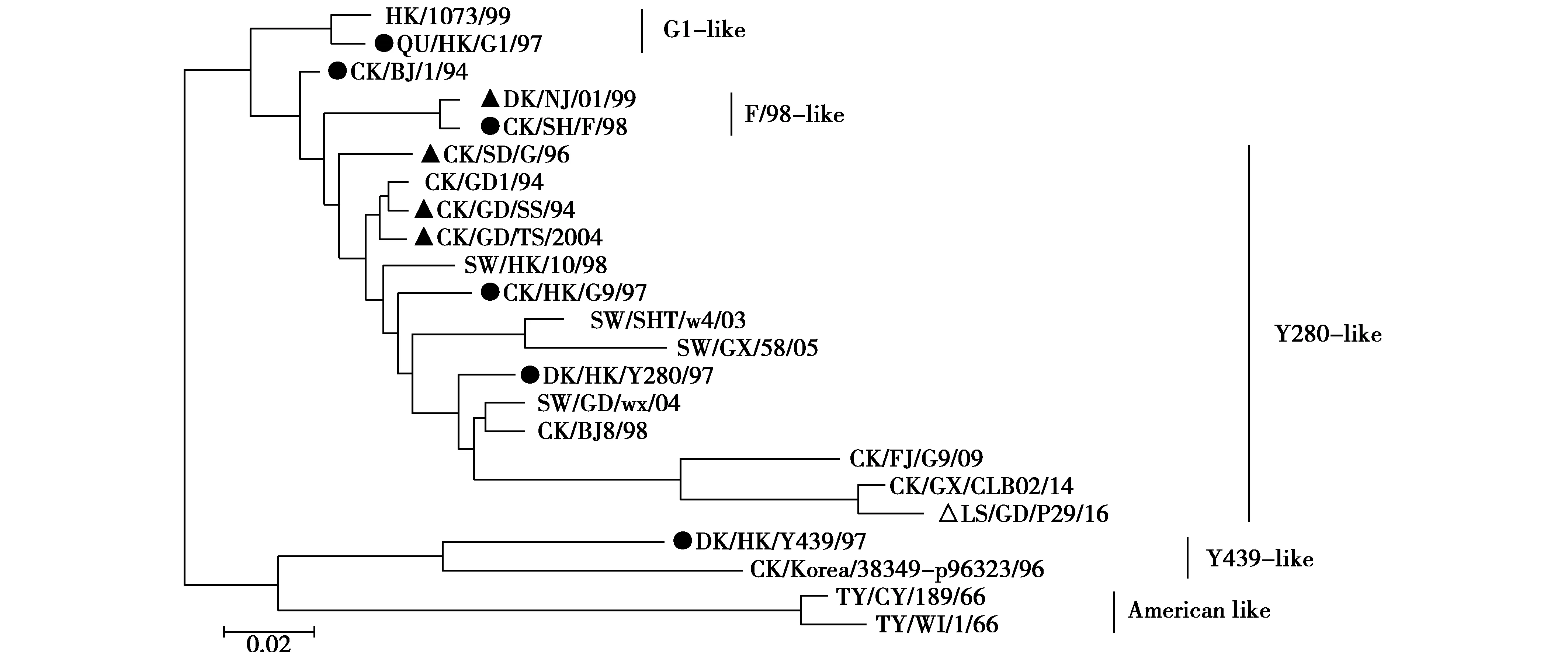
图1 HA基因进化树Fig.1 HA gene evolutionary tree 注:本研究毒株用△标记;H9N2亚型疫苗株用▲标记;欧亚分支亚系内代表毒株用●标记 Note:The strain in this study was noted by △;H9N2 subtype AIV vaccine strain was noted by ▲;and the representative strains of Eurasian clade were noted by ●
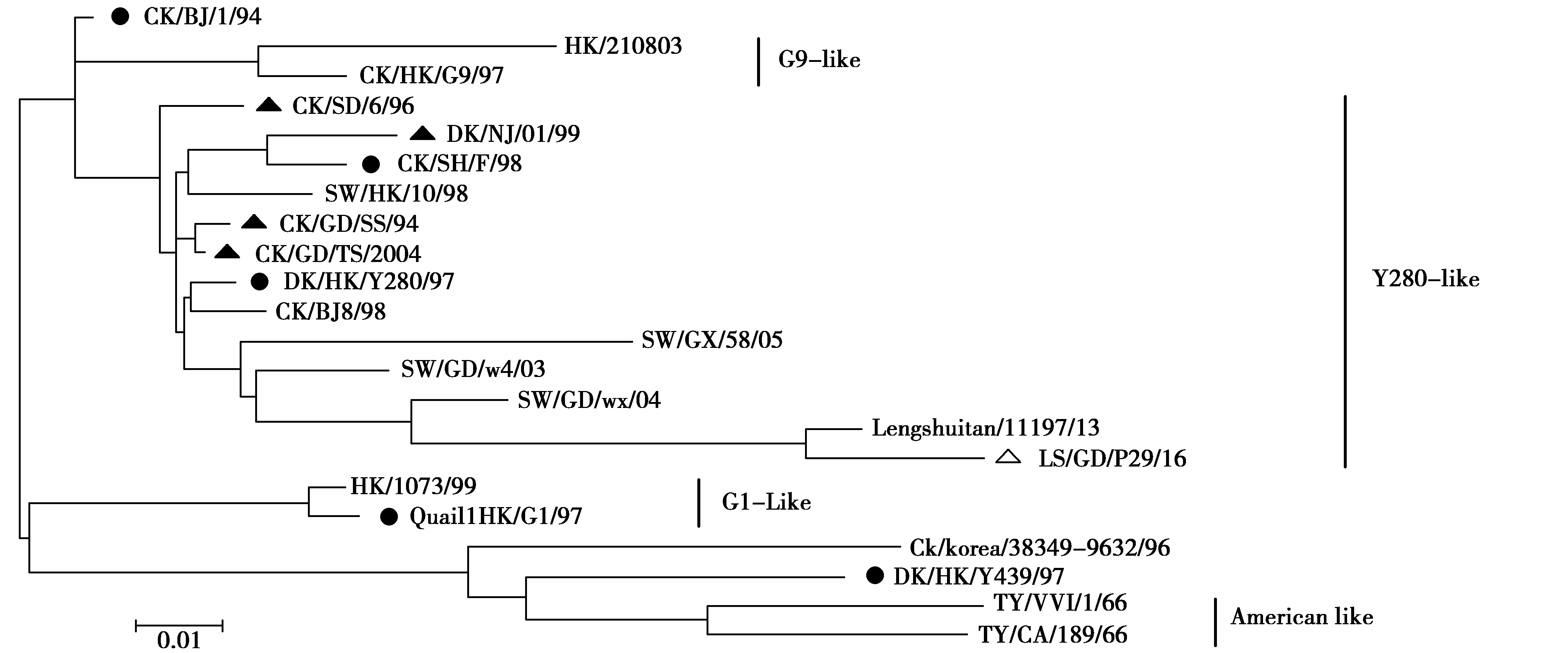
图2 NA基因进化树Fig.2 NA gene evolutionary tree 注:本研究毒株用△标记;H9N2亚型疫苗株用▲标记;欧亚分支亚系内代表毒株用●标记 Note:The strain in this study was noted by △;H9N2 subtype AIV vaccine strain was noted by ▲;and the representative strains of Eurasian clade were noted by ●
早期的H9N2亚型分离株中HA蛋白的L226与Q226并存,而近年来国内的H9N2病毒几乎均突变为L226,并且多数具有与SAα2-6Gal 结合的能力[11]。此外,有研究证实含L226的H9N2亚型AIV能够在人的气管上皮细胞中进行有效的复制。本研究的毒株也具有L226的特点,因此,有必要对此类病毒引起大流行的潜力进行评估。
NA蛋白作为AIV病毒粒子表面的一种重要糖蛋白,能帮助成熟的病毒粒子脱离宿主细胞并感染新的细胞。NA蛋白还能协助病毒穿透宿主呼吸道表面黏膜层,具有较高的变异性,易发生抗原漂移以及产生耐神经氨酸酶抑制剂的毒株,同时抗原决定簇的变异可能与抗体选择有关。不同亚型AIV之间NA颈部氨基酸序列差异明显,NA蛋白长度变化影响NA的活性,引起病毒复制能力的差异[12-13]。研究中Y280-like分支的分离株在颈部63~65缺失3个氨基酸残基,而G1-like分支的毒株则在38、39位缺失2个氨基酸,本研究分离株也符合Y280-like分支的特点,在63~65位缺失3个氨基酸。
NA蛋白潜在糖基化位点分析,发现本分离株NA蛋白具有7个潜在的糖基化位点,与其他参考毒株相比因63~65位氨基酸的缺失造成了61位潜在糖基化位点丢失,而在H264N的突变,与其他毒株相比在此位点增加了一个潜在糖基化位点。对NA蛋白红细胞结合位点(HB)分析,与其他参考毒株相比主要在367~369位、400~402位发生明显变异,而431~433位点则相对保守均为PQE氨基酸基序。对NA蛋白7个抗原决定簇进行氨基酸序列比对,分析抗原决定簇中的199、342、368、369、402、434等位置氨基酸残基变异较为活跃,这些糖基化位点、受体结合位点、抗原决定簇氨基酸的变异究竟是否与毒株的毒力直接相关还有待进一步的研究证明。
H9N2亚型AIV分为北美系和欧亚系两大分支,其中欧亚系进一步衍生出G1-like、F/98-like、Y439-like、Y280-like、BJ/94-like等小分支[14],对本研究分离株的HA和NA进行进化树分析发现本分离株属于欧亚系分支中的Y280-like,这与文献中报道的大陆近年流行株的进化分支特点一致,但是进化树分析也发现,虽然隶属Y280-like小分支中,但是与目前使用的一些疫苗株相比亲缘关系较远,除了病毒抗原漂移和漂变造成的变异之外,高强度的疫苗免疫压力,也是越来越多的免疫逃避病毒株出现的重要原因。
世界范围内发生了多次H9N2亚型AIV突破种间屏障感染人的事件,由于基因组本身的特点增加了自身基因的突变和与其他亚型流感病毒基因重组的机会,极有可能出现可以人际间传播的重组病毒株。WHO已经发出预警,认为H9N2亚型经过抗原漂移和抗原转变,是潜在可能引起全球范围内下一轮新流感爆发的病原[15]。本研究毒株的NA基因序列与湖南省上传的人源分离株的NA核苷酸序列一致性高达97%,随着病毒的持续变异与不断重组,致病力增强的毒株还不定期地出现,因此要加强流行病学监测和平时的预防接种,建立良好的生物安全体系就显得尤为重要。
[1] Homme P J,Easterday B C.Avian influenza virus infections.I.Characteristics of influenza A-turkey-Wisconsin-1966 virus[J].Avian Dis,1970,14(1):66-74.
[2] 郭元吉,王敏,董婕,等.从我国人群中再次分离到H9N2亚型流感病毒[J].中华实验和临床病毒学杂志,2000,14(3):209-212.
[3] Zhang P,Tang Y,Liu X,et al.A novel genotype H9N2 influenza virus possessing human H5N1 internal genomes has been circulating in poultry in eastern China since 1998[J].Journal of Virology,2009,83(17):8428-8438.
[4] 顾敏,刘文博,曹军平,等.1株含H9N2内部基因的H5N1亚型基因重配禽流感病毒的全基因测序及遗传进化分析[J].病毒学报,2010,26(4):298-304.
[5] Matrosovich M,Tuzikov A,Bovin N,et al.Early alterations of the receptor-binding properties of H1,H2,and H3 avian influenza virus hemagglutinins after their introduction into mammals[J].Journal of Virology,2000,74(18):8502-8512.
[6] 陈陆,刘守川,赵军,等.不同H9N2亚型禽流感病毒分离株致病力研究及HA抗原性变异分析[J].中国农业科学,2011,44(24):5100-5107.
[7] Park K J,Kwon H I,Song M S,et al.Rapid evolution of low-pathogenic H9N2 avian influenza viruses following poultry vaccination programmes[J].Journal of General Virology,2011,92(1):36-50.
[8] 刘红旗,黄勇,程坚,等.在疫苗免疫选择压力下H9N2亚型禽流行性感冒病毒HA基因的遗传变异[J].病毒学报,2002,18(2):149-154.
[9] Dong G,Luo J,Zhang H,et al.Phylogenetic diversity and genotypical complexity of H9N2 influenza A viruses revealed by genomic sequence analysis[J].PLoS One,2011,6(2):e17212.
[10] Wan H,Sorrell E M,Song H,et al.Replication and transmission of H9N2 influenza viruses in ferrets:evaluation of pandemic potential[J].PLoS One,2008,3(8):e2923.
[11] Vines A,Wells K,Matrosovich M,et al.The role of influenza A virus hemagglutinin residues 226 and 228 in receptor specificity and host range restriction[J].Journal of Virology,1998,72(9):7626-7631.
[12] Hossain M J,Hickman D,Perez D R.Evidence of expanded host range and mammalian-associated genetic changes in a duck H9N2 influenza virus following adaptation in quail and chickens[J].PLoS One,2008,3(9):e3170.
[13] Wan H,Perez D R.Amino acid 226 in the hemagglutinin of H9N2 influenza viruses determines cell tropism and replication in human airway epithelial cells[J].Journal of Virology,2007,81(10):5181-5191.
Sequence Analysis of the Neuraminidase Gene and the Hemagglutinin Gene of H9N2 Subtype Avian Influenza Viruses from Swinhoe’s Pheasant(Lophuraswinhoii)
Shan Fen1Wang Nianchen2Wang Wenqing2Jiao Peirong2Chen Wu1*Li Wanping1Peng Shiming1
(1.Guangzhou Zoo,Guangzhou,510070,China;2.South China Agricultural University,Guangzhou,510642,China)
We studied genetic mutations of the genes for neuraminidase and hemagglutinin and the molecular epidemiology of H9N2 subtype Avian Influenza Virus(AIV)in captive swinhoe’s pheasant(Lophuraswinhoii).H9N2 AIV strain was isolated from captive swinhoe’s pheasant in Guangdong province.The entire NA and HA gene fragments of the strain were amplified by PCR and sequences were analyzed.The similarity of the strain HA gene was 99% compared with H9N2 AIV A/chicken/Guangxi/CLB02/2014(H9N2)(KT023046)which was the most similar sequence in Genbank.The similarity of the strain NA gene was 97% compared with H9N2 AIV A/Lengshuitan/11197/2013(H9N2)(KM455874)which was the most similar sequence in Genbank.The sequence analysis of the strain HA protein showed that the amino residue at site 226 was leucine.It had the characteristics of the mammal SAα2-6 receptor binding,namely one basic amino had obvious variation in the cleavage site motif PSRSSR↓GL.The HA protein contained 9 potential glycosylation sites at sites 29,82,141,218,298,305,313,492,and 551.The amino acids at the HA receptor binding sites varied widely.The sequence analysis of the strain NA protein showed that the NA neck protein lost amino acids at site 63-65,and contained 7 potential glycosylation sites at sites 69,86,146,200,234,264 and 368.It showed,amino acid residues at hemadsorbing sites and antigenic determinants of NA protein varied.The HA and NA phylogeny analysis showed the isolated strain belonging to a small evolutionary branch of Y280-Like in the Eurasian clade which was in the same branch with the AIV vaccine strain but had a distant genetic relationship with it.The HA and NA genes of the H9N2 AIV strain(LS/GD/P29/16)from swinhoe’s pheasant showed some variation and attention should be paid to strengthen epidemiological surveillance.
Avian influenza virus;H9N2 subtype;Sequence analysis;Evolutionary tree
稿件运行过程
2016-11-28
修回日期:2017-01-22
发表日期:2017-08-10
S852.65 Q7
A
2310-1490(2017)03-441-06
单芬,女,34岁,兽医师;主要从事圈养野生动物保育与疾病防控工作。
*通讯作者:陈武,E-mail:guangzhouchenwu@sina.com
