虎皮鹦鹉全基因组中微卫星分布规律研究
2017-11-17原宝东杨承忠
黄 杰 原宝东 杨承忠
(1.商丘师范学院生物与食品学院,商丘,476000;2.重庆师范大学生命科学学院,重庆,401331)
虎皮鹦鹉全基因组中微卫星分布规律研究
黄 杰1原宝东1杨承忠2*
(1.商丘师范学院生物与食品学院,商丘,476000;2.重庆师范大学生命科学学院,重庆,401331)
对虎皮鹦鹉(Melopsittacusundulatus)全基因组中微卫星分布特征进行了分析,查找到l~6个碱基重复类型的微卫星序列共90 346个,约占整个基因组总长度的序列(1.1 Gb)的0.41%,分布频率为82.9/Mb。不同类型微卫星中,单碱基重复类型数目最多,为50 349个,占总数的55.7%;其次是二、四、三、五、六碱基重复单元序列,分别占到总数的16.3%,13.7%,10.8%,2.9%,0.5%。单碱基微卫星中A重复类型数量最多,二碱基中AT最多,三碱基中AAT,四碱基中AAAC最多,五碱基中AAAGA最多,六碱基中AACCCT最多。A、AT、C、AC、AAT、AAAC、AAAT、AGG、AGC、AG、AAAG、AAC、ATCC、AAGG、AGAT、ATC、AACC、ACC依次是虎皮鹦鹉基因组中分布数量最多的18种微卫星重复拷贝类型。本研究将为虎皮鹦鹉微卫星标记的分离筛选、遗传多样性的研究以及不同物种微卫星的比较分析奠定了基础。
虎皮鹦鹉;基因组;微卫星
微卫星标记(microsatellite),又称为简单序列重复(simple sequence repeats,SSRs),一般由1~6个核苷酸的串联重复片段构成。作为一种优良的分子遗传标记,因其具有多态信息含量高、分布广泛、共显性遗传、易于检测等优点而倍受研究者们的青睐。近年来常被用于动物的遗传疾病研究[1],以及个体识别(包括亲子鉴定)[2-3],种群遗传多样性分析[4-5],基因连锁分析、系统发育[6]等方面的研究。
虎皮鹦鹉(Melopsittacusundulatus)是隶属鹦形目(Psittaciformes)鹦鹉科(Psittacidae)的鸟类。这种鸟原产澳大利亚,其羽毛华丽,易于饲养,可大量繁殖,经人工培育已成为世界性笼养鸟,深受大众喜爱,全世界的总数量超过500万只[7]。目前,虎皮鹦鹉的研究主要集中在其人工繁殖、发声行为、疾病、地震预警等方面的研究[8-11],在分子生物学方面研究相对较少,主要集中于系统进化等方面[12-14]。本研究采用生物信息学的方法,通过微卫星搜索软件首次查找虎皮鹦鹉全基因组中的微卫星序列,并分析微卫星的丰度及其分布规律,为进一步筛选微卫星位点、群体遗传多样性、品种选育及亲缘关系等研究提供数据基础。
1 研究方法
1.1 虎皮鹦鹉基因组序列的获取
虎皮鹦鹉全基因组序列由 http://www.diark.org/diark/species网站下载,基因组总长度约1.1 Gb,所有序列均以FASTA文件格式保存。
1.2 数据分析
本研究采用微卫星搜索及统计软件MSDB v2.4(http://msdb.biosv.com)[15],从下载得到的虎皮鹦鹉基因组序列中寻找微卫星序列。本研究搜索微卫星的标准是:(1)要求单碱基12次重复以上,二碱基7次重复以上,三碱基5次重复以上,四碱基、五碱基和六碱基4次重复以上,且搜索的微卫星均为完美型微卫星[15-16];(2)考虑到碱基互补配对原则以及起始碱基的排列差异,将所有可循环的序列及其互补序列归为一类,比如ATC核心重复序列代表所有TCA、CAT、TAG、AGT和GTA的微卫星。总体来说,单碱基重复有A、C两种独特类型;二碱基重复有AC、AG、AT、GC 4种独特类型;三碱基重复有10种独特类型;四碱基重复有33种独特类型,五碱基重复有102种独特类型;六碱基重复有350种独特类型。
关于微卫星序列分布特征的统计术语定义参考黄杰等[17],具体如下:(1)重复类型:指微卫星序列中核心重复序列是由几个碱基组成,按照核心重复序列的碱基组成数量,微卫星序列可以分为单碱基(mononucleotide)、二碱基(dinucleotide)、三碱基(trinucleotide)、四碱基(tetranucleotide)、五碱基(pentanucleotide)以及六碱基(hexanucleotide)这6种重复类型。(2)重复拷贝类别:指各重复类型具体由哪些碱基构成,例如三碱基重复类型ATC和ACT 分别属于不同的重复拷贝类别,而TCA、CAT 和ATC 则属于相同的重复拷贝类别。(3)重复拷贝数:指一个微卫星序列中其核心序列的重复次数,例如(ACCG)15,该微卫星的重复拷贝数是15。(4)丰度(个/Mb):指1个物种基因组中每Mb序列包含的微卫星序列数量。
2 结果
2.1 各类型微卫星重复序列数目和相应的百分比
在公共数据库下载到已公布的虎皮鹦鹉基因组1.1 Gb(1,117,355,426 b)序列中,用MSDB v2.4微卫星搜索软件共搜索到90 346个不同类型重复序列,其出现频率是82.9/Mb,合计长度达1.47 Mb,占到整个基因组总长度的0.41%。 所有的重复类型如表1所示,单碱基重复数目最多,为50 349个,丰度为45.1个/Mbp,占重复序列总数目的55.7%;其次是二碱基为14 702个,丰度为13.2个/Mbp,占16.3%;四碱基重复类型,为12 408个,丰度为13.1个/Mbp,占有微卫星序列总数量的13.7%;三碱基为9 795个,丰度为8.8个/Mbp,占10.8%;五碱基为2 609个,丰度为2.3个/Mbp,占2.9%;最少的是六碱基重复,为15 558个,丰度为0.4个/Mbp,仅占微卫星序列总数量的0.5%(表1)。
表1 不同重复类型的微卫星在虎皮鹦鹉基因组的分布情况

Tab.1 Distribution of SSR for different repeat types in genomic of Melopsittacus undulatus
2.2 各种重复拷贝类别的数量和相应的比例
同一种类型的微卫星重复序列中,不同重复拷贝类别占该重复类型微卫星总量的比例也有所差异(表2)。单碱基重复类型中,拷贝类别为A的重复类型数目最为丰富,有44 144个,占单碱基重复序列总数目的87.7%,C重复拷贝类别的数量很少,只有6 205个,仅占了12.3%;二碱基重复类型中,AT重复拷贝类别最多,占二碱基总重复序列数目的53.2%,有7 816个,其次是AC(36.9%)和AG(4.8%),CG类别数量最少,仅有26个,占0.2%;三碱基重复类型中,10种重复拷贝类别中数量最多的是AAT,有3 563个,占三碱基微卫星序列总数的36.4%;CCG类别数量最少,仅有9个,占0.1%,其他类型如表2所示。由于四、五和六碱基重复类型的重复拷贝类别较多,因此我们仅列举了数量较多的部分拷贝类别。四碱基微卫星重复类型中,AAAC重复拷贝类别数量最多,为3 207个,占四碱基类型总数的25.8%,其次 AAAT(23.3%)、AAAG(8.3%)、ATCC(7.1%)。其他的类别数量相对较少,数量均小于800个。五碱基中,AAAGA重复拷贝类别数量最多,有383个,占14.7%,其次是AAACA(13.5%)、AAATA(10.6%)、AAAGG(6.3%)、ATCCC(4.5%)和AATGG(4.1%),其他重复拷贝类别数量均不到100个。六碱基微卫星中,重复拷贝类别数量最多的是AACCCT,有97个,占了六碱基重复的20.1%。
表2 虎皮鹦鹉基因组中分布频率最高的微卫星重复类型
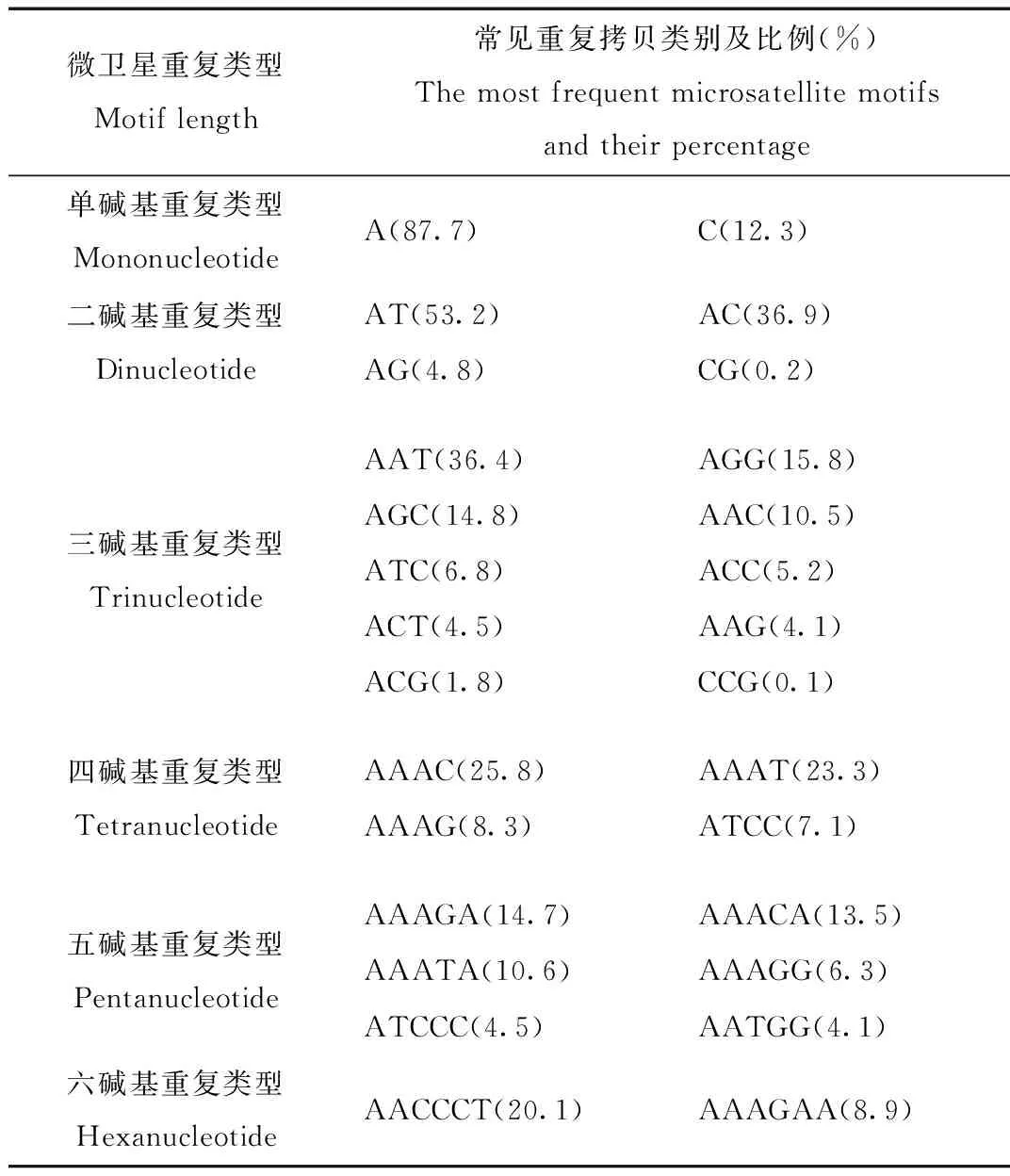
Tab.2 The most frequent microsatellite motifs found in the Melopsittacus undulatus genome
2.3 虎皮鹦鹉基因组中含量丰富的重复拷贝类别及其拷贝数的分布
如图2所示,在虎皮鹦鹉基因组中微卫星数量最多的前18种重复拷贝类别依次为A、AT、C、AC、AAT、AAAC、AAAT、AGG、AGC、AG、AAAG、AAC、ATCC、AAGG、AGAT、ATC、AACC、ACC。其中均为单碱基、二碱基、三碱基和四碱基重复类型,而且这些重复拷贝类别的微卫星数量都在500个以上,总数是83 968个,占所搜索到的微卫星重复拷贝类别总数的92.94 %,而其他所有拷贝类别仅占7.06%,而较长的重复单元并未出现在数量最多的18重复拷贝类别中。另外,在基因组中出现次数超过100次的微卫星有36种重复拷贝类别,总数占基因组全部微卫星总数的97.57%。
对于不同重复拷贝类型微卫星而言,它们的重复次数分布情况也各不相同。虎皮鹦鹉基因组重复拷贝类别中,(A)n是分布最多的微卫星类型,重复拷贝次数集中分布在12~30次,最多重复次数是39次。其次是二碱基重复拷贝类型(AT)n,最长重复次数是30次,此种重复拷贝类型的微卫星重复拷贝次数集中分布在7~11次。短的重复类型(单碱基、二碱基)比长的重复单元(三碱基、四碱基)的分布更加广泛。不同重复拷贝类型微卫星重复次数的分布情况虽然各不相同,但是不同重复拷贝类型中,均可以发现重复拷贝次数少的类型所占该类型微卫星总数的比例最高。
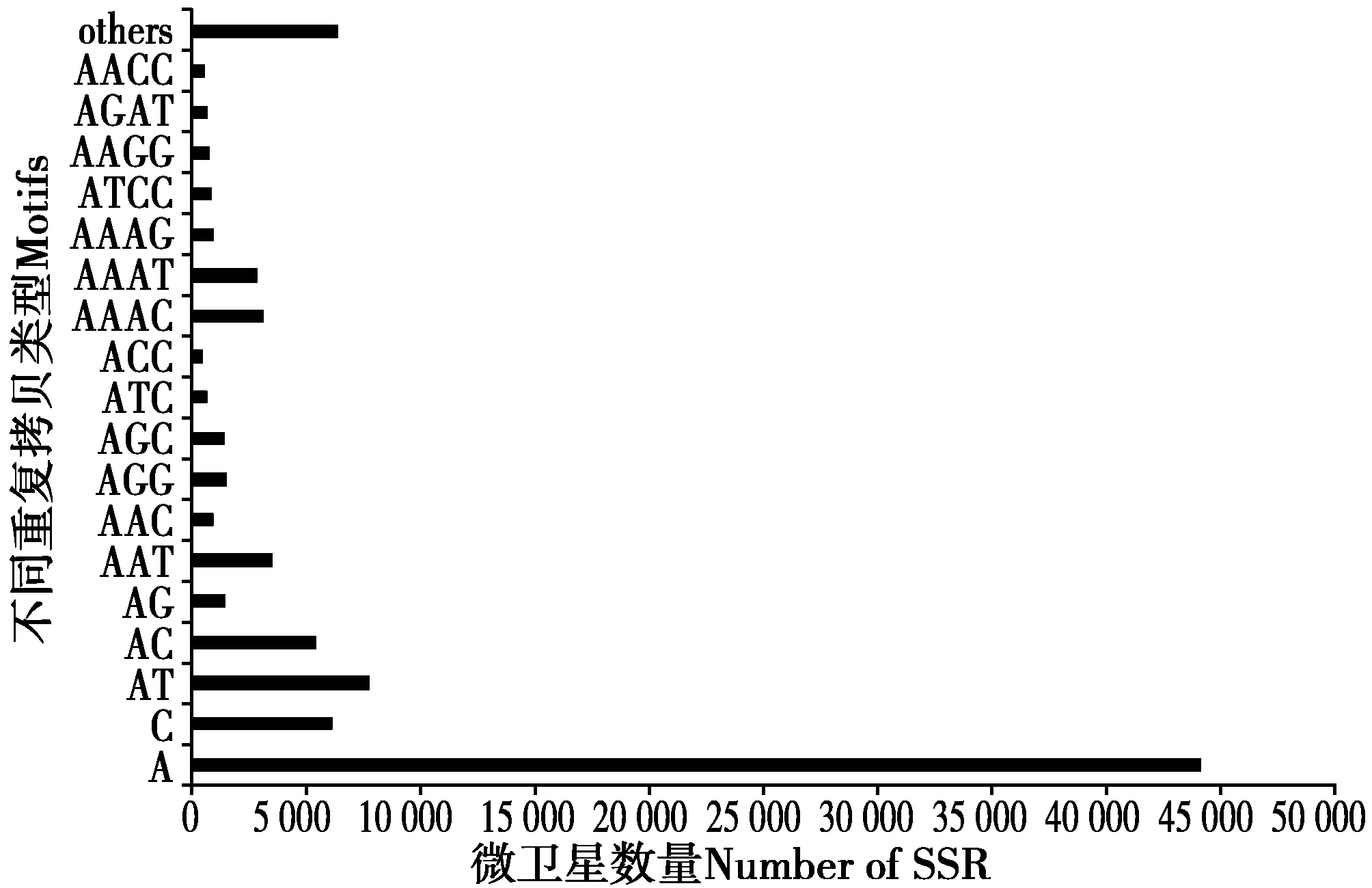
图2 虎皮鹦鹉基因组中分布最多的不同 重复拷贝类型微卫星Fig.2 Distribution of the most frequent microsatellite motifs of Melopsittacus undulatus
表3 分布频率最高的重复拷贝类型微卫星的拷贝数分布情况

Tab.3 Distribution of the copy repeats number of the most frequent microsatellite motifs
3 讨论
本文以虎皮鹦鹉基因组序列为基础,利用微卫星搜索软件搜索并统计分析了该鸟的基因组中完美型微卫星的分布特征和彼此之间的差异性。分析发现,1.1 Gb的虎皮鹦鹉基因组序列中搜索到不同的微卫星总数有90 346个,全部微卫星序列总长度达1.47 Mb,占到整个基因组序列长度的0.41%。与人类(3%),小鼠(2.85%),牛(4.7%)、绵羊(4.8%),大熊猫(0.64%),北极熊(0.79%)[18-21]等多种高等脊椎动物基因组中相比,微卫星序列在虎皮鹦鹉基因组中分布相对较少。这与Primmer等[22]预测鸟类基因组中微卫星的密度会比人基因组中微卫星的密度低很多的观点相一致。但该比例与其他鸟类,如原鸡(Gallusgallus)(0.49%),四川山鹧鸪(Arborophilarufipectus)(0.43%)等相差不大[17,23]。
不同物种基因组中的微卫星序列分布特征的差异可以作为衡量不同物种基因组差异的一个指示[24]。微卫星类型在不同物种间丰度有一定差异,不同物种在基因组中微卫星密度和碱基组成上也存在着较大差异[23]。本研究发现,在不同重复类型的虎皮鹦鹉微卫星中,单碱基微卫星占主导地位(55.7%),这一结果进一步支持了黄杰等[17]发现的高等生物基因组倾向于单碱基微卫星类型占主导地位这一特点。如果不将单碱基类型考虑在内,虎皮鹦鹉基因组微卫星中二碱基微卫星类型数量占优势,这与已公布的四川山鹧鸪和红原鸡已四碱基重复类型占主导不同[17,23]。所以,对于鸟类基因组是否具有四碱基微卫星的偏好需要进一步的研究证据。但虎皮鹦鹉四碱基重复微卫星类型中,以AAAB(B代表除A以外的任何碱基)重复拷贝类别含量最丰富,这一结果与啮齿类、灵长类、维管束植物、真菌类以及原鸡、四川山鹧鸪的研究结果一致[17,23,25]。
虎皮鹦鹉单碱基重复类型以(A)n为主(占单碱基重复序列碱基数的87.7%);二、三、四、五、六碱基重复类型分别以AT,AAT,AAAC,AAAGA,AACCCT重复单元分布最多,这表明微卫星的分布对A和T碱基具有偏好性。在中国对虾、蜜蜂、蚊子、果蝇等基因组中也出现了相似结果[26-29]。虽然不同物种基因组微卫星分布特征有差异,但均发现富含G和C的微卫星出现频率较低。研究也发现,GC含量越高的序列中微卫星越少[30]。普遍认为,微卫星是DNA复制过程中滑移而产生的[31-32],但可能由于C≡G 之间的键比A=T难断裂,GC含量高的序列复制过程中不易发生滑动,因而DNA序列发生滑动复制的概率与序列GC含量成负相关。
对基因组认识水平的提高以及生物信息学的发展,使微卫星的研究也越来越方便高效。本研究通过对虎皮鹦鹉基因组微卫星序列的扫描和分布特征分析,一方面,扫描出大量的微卫星序列为该种鸟微卫星标记的开发提供了充足的数据基础;另一方面,为研究虎皮鹦鹉连锁图谱以及种群遗传学等方面的研究奠定了基础。
[1] Ludovini V,Pistola L,Gregorc V,et al.Plasma DNA,microsatellite alterations,and p53 tumor mutations are associated with disease-free survival in radically resected non-small cell lung cancer patients:a study of the perugia multidisciplinary team for thoracic oncology[J].Journal of Thoracic Oncology,2008,3(4):365-373.
[2] Huang J,Li Y Z,Du L M,et al.Genome-wide survey and analysis of microsatellites in giant panda(Ailuropodamelanoleuca),with a focus on the applications of a novel microsatellite marker system[J].BMC Genomics,2015,16(1):61.
[3] Beacham T D,McIntosh B,Wallace C.A comparison of stock and individual identification for sockeye salmon(Oncorhynchusnerka)in British Columbia provided by microsatellites and single nucleotide polymorphisms[J].Canadian Journal of Fisheries and Aquatic Sciences,2010,67(8):1274-1290.
[4] Huang J,Zhu D,Song X,et al.High-throughput microsatellite markers discovery for the Sichuan hill partridge(Arborophilarufipectus)and assessment of genetic diversity in the Laojunshan population[J].Biochemical Systematics and Ecology,2015,60:266-272.
[5] Page R B,Sankamethawee W,Pierce A J,et al.High throughput sequencing enables discovery of microsatellites from the puff-throated bulbul(Alophoixuspallidus)and assessment of genetic diversity in Khao Yai National Park,Thailand[J].Biochemical Systematics and Ecology,2014,55:176-183.
[6] Kucuktas H,Wang S,Li P,et al.Construction of genetic linkage maps and comparative genome analysis of catfish using gene-associated markers[J].Genetics,2009,181(4):1649-1660.
[7] Perrins C.The new encyclopedia of birds[M].Oxford:Oxford University Press,2003.
[8] Ramis A,黄瑜.虎皮鹦鹉同时暴发喙羽病病毒和多瘤病毒感染[J].国外畜牧科技,1999,26(6):37-40.
[9] 钟福生,曾元根,莫小林,等.虎皮鹦鹉人工繁殖的研究[J].经济动物学报,2001,5(2):43-46.
[10] 江文科,李东风.鸣管神经支对虎皮鹦鹉鸣叫声的支配[J].华南师范大学学报:自然科学版,2015,47(2):90-95.
[11] 李均之.虎皮鹦鹉异常行为与临震预测[J].北京工业大学学报,2008,34(2):216-219.
[12] Schweizer M,Seehausen O,Güntert M,et al.The evolutionary diversification of parrots supports a taxon pulse model with multiple trans-oceanic dispersal events and local radiations[J].Mol Phylogenet Evol,2010,54(3):984-994.
[13] Wright T F,Schirtzinger E E,Matsumoto T,et al.A multilocus molecular phylogeny of the parrots(Psittaciformes):support for a Gondwanan origin during the Cretaceous[J].Mol Biol Evol,2008,25(10):2141-2156.
[14] Tokita M,Kiyoshi T,Armstrong K N.Evolution of craniofacial novelty in parrots through developmental modularity and heterochrony[J].Evol Dev,2007,9(6):590-601.
[15] Du L M,Li Y Z,Zhang X Y,et al.MSDB:a user-friendly program for reporting distribution and building databases of microsatellites from genome sequences[J].Journal of Heredity,2012,104(1):154-157.
[16] 汪自立,黄杰,杜联明,等.二斑叶螨和肩突硬蜱基因组微卫星分布规律研究[J].四川动物,2013,32(4):481-486.
[17] 黄杰,周瑜,刘与之,等.基于454 GS FLX高通量测序的四川山鹧鸪基因组微卫星特征分析[J].四川动物,2015,34(1):8-14.
[18] Subramanian S,Mishra R K,Singh L.Genome-wide analysis of microsatellite repeats in humans:their abundance and density in specific genomic regions[J].Genome Biology,2003,4(2):R13.
[19] 童晓玲,代方银,李斌,等.小鼠基因组中的微卫星重复序列的数量、分布和密度[J].动物学报,2006,52(1):138-152.
[20] 戚文华,蒋雪梅,肖国生,等.牛和绵羊全基因组微卫星序列的搜索及其生物信息学分析[J].畜牧兽医学报,2013,44(11):1724-1733.
[21] 李午佼,李玉芝,杜联明,等.大熊猫和北极熊基因组微卫星分布特征比较分析[J].四川动物,2014,33(6):874-878.
[22] Primmer C R,Raudsepp T,Chowdhary B P,et al.Low frequency of microsatellites in the avian genome[J].Genome Res,1997,7(5):471-482.
[23] 黄杰,杜联明,李玉芝,等.红原鸡全基因组中微卫星分布规律研究[J].四川动物,2012,31(3):358-363.
[24] 马秋月,戴晓港,陈赢男,等.枣基因组的微卫星特征[J].林业科学,2013,49(12):81-87.
[25] Tóth G,Gáspári Z,Jurka J.Microsatellites in different eukaryotic genomes:survey and analysis[J].Genome Res,2000,10(7):967-981.
[26] 高焕,刘萍,孟宪红,等.中国对虾(Fenneropenaeuschinensis)基因组微卫星特征分析[J].海洋与湖沼,2004,35(5):424-431.
[27] 魏朝明,孔光耀,廉振民,等.蜜蜂全基因组中微卫星的丰度及其分布[J].昆虫知识,2007,44(4):501-504.
[28] Yu Quanyou,Li Bin,Li Guanrong,et al.Abundance and distribution of microsatellites in the entire mosquito genome[J].Prog Biochem Biophys,2005,32(5):435-441.
[29] Kim T S,Booth J G,Gauch H G,et al.Simple sequence repeats inNeurosporacrassa:distribution,polymorphism and evolutionary inference[J].BMC Genomics,2008,9(1):31.
[30] Schlötterer C.Genome evolution:are microsatellites really simple sequences?[J].Curr Biol,1998,8(4):R132-R134.
[31] Schlötterer C,Tautz D.Slippage synthesis of simple sequence DNA[J].Nucleic Acids Res,1992,20(2):211-215.
[32] Zhao X,Tan Z,Feng H,et al.Microsatellites in differentPotyvirusgenomes:survey and analysis[J].Gene,2011,488(1/2):52-56.
Distribution of Microsatellites in Genome of Budgerigar(Melopsittacusundulatus)
Huang Jie1Yuan Baodong1Yang Chengzhong2*
(1.College of Biology and Food Sciences,Shangqiu, 476000,China;2.College of Life Science,Chongqing Normal University, Chongqing,401331,China)
We analyzed the distribution of microsatellites in the entire genome of budgerigar(Melopsittacusundulatus).A total of 90 346 simple sequence repeats(SSRs)with 1~6 bp nucleotide motifs were identified,means that about 0.41% of the budgerigar genome was occupied by SSRs.And the frequency of SSR was 82.9/Mb.In the different repeat types of the microsatellites,mononucleotide repeat motifs was the most abundant type(55.7%),while the tetranucleotide,dinucleotide,trinucleotide,pentanucleotide and hexanucleotide repeats were 16.3%,13.7%,10.8%,2.9% and 0.5%,respectively.Moreover,A,AT,AAT,AAAC,AAAGA,AACCCT were the most frequent repeat copy units of the six motif types,respectively.In the budgerigar genome,A,AT,C,AC,AAT,AAAC,AAAT,AGG,AGC,AG,AAAG,AAC,ATCC, AAGG,AGAT,ATC,AACC,and ACC were the predominant repeat types. This study will facilitate the isolation of microsatellites and the genetic diversity analysis of budgerigar,and serve as a foundation for comparative studies of microsatellites in different species.
Melopsittacusundulates;Genome;Microsatellite
稿件运行过程
2016-12-07
修回日期:2017-02-11
发表日期:2017-08-10
Q78 Q953
A
2310-1490(2017)03-422-05
国家自然科学基金项目(No.31501845),商丘师范学院高层次人才科研启动项目(No.50013901;50014001)
黄杰,女,30岁, 博士; 主要从事动物学方面研究。
*通讯作者:杨承忠, E-mail:drczyang@126.com
