兰花磷酸肌醇特异性磷脂酶C基因家族生物信息学分析
2017-11-16胡广隆陈亚娟魏建华王宏芝张杰伟
胡广隆,陈亚娟,魏建华,王宏芝,张杰伟
(北京市农林科学院 农业基因资源与生物技术北京市重点实验室,北京 100097)
兰花磷酸肌醇特异性磷脂酶C基因家族生物信息学分析
胡广隆,陈亚娟,魏建华,王宏芝*,张杰伟*
(北京市农林科学院 农业基因资源与生物技术北京市重点实验室,北京 100097)
【目的】为研究兰花(Phalaenopsisequestris)中PI-PLC基因家族各成员在兰花中的生理功能。【方法】利用兰花基因组数据库,经过生物信息学分析,获得兰花PI-PLC基因家族成员的基因结构、染色体定位和编码蛋白,经过多重序列比对进行进化和分类分析。【结果】结果表明:兰花基因组中含有3个PI-PLC家族基因成员,分别含有9~13个外显子。TMHMM跨膜区结构分析显示,兰花PI-PLC蛋白均不含有跨膜区;MEME保守结构域分析显示,兰花PI-PLC 蛋白均含有4个保守的EF、X、Y和C2结构域。【结论】以上结果对解析兰花PI-PLC蛋白的生物学功能提供重要的线索。
磷酸肌醇特异性磷脂酶C; 兰花; 基因家族; 进化分析
【研究意义】磷酸肌醇特异性磷脂酶C(phosphoinositide-specific phospholipase C,PI-PLC)是一类能特异性水解4,5-二磷酸磷脂酰肌醇(PIP2)的磷脂酶,在其催化下,生成两个信号转导中重要的第二信使——1,2-二酰甘油(DAG)和1,4,5-三磷酸肌醇(IP3)[1-2]。最新的研究表明:在植物细胞中,脂溶性DAG停留在细胞膜上,在二酰甘油激酶的作用下生成磷脂酸(phosphatidic acid, PA),PA与特定的蛋白激酶、脂激酶和NADPH氧化酶等蛋白相互作用[3];而水溶性IP3进入细胞质,在一系列磷酸激酶的作用下可以快速转化为IP4、IP5,最终转化为IP6, 促进细胞内钙库中Ca2+内流,从而调节Ca2+浓度及Ca2+依赖的酶类或Ca2+依赖的通道[1-2];进而调节植物生长发育、非生物胁迫和细胞死亡等生理过程[1-3]。【前人研究进展】高等植物中,拟南芥PI-PLC蛋白生物功能解析较为清晰。拟南芥PI-PLC基因家族有9个成员[4-5]。其中拟南芥AtPLC1(At5g58670)在不依赖ABA的高渗胁迫中发挥着重要作用[6];拟南芥AtPLC2(At3g08510)在生长素调节的雌雄配子体的发育中发挥着重要作用[7];拟南芥AtPLC3(At4g38530)和AtPLC9(At3g47220)在植株耐热性方面发挥着重要作用[1,8],在拟南芥中过量表达拟南芥AtPLC5(At5g58690)引起拟南芥叶片早衰,在烟草中过量表达拟南芥AtPLC5(At5g58690)引起烟草点状死亡[9]。【本研究切入点】兰科是被子植物门中第三大科,其具有重要的商品特性。2014年11月24日小兰屿蝴蝶兰[Phalaenopsisequestris(Schauer) Rchb.f.]的全基因组以封面文章的形式在Nature Genetics杂志公开发表[10],为后续利用生物信息学分析和鉴定兰花基因组奠定了序列基础。【拟解决的关键问题】本研究充分利用兰花基因组数据库,着重分析兰花PI-PLC基因家族成员的基因结构、染色体定位和编码蛋白、跨膜区结构特性及编码蛋白保守结构域,为克隆兰花PI-PLC基因(PePLC)提供重要的序列信息,为深入研究PePLC 蛋白的功能奠定重要基础。
1 材料与方法
1.1 兰花PI-PLC基因家族全基因组、cDNA和蛋白序列的获得
拟南芥(Arabidopsisthaliana)PI-PLC基因及蛋白序列下载自TAIR数据库(http://www.arabidopsis.org),水稻(OryzasativaL. spp.Japonicacv.Nipponbare)PI-PLC基因及蛋白序列下载自RAP数据库(http://rapdb.dna.affrc.go.jp/),兰花(P.equestris)PI-PLC基因及蛋白序列均下载自P. equestris Genome数据库(http://orchidbase.itps.ncku.edu.tw/est/P_Equestris_Genome.aspx)。
以水稻(O.sativa)PI-PLC基因家族蛋白质序列OsPLC1(Os07g49330)、OsPLC2 (Os03g18010)、OsPLC3(Os12g37560)和OsPLC4 (Os05g03610)为参比序列,利用兰花P. equestris Genome数据库分别进行BlastP检索,检索结果的全序列E值大于e-8的舍去,再利用NCBI提供的在线CDS 软件(http://www.ncbi.nlm.nih.gov/cdd)预测这些蛋白有无PI-PLC 结构域,存在PI-PLC 结构域的蛋白序列属于PI-PLC基因家族。
1.2 兰花PI-PLC基因家族结构分析
利用P.equestrisGenome数据库,获取兰花(P.equestris)中PI-PLC基因家族成员的基因注释,根据各成员的内含子和外显子的大小和数目,用手绘制其各自基因结构示意图。
1.3 兰花PI-PLC基因家族二级结构及跨膜区结构预测
利用在线Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/)工具对PI-PLC 基因家族各成员编码蛋白的氨基酸相似性进行分析[11],利用在线二级结构预测软件SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对兰花(P.equestris)PI-PLC蛋白的α-螺旋、β-折叠和无规则卷曲等二级结构进行分析[12]。利用在线软件TMHMM Server v. 2. 0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)对兰花(P.equestris)PI-PLC蛋白的跨膜区结构进行预测分析[13]。
1.4 兰花PI-PLC基因家族保守结构域分析
应用在线软件MEME(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)对拟南芥、水稻和兰花(P.equestris)PI-PLC蛋白的保守结构域进行分析[14]。
1.5 兰花PI-PLC蛋白三级结构预测
应用在线三级结构预测软件Swiss-Model (https://swissmodel.expasy.org/) ,选取其自动建模功能对兰花(P.equestris)PI-PLC蛋白质的空间结构模型进行同源建模分析[15]。
1.6 兰花PI-PLC蛋白系统进化树的构建
通过Clustal X(2.0)[16]软件对拟南芥、水稻和兰花(P.equestris)的PI-PLC蛋白进行多重序列比对分析,使用MEGA 6.0[17]软件将序列比对结果采用Neighborjoining法(bootstrap值设为1000)生成PI-PLC蛋白的系统进化树。
2 结果与分析
2.1 兰花PI-PLC 基因家族成员的鉴定和命名
根据水稻中已经鉴别出来的4个PI-PLC蛋白序列,分别利用P.equestrisGenome数据库提供的Blast 程序中进行BlastP,得到8个兰花(P.equestris)候选PI-PLC基因。利用保守域在线预测软件CDS进行验证PI-PLC保守结构域的存在,初步确定了兰花(P.equestris)中含有3个PI-PLC基因。根据兰花(P.equestris)PI-PLC基因在染色体上的定位分别命名为PePLC1-3,其分别含有9~13个外显子(图1),各成员的开放阅读框长度、氨基酸长度、染色体定位和外显子个数等详细信息见表1。
2.2 兰花PI-PLC 基因家族结构分析
利用在线Clustal Omega对PI-PLC基因家族各成员编码蛋白的氨基酸相似性进行分析,分析表明,兰花PePLC1蛋白和PePLC3蛋白相似性最高,为70.07 %,兰花PePLC2蛋白和PePLC3蛋白相似性最低,为59.52 %(表2)。利用在线二级结构预测软件SOPMA 对兰花PI-PLC基因家族各成员编码蛋白的二级结构进行分析。分析表明:兰花PI-PLC蛋白中无规则卷曲的比例最高(33.81 %~37.50 %),其次为α-螺旋(30.48 %~33.39 %),再次为β-折叠(20.16 %~23.02 %),β-转角最低(9.53 %~12.70 %)(表3)。利用在线软件TMHMM Server v. 2. 0对兰花PI-PLC蛋白的跨膜区结构进行分析,结果显示:兰花PI-PLC蛋白均不含有跨膜区(图2)。
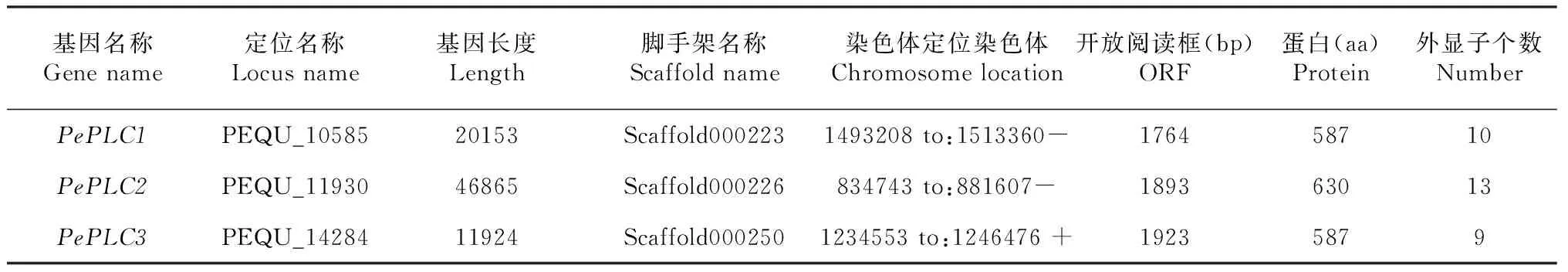
表1 兰花PI-PLC基因家族的基本特征
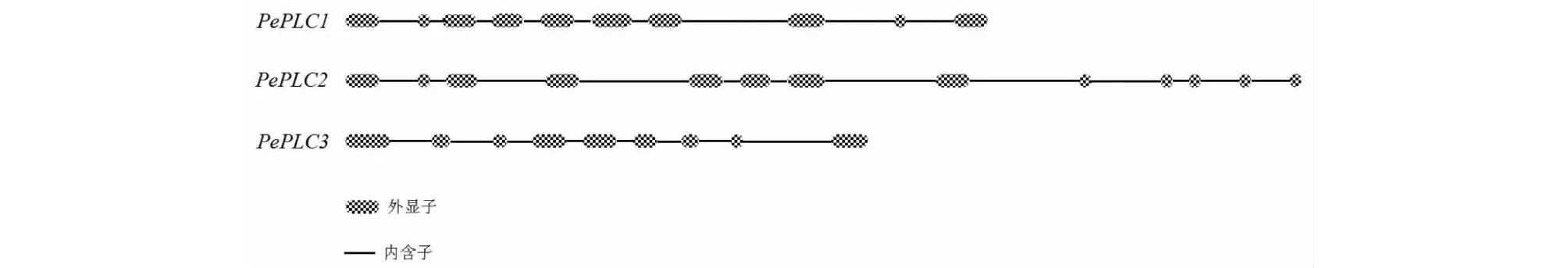
图1 兰花PI-PLC基因家族结构分析Fig.1 Gene structure of the orchid PI-PLC gene family
使用在线保守结构域预测软件MEME 对拟南芥、水稻和兰花PI-PLC基因家族保守结构域进行分析,结果表明拟南芥、水稻和兰花PI-PLC基因家族包含4个保守结构域。其中,基序1为EF结构域,相似度为8.0e-594;基序2和基序3 分别是X 和Y结构域(共同组成PI-PLC蛋白催化结构域),相似度为分别为5.7e-509和3.9e-348;基序4为C2结构域,相似度为3.2e-371(图3)。无论在单子叶植物水稻和兰花中,还是在双子叶植物拟南芥中,PI-PLC基因家族均含有EF、X、Y和C2 4个结构域,这表明PI-PLC蛋白在进化过程中存在高度保守的结构域(图3)。

图2 兰花PI-PLC蛋白跨膜结构预测Fig.2 Prediction of transmembrane structure of PI-PLC proteins in Orchid

PI-PLC家族PePLC2PePLC3PePLC165.2970.07PePLC259.52
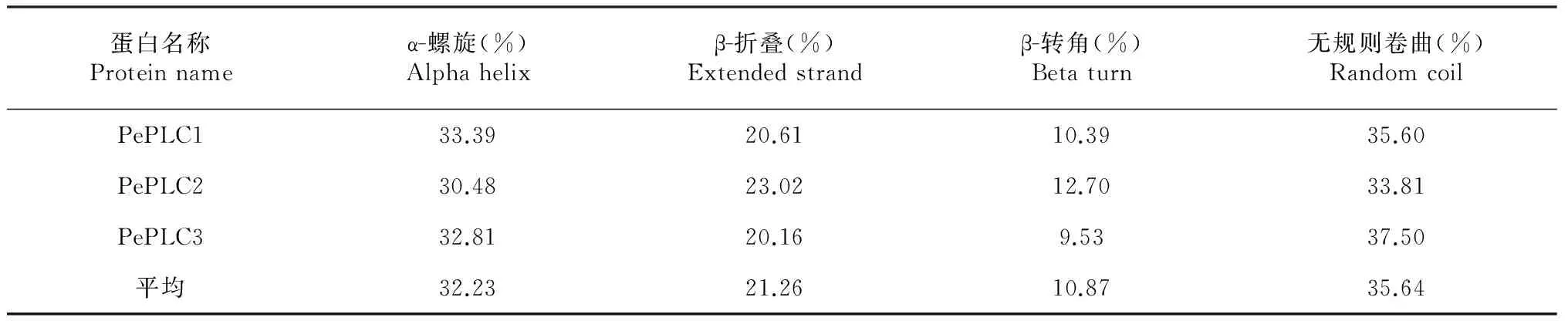
表3 兰花PI-PLC蛋白的二级结构信息

图3 拟南芥、水稻和兰花的PI-PLC家族基因编码蛋白保守结构域分布Fig.3 Distribution of conserved motifs in Arabidopsis, rice and orchid PI-PLC proteins

图4 兰花3条PI-PLC蛋白质三级结构Fig.4 Tertiary structure of 3 orchid PI-PLC proteins
蛋白质三级结构是在二级结构的基础上借助各种次级键卷曲折叠成特定的球状分子结构的构象,本研究采用Swiss-Model对兰花PI-PLC蛋白进行同源建模,图4表明:兰花PI-PLC蛋白最终形成的三级结构在空间结构非常相似,然而无规则卷曲的比例最高,暗示不同兰花PI-PLC蛋白在生物学功能上存在很高的保守性,不过特定的PI-PLC蛋白在特定的环境及时空中又具有各自的特异性。
2.3 兰花PI-PLC 家族蛋白相似性和系统进化分析
利用进化树分析软件MEGA 6.0 对9个拟南芥PI-PLC蛋白、4个水稻PI-PLC蛋白、3个兰花PI-PLC蛋白序列进行系统进化分析。结果显示:这16
个PI-PLC蛋白并没有完全按照物种聚类在一起,而是混合分布在一起。PI-PLC蛋白系统进化树基本上按照单子叶植物和双子叶植物聚类在一起,推测PI-PLC蛋白在单子叶植物和双子叶植物中具有各自特异的生物学功能,暗示PI-PLC蛋白在植物进化过程中可能具有不同的进化过程。水稻OsPLC4与拟南芥AtPLC1(At5g58670)和AtPLC3(At4g38530)聚类在一起,暗示水稻OsPLC4可能具有与双子叶植物拟南芥AtPLC1(At5g58670)和AtPLC3(At4g38530)相似的进化过程和生物学功能(图5)。兰花PePLC1~3和水稻的OsPLC1~3聚类在一起,暗示他们具有相似的生物学功能(图5)。
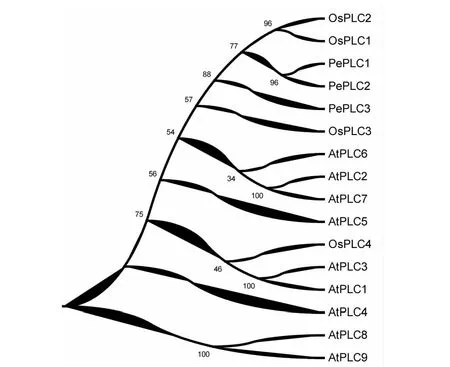
图5 拟南芥、水稻和兰花的PI-PLC蛋白系统进化树Fig.5 Phylogenetic tree for Arabidopsis, rice and orchid PI-PLC proteins
3 讨 论
PI-PLC是磷酸肌醇信号转导途径中的一类关键水解酶,研究表明:植物PI-PLC参与了生物胁迫、非生物胁迫、调节细胞骨架等多个生理过程[1-9,18-20]。随着分子生物学技术的不断发展,PI-PLC基因家族已经陆续在拟南芥[4]、水稻[5]、番茄[21]中鉴定,但兰花PI-PLC基因家族尚未鉴定。本研究鉴定兰花PI-PLC基因家族含有3个成员,而已鉴定的拟南芥、水稻和番茄PI-PLC基因家族分别含有9个、4个和6个成员,这说明在进化过程中,PI-PLC基因可能经历了不断发生谱系的特异扩张和拷贝丢失。兰花PI-PLC基因家族的3个成员分别含有9~13个外显子,均不含有跨膜区,均含有EF、X、Y和C2 4个结构域,这表明不同物种PI-PLC基因家族中重要结构域的高度保守性。推测他们在植物诸多生理过程中发挥相似的作用。而不同物种中含有的成员数目各不相同,推测他们在特定的时空和组织响应不同的刺激从而精细调节各生理过程。
进化分析表明,兰花PePLC1~3和水稻的OsPLC1~3聚类在一起,暗示他们可能具有相似的生物学功能(图5)。其中兰花PePLC1、PePLC2和水稻OsPLC1、OsPLC2,兰花PePLC3和OsPLC3同源性较高。与水稻OsPLC2同源性最高的籼稻OsIPI-PLC1(OryzasativaL. Spp.indicaS.Katocv.yuanfengzao)参与了水稻抗病的信号转导过程[22],水稻OsPLC2和OsPLC3均被证明具有典型的PI-PLC生物学功能,且均定位于质膜上,OsPLC3正向调节水稻的耐盐性[23]。据此推测兰花PePLC1和PePLC2可能参与兰花的抗病过程,PePLC3可能参与兰花的耐盐过程。
兰花PePLC1和PePLC2、水稻OsPLC1和OsPLC2、拟南芥AtPLC1(At5g58670)和AtPLC3(At4g38530)、AtPLC2(At3g08510)和AtPLC7(At3g55940)、AtPLC8(At3g47290)和AtPLC9(At3g47220)高度同源(图5),研究表明:AtPLC1(At5g58670)响应了干旱、盐胁迫、低温胁迫[24]和ABA的次级信号转导[25]。AtPLC3(At4g38530)不仅参与了拟南芥的开花过程,而且参与了拟南芥的耐热反应[1,8]。表明聚类在一起的同源基因可能具有不同的生物学功能,因此不能简单推测PePLC1和PePLC2参与兰花的抗病过程。兰花PePLC各成员特定的生物学功能还需要从兰花中克隆后逐一解析其功能。
4 结 论
兰花基因组中鉴定出3个PI-PLC家族基因成员,含有9~13个外显子。兰花PePLCs蛋白均不含跨膜区、均含有4个高度保守的(EF、X、Y和C2)结构域。
[1]Zheng S Z, Liu Y L, Li B, et al. Phosphoinositide-specific phospholipase C9 is involved in the thermotolerance inArabidopsis[J]. Plant J, 2012, 69(4):689-700.
[2]Rupwate S D, Rajasekharan R. Plant phosphoinositide-specific phospholipase C: an insight[J]. Plant Signa Behav, 2012, 7(10):1281-1283.
[3]Wang X, Devaiah S P, Zhang W, et al. Signaling functions of phosphatidic acid[J]. Prog Lipid Res, 2006, 45 (3): 250-278.
[4]Tasma I M, Brendel V, Whitham S A, et al. Expression and evolution of the phosphoinositide-specific phospholipase C gene family inArabidopsisthaliana[J]. Plant Physiol Bioch, 2008, 46(7):627-637.
[5]Singh A, Kanwar P, Pandey A, et al. Comprehensive genomic analysis and expression profiling of phospholipase C gene family during abiotic stresses and development in rice[J]. PLoS One, 2013, 8(4): e62494.
[6]Takahashi S, Katagiri T, Hirayama T, et al. Hyperosmotic stress induces a rapid and transient increase in inositol 1, 4, 5-trisphosphate independent of abscisic acid inArabidopsiscell culture[J]. Plant Cell Physiol, 2001, 42(2):214-222.
[7]Li L, He Y, Wang Y, et al.ArabidopsisPLC2 is involved in auxin-modulated reproductive development[J]. Plant J, 2015, 84(3): 504-515.
[8]Gao K, Liu Y L, Li B, et al.Arabidopsisthalianaphosphoinositide-specific phospholipase C isoform 3 (AtPLC3) and AtPLC9 have an additive effect on thermotolerance[J]. Plant Cell Physiol, 2014, 55(11): 1873-1883.
[9]Zhang J, Xia K, Yang Y, et al. Overexpression ofArabidopsisphosphoinositide-specific phospholipase C5 induces leaf senescence[J]. Plant Cell Tiss Organ Cult, 2015 120(2): 585-595.
[10]Cai J, Liu X, Vanneste K, et al. The genome sequence of the orchidPhalaenopsisequestris[J]. Nat Genet, 2014, 47(1):186-186.
[11]Sievers F, Wilm A, Dineen D, et al. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega[J]. Mol Syst Biol, 2011, 7(1):1429-1432.
[12]Geourjon C, Deléage G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Comput Appl Biosci, 1995, 11(6): 681-684.
[13]张演义, 吕福堂, 张全军, 等. 葡萄MAPKK基因家族的识别与分析[J]. 西南农业学报, 2015, 28(4):1791-1797.
[14]Bailey T L, Boden M, Buske F A, et al. MEME SUITE: Tools for motif discovery and searching[J]. Nucleic Acids Res, 2009, 37(S2):202-208.
[15]Arnold K, Bordoli L, Kopp J T. The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling[J]. Bioinformatics, 2006, 22(2):195-201.
[16]Larkin M A, Blackshields G, Brown N P, et al. Clustal W and clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948.
[17]Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729.
[18]Tripathy M K, Tyagi W, Goswami M, et al. Characterization and functional validation of tobacco PLCδ for abiotic stress tolerance[J]. Plant Mol Biol Rep, 2012,30(2):488-497.
[19]Di Paolo G, De Camilli P. Phosphoinositides in cell regulation and membrane dynamics[J]. Nature, 2006, 443(7112):651-657.
[20]Coursol S, Giglioli-Guivarc'h N, Vidal J, et al. An increase in phosphoinositide-specific phospholipase C activity precedes induction of C4 phosphoenolpyruvate carboxylase phosphorylation in illuminated and NH4Cl-treated protoplasts fromDigitariasanguinalis[J]. Plant J, 2000, 23(4):497-506.
[21]Vossen J H, Abd-El-Haliem A, Fradin E F, et al. Identification of tomato phosphatidylinositol-specific phospholipase-C (PI-PLC) family members and the role of PLC4 and PLC6 in HR and disease resistance[J]. Plant J, 2010, 62(2):224-239.
[22]Song F, Goodman R M. Molecular cloning and characterization of a rice phosphoinositide-specific phospholipase C gene,OsPI-PLC1, that is activated in systemic acquired resistance[J]. Physiol Mol Plant Pathol, 2002, 61(1):31-40.
[23]王法微. 水稻磷脂酶C调节耐盐性以及磷脂酶D参与种子老化的研究[D]. 南京:南京农业大学, 2011.
[24]Hirayama T, Ohto C, Mizoguchi T, et al. A gene encoding a phosphatidylinositol-specific phospholipase C is induced by dehydration and salt stress inArabidopsisthaliana[J]. Proc Natl Acad Sci USA, 1995,92(9):3903-3907.
[25]Sanchez J P, Chua N H.ArabidopsisPLC1 is required for secondary responses to abscisic acid signals[J]. Plant Cell, 2001, 13(5):1143-1154.
BioinformaticsAnalysisofPhosphoinositide-specificPhospholipaseCGeneFamilyinOrchid[Phalaenopsisequestris(Schauer)Rchb.f.]
HU Guang-long, CHEN Ya-juan, WEI Jian-hua, WANG Hong-zhi*, ZHANG Jie-wei*
(Beijing Academy of Agriculture and Forestry Sciences, Beijing Key Laboratory of Agricultural Genetic Resources and Biotechnology, Beijing 100097, China)
【Objective】The study aims to study the function ofPhalaenopsisequestrisPI-PLCgene family members in Orchid. 【Method】On the basis of orchid genome database and bioinformatics analysis, we obtained orchidPI-PLCgene family structure and their positions on chromosome and duplication information. 【Result】The results showed that 3PePLCgenes existed in the orchid genome, contained 9-13 extrons. TMHMM results showed that all PePLC proteins don’t contain transmembrane structure. Multiple alignment and motif display results indicated that all PePLC proteins contain four conserved domains (EF, X, Y and C2). 【Conclusion】The results would provide the important clues for functional studies of orchid PI-PLC proteins.
PI-PLC;Phalaenopsisequestris; Gene family; Phylogeny analysis
1001-4829(2017)10-2218-06
10.16213/j.cnki.scjas.2017.10.010
2016-12-29
北京市科技计划(Z171100001517011);北京市农林科学院青年科研基金项目(QNJJ201416);北京市农林科学院科技创新能力建设专项(KJCX20170203)
胡广隆(1981-),男,助理研究员,研究方向为分子生物学,E-mail:532476467@qq.com,*为通讯作者:王宏芝,副研究员,研究方向为林木分子生物学,E-mail:wanghongzhi@baafs.net.cn,张杰伟,副研究员,研究方向为植物抗逆生理与分子生物学,E-mail:jwzhang919@163.com。
S682.31
A
(责任编辑 李 洁)
