西藏牛Y染色体USP9Y基因与mtDNA D-loop区遗传多态性研究
2017-11-16夏小婷马志杰张成福蓝贤勇雷初朝
夏小婷,马志杰,张成福,蓝贤勇,陈 宏,雷初朝*
(1.西北农林科技大学动物科技学院,陕西 杨凌 712100;2. 青海大学畜牧兽医科学院,青海 西宁810016;3.西藏自治区农牧科学院畜牧兽医研究所,西藏 拉萨 850000)
西藏牛Y染色体USP9Y基因与mtDNA D-loop区遗传多态性研究
夏小婷1,马志杰2,张成福3,蓝贤勇1,陈 宏1,雷初朝1*
(1.西北农林科技大学动物科技学院,陕西 杨凌 712100;2. 青海大学畜牧兽医科学院,青海 西宁810016;3.西藏自治区农牧科学院畜牧兽医研究所,西藏 拉萨 850000)
本研究旨在探究西藏牛的Y染色体与mtDNA D-loop区的遗传多态性和起源.采用PCR扩增和限制性内切酶酶切的方法测定30头西藏牛Y染色体USP9Y基因的多态性;采用PCR扩增和测序技术测定30头西藏牛的mtDNA D-loop区序列,利用生物信息学的方法对西藏牛44条D-loop序列(其中14条从GenBank下载)进行遗传多态性分析.对30头西藏牛USP9Y基因的PCR产物进行电泳,共检测到471 bp和552 bp 2个片段,且552 bp片段不能被限制性内切酶Ssp I切开,说明西藏牛具有普通牛Y1和Y2 2种单倍型组,其频率分别为0.067和0.933.通过对44条西藏牛mtDNA D-loop全序列进行比对,发现本研究测定的30条西藏牛mtDNA D-loop全序列中,有8条为牦牛序列,说明西藏牛群体中有牦牛的基因渗入.对36条西藏牛mtDNA D-loop全序列进行分析,共检测到62个变异位点,界定了21个mtDNA单倍型,单倍型多样度为0.956 0,核苷酸多样度为0.006 4,表明西藏牛具有丰富的母系遗传多态性.系统发育树显示, 西藏牛具有普通牛和瘤牛混合母系起源,且普通牛的单倍型在西藏牛群体中占绝对优势(95.24%).西藏牛具有丰富的母系遗传多态性,由2个普通牛父系单倍型组构成,应归于普通牛一类,且群体中存在牦牛和瘤牛的基因渗入现象.
西藏牛;Y染色体;USP9Y;mtDNA D-loop;遗传多态性;起源
黄牛是指除牦牛和水牛之外的家牛的总称. 家牛属包括欧洲普通牛(Bos taurus)和瘤牛(Bos indicus)2个牛种.西藏牛主要分布在西藏农区和半农半牧区,是世界上适应高海拔地区的普通牛种之一,具有体型小、耐寒、适应性和抗逆性强等特点[1].目前,对西藏牛品种的起源和遗传多态性研究,研究者已在蛋白多态性[2]、染色体[3]及体型外貌[4]等多个不同层面开展了大量研究工作.
Y染色体分子遗传多态性和线粒体DNA(mtDNA)的研究结果,均支持中国地方黄牛属于普通牛和瘤牛的混合起源,提出北方黄牛受普通牛影响大,南方黄牛受瘤牛的影响大,中原黄牛同时受到普通牛和瘤牛影响的观点[5-11].基于Y染色体单核苷酸多态性(Y-SNPs)的研究,提出普通牛起源的家牛具有Y1和Y2单倍型组,瘤牛起源的家牛具有Y3单倍型组[6-7].Bonfiglio等[7]首次利用PCR技术对家牛Y染色体上USP9Y基因片段进行扩增,仅通过琼脂糖凝胶电泳和限制性内切酶处理该基因的PCR产物,即可有效地区分家牛的3种Y染色体单倍型组.mtDNA因其遗传过程遵循严格的母系遗传方式,不受公畜杂交改良的影响,可真实地反映家畜的母系起源,近10年来一直是研究家畜起源进化的重要分子标记[8-11].
由于西藏牛是比较特殊的一支,分布在高海拔地区,其父系和母系遗传多态性的研究尚未见大量报道.本研究利用USP9Y标记对西藏牛进行Y染色体单倍型组分析,并测定其mtDNA D-loop全序列,分别探究西藏牛的父系和母系起源及分子遗传多态性,为西藏牛的保种和改良提供理论依据.
1 材料与方法
1.1 样本采集与基因组DNA的提取 从西藏自治区昌都地区采集30头西藏牛耳组织样本,于-80℃保存.采用基因组DNA快速提取试剂盒(北京艾德莱生物科技有限公司)提取基因组DNA,-20℃保存备用.
1.2 USP9Y基因PCR扩增与酶切分析 引用Bonfiglio等[7]发表的家牛Y染色体的USP9Y基因的1对引物,上游引物:5′-AAACCCTTCAAGGT AATAAAACAAAA-3′,下游引物:5′-CACAGCTC CTCAAAACCAGA-3′,由生工生物工程(上海)股份有限公司合成.PCR反应体系为12.5 μL,其中上、下游引物各0.25 μL(10 pmol/L),2XTaq MasterMix 6.25 μL, 模 板 DNA 1 μL,ddH2O 5.25 μL.扩增条件为95℃预变性4 min;94℃变性30 s,57℃退火45 s,72℃延伸30 s,36个循环;最后72℃延伸10 min,4℃保存.
用1%的琼脂糖凝胶对西藏牛USP9Y基因的PCR产物进行电泳检测,根据扩增条带对单倍型做出初步分析.再用Ssp I限制性内切酶对PCR产物进行酶切.Ssp I酶切体系为8 μL,其中Ssp I酶(10 units/μL)(TaKaRa)0.4 μL,10XSsp I Buffer 0.8 μL,PCR 产物 2.4 μL,ddH2O 4.4 μL.置于37℃条件下酶切2~3 h后,用1%琼脂糖凝胶对酶切产物进行电泳检测,再根据条带大小进行单倍型分析和统计.
1.3 mtDNA D-loop的扩增、序列测定与数据处理 根据雷初朝等[12]发表的黄牛mtDNA D-loop全序列的引物序列合成正反向引物.正向引物:5′-CTGCAGTCTCACCATCAACC-3′,反向引物:5′-GGGGTGTAGATGCTTGC-3′.PCR 反应体系为20 μL.扩增条件为95℃预变性4 min;94℃变性30 s,59℃退火60 s,72℃延伸90 s,40个循环;最后72℃延伸10 min,4℃保存.采用1.0% 琼脂糖凝胶检测PCR产物,将符合要求的PCR产物送至西安擎科泽西生物科技有限责任公司测序.
在本研究中,44条西藏牛mtDNA D-loop序列被用于系统发育关系的分析,其中30条序列是本研究所测序,其余14条是从GenBank下载的( 登 陆 号:AY378114 ~ AY378120、EU281505 ~EU281511).所测序列利用DNASTAR软件进行同源序列比对和编辑,以确保所测序列的准确性.利用MEGA5.0和DnaSP5.0软件统计单倍型及变异位点,并构建邻接系统发育树(Neighbor-Joining Tree).
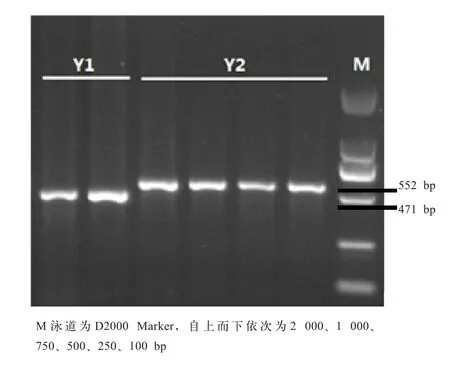
图1 西藏牛USP9Y基因PCR产物酶切后电泳图谱
2 结果与分析
2.1 西藏牛USP9Y基因扩增多态性与Y染色体单倍型组频率 通过对西藏牛USP9Y基因部分片段扩增结果显示,在30个样本中,有2头牛的PCR产物为471 bp,其余28头牛的PCR产物均为552 bp,且不能被Ssp I酶切开(图1).根据Bonfiglio等[7]的判定标准,Y1单倍型组的PCR产物(471 bp)可直接通过琼脂糖凝胶电泳与Y2、Y3单倍型组(552 bp)区分开;而Y3单倍型组的PCR产物能被Ssp I酶切成2条带(215、338 bp),Y2单倍型组不能被Ssp I酶切开,以此进行Y2和Y3的鉴别.依据以上判定标准,30头西藏牛中,有2头属于普通牛Y1单倍型组,另外28头均为普通牛Y2单倍型组.对30头西藏牛个体进行Y1和Y2单倍型组频率统计表明,30头西藏牛中,Y1单倍型组个体数为2头,所占频率为0.067;Y2单倍型组的个体数为28头,所占频率为0.933.
2.2 西藏牛mtDNA D-loop区核苷酸变异与单倍型多样度 对测定的30条西藏牛mtDNA D-loop区序列以及从GenBank下载的14条西藏牛mtDNA D-loop区序列分析表明,本研究测得的30头西藏牛中有8个个体显示为牦牛序列,比对时将其排除在外.由于碱基插入与缺失的存在,其余36个个体的mtDNA D-loop区全长为909~911 bp(图2).共检测到多态位点62个,占全序列的6.81%(62/910),其中转换59个(A/G转换22个,C/T转换37个),颠换2个(C/G颠换),在16 057 bp处发现G/C/A转换与颠换共存.
以Anderson等[13]测定的欧洲牛mtDNA D-loop全序列(GenBank登录号:V00654)为标准,利用DnaSP5.0软件对西藏牛36个个体的mtDNA单倍型进行多样性分析(图2),结果表明,西藏牛共有21种单倍型,单倍型多样度(Hd)为0.956 0,平均核苷酸多样度(Pi)为0.006 4,表明西藏牛的遗传多态性非常丰富.对比检测到的21种单倍型,只有1种属于瘤牛单倍型,其包含个体数为1;其余20种都是普通牛单倍型,共包含35个个体,说明普通牛的mtDNA D-loop单倍型在西藏牛群体中占绝对优势(95.24%).
2.3 西藏牛mtDNA单倍型的系统发育树的构建 以Anderson等[13]测定的欧洲普通牛(GenBank登录号:V00654)和Loftus等[8]测定的印度瘤牛(GenBank登录号:L27733)为标准序列,用MEGA软件采用Kimura(1980)双参数模型计算西藏牛mtDNA单倍型间的遗传距离,构建各单倍型间的邻接树(NJ-Tree)(图3).结果显示,西藏牛明显分为普通牛和瘤牛两大支系,普通牛单倍型所占比例为20/21,只有1个单倍型属于瘤牛单倍型(1/21),提示西藏牛为普通牛和瘤牛混合母系起源.
3 讨 论

图2 西藏牛36个mtDNA D-loop单倍型及其多态位点
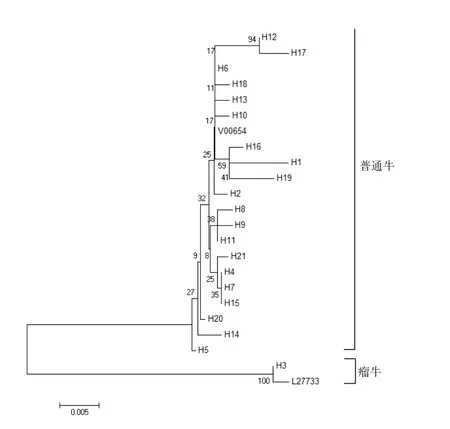
图3 西藏牛21个mtDNA D-loop单倍型构建的NJ系统树
通过Y-SNPs和Y-STRs的研究,表明家牛有Y1、Y2、Y3 3种单倍型组[5~7,14].Bonfiglio等[7]发现,利用Y染色体USP9Y基因内含子26上的1个81 bp的插入和1个限制性酶切位点进行琼脂糖电泳能够直接对这3种Y染色体单倍型组进行分型,不需要测序,既分型方便又大大降低了研究成本.本研究利用这一方法对西藏牛30个个体进行父系遗传多态性检测的结果表明,在30头西藏牛中,2头为普通牛Y1单倍型组,28头为普通牛Y2单倍型组,Y1和Y2所占频率分别为0.067和0.933,Y2单倍型组明显占优势,说明西藏牛有2个父系起源,且都属于普通牛血统.在世界现存的普通牛中,中东和欧洲南部的普通牛大都属于Y2单倍型组,而Y1单倍型组在欧亚大陆北部更为普遍[6].而中国黄牛主要包含Y2和Y3 2种Y染色体单倍型组[15],西藏牛Y1单倍型组的发现可能与近些年来藏区人民通过引进大量国外品种与西藏本地牛进行杂交有关[16].
对30条西藏牛mtDNA D-loop区全序列以及从GenBank下载的14条D-loop序列进行比对后发现,本研究所测得的30条序列中有8条属于牦牛序列.将这8个mtDNA D-loop区显示为牦牛序列个体的Y染色体单倍型组与之对应后分析发现,其全部属于Y2单倍型组.由于西藏牛主要分布在西藏雅鲁藏布江中下游、喜马拉雅山东段和三江流域下游地区,而西藏也是驯养牦牛最早的地区,根据检测出的8个牦牛序列推测西藏牛与当地的牦牛存在较多的基因交流.本研究对其余36条西藏牛D-loop区909~911 bp序列进行分析,共发现62个核苷酸变异位点,其中转换59个,颠换2个,转换与颠换共存位点1个.雷初朝等[12]报道的中国8个黄牛品种mtDNA D-loop区序列的核苷酸多样度(Pi)为0.005 5~0.053 9.本研究检测到西藏牛Pi为0.006 4,说明西藏牛mtDNA D-loop区核苷酸多态性比较丰富.西藏牛单倍型多样度为0.956 0,与蔡欣等[17]研究的中国17个黄牛品种的单倍型多样度(0.919)相比要高,表明西藏牛线粒体DNA遗传多态性丰富.在西藏牛36个个体中共检测到21种单倍型,只有1条序列(GenBank登录号:AY378119)属于瘤牛单倍型,其余35个个体共享20个普通牛单倍型,且本研究所测得的西藏牛都属于普通牛单倍型,提示西藏牛为普通牛和瘤牛的混合母系起源,但以普通牛为主.Lei等[11]基于mtDNA D-loop区序列变异的研究,也证实了西藏牛有普通牛和瘤牛2种母系起源,但瘤牛所占数量同样较少(12.5%、1/8).陈幼春等[4]认为,西藏牛是无肩峰的小个子牛,在瘤牛进入印度前就已存在,是原牛保留在南亚边缘未迁移去西方的古老牛种的后裔.陈智华等[2~3]通过对西藏牛血液蛋白多态性研究表明,西藏牛与蒙古牛、安西牛为一类,属于普通牛种,但与中国北方黄牛品种并不同源;西藏牛Y染色体为中部着丝粒染色体,与普通牛的Y染色体一致,判断其属于普通牛型.而西藏地区的黄牛品种主要包括西藏牛、阿沛甲咂牛、日喀则驼峰牛和樟木牛,除西藏牛外,其他3个黄牛品种都含有瘤牛血统[1].所以本研究认为,西藏牛mtDNA检测到少量瘤牛血统,可能与当地含有瘤牛血统的黄牛存在基因交流所致.
西藏以其特殊的地理位置、高海拔的生态条件孕育了极其丰富的牛种资源,包括黄牛和牦牛2个家养牛种.本文首次从父系和母系2个角度探讨了西藏牛的起源和遗传多态性,基于Y-SNPs和mtDNA D-loop研究,认为西藏牛具有丰富的遗传多态性,应归于普通牛一类,且群体中存在牦牛和瘤牛的基因渗入现象.西藏牛虽在适应严酷的自然条件、耐粗饲和抗病力强等方面有较多优点,但属原始低产品种,产乳和产肉等方面都不能满足人们的生活需要.西藏牛产区的牧民比较注重对母牛的选择,而不重视公牛的选育,且公母混群放牧,近亲繁殖现象较严重[1].近年来人们为了改良西藏牛品种,通过引进大量国外品种与西藏本地牛进行杂交[16],加上人们对种质资源保护的意识淡薄,使得西藏牛的群体遗传组成发生变化,因此加大西藏牛的保种力度迫在眉睫.
[1] 张沅. 中国畜禽遗传资源志‐牛志[M]. 北京: 中国农业出版社, 2011.
[2] 陈智华, 钟金城, 邓晓英, 等. 西藏牛血液蛋白多态性的研究[J]. 中国牛业科学,1995, (S1): 80‐83.
[3] 陈智华, 钟金城, 邓晓莹, 等. 西藏牛染色体的研究[J]. 中国牛业科学, 1995, (4): 1‐2.
[4] 陈幼春, 王毓英, 曹红鹤, 等. 中国黄牛生态种特征及其利用方向[M]. 北京: 农业出版社, 1990: 1‐124.
[5] Ginja C, Telo d G L, Penedo M C. Y chromosome haplotype analysis in Portuguese cattle breeds using SNPs and STRs[J]. J Hered, 2009, 100(2): 148‐157.
[6] Götherström A, Anderung C, Hellborg L, et al. Cattle domestication in the Near East was followed by hybridization with aurochs bulls in Europe[J]. P Roy Soc Lond B Bio, 2005, 272(1579): 2345‐2350.
[7] Bonfiglio S, De G A, Tesfaye K, et al. A novel USP9Y polymorphism allowing a rapid and unambiguous classification of Bos taurus Y chromosomes into haplogroups[J]. Anim Genet, 2012, 43(5): 611‐613.
[8] Loftus R T, Cunningham P. Evidence for two independent domestications of cattle[J]. PNAS, 1994, 91(7): 2757‐2761.
[9] Jia S G, Zhou Y, Lei C Z, et al. A new insight into cattle′s maternal origin in six Asian countries[J]. J Genet Genomics, 2010, 37(3): 173‐180.
[10] Achilli A, Bonfiglio S, Olivieri A, et al. The multifaceted origin of taurine cattle reflected by the mitochondrial genome[J]. PLoS One, 2009, 4(6): e5753.
[11] Lei C Z, Chen H, Zhang H C, et al. Origin and phylogeographical structure of Chinese cattle[J]. Anim Genet, 2006, 37(6): 579‐582.
[12] 雷初朝, 陈宏, 杨公社, 等. 中国部分黄牛品种mtDNA遗传多态性研究[J]. 遗传学报, 2004, 31(1): 57‐62.
[13] Anderson S, de Bruijn M H, Coulson A R, et al. Complete sequence of bovine mitochondrial DNA conserved features of the mammalian mitochondrial genome[J]. J Mol Biol,1982, 156(4): 683‐717.
[14] Edwards C J, Gaillard C, Bradley D G, et al. Y‐specific microsatellite polymorphisms in a range of bovid species[J]. Anim Genet, 2000, 31(2): 127.
[15] Li R, Xie W M, Chang Z H, et al. Y chromosome diversity and paternal origin of Chinese cattle[J]. Mol Biol Rep,2013, 40(12): 6633‐6636.
[16] 晋美加措. 西藏牛改良工作现状及建议[J]. 中国畜牧业,2014, (10): 37‐39.
[17] 蔡欣, 陈宏, 雷初朝, 等. 中国17个黄牛品种mtDNA变异特征与多态性分析[J]. 中国生物化学与分子生物学报,2007, 23(8): 666‐674.
Genetic Diversity of Y Chromosome USP9Y Gene and Mitochondrial DNA D-loop Region in Tibetan Cattle
XIA Xiao‐ting1, MA Zhi‐jie2, ZHANG Cheng‐fu3, LAN Xian‐yong1, CHEN Hong1, LEI Chu‐zhao1*
(1. College of Animal Science and Technology, Northwest A&F University, Shaanxi Yangling 712100, China; 2.Academy of Animal Science and Veterinary Medicine, Qinghai University, Qinghai Xining 810016, China; 3. Institute of Animal Science, Tibet Academy of Agricultural and Animal Husbandry Sciences, Tibet Lhasa 850009, China)
This study is to investigate the genetic diversity and the origins of Y chromosome and mitochondrial DNA D‐loop region in Tibetan cattle. Using PCR amplification and restriction enzyme digestion methods to detect the polymorphism of Y chromosome USP9Y gene of 30 Tibetan cattle; the complete D‐loop region sequences of mitochondrial DNA from 30 individuals in Tibetan cattle were determined by PCR amplification and sequencing methods. Bioinformatics method was used to analyze the genetic polymorphism of 44 D‐loop sequences in Tibetan cattle (of which 14 sequences of Tibetan were obtained from GenBank). 30 individuals of Tibetan cattle were analyzed using a recently discovered USP9Y marker that could distinguish taurine from zebu more efficiently. The results showed that the USP9Y sequence‐tagged sites of Tibetan cattle were detected two DNA fragments, 471 bp and 552bp, and the 552 bp DNA fragment can't be digested by SspI enzyme,indicating that Tibetan cattle only had Y1 and Y2 haplogroups. The haplogroup frequencies of Y1 and Y2 were 0.067 and 0.933, respectively. To compare with 44 individuals of the complete mitochondrial DNA D‐loop region in Tibetan cattle, 8 individuals were proved to belong to yak which detected in this study, indicating the introgression of genes from the yak. 62 mutation sites and 21 haplotypes were found in 36 individuals of complete mitochondrial DNA D‐loop sequences of Tibetan cattle. Nucleotide diversity was 0.0064 and haplotype diversity was 0.9560, suggesting an abundant mitochondrial genetic diversity in Tibetan cattle. Phylogenetic analysis showed that the maternal origins of Tibetan cattle were from Bos taurus and Bos indicus, and the taurine haplotypes dominated the Tibetan cattle population (95.24%). Our results demonstrated that Tibetan cattle have rather abundant mitochondrial genetic diversity, consisting of two taurine paternal haplogroups. The Tibetan cattle should be classified as Bos taurus, and our study also revealed a female yak and zebu introgression in Tibetan cattle.
Tibetan cattle; Y chromosome; USP9Y; Mitochondrial DNA D‐loop; Genetic diversity; Origins
S823.2
A
10.19556/j.0258-7033.2017-11-030
2017-04-10;
2017-05-18
国家肉牛牦牛产业技术体系资助(CARS-37)
夏小婷(1991-),女,山东威海人,硕士生,主要从事动物遗传资源研究,E-mail:xiaxiaoting1991@163.com
*通讯作者:雷初朝(1968-),男,湖南常宁人,教授,博导,主要从事牛遗传资源研究, E-mail:leichuzhao1118@126.com
