通过CRISPR/Cas9技术创制基因编辑山羊和绵羊模型
2017-11-16王小龙陈玉林
黄 玉,蔡 蓓,王小龙,陈玉林
(西北农林科技大学动物科技学院,陕西 杨凌 712100)
编者按

2017年《中国畜牧杂志》全面改版.为更好展示畜牧学领域的科技前沿,促进学术交流,本刊特别邀请不同领域的专家或青年骨干,围绕各自的研究领域,介绍自己、团队或国际研究机构近几年的研究成果、存在问题及研究展望,或就当前的研究热点问题进行讨论和评论.2017年第一期自副主编开始,将为读者带来相关综述,希望能引发读者对相关热点、难点的思考,并进行深入探讨.
通过CRISPR/Cas9技术创制基因编辑山羊和绵羊模型
黄 玉,蔡 蓓,王小龙,陈玉林*
(西北农林科技大学动物科技学院,陕西 杨凌 712100)
山羊和绵羊不仅是重要的经济动物,更是基因编辑的模式动物.基因编辑山羊、绵羊的研究对动物生产性能改善、生物医学和生物制药的研究都具有重要的意义.羊的基因编辑技术兴起于20世纪80年代,Hamme等[1]将鼠的金属硫蛋白基因和人生长激素基因通过显微注射技术导入绵羊原核期胚胎中,成功获得了世界上第1只用显微注射法制备的转基因绵羊,这种显微注射DNA片段的方法建立了绵羊基因编辑的概念,但该方法的基因编辑效率较低、靶基因的整合具有随机性、表型不可预知;至20世纪90年代,体细胞核移植技术的出现,再次掀起了基因编辑的热潮,Wilmut等[2]通过成年动物体细胞克隆法获得了第1只基因编辑绵羊;2000年以后,慢病毒系统、转座子、RNA干扰、精子介导法以及位点特异性重组酶等新技术的兴起和应用,大大推进了动物基因编辑研究与应用的进程;迄今,已发现ZFNs、TALENs和CRISPR/Cas9等限制性核酸内切酶可在基因组内特定位点造成双链断裂,尤其是2012年CRISPR/Cas9系统的广泛应用,让生产靶向基因编辑的动物成为了可能[3].截至2015年,基于CRISPR/Cas9系统在小鼠[4]、猪[5]、羊[6-7]等哺乳动物上已有一些成功案例.本文主要综述2015年以来本课题组基于CRISPR/Cas9系统开展基因编辑山羊和基因编辑绵羊的研究情况,并与国内外的同类研究进行了比较和前景分析.
1 山羊、绵羊基因编辑靶基因的选择
2014年,本课题组选择陕北白绒山羊和宁夏肉裘兼用的滩羊(绵羊)作为基因编辑模式动物,旨在通过CRISPR/Cas9系统制备具备多个优良性状的基因编辑山羊和绵羊,构建相应的核心育种群.在山羊靶基因选择时,选取了具有抑制绒毛生长功能的成纤维细胞生长因子5(Fibroblast Growth Factor‐5,FGF5)基因[8]和已在小鼠[9]、牛[10]等多个物种上证实具有抑制肌肉发育功能的肌肉生长抑制素(Myostatin,MSTN)基因作为靶向敲除的目标基因;在绵羊靶基因选择时,选择了MSTN基因、影响脂肪颜色的β-胡萝卜素双脱氧加氢酶(β‐carotene‐9′,10′‐dioxygenase,BCO2)基因和影响毛色的刺鼠信号蛋白(Agouti Signal Protein,ASIP)作为目标基因.
2 基因编辑山羊、绵羊的制备效率
针对上述山羊、绵羊的基因编辑靶基因,本课题组设计了相应的sgRNA,经过sgRNA筛选,进行细胞水平切割效率实验、胚胎sgRNA和Cas9 mRNA显微注射效果实验,证明靶基因的sgRNA在细胞水平的切割效率可达50%~80%,羊原核胚胎(受精卵1细胞期)sgRNA和Cas9 mRNA的显微注射浓度分别为5、20 ng/μL时切割效果较为理想.2014年10月-11月,按照图1所示,技术路线进行了原核胚胎微注射和胚胎移植.2015年3月-5月,分别获得山羊(陕北白绒山羊)羔羊98只(活羔93只),绵羊(宁夏滩羊)羔羊54只(活羔36只).在基因编辑山羊羔中,FGF5基因编辑阳性羊占21.4%(21/98,包括5只流产死羊),MSTN基因编辑阳性羊为15.3%(15/98),FGF5和MSTN双基因突变的阳性羊为10.2%(10/98)[11];在基因编辑绵羊羔中,MSTN阳性羊27.8%(10/36),ASIP阳性羊33.3%(12/36),BCO2阳性羊27.8%(10/36)[12].同时还对基因编辑阳性羊的生殖细胞进行了PCR扩增、T7E1酶切和Sanger测序分析,充分证明了基因编辑个体可以稳定地将突变遗传给下一代[13].
2014年陈创夫团队利用CRISPR/Cas9系统结合体细胞核移植技术获得了3只基因编辑的山羊,这3只羔羊均为基因编辑羊;在此之前连正兴课题组[5]也用此方式获得了35只绵羊,但MSTN单基因编辑的阳性率仅为5.7%(2/35).分析表明,通过胚胎显微注射法获得基因编辑羊的成功率较高、制作成本较低,详见表1.通过将CRISPR/Cas9系统显微注射到1细胞期原核胚胎后可在24 h内进行胚胎移植,此方法可用于大规模创制基因编辑羊,值得关注的问题是获得的基因编辑阳性个体中嵌合体羔羊比例较高.相比较而言,基因编辑结合体细胞核移植的方法的基因编辑效率很低,一旦胚胎移植获得成功,其基因编辑的阳性率可达100%.
上述基因编辑效率实验结果低于在小鼠[4]、兔子[14]、猪[5]、狗[15]、猴子[16-17]等哺乳动物中的编辑效率.2013年使用Cas9 mRNA和sgRNA进行小鼠基因编辑的单基因编辑个体阳性率已达到60%~95%,2014年在基因编辑猪的创制中达到63%的单基因阳性率,2015年创制基因编辑猴子的单基因阳性率为23%~61%,2015年国外的团队创制基因编辑绵羊的阳性率为45.45%(10/22)[6],而本研究的单基因编辑阳性率仅为15%~33%.目前研究人员不断优化Cas9系统使其具备更高的特异性和编辑效率[18],在小鼠上的研究表明使用Cas9蛋白替代Cas9 mRNA进行胚胎注射能大幅度提高编辑效率[19],2016年在猪上的研究发现使用电穿孔转染Cas9蛋白的方式不仅可以提高编辑效率,减小胚胎注射对胚胎的损伤,还可获得纯合的阳性个体[20].基因的改变、sgRNA的效率等因素都会影响创制基因编辑效率,与小鼠、兔子、猪等一胎多仔的动物相比,获得基因编辑羊的成本会更高,提高编辑效率是基因编辑羊成本下降的重要途径.
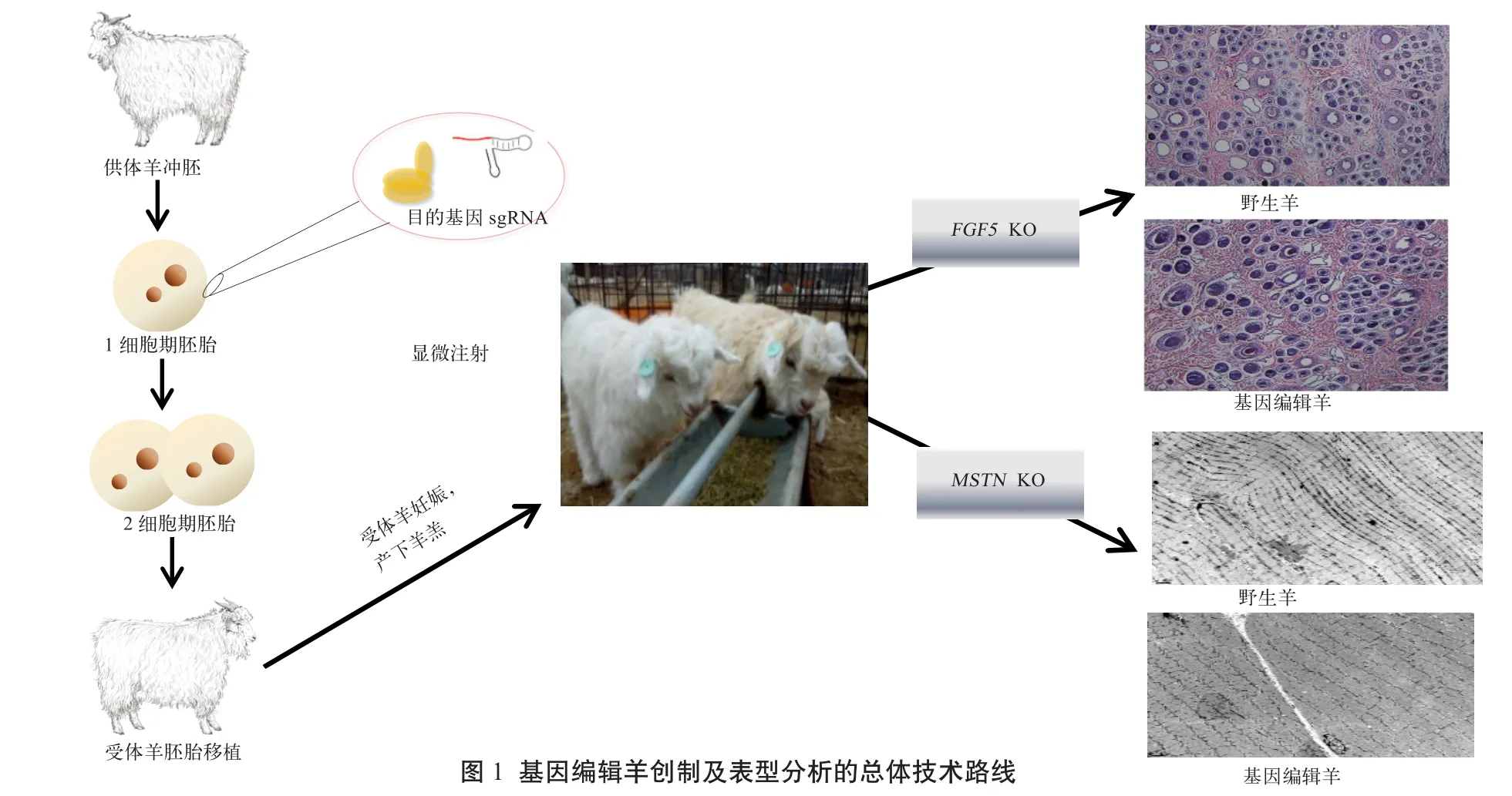
图1 基因编辑羊创制及表型分析的总体技术路线
3 基因编辑山羊、绵羊的脱靶效应
在动物基因编辑过程中,CRISPR/Cas9系统的脱靶效应问题是备受重视的技术问题和生物安全问题.理论上,脱靶效应主要由sgRNA中邻近"PAM"位点的12个碱基(称为"种子区")序列所决定.将种子序列与基因组序列进行比对,一般可筛选出0~5个错配位点,错配数越低的位点,发生脱靶的可能性越大.脱靶效应的检测是通过PCR、T7E1酶切和测序法对潜在脱靶位点进行验证,在利用CRISPR/Cas9系统制备绵羊[6]的过程中也检测到了脱靶效应的存在.有研究表明,通过选择合适的sgRNA可以有效提高CRISPR/Cas9系统的特异性,降低脱靶率.在基因编辑小鼠[4]、猪[5]、狗[15]、猴子[16-17]等动物的制备中均未检测到脱靶效应.
本研究表明,在基因编辑羊的制备过程中,脱靶效应是存在的,在MSTN基因的OT5位点和FGF5基因的OT7位点检测到脱靶效应,而基因编辑绵羊制备过程中,由于重视研究了靶基因sgRNA脱靶效应问题,事先使用SeqMap[20]对sgRNA的靶位点进行了预测,先后3批次的实验结果表明,在潜在脱靶位点未检测到脱靶突变[12],没有出现脱靶现象.
4 基因编辑山羊、绵羊的表型
在经济动物的品种培育过程中,基因工程的技术手段有助于缩短育种时间、快速提高和改善动物生产性能[21].本课题组通过CRISPR/Cas9系统创制了基因编辑阳性羊群体,针对不同基因的功能分别对不同的基因编辑阳性羊展开了研究.对10只MSTN阳性羊和10只野生型羊从出生至240 d的体重监测结果表明,MSTN阳性山羊、绵羊的体重均显著高于野生型羊,且阳性羊的肌纤维直径更宽、肌纤维横截面积更大,表明MSTN基因突变造成基因编辑羊肌肉发育能力的提高[11].这一研究结果与MSTN基因在鼠、兔子、猪、狗以及其他山羊和绵羊的研究结果相符.对FGF5阳性绒山羊0~120 d的羊绒长度的检测表明,其绒毛长度和密度指标均显著高于野生型绒山羊[13].
CRISPR/Cas9系统创制基因编辑羊的应用,也有助于建立动物疾病模型,为某些与基因型相关的动物疾病提供新的解决思路.Whitworth等[22]的研究发现,通过CRISPR/Cas9系统对CD163基因进行破坏后的猪能抵抗猪繁殖与呼吸障碍综合征病毒的感染.有研究表明,羊的黄脂性状和某些代谢疾病存在一定关联.本课题组对BCO2阳性绵羊脂肪颜色进行观测,将其与杂合型和野生型个体进行比较,发现BCO2双等位基因突变组的绵羊脂肪呈黄脂性状,表明BCO2发生双等位基因突变导致了黄脂的产生[23].
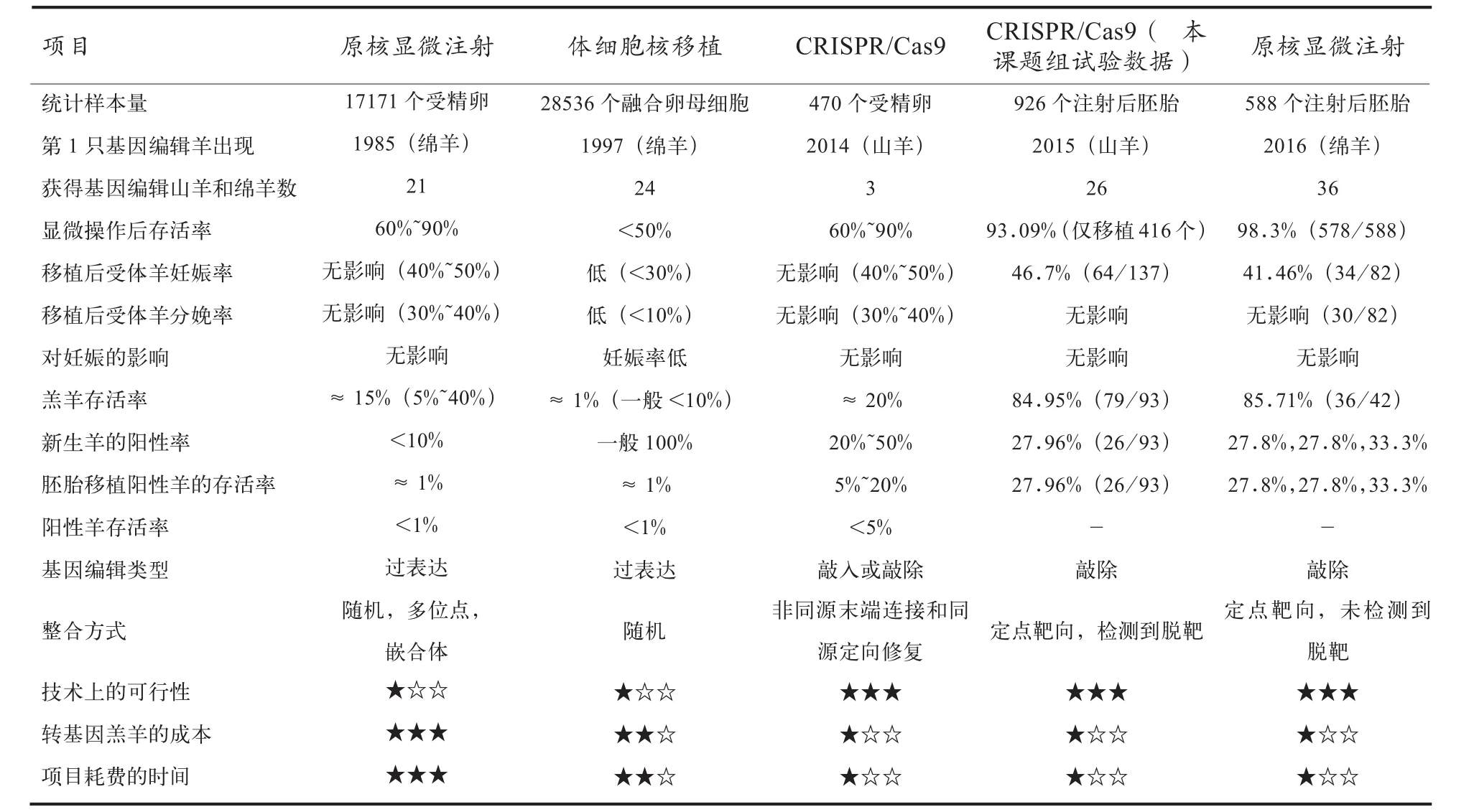
表1 基因工程产生山羊和绵羊的预期结果
如今一些借助基因工程技术生产的转基因动物已被批准用于生物医药的生产,其中包括山羊产生的生物药品ATryn1(人抗凝血酶-III);美国食物药品管理局(FDA)于2015年批准了通过基因编辑改造后生长更迅速的转基因三文鱼的上市[24].
5 前景展望
综上,CRISPR/Cas9系统的优点:一是可对特定基因进行靶向编辑;二是可同时对多个基因进行定向编辑以实现多基因聚合;三是可提高基因编辑效率和降低制备成本.不断优化CRISPR/Cas9技术体系提高基因编辑效率,结合诸如体细胞核移植技术解决基因编辑个体的嵌合体问题是基因编辑技术在羊分子聚合育种中得以推广应用的关键.除此之外,加快山羊、绵羊重要性状相关功能基因的研究是基因编辑技术应用取得实效的前提与基础.
[1] Hammer R E, Pursel V G, Jr R C, et al. Production of transgenic rabbits, sheep and pigs by microinjection[J].Nature,1985, 315(6021):680‐683.
[2] Schnieke A E, Kind A J, Ritchie W A, et al. Human factor IX transgenic sheep produced by transfer of nuclei from transfected fetal fibroblasts[J]. Science,1997,278(5346):2130‐2133.
[3] Menchaca A, Anegon I, Whitelaw C B, et al. New insights and current tools for genetically engineered (GE) sheep and goats[J]. Theriogenology, 2016, 86(1):160‐169.
[4] Wang H, Yang H, Shivalila C S, et al. One‐step generation of mice carrying mutations in multiple genes by CRISPR/Cas‐mediated genome engineering[J]. Cell, 2013,153(4):910‐918.
[5] Hai T, Teng F, Guo R, et al. One‐step generation of knockout pigs by zygote injection of CRISPR/Cas system[J]. Cell Res, 2014, 24(3):372.
[6] Crispo M, Mulet A P, Tesson L, et al. Efficient generation of myostatin knock‐out sheep using CRISPR/Cas9 technology and microinjection into zygotes[J]. PLoS One,2015, 10(8):e0136690.
[7] Ni W, Qiao J, Hu S, et al. Efficient gene knockout in goats using CRISPR/Cas9 system[J]. PLoS One, 2014,9(9):e106718.
[8] He X, Chao Y, Zhou G, et al. Fibroblast growth factor 5‐short (FGF5s) inhibits the activity of FGF5 in primary and secondary hair follicle dermal papilla cells of cashmere goats[J]. Gene, 2016, 575(2):393‐398.
[9] Hao G, Yong C, Qiu B, et al. Establishment and phenotypic analysis of an Mstn knockout rat[J]. Biochem Bioph Res Co, 2016, 477(1):115.
[10] Proudfoot C, Carlson D F, Huddart R, et al. Genome edited sheep and cattle[J]. Transgenic Res, 2015, 24(1):147‐153.
[11] Wang X, Yu H, Lei A, et al. Generation of gene‐modified goats targeting MSTN and FGF5 via zygote injection of CRISPR/Cas9 system[J]. Sci Rep U K, 2015, 5:13878.
[12] Wang X, Niu Y, Zhou J, et al. Multiplex gene editing via CRISPR/Cas9 exhibits desirable muscle hypertrophy without detectable off‐target effects in sheep[J]. Sci Rep U K, 2016, 6:32271.
[13] Lv Q, Yuan L, Deng J, et al. Efficient generation of myostatin gene mutated rabbit by CRISPR/Cas9[J]. Sci Rep‐UK, 2016, 6:25029.
[14] Zou Q, Wang X, Liu Y, et al. Generation of gene‐target dogs using CRISPR/Cas9 system[J]. J Mol Cell Biol, 2015,7(6):580‐583.
[15] Niu Y, Shen B, Cui Y, et al. Generation of gene‐modified cynomolgus monkey via Cas9/RNA‐mediated gene targeting in one‐cell embryos[J]. Cell, 2014, 156(4):836‐843.
[16] Chen Y, Zheng Y, Yu K, et al. Functional disruption of the dystrophin gene in rhesus monkey using CRISPR/Cas9[J].Hum Mol Cenet, 2015, 24(13):3764.
[17] Zhang X, Liang P, Ding C, et al. Efficient production of gene‐modified mice using Staphylococcus aureusCas9[J].Sci Rep U K, 2016, 6:32565.
[18] Qin W, Dion S L, Kutny P M, et al. Efficient CRISPR/Cas9‐mediated genome editing in mice by zygote electroporation of nuclease[J]. Genetics, 2015, 200(2):423‐430.
[19] Tanihara F, Takemoto T, Kitagawa E, et al. Somatic cell reprogramming‐free generation of genetically modified pigs[J]. Sci Adv, 2016, 2(9):e1600803‐e1600803.
[20] Hruscha A, Krawitz P, Rechenberg A, et al. Efficient CRISPR/Cas9 genome editing with low off‐target effects in zebrafish[J]. Development, 2013, 140(24):4982‐4987.
[21] Van Eenennaam A L. Genetic modification of food animals[J]. Curr Opin Biotech, 2017, 44:27‐34.
[22] Whitworth K M, Rowland R R, Ewen C L, et al. Gene‐edited pigs are protected from porcine reproductive and respiratory syndrome virus[J]. Nat Biotechnol, 2015,34(1):20.
[23] Niu Y, Jin M, Li Y, et al. Biallelic β‐carotene oxygenase 2 knockout results in yellow fat in sheep via CRISPR/Cas9[J]. Anim Genet, 2016, 48(2):242‐244.
[24] Wang X, Cai B, Zhou J, et al. Disruption of FGF5 in cashmere goats using CRISPR/Cas9 results in more secondary hair follicles and longer fibers[J]. PLoS One,2016, 11(10):e0164640.
10.19556/j.0258-7033.2017-11-001
宁夏农牧厅滩羊育种专项(NXTS201601);国家自然科学基金(31372279、31402038、31572369);陕西省重点研发计划(2017NY-072)
黄玉(1994-),女,硕士研究生,研究方向为动物遗传育种与繁殖,E-mail:1215234902@qq.com
*通讯作者:陈玉林(1964-),男,教授,研究方向为动物遗传资源研究,E-mail:chenyulin@nwafu.edu.cn
