基于线粒体COI基因的泰国近海有毒水母遗传多样性研究
2017-11-14缪晓翔张学雷刘瑞娟AUNGTONYACharatsee
缪晓翔,肖 洁,张学雷*,刘瑞娟,AUNGTONYA Charatsee
(1.国家海洋局第一海洋研究所,山东青岛266061;2.海洋生态环境科学与工程国家海洋局重点实验室,山东青岛266061;3.Phuket Marine Biological Center,Phuket 83000,Thailand)
基于线粒体COI基因的泰国近海有毒水母遗传多样性研究
缪晓翔1,2,肖 洁1,2,张学雷1,2*,刘瑞娟1,2,AUNGTONYA Charatsee3
(1.国家海洋局第一海洋研究所,山东青岛266061;2.海洋生态环境科学与工程国家海洋局重点实验室,山东青岛266061;3.Phuket Marine Biological Center,Phuket 83000,Thailand)
利用编码线粒体细胞色素氧化酶I(COI)的基因片段序列,对泰国近海有毒水母69个样本进行遗传多样性研究,并探讨该片段作为DNA条形码对泰国近海有毒水母类进行快速分类鉴定的可行性,以期为后续有毒水母的监测和预警、生物学和生态学等的研究提供技术支撑。基因碱基组成分析表明,立方水母COI序列GC含量为42.1%,明显高于钵水母(37.1%)和水螅水母(36.9%)。COI基因编码氨基酸的密码子第3位点GC含量(30.2%)显著低于第1,2位点(分别为47.4%,42.1%),且该位点碱基替换频率最高,转换/颠换比值为1.0,表明该位点碱基突变趋于饱和;但去除密码子第3位点,对系统进化树的构建无显著影响。根据COI序列计算3个纲水母的K2P(Kimura 2-parameter)遗传距离,得出种内遗传差异为0.000~0.151,均值为0.036,同纲种间遗传差异为0.167~0.321,均值为0.263,纲间遗传差异为0.246~0.385,均值为0.334。该COI基因片段能够快速有效区分这些有毒水母种类。研究表明,泰国沿岸水母多样性较高,所获69个样品可以分为13个种,包括5种钵水母、6种立方水母和2种水螅水母,其中Carukiidae科存在新属新种。立方水母的地理分布具有明显的区域特征,马来半岛东西两岸存在物种和遗传多样性差异,其产生原因还有待于进一步研究。
立方水母;钵水母;DNA条形码;COI序列;泰国
刺胞动物门(Cnidaria)中的水母亚门(Medusozoa)包含水螅虫纲(Hydrozoa)、钵水母纲(Scyphozoa)、立方水母纲(Cubozoa)和十字水母纲(Staurozoa)四个纲。其中,立方水母虽然是种类数量最少的一个纲(目前已知仅有约36种[1]),但许多种类毒性极强,例如:Chironex fleckeri被称为“海黄蜂(Sea wasp)”,人被大面积螫伤后可导致几分钟内死亡[2]。这些剧毒立方水母的广泛分布,严重影响着沿海居民生命安全及海滨旅游业的发展,在印度洋-西太平洋热带地区,剧毒立方水母蜇伤甚至使游客致死的事件频繁发生,引起了当地政府的重点关注[3-4]。仅在菲律宾沿岸的立方水母蜇死游客或渔民的发生率,每年可达20~50例,由于一些偏远地区没有统计数据,实际发生率应远高于此[4]。另外,近年来由于受人类活动及气候变化的影响,水母数量剧增引发的重大事件在全球范围内频繁爆发,例如:水母爆发堵塞发电厂的冷却系统,导致中国河北、山东等多地沿海发电厂被迫临时关闭[5-6];在日本海[7]以及地中海、大西洋等某些海域[8],近海海域水母频繁爆发严重破坏了当地浮游生态系统平衡,使海洋渔业大规模减产,造成了严重的经济损失。因此,掌握水母种群和数量的动态分布与变化,尤其是对有毒水母进行快速鉴别和区分具有重要意义。
然而,仅依据传统形态学来鉴别和区分不同的水母种类存在一定的局限性,不能直接应用于水母生物多样性和种群监测等的研究中。首先,目前水母的分类系统还不够完善,一些种类原始标本的遗失、形态描述不充分、隐种(Cryptic species)的存在等,使得一些水母科属还未明确其分类地位[9];随着研究的深入,一些新的水母种类不断被发现[10-11],但仍有许多种类没有充分描述或正式命名[12]。其次,已有研究表明一些水母的形态特征具有不确定性,例如:不同生活史时期差异甚大[13],水母的形态在不同的海域也往往有变化。而且实际调查中的很多限制因素如样品含水多,采集后易变形失真等,也增加了形态鉴定的难度。相对于传统形态学鉴定方法,DNA条形码的优势在于能以较低的成本快速获取DNA信息,大大缩短了鉴定时间,且可以监测水母从浮浪幼虫到成体的全生活阶段[14]。
自20世纪90年代开始,随着测序技术的飞速发展和广泛应用,一些水母类的核糖体基因和线粒体基因片段也逐渐被开发,并应用于系统发育和进化研究[15-17]。线粒体DNA具有结构简单、碱基替换率高、无组织特异性等优点,适于物种分类、进化关系等的分析,其中线粒体细胞色素c氧化酶I(COI)在海洋生物分子系统学研究中应用广泛。已有的许多研究证明基于COI的DNA条形码在物种鉴定、隐存种发现等方面具有强大的功能[18-20],这在水母研究中得到应用[21-22]。
近年来,泰国有毒水母蜇伤游客的事故频发[4],引起泰国政府部门的高度重视,初步调查发现泰国海域有毒水母多属于立方水母纲和钵水母纲,尤其是数个剧毒的立方水母种类尚难准确鉴定并区分其种类[23]。另外,这些水母生活史复杂、形态多变,严重影响了对其种群分布、生活史以及环境影响要素等的生态学调查研究。基于形态分类的不足和DNA条形码在物种分类上的优势,我们分析了泰国近海有毒水母COI基因片段,探讨其在水母种类鉴定方面的应用价值,以期推动线粒体COI序列片段在泰国有毒水母种群监测和研究中的应用。
1 材料与方法
1.1 样品的采集与保存
2014-2015年,西南季风季(5-10月)、东北季风季(11月至翌年4月)各采样2~3次。由于泰国近岸的剧毒水母(如立方水母)比较多,采样工作均由当地有经验的监测人员和渔民负责,大部分是利用大型浮游生物网采集,另有少量样本来自当地渔民的零星采集。
甄选有毒水母样本69个用于遗传多样性研究,其中12个采自泰国湾西部沿岸,57个来自安达曼海东部沿岸(图1)。取小块伞部或触手组织,用体积分数为95%的酒精固定,4℃保存于冰箱中,运送至实验室,2015-10开始进行DNA分析。其余组织保存于体积分数为5%的甲醛中,用于形态学的鉴定,泰方负责的形态学研究结果另文追述,表4仅列出我们研究中69个样品的形态鉴定相关信息。
1.2 DNA的提取以及PCR扩增和测序
切取每个组织样本约3 mg,用蒸馏水冲洗数次,无菌滤纸汲取样品中的水分后,用灭菌刀片将组织切碎并研磨,使用动物组织DNA提取试剂盒(美国Omega Bio-Tek公司生产E.Z.N.A.TM型)提取基因组DNA[24]。
PCR扩增在PCR仪(杭州博日科技有限公司生产Gene Touch型)上进行,COI反应体系为50μL,包括5μL 10×buffer,4μL Mg2+(25 mmol/L),4μL d NTP(10 mmol/L),2μL每条引物(10μmol/L),0.25 U Taq酶(大连宝生物工程有限公司生产),0.75μL BSA(10 mg/m L,牛血清白蛋白),2μL基因组DNA模版(约50 ng),其余用超纯水补足。扩增COI片段的反应程序:94℃预变性2 min,然后35个循环包括94℃变性45 s,48℃退火60 s,72℃延伸2 min,最后72℃延伸10 min。反应引物(表1)主要使用引物CNCOXF/R扩增,效果不佳时,采用其他2对引物扩增。

图1 采集地点和样品数量(括号内数字)Fig.1 The collecting locations and sample sizes(numbers in brackets)

表1 所用引物序列及参考文献Table 1 Sequences and references of the primers used in the study
扩增产物经琼脂糖凝胶电泳检测,验证无误后使用割胶回收试剂盒(美国Omega Bio-Tek公司生产E.Z.N.A.TM型)进行回收纯化[24]。纯化产物直接送青岛擎科梓熙生物技术有限公司使用PCR引物双向测序。部分测序困难的样品采用PMD TM 19-T(大连宝生物工程有限公司生产)连接,并转化至感受态大肠杆菌DH5α,采用麦康凯选择性培养基筛选阳性克隆,经PCR检测确认无误后,选择4~5个阳性克隆进行双向测序。
1.3 数据处理
所得原始峰图文件先经Seqman软件(DNAstar软件包)处理,进行序列拼接,并人工校正。使用Bioedit v7.0.9.0软件中内置的Clustal W对核苷酸序列进行比对分析,参数为默认值。所得序列去除引物和两端质量不高的部分,并双向进行比对,对于正反向存在不一致的序列,会重复测序,以确定正确的序列。部分样品进行连接转化后测序,所得的重复序列进行比对,并以一致共有的序列作为该样品的代表性序列,与其它样品进行比较。研究中获得水母COI片段69条,结合从GenBank数据库中下载的19条相关代表性COI序列(表2),对所得样品进行系统发育研究。比对后,去除两端较长的序列,保留621 bp可比对位点。使用MEGA 6.0软件统计碱基组成、碱基替换。计算基于K2P(Kimura 2-parameter)模型的样品间相对遗传距离(d),并按各样品的分类组别分别统计和比较种内、种间和纲间遗传距离的变化。利用无脊椎动物线粒体遗传密码翻译COI核苷酸序列,并与GenBank中已知序列进行比对,寻找正确的翻译密码框;两两比较不同序列间转换、颠换数量和转换/颠换比值(R),进一步分析R值与d之间的关系;分别计算密码子不同位点(第1,2,3位点)上的碱基含量、替换频率、R值,以及与d的关系。分别采用邻接法NJ(Neighbor-joining)和最大似然法ML(Maximum Likelihood)构建系统发育树。邻接法通过确定距离最近或相邻的成对分类单位来使系统树的总距离达到最小,而最大似然法根据碱基替换模型计算每个系统树的似然值,以似然率最大的拓扑结构作为最优树,2种方法所建的系统树可相互比较印证,并用1 000次bootstrap检验系统发育树各节点的可信度。
1.4 统计分析
分别统计各纲中COI序列的GC含量(%)及标准误差(SE),并利用SPSS16.0软件包中的单因素方差分析(one-way ANOVA)方法,检验不同纲之间GC含量是否具有显著差异。为检验不同样本间(种内、种间和纲间)R值是否趋于饱和,做R值随着遗传距离(d)增加而变化的散点图,并利用多项式回归模型做出两者之间的趋势线。

表2 从Genebank下载的水母COI基因序列信息,包括序列号、种类和资料来源Table 2 Information of the COI sequences retrieved from the GenBank database,including accession numbers,species and the references
2 结果与分析
2.1 COI基因序列碱基组成与替换统计分析
3个纲(钵水母、立方水母、水螅水母)的GC含量平均分别为37.1%(标准误差SE=0.3)、42.1%(SE=0.2)、36.9%(SE=2.2),并且不同纲间差异显著(one-way ANOVA,P<0.01),立方水母的COI序列GC含量明显高于钵水母和水螅水母(表3)。3个密码子位点的碱基组成差异也较大,第1位点较高,平均为47.4%,第2位点次之,为42.1%,第3位点最低,为30.2%,且3个纲的趋势一致。可见,密码子碱基使用频率存在明显的偏向性。
总共621 bp比对位点中,保守位点C(Conserved sites)322个,约占总数的51.9%;变异位点V(Variable sites)299个,其中简约信息位点Pi(Parsim-info sites)293个,自裔位点Si(Singleton sites)6个,分别占47.2%,1.0%。进一步对密码子各位点分别进行分析发现:碱基替换主要发生在密码子的第3位点,总共299个变异位点中,128个发生在第3位点上,占总变异位点的42.8%;对比密码子第1,2位点,第3位点颠换的发生频率较高,转换与颠换的比值(R)为1.00;密码子第1,2位点的碱基替换频率分别为4.19%,0.97%,R值分别是1.43,0.37。3个纲间的R值波动较大,立方水母和水螅水母的转换明显高于颠换,R值分别为1.34,1.33,钵水母的COI序列中,颠换较多,R值为0.93。

表3 COI基因片段GC含量(%)和转换/颠换比值(R)Table 3 G and C nucleotide composition(%)and Ts vs.Tv(R)of partial COI sequences
2.2 遗传距离计算
以K2P模型计算COI序列之间遗传距离,按照分类阶元进行统计分析(图2)。种内差异明显小于种间和纲间差异。在种内,COI序列的差异为0.000~0.151(平均为0.036),中位数为0.006。同纲种间,序列差异为0.167~0.321(平均0.263)。不同的纲间遗传差异为0.246~0.385(平均为0.334)。同纲种间遗传距离是种内遗传距离的7.3倍,而不同纲间的遗传距离是种内的9.3倍(图2)。
进一步分析密码子不同位点的碱基突变与遗传距离估算之间的关系,发现遗传距离d<0.100时,序列间转换/颠换比值(R)波动较大,且密码子第3位点的R值明显低于3个位点的综合结果;遗传距离d>0.150时,R值较小,且波动不大,第3位点的R值与综合密码子3个位点的结果无明显区别(图3)。

图2 水母COI序列不同分类阶元水平的遗传距离(d)范围箱式图Fig.2 Boxplot on the range of pairwised genetic distances(d)at the levels of intra-,inter-species and among classes
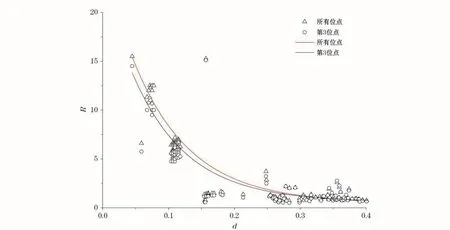
图3 R值与遗传距离(d)的散点图Fig.3 Estimated R values plotted as a function of the genetic distance(d)from pairwise comparisons of COI genes
2.3 基于COI序列的系统树分析
用NJ和ML法对本研究所得69条COI序列及19条Gen Bank数据库中代表性序列(表2)构建系统进化树,2种方法所得的进化树基本一致(图3):88条序列可以分为3大组,39条序列与6条立方水母纲下载序列(FJ665180,KT223648,JN642335,JN700970,HQ824530,JN700960)组成立方水母纲(Cubozoa);另有26条本研究所得序列和9条下载序列聚合在一起,形成钵水母纲(Scyphozoa);其余8条序列聚成水螅水母纲(Hydrozoa)。钵水母纲可进一步分为5个独立分支(图4),立方水母亦可分成6个分支,分别代表本研究中所检测获得的不同种类。去除突变率较高的密码子第3位点,仅根据第1,2位点构建系统发育树(图5),结果与保留所有位点构建的系统发育树基本相同。
2.4 泰国有毒水母的种类及分布
对研究中69个泰国近海常见有毒水母样本的分类及分布范围的统计见表4。钵水母纲26个样品中共有5个种:金黄水母(Chrysaora sp.)、海月水母(Aurelia sp.)、珍珠水母(Phyllorhiza puncatata)、沙海蜇(Stomolophus meleagris)和端棍水母(Catostylus sp.);立方水母纲39个样品,分别属于3科(Carukiidae,Chirodropidae和Chiropsalmidae),6个种(表4);水螅水母样品共有4个,属于2个种:多管水母(Aequorea sp.)和僧帽水母(Physalia sp.)。Chiropsoides buitendijki存在种内分化,根据COI序列,20个样本明显分为2个群体(图4和图5),而这2个群体的18S序列完全一致[23],属于同一种,其COI序列的分化代表种内不同遗传群体间的差异。金黄水母、Chiropsoides buitendijki数量较多,且分布范围较广,在安达曼海和泰国湾均有分布。

图4 基于COI序列的系统树(括号外为ML法检验值,括号内为NJ法检验值)Fig.4 Phylogenetic tree based on COI sequences(bootstrapping support values of MJ tree are labeled outside the brackets and values of NJ tree are inside the brackets)
2.5 立方水母纲新种的发现
立方水母纲Carukiidae科的16个样本分为4个小支,结合16S和18S数据[23],其中7个样本(NO.C1~NO.C6,NO.L3)属于Morbakka属;其余9个样本与该科内已知的4个属(Malo,Morbakka,Gerongia和Carukia)存在明显的遗传分化,结合形态学数据(Charatsee Aungtonya和肖洁未发表数据),这9个样本组成一新属,并可分为2个新种(表4)。然而,GenBank数据库中关于立方水母的基因数据不够完整,目前已有基因数据的种类数量不足总数的40%[38],而东南亚热带地区立方水母的序列数据尤为匮乏,一些样本仅依赖分子序列,并不能完全确定其种属地位,需要结合形态学数据进行系统的研究。在后续研究中,我们将与泰方研究人员合作,对一些新种进行逐个描述和命名。

图5 基于COI序列的系统树(只包含密码子第1,2位点)Fig.5 Phylogenetic tree based on COI sequences with only 1st,2nd condon positions included

表4 泰国沿海有毒水母种类和分布范围Table 4 Species and distribution of the venomous jellyfish collected from the coasts of Thailand
3 讨 论
我们对采自于泰国沿海的69个有毒水母样本的线粒体COI基因片段进行分析,碱基组成分析结果表明,3个纲线粒体COI基因的GC含量都小于50%,这符合后生动物的线粒体基因组碱基组成的特点[39],立方水母的GC含量显著高于钵水母和水螅水母纲,这与前人的结果[27]基本一致。3个密码位点的碱基组成结果表明,密码子第3位点的GC含量最小,这表明COI序列核苷酸碱基构成出现了明显的偏倚现象;并且,密码子第3位点的碱基替换速率明显高于第1,2位点,符合氨基酸编码基因第3位点进化最快的规律[40];该位点上,转换与颠换比值(R)也低于第1,2位点,尤其是水螅水母和钵水母,R<2(表3),说明该位点上碱基突变已趋于饱和(图3)。为减少由于碱基突变过饱和,而低估物种之间的遗传差异,我们采用2种方法(保留所有位点和只包含密码子第1,2位点)构建COI基因序列的系统发育树。结果表明2种方法所得的系统发育树基本一致(图4和图5),说明密码子第3位点的高突变率未对遗传距离的估算和遗传关系分析造成明显影响,该段基因适用于不同水平的物种区分和进化树构建。
通过COI基因片段的序列分析,在泰国沿海采集的69个有毒水母样本可区分为13个种,在纲的水平上,利用分子生物学与形态学手段所得的结果一致,但在种的水平上存在一些差异(表4),例如:钵水母纲中,有9个样品被鉴定为同一种向心水母(Lychnorhiza sp.),然而根据其COI序列可分为3种,分别为海月水母(Aurelia sp.)、端棍水母(Catostylus sp.)和金黄水母(Chrysaora sp.)。立方水母纲中有16个样本被鉴定为Morbakka sp.,但其COI基因序列存在明显分化,可分为3个独立分支。结合18S与16S基因结果[23],其中一支与来自于日本的Morbakka virulenta[38]相近,虽然18S基因与M.virulenta一致,但16S与COI基因存在明显差异,16S遗传距离为0.000~0.050,均值为0.009[23],研究中COI遗传距离为0.000~0.151,均值为0.036;另外,该水母的足叶管等形态特征(Charatsee Aungtonya未发表数据)与已知的M.virulenta和M.fenner[41]不同;因此,该水母可能为Morbakka属中的新种。另2个分支也属于Carukiidae科,但16S和18S序列数据表明其与该科内已知的4个属(Malo,Morbakka,Gerongia和Carukia)明显不同[23,40,42];形态分析进一步发现其眼点(rhopalia)形状和假缘膜(velarium)花纹比较特殊(Charatsee Aungtonya未发表数据)[42],也与该科内的其他4个属不同,因此,该水母为Carukiidae科中的新属,系统的形态描述和种属的建立正在进行中。上述结果也表明,研究所用的线粒体COI基因片段,可以有效区分泰国沿海常见的水母种类,这种基于线粒体COI基因片段的DNA条形码技术可以为后续有毒水母种类的生活史、监测和生态学等方面的研究提供快捷有效的技术手段。
4 结 语
我们利用线粒体COI序列对泰国沿海习见有毒水母进行遗传多样性分析,研究表明泰国沿海水母多样性较高,其中剧毒的立方水母有5种,且立方水母的地理分布呈明显的区域特征。例如:Chiropsoides buitendijk明显分为2个遗传群体(图4和图5),其中一个群体集中于攀牙府(Phany-nga Province)的Panwa Bay,而另一个群体在安达曼海东部沿岸和泰国湾均有分布;Carukiidae科新属中的2个相近种,一种只出现于安达曼海东部沿岸,另一种在泰国湾和安达曼海东部沿岸均有发现,而Morbakka sp.也只在泰国湾沿岸发现。与泰国湾海域相比,安达曼东部沿岸的水母多样性更高,其中,攀牙府附近海域的水母多样性最高,约有8种。我们的研究中来自安达曼东部沿海的样本数量高于泰国湾的,这可能对评估2个海域的物种遗传多样性产生一定的影响,后续研究中应加强对泰国湾海域的样品采集和分析。然而,一些种类或群体呈明显的局域性分布,结合往年对于泰国近岸有毒水母分布的调查数据[43]分析,马来半岛(Malay Peninsula)东西两岸的水母存在物种和遗传多样性差异。这种差异是否在浮游生物中普遍存在,是否是2海域的地理隔离造成的,有待于进一步研究。
[1] DALY M,BRUGLER M R,CARTWRIGHT P,et al.The phylum Cnidaria:A review of phylogenetic patterns and diversity 300 years after Linnaeus[J].Zootaxa,2007,1668:127-182.
[2] FENNER P J,HARRISON S L.Irukandji and Chironex fleckeri jellyfish envenomation in tropical Australia[J].Wilderness and Environmental Medicine,2000,11:233-240.
[3] XIAO L,ZHANG L M.Research development of jellyfish toxins[J].Chinese Journal of Marine Drugs,2007,26(6):40-43.肖良,张黎明.水母毒素的研究进展[J].中国海洋药物,2007,26(6):40-43.
[4] FENNER P J,LIPPMANN J,GERSHWIN L A.Fatal and nonfatal severe jellyfish stings in Thai waters[J].Journal of Travel Medicine,2010,17(2):133-138.
[5] LIU X,LI D G.When the jellyfish hit rough[J].North China Power,2008(4):66-69.刘旋,李德刚.当海蛰群汹涌袭来[J].华北电业,2008(4):66-69.
[6] DONG Z,LIU D,KEESING J K.Jellyfish blooms in China:dominant species,causes and consequences[J].Marine Pollution Bulletin,2010,60(7):954-963.
[7] UYE S I,FUJII N,TAKEOKA H.Unusual aggregations of the scyphomedusa Aurelia aurita in coastal waters along western Shikoku,Japan[J].Plankton Biology and Ecology,2003,50(1):17-21.
[8] LICANDRO P,CONWAY D V P,DALY YAHIA M N,et al.A blooming jellyfish in the northeast Atlantic and Mediterranean[J].Biology Letters,2010,6(6):688-691.
[9] DAWSON M N.Some implications of molecular phylogenetics for understanding biodiversity in jellyfishes,with emphasis on Scyphozoa[J].Hydrobiologia,2004(530/531):249-260.
[10] HUANG J Q,XU Z Z,LIN M,et al.Two new species of genus Nubiella from the Taiwan Strait,China(Filifera,Bougainvillidae)[J].Journal of Xiamen University(Natural Science),2012,51(1):130-133.黄加祺,许振祖,林茂,等.台湾海峡单肢水母属二新种(丝螅水母目,高手水母科)[J].厦门大学学报(自然科学),2012,51(1):130-133.
[11] HUANG J Q,XU Z Z,LIN M,et al.Two new species of Suborder Tubulariida from the South China Sea(Anthomedusae,Capitata)[J].Journal of Xiamen University(Natural Science),2015,54(6):825-828.黄加祺,许振祖,林茂,等.南海筒螅水母亚目二新种(花水母亚纲,头螅水母目)[J].厦门大学学报(自然科学),2015,54(6):825-828.
[12] BENTLAGE B.Phylogenetic systematics,taxonomy and biogeography of jellyfish(Cnidaria:Medusozoa)[D].Larence:The University of Kansas,2012.
[13] ORTMAN B D,BUCKLIN A,PAGES F,et al.DNA Barcoding the Medusozoa using mtCOI[J].Deep Sea Research Part II:Topical Studies in Oceanography,2010,57(24):2148-2156.
[14] CHENG F P,WANG X M,WANG Y T,et al.DNA barcoding of common Medusozoa in northern China based on mtCOI sequence[J].Oceanologia et Limnologia Sinica,2012,43(3):451-459.程方平,王晓敏,王彦涛,等.中国北方习见水母类的DNA条形码分析[J].海洋与湖沼,2012,43(3):451-459.
[15] BRIDGE D,CUNNINGHAM C W,SCHIERWATER B,et al.Class-level relationships in the phylum Cnidaria:evidence from mitochondrial genome structure[J].Proceedings of the National academy of Sciences,1992,89(18):8750-8753.
[16] HORI H,SATOW Y.Dead-end evolution of the Cnidaria as deduced from 5S ribosomal RNA sequences[J].Hydrobiologia,1991,216(1):505-508.
[17] ODORICO D M,MILLER D J.Variation in the ribosomal internal transcribed spacers and 5.8S r DNA among five species of Acropora(Cnidaria;Scleractinia):patterns of variation consistent with reticulate evolution[J].Molecular Biology and Evolution,1997,14(5):465-473.
[18] BALL S L,HEBERT P D N,BURIAN S K,et al.Biological identifications of mayflies(Ephemeroptera)using DNA barcodes[J].Journal of the North American Benthological Society,2005,24(3):508-524.
[19] JOHNSON S B,WAREN A,VRIJENHOEK R C.DNA barcoding of Lepetodrilus limpets reveals cryptic species[J].Journal of Shellfish Research,2008,27(1):43-51.
[20] SCHLEI O L,CREETE-LAFRENLERE A,WHITELEY A R,et al.DNA barcoding of eight North American coregonine species[J].Molecular Ecology Resources,2008,8(6):1212-1218.
[21] ZHANG S,ZHANG F,LIU Y,et al.Molecular identification of two macro-jellyfish in China[J].Oceanologia et Limnologia Sinica,2009,40(1):94-101.张姝,张芳,刘媛,等.我国海域两种大型水母的分子鉴定[J].海洋与湖沼,2009,40(1):94-101.
[22] ZHENG L M,LIN Y S,LI S J,et al.Morphological and molecular evidences of Aequorea taiwanensis n.sp.from Taiwan Strait,with mtCOI sequence analysis for genus Aequorea[J].Haiyang Xuebao,2008,30(4):139-146.郑连明,林元烧,李少菁,等.台湾海峡多管水母属——新种及基于线粒体COI序列分析鉴定多管水母[J].海洋学报,2008,30(4):139-146.
[23] LIU R J,XIAO J,ZHANG X L,et al.Genetic analysis of common venomous Cubozoa and Scyphozoa in Thai waters based on 16Sand 18S r DNA sequences[J].Haiyang Xuebao,2016,38(6):51-61.刘瑞娟,肖洁,张学雷,等.泰国近海习见有毒立方水母和钵水母的遗传分析[J].海洋学报,2016,38(6):51-61.
[24] OMEGA BIO-TEK.E.Z.N.ATMprotocol for tissue[EB/OL].(2012-03)[2016-08-15].https:∥www.omegbiotek.com.
[25] FOLMER O,BLACK M,HOEH W,et al.DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J].Molecular Marine Biology and Biotechnology,1994,3(5):294-299.
[26] SMITH D R,KAYAL E,YANAGIHARA A A,et al.First complete mitochondrial genome sequence from a box jellyfish reveals a highly fragmented linear architecture and insights into telomere evolution[J].Genome Biology and Evolution,2012,4(1):52-58.
[27] KAYAL E,BENTLAGE B,COLLINS A G,et al.Evolution of linear mitochondrial genomes in medusozoan cnidarians[J].Genome Biology and Evolution,2011,4(1):1-12.
[28] NCBI(National Center for Biotechnology Information).GenBank.[DB/OL].[2016-08-15].https:∥www.ncbi.nlm.nih.gov/genbank.
[29] LEWIS C,BENTLAGE B.Clarifying the identity of the Japanese Habu-kurage,Chironex yamaguchii,sp.nov.(Cnidaria:Cubozoa:Chirodropida)[J].Zootaxa,2009,513(2030):59-65.
[30] COLLINS A G,SCHUCHERT P,MARQUES A C,et al.Medusozoan phylogeny and character evolution clarified by new large and small subunit r DNA data and an assessment of the utility of phylogenetic mixture models[J].Systematic Biology,2006,55(1):97-115.
[31] KI J S,HWANG D S,SHIN K,et al.Recent moon jelly(Aurelia sp.1)blooms in Korean coastal waters suggest global expansion:examples inferred from mitochondrial COI and nuclear ITS-5.8 S rDNA sequences[J].ICES Journal of Marine Science,2008,65(3):443-452.
[32] DAWSON M N,CIECIEL K,DECKER M B,et al.Population-level perspectives on global change:genetic and demographic analyses indicate various scales,timing,and causes of scyphozoan jellyfish blooms[J].Biological Invasions,2015,17(3):851-867.
[33] ARMANI A,TINACCI L,GIUSTI A,et al.What is inside the jar?-Forensically informative nucleotide sequencing(FINS)of a short mitochondrial COI gene fragment reveals a high percentage of mislabeling in jellyfish food products[J].Food Research International,2013,54(2):1383-1393.
[34] BAYHA K M,GRAHAM W M.First confirmed reports of the rhizostome jellyfish Mastigias(Cnidaria:Rhizostomeae)in the Atlantic basin[J].Aquatic Invasions,2011,6(3):361-366.
[35] KOCOT K M,TODT C.Three new meiofaunal solenogaster species(Mollusca:Aplacophora)from the north-east Pacific[J].Journal of Natural History,2014,48(45-48):3007-3031.
[36] ZHENG L,HE J,LIN Y,et al.16S r RNA is a better choice than COI for DNA barcoding hydrozoans in the coastal waters of China[J].Acta Oceanologica Sinica,2014,33(4):55-76.
[37] KAYAL E,BENTLAGE B,CARTWRIGHT P,et al.Phylogenetic analysis of higher-level relationships within Hydroidolina(Cnidaria:Hydrozoa)using mitochondrial genome data and insight into their mitochondrial transcription[J].PeerJ,2015,3:e1403.
[38] BENTLAGE B,CARTWRIGHT P,YANAGIHARA A A,et al.Evolution of box jellyfish(Cnidaria:Cubozoa),a group of highly toxic invertebrates[J].Proceedings of the Royal Society of London B:Biological Sciences,2010,277(1680):493-501.
[39] BEARD C B,HAMM D,COLLINS F H.The mitochondrial genome of the mosquito Anopheles gambiae:DNA sequence,genome organization,and comparisons with mitochondrial sequences of other insects[J].Insect Molecular Biology,1993,2(2):103-124.
[40] IRWIN D M,KOCHER T D,WILSON A C.Evolution of the cytochrome b gene of mammals[J].Journal of Molecular Evolution,1991,32(2):128-144.
[41] GERSHWIN L A.MORBAKKA FENNERI.A new genus and species of Irukandji jellyfish(Cnidaria:Cubozoa)[J].Memoirs of the Queensland Museum-Nature,2008,54(1):23-33.
[42] BENTLAGE B,LEWIS C.An illustrated key and synopsis of the families and genera of carybdeid box jellyfishes(Cnidaria:Cubozoa:Carybdeida),with emphasis on the"Irukandji family"(Carukiidae)[J].Journal of Natural History,2012,46(41-42):2595-2620.
[43] AUNGTONYA C,CHANACHON K.Species and distribution of venomous jellyfish in coastal areas of Phuket Province[R]∥Technical Paper.Phuket:Phuket Marine Biological Center Press,2012.
Genetic Diversity of the Venomous Medusae in Thai Waters Based on the Mitochondrial COI Gene Sequences
MIAO Xiao-xiang1,2,XIAO Jie1,2,ZHANG Xue-lei1,2,LIU Rui-juan1,2,AUNGTONYA Charatsee3
(1.The First Institute of Oceanography,SOA,Qingdao 266061,China;2.Key Laboratory of Science and Engineering for Marine Ecology and Environment,SOA,Qingdao 266061,China;3.Phuket Marine Biological Center,Phuket 83000,Thailand)
To explore the genetic diversity of the venomous medusae,the partial mitochondrial COI gene sequences were generated from 69 jellyfish specimens collected from the coastal waters of Thailand.And the feasibility of DNA barcoding using this gene fragment was further investigated,in order to provide technique supporting for the early warning of venomous medusae in Thailand,and for the future work on ecology and biology of these venomous jellyfish as well.The analysis of nucleotide composition of the sequences showed that G and C nucleotide percentage(GC%)of Cubozoa(42.1%)was significantly higher than those of Scyphozoa(37.1%)and Hydrozoa(36.9%).GC%of the 3rd condon position(30.2%)was much lower than those of the 1st and 2nd positions(47.4%and 42.1%,respectively).Besides,the frequency of nucleotide substitution was highest at the 3rd positions with the R value(transition/transversion)of 1.0,indicating that the mutational rate at this position was prone to be saturated.Whereas,the topology of the phylogenetic tree was not significantly changed after the 3rd condon positions were excluded from the analyses.The K2P(Kimura 2-parameter)genetic distance was evaluated within and among the species based on the sequences of COI gene.The intra-specific genetic distances ranged from 0.000 to 0.151,with the average of 0.036;the inter-specific distances varied from 0.167 to 0.321,with the average of 0.263,and inter-classes were from 0.246 to 0.385 with the average of 0.334.The COI gene fragments were proved to be able to discriminate these venomous jellyfish species fast and efficiently.Additionally,a high species diversity of the venomous jellyfish along the coasts of Thailand was revealed by this research.There were total 13 species identified from the 69 samples,including 5 Cubozoa species,6 scyphozoans and 2 hydrozoans.A new genus of the family Carukiidae was suggested by this study.A regional distribution pattern of the cubozoan jellyfish in Thai waters was observed,which showed the species and genetic divergence between east and west coasts of Malaya Peninsula.It needs further investigation whether there is a geographic barrier for the divergence between these two regional groups of cubozoan jellyfish.
Cubozoa;Scyphozoa;DNA barcoding;COI sequences;Thailand
September 1,2016
Q344
A
1671-6647(2017)04-0535-12
10.3969/j.issn.1671-6647.2017.04.010
2016-09-01
中国-东盟海上合作基金项目——濒危海洋物种合作研究;国家自然科学青年基金项目——我国海水甲壳类中寄生性甲藻(Hematodinium)的种群遗传结构及其分子系统分类地位的研究(41206162)
缪晓翔(1991-),女,江苏南通人,硕士研究生,主要从事近海水母和大型藻类的生态学方面研究.E-mail:miaoxiaoxiang@fio.org.cn
*通讯作者:张学雷(1973-),男,山东淄博人,博士,研究员,主要从事海洋生物、环境科学和生态学方面研究.E-mail:zhangxl@fio.org.cn
(高 峻 编辑)
