酱油发酵醪中谷氨酰胺酶活性蛋白序列分析
2017-11-13邹谋勇朱新贵
邹谋勇,朱新贵
(李锦记(新会)食品有限公司,广东 江门 529100)
酱油发酵醪中谷氨酰胺酶活性蛋白序列分析
邹谋勇,朱新贵
(李锦记(新会)食品有限公司,广东 江门 529100)
为研究酱油发酵醪中微生物菌群谷氨酰胺酶活性蛋白的分布和基本特性,采用生物信息学方法对发酵醪中已鉴定微生物种属的谷氨酰胺酶活性蛋白进行了分类、筛选、信号肽分析、系统进化树分析、理化特性预测和3-D结构同源建模。发现酱油发酵醪中具有谷氨酰胺酶活性的蛋白分为3 类,分别参与谷氨酰胺分解代谢、生物合成和谷氨酰胺转氨基作用。分析显示4 种谷氨酰胺酶具有信号肽,其余谷氨酰胺酶活性蛋白序列均不含信号肽。系统进化树分析显示细菌类谷氨酰胺酶与真菌类谷氨酰胺酶呈现两个不同的进化方向。分泌型谷氨酰胺酶均有一定的亲水性,等电点在4.81~5.99之间。3-D结构同源模建显示阴沟乳杆菌和弗氏柠檬酸杆菌分泌的谷氨酰胺酶与大肠杆菌、枯草芽孢杆菌谷氨酰胺酶具有结构相似性。
谷氨酰胺酶;酱油;微生物菌群;序列分析
发酵调味品主要原料大豆的贮藏蛋白中11S球蛋白含谷氨酰胺质量分数在8.8%以上,7S β-伴球蛋白谷氨酰胺质量分数为7.5%[1],谷氨酰胺酶具有将无鲜味的谷氨酰胺转化为具有鲜味的谷氨酸的能力[2-3],因此其在食品[4],特别是发酵调味品(如酱油)中的应用越来越受到人们的关注[5-7],叶茂等[8]通过谷氨酰胺酶粗酶添加酱油的方法证实了其对酱油风味的改善作用,日本学者也从枯草杆菌中分离到谷氨酰胺酶,并应用到酱油品质优化中[9],Ye M ao等[10]将来源于地衣芽孢杆菌的谷氨酰胺酶应用于酱油发酵中,收到了良好效果。
酱油发酵醪中谷氨酰胺酶的来源主要有曲料带入和发酵微生物菌群合成。曲料中谷氨酰胺酶主要来源于米曲霉,米曲霉产谷氨酰胺酶方面已经有较多的研究报道。陈红玲[11]通过诱变育种获得高产谷氨酰胺酶米曲霉菌株3.422;马永强等[12]分析了米曲霉制曲过程中谷氨酰胺酶积累的主要影响因素,并确定了最优工艺条件。酱油发酵微生物菌群组成方面已有较多报道,汇总这些数据发现酱油发酵醪中主要微生物种属有28 个[13-20]。詹寿年等[21]筛选到一株产谷氨酰胺酶的枯草芽孢杆菌(Bacillus subtilis GA713),其谷氨酰胺酶活力(37 ℃)为68.407 mg NH3/(g曲·h);康维民[9]、周池虹伶[22]等也对芽孢杆菌属产谷氨酰胺酶细菌进行了酱油发酵应用研究;韩铭海等[23]筛选到一株高产谷氨酰胺酶菌株,并通过生理生化实验和菌体形态特性分析,鉴定其为柠檬酸杆菌属细菌(Citrobacter)。但是,以酱油发酵醪微生物菌群为目标分析谷氨酰胺酶的分布和序列特性的研究却鲜有报道。
微生物细胞中含有多种具备谷氨酰胺酶活性的蛋白,存在于不同代谢途径中,为更直观清晰地研究谷氨酰胺酶活性蛋白,依据其参与细胞代谢的类型及代谢产物的差异,对酱油发酵醪中微生物菌群的谷氨酰胺酶活性蛋白进行分类。本研究从已报道的酱油发酵醪微生物菌群出发,逐一分析了各种属微生物具有谷氨酰胺酶活性的蛋白种类及其信号肽;对不同来源蛋白序列进行了系统进化树分析;对具有信号肽的谷氨酰胺酶进行了主要理化特性预测和同源模建。通过研究以期为基于谷氨酰胺酶的酱油风味优化及微生物选育、添加和控制提供理论参考。
1 材料与方法
1.1 材料
酱油发酵醪微生物的谷氨酰胺酶蛋白序列来源于UniPro数据库[24],已报道的酱油发酵醪微生物如表1所示,以这些微生物的基因组和已注释蛋白为对象,筛选具有谷氨酰胺酶活性的蛋白序列。

表1 已报道酱油发酵醪中微生物种属Tab le 1 Reported m icrobial genera and species from fermented mash of soy sauce
1.2 仪器与设备
生物信息学数据采用ThinkPad E40(0578B31)计算机进行分析,所用生物信息学软件有:Clustal X、DNAMAN、MEGA 4、PyMOL、Endnote;其他数据均采用在线工具进行分析。
1.3 方法
1.3.1 谷氨酰胺酶活性蛋白信号肽预测
酱油发酵醪中微生物菌群谷氨酰胺酶信号肽采用在线分析工具SignalP 4.1 Server[25]进行预测。
1.3.2 谷氨酰胺酶系统进化树分析
采用Clustal X[26]对酱油发酵醪中微生物菌群谷氨酰胺酶活性蛋白序列进行多重比对,比对结果导入MEGA 4软件[27],采用邻接法构建系统进化树。Bootstrap值设为1 000,其他参数为默认值。
1.3.3 分泌型谷氨酰胺酶生物信息学分析
分泌型谷氨酰胺酶疏水性、等电点、分子质量、蛋白理化特性采用在线工具ProtParam[28]进行分析。谷氨酰胺酶空间结构采用SW ISS-MODEL的自动模式进行同源模建,从PDB数据库中比对筛选相似度最高的蛋白结构作为同源模建模板。
2 结果与分析
2.1 谷氨酰胺酶活性蛋白种类分析
依据谷氨酰胺酶活性蛋白参与代谢途径的类型,酱油发酵醪中微生物菌群的谷氨酰胺酶活性蛋白大致可以分为5种(表2),其中参与谷氨酰胺分解代谢的1 种,参与生物合成的3 种,参与转氨基作用的1 种。从蛋白分子质量范围分析,参与生物合成和转氨基作用的谷氨酰胺酶活性蛋白分子质量变化不大,而参与谷氨酰胺分解代谢的谷氨酰胺酶分子质量大小差异较大,真菌的谷氨酰胺酶蛋白分子质量(如酵母101 kD、米曲霉76 kD)明显大于细菌来源的谷氨酰胺酶(如肠杆菌属36 kD)。
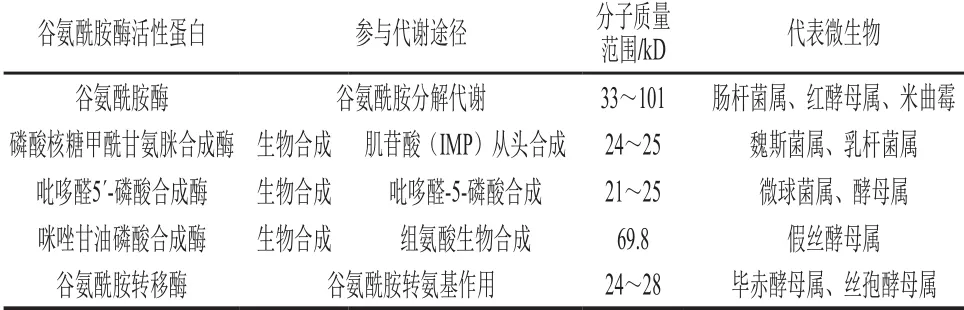
表2 酱油发酵醪中微生物菌群谷氨酰胺酶活性蛋白分类Tab le 2 Classification of proteins w ith glutam inase activity from m icrobial flora in fermented mash of soy sauce
2.2 微生物菌群谷氨酰胺酶活性蛋白分析
为更加全面掌握酱油发酵醪中微生物菌群合成谷氨酰胺酶活性蛋白的情况,对目前已报道的酱油发酵主要微生物菌群进行了汇总(表1),并通过蛋白数据库(Uniport)对每一种属微生物可能存在的谷氨酰胺酶活性蛋白进行分析,结果如表3所示。细菌类微生物的谷氨酰胺酶活性蛋白主要存在于谷氨酰胺分解代谢、肌苷酸合成以及吡哆醛-5-磷酸合成途径中,真菌类微生物的谷氨酰胺酶活性蛋白主要存在于谷氨酰胺分解代谢、氨基酸氨基转移和组氨酸合成途径中。部分微生物存在2~3 种谷氨酰胺酶活性蛋白,如微球菌属、气球菌、枯草芽孢杆菌、地衣芽孢杆菌、丝孢酵母属和红冬孢酵母属等。Li Youbao等[29]在酱醪宏基因组中发现了新的谷氨酰胺酶基因,Ito等[30]在隐球菌中发现了耐盐耐热的谷氨酰胺酶新家族。但是,基于现有的蛋白注释分析,酱油发酵中微生物也有部分菌群不表达具有谷氨酰胺转氨酶活性的蛋白,如厚壁菌属(Fimicutes)、色盐杆菌属(Chromohalobacter)、四联球菌属(Tetragenococcus)和球拟酵母属(Torulopsis)等均未筛选到谷氨酰胺酶活性蛋白的存在;相反此类微生物一般含有天冬酰胺酶活性蛋白,这类酶催化天冬酰胺生成天冬氨酸和氨,亦可以满足细胞某些重要代谢活动的需求。

表3 酱油发酵醪微生物菌群谷氨酰胺酶活性蛋白分析Tab le 3 Analysis of p roteins w ith glutam inase activity from m icrobial flora in fermented mash of soy sauce
2.3 微生物菌群谷氨酰胺酶活性蛋白信号肽分析
在酱油发酵过程中,谷氨酰胺酶须分泌到微生物细胞外或者在细胞自溶后才能发挥分解谷氨酰胺的作用。由于细胞自溶伴随大量蛋白酶的水解作用,谷氨酰胺酶存留量较小,酱油发酵过程谷氨酰胺的转化主要依赖于发酵微生物的合成和分泌。采用在线分析工具SignalP 4.1 Server对已报道的酱油发酵醪主要微生物的谷氨酰胺酶活性蛋白进行了信号肽分析,结果如表4所示。肠杆菌属、弗氏柠檬酸杆菌、红冬孢酵母属和米曲霉表达的谷氨酰胺酶均具有信号肽序列;细菌类微生物肠杆菌属和弗氏柠檬酸杆菌的信号肽长度为23 个氨基酸,真菌类微生物红酵母属和米曲霉的信号肽长度为20 个氨基酸。另外15 个种属酱醪微生物的谷氨酰胺酶活性蛋白均没有检测到信号肽序列,分析发现表4中多数蛋白主要参与细胞生物合成和转氨基代谢,为胞内酶;而谷氨酰胺酶则因微生物种类的差异可存在胞内,也可以分泌至胞外。

表4 酱油发酵醪中微生物菌群谷氨酰胺酶活性蛋白信号肽分析Table 4 Analysis of signal pep tides of proteins w ith glutam inase activity from m icrobial flora in fermented mash of soy sauce
2.4 微生物菌群谷氨酰胺酶系统进化树分析

图1 酱油发酵醪中微生物菌群谷氨酰胺酶系统进化树分析Fig. 1 Phylogenetic tree analysis of glutam inases from m icrobial flora in fermented mash of soy sauce
由信号肽分析结果可知,可以分泌到细胞外的谷氨酰胺酶活性蛋白均为谷氨酰胺酶,对酱油发酵醪已鉴定的微生物菌属或种所表达的谷氨酰胺酶进行系统进化树分析,结果如图1所示。细菌来源的谷氨酰胺酶和真菌来源的谷氨酰胺酶分别聚为一支,肺炎克雷伯菌亚种(K. pneum on iae subsp. pneum on iae)和泛氏菌(P. vagans)聚为一支,亲缘关系较近;枯草芽孢杆菌(B. subtilis)和地衣芽孢杆菌(B. licheniform is)聚为一支;阴沟肠杆菌(E. lign o ly ticu s,属于E. cloacae complex)和弗氏柠檬酸杆菌(C. freundii)聚为一支,进化距离较近;三小支之间遗传出现一定程度的分化,进一步聚为一支;圆红冬孢酵母(R. toruloides)和米曲霉(A. oryzae)单独聚为一支。推测谷氨酰胺酶在细菌和真菌中单独进化,而且来自于不同的祖先。具有信号肽序列的谷氨酰胺酶蛋白聚为两支,两支内部亲缘关系较近。
2.5 分泌型谷氨酰胺酶理化性质预测

表5 谷氨酰胺酶理化性质预测Tab le 5 Predicted physicochem ical properties of glutam inases
对酱油发酵醪已鉴定的微生物菌属或种所表达的谷氨酰胺酶进行理化性质分析,结果如表5所示。除肺炎克雷伯菌和地衣芽孢杆菌之外,其他菌株的谷氨酰胺酶均具有一定的亲水性。细菌来源的谷氨酰胺酶蛋白分子质量大小相近,等电点在5.25~5.99之间;真菌来源的谷氨酰胺酶分子质量变化较大,圆红冬孢酵母谷氨酰胺酶分子质量达到了100.9 kD,等电点为5.53;米曲霉谷氨酰胺酶分子质量为76.2 kD,等电点为4.81。
2.6 分泌型谷氨酰胺酶同源模建

表6 分泌型谷氨酰胺酶同源模建模板Table 6 Homology modeling tem p lates for secretory glutam inase proteins
如表6所示,阴沟肠杆菌和弗氏柠檬酸杆菌与模板谷氨酰胺酶序列相似度分别为46.28%和47.25%,圆红冬孢酵母和米曲霉谷氨酰胺酶在PDB数据库均未找到相似度大于20%的模板。
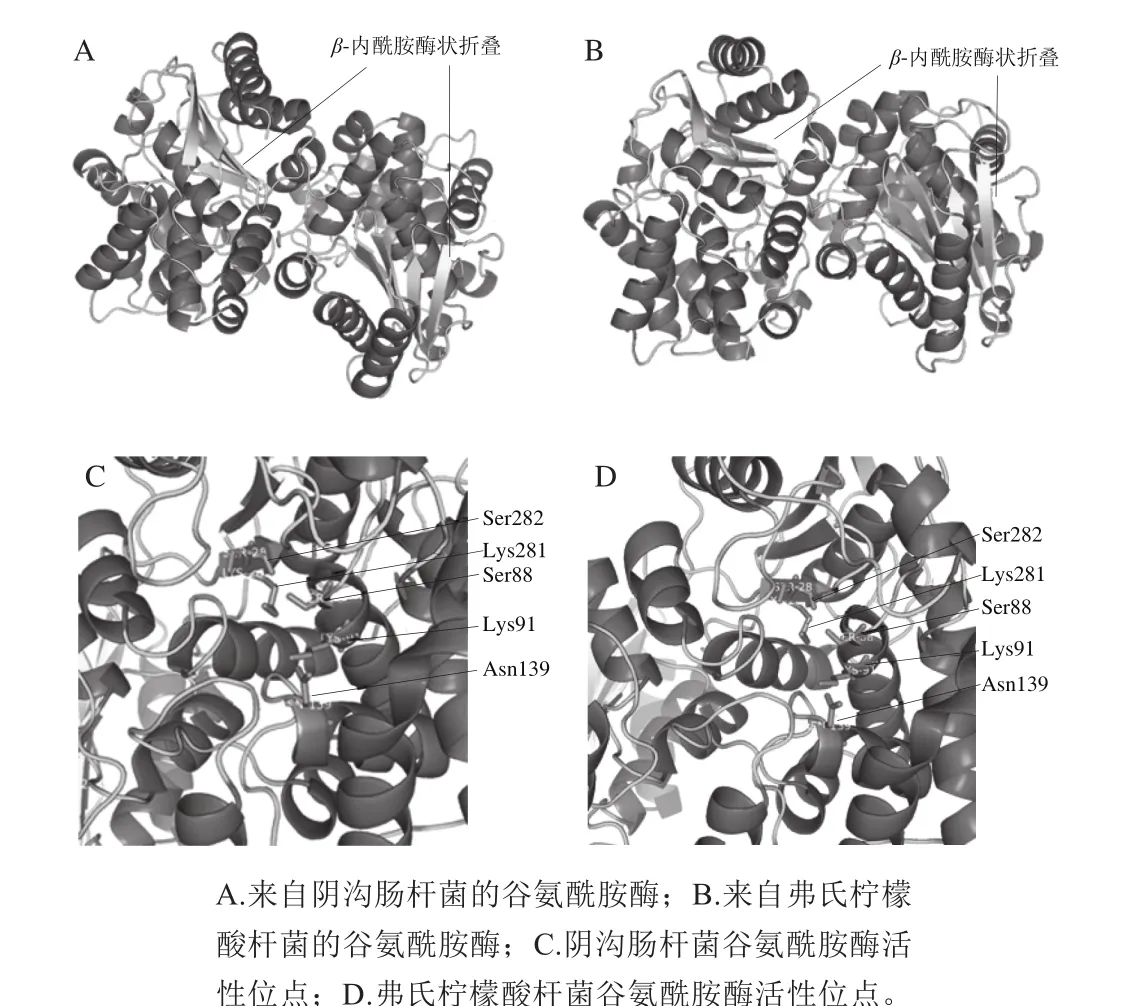
图2 分泌型谷氨酰胺酶蛋白同源模建Fig. 2 Homology modeling of secretory glutam inase proteins
以表6所列蛋白作为模板,采用SW ISS-MODEL对阴沟肠杆菌和弗氏柠檬酸杆菌的谷氨酰胺酶进行同源模建,绘制蛋白3-D图像,结果如图2A、B所示。这两株菌的谷氨酰胺酶蛋白3-D结构均以二聚体的形式存在,均具有典型的β-内胺酶状折叠,空间结构相似度较高。Brown等[31]对大肠杆菌YbaS和芽孢杆菌YbgJ的谷氨酰胺酶的晶体结构(PDB:u60.1.A)进行了解析,确定了该酶具有典型的β-内酰胺酶状折叠,并具有β-内酰胺酶催化位点所需的关键氨基酸残基(YbgJ菌株为:Ser74、Lys77、Asn126、Lys268和Ser269)。阴沟肠杆菌和弗氏柠檬酸杆菌谷氨酰胺酶活性位点如图2C、D所示,组成活性位点的氨基酸残基均为Ser88、Lys91、Asn139、Lys281、Ser282,各残基之间氨基酸间距以及在空间上的排布均显示出其与大肠杆菌YbaS和芽孢杆菌YbgJ的谷氨酰胺酶有高度同源性。不同之处在于阴沟肠杆菌和弗氏柠檬酸杆菌谷氨酰胺酶均具有信号肽,而大肠杆菌和枯草芽孢杆菌谷氨酰胺酶均无信号肽,这暗示了不同微生物在适应环境时为提升自身在营养方面的竞争能力,进化过程中为谷氨酰胺酶编码了信号肽并分泌至胞外,通过分解谷氨酰胺提高其在碳源和氮源转化、吸收方面的效率。
3 讨 论
来源于酱油发酵醪中微生物菌群的谷氨酰胺酶活性蛋白分为3 类,分别为谷氨酰胺分解代谢、生物合成和谷氨酰胺转氨基作用。含有信号肽序列的谷氨酰胺酶活性蛋白均为参与谷氨酰胺分解代谢的谷氨酰胺酶,来源于阴沟肠杆菌、弗氏柠檬酸杆菌、圆红冬孢酵母和米曲霉。细菌类来源和真菌类来源的谷氨酰胺酶在进化树中分为两支,推测其在这两大类微生物中单独进化。阴沟肠杆菌和弗氏柠檬酸杆菌谷氨酰胺酶蛋白分子质量大小相近,等电点分别为5.99和5.25;米曲霉谷氨酰胺酶等电点为4.81。阴沟肠杆菌和弗氏柠檬酸杆菌的谷氨酰胺酶蛋白均以二聚体的形式存在,均具有典型的β-内酰胺酶状折叠;活性位点氨基酸残基均为Ser88、Lys91、Asn139、Lys281、Ser282,各残基之间氨基酸间距以及在空间上的排布均显示出其与大肠杆菌YbaS和芽孢杆菌YbgJ的谷氨酰胺酶的高度同源性。
目前已报道可产生谷氨酰胺酶的微生物有枯草芽孢杆菌[21]、解淀粉芽孢杆菌[22]、柠檬酸杆菌[23]、硝基还原单胞菌[32]、腐生葡萄球菌[33]、隐球菌[30]、嗜麦芽寡养单胞菌[34]、毕赤酵母[35]、米曲霉[7]等。韩铭海等[23]研究发现柠檬酸杆菌产生的谷氨酰胺酶的最适pH值为6.2,最适温度50 ℃,在pH 6.2和40 ℃以下具有较高的稳定性,高含量NaCl对谷氨酰胺酶活性影响不显著;适合在酱油等高盐调味品生产中应用。李丹等[36]报道了酱油发酵醪在发酵10~30 d周期内pH值由5.3降低到4.8;发酵后期pH值不再显著下降,维持在4.6~4.9之间[13],非常接近米曲霉谷氨酰胺酶等电点4.81;因此米曲霉表达分泌的谷氨酰胺酶在酱油发酵中后期pH值范围内不稳定。另外,谷氨酰胺酶转化谷氨酰胺生成谷氨酸发生在发酵中后期,前期需要蛋白酶、肽酶的作用产生谷氨酰胺;而蛋白酶、肽酶的水解作用也会一定程度破坏谷氨酰胺酶。基于以上两点,制曲阶段米曲霉合成分泌的谷氨酰胺酶在发酵阶段发挥作用的空间较小。提高发酵醪中谷氨酰胺酶活力需要寻找更为有效的途径。
通过发酵条件优化和基因工程可使微生物谷氨酰胺酶产量明显提高。周驰虹伶等[22]优化解淀粉芽孢杆菌产酶条件后,谷氨酰胺酶活力达到2 690.02 U/mg干基;卢彪等[37]通过基因定点整合技术使枯草芽孢杆菌的谷氨酰胺酶活力提高了3.36 倍,通过大肠杆菌表达系统使谷氨酰胺酶活力达到了608.2 U/μg[38]。然而酱油发酵醪添加工业微生物生产的谷氨酰胺酶制剂直接导致了酱油生产成本的增加,在工业生产中实际应用较少。因此利用酱油发酵醪中本源的微生物,通过分离、选育、添加发酵提升酱油谷氨酸含量成为一种经济有效的方式。但谷氨酰胺酶在酱油发酵醪微生物菌群中的分布鲜有报道。
酱油发酵醪微生物菌群组成分布已有大量文献报道,采用生物信息学的方法从已报道的微生物种属中挖掘出可以合成分泌谷氨酰胺酶的微生物,可以大大提高产酶微生物分离筛选的针对性,提高菌种研究效率。通过序列分析初步预测谷氨酰胺酶基本理化性质、空间结构、活性位点可为酶学性质、酶分子改造和定向进化的研究提供重要参考。由于酱油发酵醪微生物菌群报道数据的有限性,本研究采用生物信息学对于酶蛋白分布的挖掘尚不够全面;谷氨酰胺酶的蛋白结构解析目前限于原核生物,对于真菌来源的谷氨酰胺酶的分析尚不充分。随着特定生境微生物菌群研究和酶蛋白性质和结构研究的深入,采用数据归纳和生物信息学挖掘、分析相结合研究微生物群体的酶分子分布和特定物质代谢流的方法将成为一个重要的研究方向。
[1] 邹谋勇. 大豆贮藏蛋白序列分析与3-D结构同源模建[J]. 食品与发酵科技, 2016, 52(4): 25-32. DOI:10.3969/j.issn.1674-506X.2016.04-006.
[2] 包启安. 大豆发酵食品与谷氨酰胺酶[J]. 中国调味品, 1983(10): 3-7.
[3] ITO K, KOYAMA Y, HANYA Y. Identification of the glutam inase genes of Aspergillus sojae involved in glutamate production during soy sauce fermentation[J]. Bioscience Biotechnology & Biochem istry,2013, 77(9): 1832-1840. DOI:10.1271/bbb.130151.
[4] NANDAKUMAR R, YOSHIMUNE K, WAKAYAMA M, et al.M icrobial glutam inase biochem istry, molecular approaches and applications in the food industry[J]. Journal of Molecular Catalysis B:Enzymatic, 2003, 23(2): 87-100. DOI:10.1016/s1381-1177(03)00075-4.
[5] 罗科丽, 彭喜春, 晏日安. 微生物谷氨酰胺酶的研究进展[J]. 中国调味品, 2010, 35(11): 30-34. DOI:10.3969/j.issn.1000-9973.2010.11.002.
[6] 周尚庭, 李沛, 郭辉. 谷氨酰胺酶和酵母抽提物对无添加酱油的品质提升研究[J]. 中国调味品, 2016, 41(5): 45-50. DOI:10.3969/j.issn.1000-9973.2016.05.010.
[7] LIANG Y, PAN L, LIN Y. Analysis o f ex tracellu lar p roteins of Aspergillus oryzae grow n on soy sauce ko ji[J]. Bioscience Biotechnology & Biochem istry, 2009, 73(1): 192-195. DOI:10.1271/bbb.80500.
[8] 叶茂, 张远平, 邓毛程. 一种耐盐性谷氨酰胺酶在高盐稀态酱油酿造过程中的应用研究[J]. 中国调味品, 2014, 39(7): 5-7; 17.DOI:10.3969/j.issn.1000-9973.2014.07.002.
[9] 康维民, 肖念新. 枯草菌产生的谷氨酰胺酶在酱油酿造中的作用[J].中国酿造, 1997, 16(4): 36-37.
[10] YE M, LIU X, ZHAO L. Production of a novel salt-tolerant L-glutaminase from Bacillus amyloliquefaciens using agro-industrial residues and its application in Chinese soy sauce fermentation[J].Biotechnology, 2013, 12(1): 25-35. DOI:10.3923/biotech.2013.25.35.
[11] 陈红玲. 谷氨酰胺酶高产菌沪酿3.422米曲霉的育种[J]. 中国酿造,1990, 9(3): 38-40.
[12] 马永强, 宫安旭, 陈晓昕, 等. 酱油制曲过程中蛋白酶和谷氨酰胺酶活力影响因素[J]. 食品科学, 2010, 31(17): 294-297.
[13] 谢显华. 酱油发酵过程微生物群落结构的动态研究[D]. 广州: 华南理工大学, 2010.
[14] 耿予欢, 谢显华, 李国基. 酱油发酵过程中细菌群落结构的动态变化[J]. 现代食品科技, 2015, 31(8): 83-87. DOI:10.13982/j.m fst.1673-9078.2015.8.015.
[15] 谢小保, 欧阳友生, 曾海燕, 等. 高盐稀醪酱油发酵原油中微生物区系研究[J]. 微生物学通报, 2007, 34(3): 504-507. DOI:10.3969/j.issn.0253-2654.2007.03.026.
[16] 梁恒宇, 邓立康, 林海龙. 传统发酵大豆食品中乳酸菌的分布、功能和应用[J]. 食品科学, 2013, 34(19): 381-385. DOI:10.7506/spkx1002-6630-201319077.
[17] 徐次刚, 周其洋, 赵丽云. PCR-DGGE技术对传统发酵酱油的菌群分析[J]. 中国调味品, 2014, 39(7): 29-32. DOI:10.3969/j.issn.1000-9973.2014.07.007.
[18] 王红涛, 罗立新, 张士伟. 一株芽孢杆菌的分离鉴定及其在发酵酱渣研究中的应用[J]. 食品工业科技, 2012, 33(7): 184-187.DOI:10.13386/j.issn1002-0306.2012.07.012.
[19] 李明霞. 酵母菌[J]. 微生物学通报, 1974(2): 9. DOI:10.13344/j.m icrobiol.china.1974.02.007.
[20] 刘婷婷, 蒋雪薇, 周尚庭, 等. 高盐稀态发酵与低盐固态发酵酱油中次生菌群分析[J]. 食品与机械, 2010, 26(6): 13-17.
[21] 詹寿年, 吴拥军, 郭倩倩, 等. 高活性谷氨酰胺酶豆豉芽孢杆菌的筛选及glsA基因的克隆[J]. 贵州农业科学, 2011, 39(6): 119-122.DOI:10.3969/j.issn.1001-3601.2011.06.032.
[22] 周池虹伶, 崔春, 赵谋明. 解淀粉芽孢杆菌SW JS22固体发酵产谷氨酰胺酶的优化研究[J]. 食品与机械, 2015, 31(6): 33-37.DOI:10.13652/j.issn.1003-5788.2015.06.007.
[23] 韩铭海, 赵娟娟, 华盛龙, 等. 一株高产谷氨酰胺酶菌株的鉴定和酶活特性的研究[J]. 大豆科学, 2008, 27(6): 1037-1040.
[24] U n iP ro数据库[D B/O L]. h ttp://w w w.u n ip ro t.o rg/contact,2016-11-01/2017-02-07.
[25] 信号肽分析工具SignalP 4.1 Server[CP/OL]. http://www.cbs.dtu.dk/services/SignalP/,2016-11-01/2017-02-07.
[26] LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948.DOI:10.1093/bioinformatics/btm404.
[27] ZHANG W, SUN Z. Random local neighbor joining: a new method for reconstructing phylogenetic trees[J]. Molecular Phylogenetics &Evolution, 2008, 47(1): 117-128. DOI:10.1016/j.ympev.2008.01.019.
[28] 蛋白理化特性采用在线工具ProtParam[CP/OL]. http://web.expasy.org/protparam/,2016-11-01/2017-02-07.
[29] LI Y B, SUN K S, YU H S, et al. Cloning and expression of glutaminase new gene in the sauce billet metagenomic[J]. Advance Journal of Food Science &Technology, 2016, 10(12): 894-898. DOI:10.19026/ajfst.10.2283.
[30] ITO K, UM ITSUK I G, OGUMA T, et al. Salt-to lerant and thermostable glutam inases of Cryptococcus species form a new glutam inase fam ily[J]. Agricultural and Biological Chem istry, 2011,75(7): 1317-1324. DOI:10.1271/bbb.110092.
[31] BROWN G, SINGER A, KUZNETSOVA E, et al. Functional and structural characterization of four glutam inases from Escherichia coli and Bacillus subtilis[J]. Biochem istry, 2008, 47(21): 5724-5735.DOI:10.1021/bi800097h.
[32] 杨成, 张涛, 江波, 等. 硝基还原假单胞菌谷氨酰胺酶的分离纯化及酶学性质[J]. 食品与发酵工业, 2012, 38(12): 16-21. DOI:10.13995/j.cnki.11-1802/ts.2012.12.015s.
[33] 于海, 王敏, 李梦璐, 等. 1 株源自如皋火腿的产谷氨酰胺酶腐生葡萄球菌RG-2催化条件的优化[J]. 食品与发酵工业, 2012, 38(9): 17-21. DOI:10.13995/j.cnki.11-1802/ts.2012.09.035.
[34] WAKAYAM A M, YAM AGATA T, KAM EM URA A, et al.Characterization of salt-tolerant glutam inase from Stenotrophomonas maltophilia NYW-81 and its app lication in Japanese soy sauce fermentation[J]. Journal of Industrial M icrobiology & Biotechnology,2005, 32(9): 383-390. DOI:10.1007/s10295-005-0257-7.
[35] ARYUMAN P, LERTSIRI S, VISESSANGUAN W, et al. Glutaminaseproducing Meyerozyma (Pichia) guilliermondii isolated from Thai soy sauce fermentation[J]. International Journal of Food M icrobiology,2015, 192: 7-12. DOI:10.1016/j.ijfoodm icro.2014.09.019.
[36] 李丹, 王娅琴, 赵海锋, 等. 盐水浓度及pH对高盐稀态酱油酿造初期酱醪理化性质影响的研究[J]. 现代食品科技, 2011, 27(4): 380-383.DOI:10.13982/j.m fst.1673-9078.2011.04.022.
[37] 卢彪, 吴拥军, 刘艳敏, 等. 高活性谷氨酰胺酶基因glsA2在枯草芽孢杆菌BJ3-2染色体上的定点整合[J]. 食品科学, 2014, 35(1): 141-144.DOI:10.7506/spkx1002-6630-201401027.
[38] 卢彪, 吴拥军, 吴玉俊, 等. 谷氨酰胺酶基因原核表达载体的构建与表达[J]. 食品科学, 2013, 34(9): 140-142. DOI:10.7506/spkx1002-6630-201309029.
Sequence Analysis of Proteins w ith Glutam inase Activity in Fermented Mash of Soy Sauce
ZOU Mouyong, ZHU Xingui
(Lee Kum Kee (Xinhui) Foods Co. Ltd., Jiangmen 529100, China)
For a better understanding of the distribution and basic characteristics of the proteins w ith glutam inase activity from the m icrobial flora in the fermented mash of soy sauce, bioinformatics was used to conduct their classification,screening, signal peptides analysis, phylogenetic tree analysis, physicochem ical properties prediction and homology modeling of 3-dimensional (3-D) structure. It was found that the proteins w ith glutaminase activity were divided into 3 categories, participating in glutam ine catabolism, biosynthesis and transam ination of glutam ine, respectively. None of the proteins w ith glutam inase activity contained signal peptide sequence except 4 glutaminases. Two distinct evolutionary directions were found between bacterial and fungal glutaminase groups by phylogenetic tree analysis. Secretory glutaminases showed slight hydrophilicity w ith p Is of 4.81–5.99. The structural homology between glutam inases from Enterobacter lignolyticus, Citrobacter freundii and those from Escherichia coli and Bacillus subtilis was shown by 3-D homology modeling.
glutam inase; soy sauce; m icrobial flora; sequence analysis
10.7506/spkx1002-6630-201722022
TS264.2
A
1002-6630(2017)22-0143-06
邹谋勇, 朱新贵. 酱油发酵醪中谷氨酰胺酶活性蛋白序列分析[J]. 食品科学, 2017, 38(22): 143-148. DOI:10.7506/spkx1002-6630-201722022. http://www.spkx.net.cn
ZOU Mouyong, ZHU Xingui. Sequence analysis of proteins with glutaminase activity in fermented mash of soy sauce[J]. Food Science,2017, 38(22): 143-148. (in Chinese w ith English abstract)
10.7506/spkx1002-6630-201722022. http://www.spkx.net.cn
2016-12-16
邹谋勇(1987—),男,工程师,硕士,研究方向为调味品发酵技术研究与产品开发。E-mail:zoumouyong@163.com
