基于ITS和matK基因对牡丹组序列分析及其亲缘关系的研究
2017-11-11丑欢欢
丑欢欢 唐 红
(甘肃农业大学林学院,兰州 730070)
基于ITS和matK基因对牡丹组序列分析及其亲缘关系的研究
丑欢欢 唐 红*
(甘肃农业大学林学院,兰州 730070)
以芍药属牡丹组全部9个野生种、5个紫斑牡丹栽培品种及3个中原牡丹品种为试材,进行核糖体内转录间隔区(ITS)和叶绿体成熟酶K(matK)基因片段测序分析,探讨ITS和matK序列为牡丹组所有野生种种间关系提供分子证据。从GeneBank中选取了1个牡丹及3个外类群芍药、川赤芍和草芍药的ITS及matK序列。对试验样品进行DNA提取、PCR扩增并双向测序得到44条序列,人工校正后将所得44条序列进行比对;计算碱基组成频率、变异位点、简约信息位点数、转换/颠换比率、种内及种间遗传距离,以邻接法进行系统发育分析。结果表明,牡丹组所有个体ITS序列长度在750~800 bp,含有86个多态位点,74个简约信息位点,转换/颠换比率(R)为1.2;而matK序列含有20个简约信息位点,转换/颠换比率(R)为1.7。ITS序列分析将牡丹组野生种分为两大枝,稷山牡丹、紫斑牡丹、卵叶牡丹和杨山牡丹聚为一枝,狭叶牡丹、滇牡丹、黄牡丹和大花黄牡丹聚为另一枝,这两枝分别与革质花盘亚组和肉质花盘亚组相对应,而四川牡丹位于革质花盘亚组最底端,支持前人研究将四川牡丹归为革质花盘亚组。matK序列分析的牡丹组野生种间遗传距离结果不理想,未能清晰的表明野生种之间的亲缘关系。由此说明,ITS序列更适合牡丹组野生种间亲缘关系的研究分析。
牡丹组;ITS;matK;亲缘关系
牡丹组(PaeoniaSect.MoutanDC)隶属于毛茛科(Ranunculaceae)芍药属(Paeonia),为我国特有的落叶亚灌木类群,分布于我国的西南沿东北方向至中北部地区,包括云南、西藏、四川、甘肃、陕西、山西、河南和安徽等地[1],是家喻户晓的观赏植物和药用植物,其观赏价值和药用价值的利用历史已经超过了两千多年的历史[2]。开展野生牡丹种间亲缘关系研究,具有重要的学术和实践价值。然而,目前我们对野生牡丹种间亲缘关系的认识还有限。在以往的研究中,国内外学者主要分别从形态学[3]、细胞学[4]、孢粉学[5]、地理分布[6],以及分子标记[7]和基因序列[8]等多方面对野生牡丹的种间关系进行了研究。Sang等利用ITS和matK序列对芍药属部分种进行测定(包括矮牡丹和滇牡丹),对于牡丹组亲缘关系未能提供准确有用的信息[9~11];林启冰和周志钦等在Sang及Ferguson研究的基础上利用Adh基因研究牡丹8个野生种亲缘关系,结果对应Stern根据形态划分的革质花盘亚组和肉质花盘亚组[12];后来对8个野生种的GPAT基因进行测序,结果获得分辨良好的牡丹组种间关系树,这一分子证据支持的牡丹组种间关系与形态学性状建立的牡丹组种间关系相当吻合[13];张金梅等对牡丹野生种间的亲缘关系与形态学分类结果不一致[14]。然而由于样本数量有限及选择基因的不同,尤其是牡丹复合体内各种间关系及与四川牡丹的关系在现有的报道中没有得到一致的结论。因此,充分利用我国牡丹组种质资源,进一步全面地开展牡丹组种间亲缘关系的研究与分析是非常必要的。
在高等植物中,核糖体DNA的编码区序列是高度保守的,其内转录间隔区ITS(Internal transcribed spacer)由ITS1、ITS2和5.8S共3部分组成,在被子植物中,由于ITS1和ITS2非编码区受外界环境因素的影响较小,承受的选择压力较小,因而进化速度较快,核苷酸序列变异较大,且主要是以相互独立的点突变为主,可为属以下水平的研究提供较丰富的信息位点和变异位点等有效信息[15]。ITS序列可以显示亲缘关系很近的种之间的差异,表现出他们之间的系统进化关系。
matK基因是叶绿体基因组蛋白编码基因中进化速率最快的基因之一,也是最早被用于植物系统发育学研究的基因序列之一。目前,matK序列分析已成功为科、属级水平类群内部的系统重建提供了大量的信息并获得较高的支持率。同时matK序列还成功应用于某些类群的种间、种内的系统进化研究[16~20]。
由于前人利用ITS和matK基因对牡丹组关系进行研究时,所用的材料、种类不完全相同,因此并未对其进行全面的亲缘关系的探讨,本次研究利用ITS和matK基因分别对全部牡丹组野生种核苷酸进行序列分析,希望能在前人的研究基础上,为牡丹组野生种的亲缘关系及分类找到直接更加清晰可靠的分子证据。
1 材料与方法
1.1 材料
本研究所用的牡丹组野生种材料均采自甘肃省林业科学技术推广总站的野生牡丹种质资源圃,紫斑牡丹栽培品种采自临洮县洮阳镇紫斑牡丹资源圃,中原牡丹品种采自河南洛阳中原牡丹基地。详细资料见表1。除了本研究新测定的基因序列外,在构建基因树时还使用了GenBank中如下序列。
1.2 叶片总DNA的提取
取冷藏于4℃叶片200 mg,置于液氮中迅速研磨,将研磨后的样品装入灭菌的2 mL离心管后,利用植物基因组DNA提取试剂盒(北京,天根)提取总DNA,采用1%的琼脂糖凝胶电泳法检测。
1.3 ITS和matK序列的PCR扩增和测序
根据GenBank中紫斑牡丹ITS基因的序列(GenBank号,AF033592.1),设计一对引物ITS1(5′-GGAAGGAGAAGTCGTAACAAGG-3′)和ITS4(5′-CTTTTCCTCCGCTTATTGATATG-3′)及根据GenBank中紫斑牡丹matK基因的序列(GenBank号,AF033592.1),设计一对引物P1(5′-CCTATATCCGCTACTCCTTC-3′)和P2(5′-CTCAGTGAATTTCACCACG-3′),引物由上海生物工程有限公司合成。PCR扩增反应体系(20 μL):DNA模板1~1.5 μL(根据实际情况进行调整),上下游引物各0.4 μL,2×Tag Plus PCR Master Mix 9.8 μL,ddH2O(7.9~8.4 μL),扩增程序:95℃预变性5 min,(95℃变性30 s,退火温度43℃持续45 s,72℃延伸30 s,35个循环),72℃延伸7 min(引物序列及扩增程序如表)。用1%琼脂糖胶对PCR结果进行电泳检测,获得扩增产物的PCR被送往金唯智生物科技有限责任公司(苏州)进行纯化及测序。为保证测序的准确性,采用正反双向测序。序列利用软件BioEdit 5.0及MEGA(ver5.05)进行人工校正,对齐并除去引物区及测序质量较差区域的片段,最终获得干净、长度一致的序列用于后续数据分析。利用MEGA 5.05软件分析序列间的变异位点,计算种间及品种间遗传距离。
将芍药、草芍药及川赤芍设为外类群,利用MEGA 5.05分析软件的K2P距离对所有实验样品的序列以及来自GeneBank的5条序列分别构建ITS和matK序列的NJ系统发育树(图1~2)。
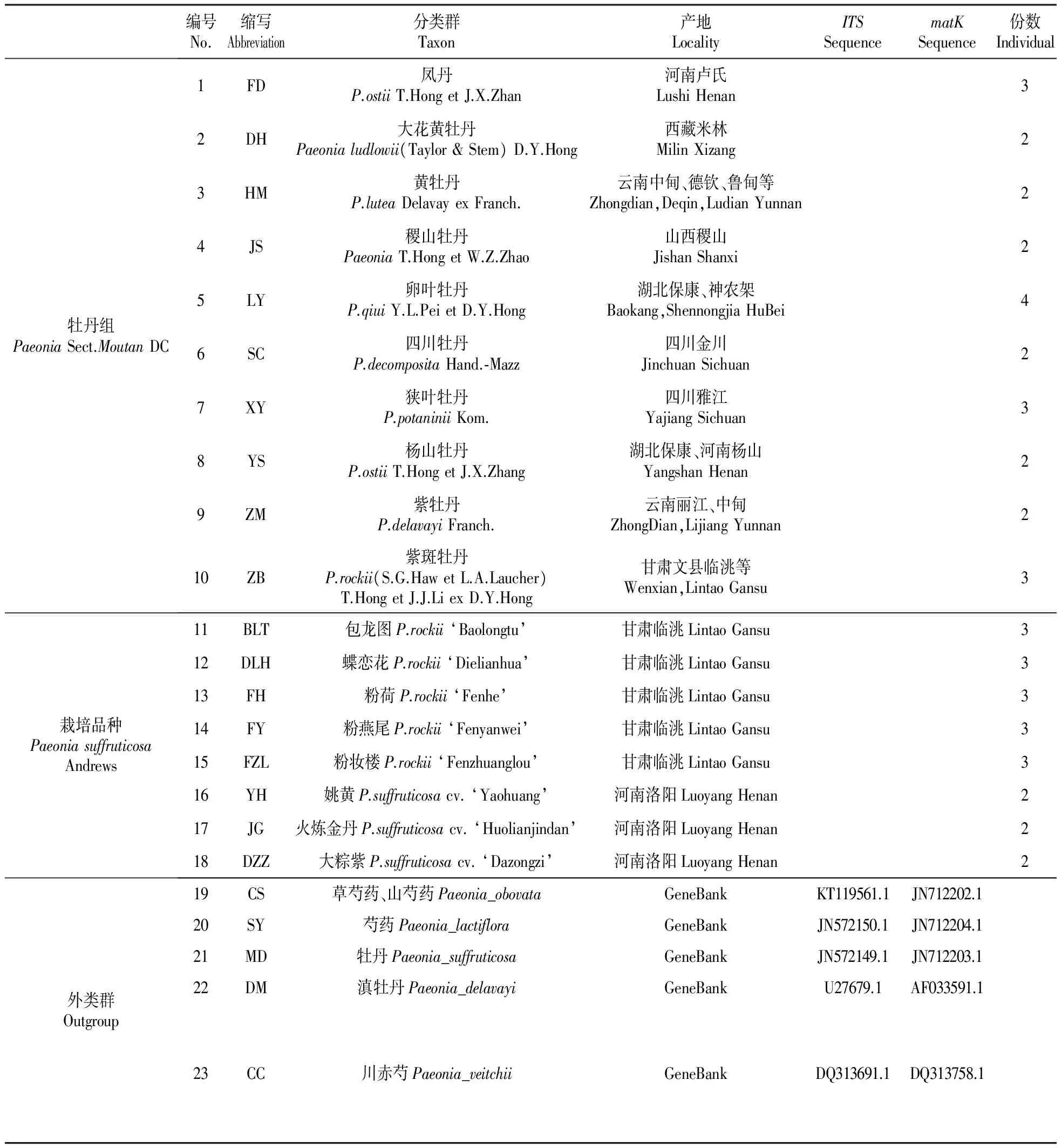
表1 实验材料及其来源

图1 基于牡丹组野生种及部分栽培品种ITS序列和邻接法(NJ)构建的系统发育树Fig.1 A phylogenetic tree constructed by neighbor-joining method with ITS sequence of Paeonia Sect. Moutan and part of P.suffruticosa Andr.

图2 基于牡丹组野生种及部分栽培品种matK序列和邻接法(NJ)构建的系统发育树Fig.2 A phylogenetic tree constructed by neighbor-joining method with matK sequence of Paeonia Sect.Moutan DC and part of P.suffruticosa Andr.
1.4 ITS和matK序列的数据分析和系统学分析
测序获得的DNA序列在MEGA(5.05)上进行Blast比对,选择芍药、草芍药及川赤芍作为外类群,进行系统发育分析。不同序列间的碱基组成频率、变异位点、简约信息位点数点,碱基转换/颠换比率、种内种间遗传距离等用MEGA(5.05)计算。分别用邻接法NJ(Neighbor-joining method)构建系统发育树。
2 结果与分析
2.1 ITS序列及系统发育分析
牡丹组全部9个野生种及紫斑牡丹和中原牡丹栽培品种的44条及来自GeneBank的5条序列经MEGA软件比对,分析其碱基序列差异,统计分析。根据Kimura-2参数的遗传距离模型计算各样本序列间的遗传距离,构建系统发育树。以芍药(Paeonialactiflora)、川赤芍(Paeoniaveitchii)和草芍药(Paeoniaobovata)作为外类群,构建NJ系统发育树。
所采集的牡丹野生种与栽培品种的ITS序列长度在750~800 bp,A、T、C、G平均含量分别为23.6%、20.8%、28.3%和27.3%,G和C含量超过了44.4%,明显高于A和T含量。序列含有86个多态位点、12个单一位点(singleton sites)和74个简约信息位点,转换/颠换比率(R)为1.2(表2)。
牡丹组大多数个体间的ITS序列差异较大,遗传距离在0.000~0.078,牡丹组全部野生种间遗传距离在0.000~0.020,最小为杨山牡丹与紫斑牡丹,其次是杨山牡丹与稷山牡丹、卵叶牡丹和四川牡丹遗传距离,为0.002,以及紫斑牡丹与稷山牡丹、卵叶牡丹和四川牡丹遗传距离,也为0.002;组间最大遗传距离为大花黄牡丹与稷山牡丹和卵叶牡丹之间,为0.020,其次为大花黄牡丹与杨山牡丹与紫斑牡丹之间。而各栽培品种与种间遗传距离最大的“粉妆楼”与大花黄牡丹,为0.069;其次是“粉妆楼”与狭叶牡丹,为0.064;表明了牡丹栽培品种与野生种之间的遗传多样性。“Paeoniasuffruticosa”(JN572149.1)与卵叶牡丹的遗传距离最小为0.000,表现很近的遗传关系;与稷山牡丹、四川牡丹、杨山牡丹和紫斑牡丹的遗传关系也较近,也说明它们之间的亲缘关系较近。来自GeneBank的Paeonia_delavayi(U27679.1)与实验材料中黄牡丹、狭叶牡丹和滇牡丹的遗传距离较小,也表现较近的亲缘关系。来自GeneBank的Paeonia_suffruticosa与卵叶牡丹遗传距离最小为0.000,而与稷山牡丹、四川牡丹、杨山牡丹、紫斑牡丹及栽培品种“包龙图”、“蝶恋花”、“粉荷”、“粉燕尾”也较近,遗传距离为0.002~0.003。在所有外类群与牡丹组中,遗传距离最大的是“粉妆楼”与Paeonia_lactiflora(JN572150.1),其次为“粉妆楼”与Paeonia_veitchii(DQ313691.1)。外类群芍药、草芍药、川赤芍在发育树最底端,明显和牡丹组物种差距很大(表3)。

表2 牡丹组ITS序列特异位点的变异式样

表3 基于牡丹组ITS序列的遗传距离矩阵
ITS的NJ进化树上,A组主要包括凤丹、稷山牡丹、紫斑牡丹、杨山牡丹及紫斑牡丹的栽培品种“蝶恋花”、“包龙图”和“粉荷”。B组中的一个进化枝为卵叶牡丹、四川牡丹、来自GengBank的P.suffruticosa及紫斑牡丹栽培品种“粉燕尾”、“粉妆楼”。C组包括中原牡丹的三个栽培品种“大粽紫”、“姚黄”和“火炼金丹”。D组包括狭叶牡丹、滇牡丹、黄牡丹及大花黄牡丹和P.delavayi。A组、B组、C组、D组具有很强的自展支持率。
2.2 matK序列及系统发育树分析
利用引物扩增的所有牡丹组野生种及栽培品种的matK序列,长度为757~780 bp,含有20个变异位点数、20个简约信息位点,没有单一位点,转换/颠换比率(R)为1.7,序列间平均距离为0.005 2(表4)。

表4 牡丹组matK序列特异位点的变异式样
所测序的matK序列及GenBank获取的牡丹组野生种和紫斑牡丹matK序列共49个,用MEGA(5.05)软件进行序列比对并计算种内种间遗传距离,进行系统发育分析。
牡丹组大多数个体间的matK序列差异较小,遗传距离在0.000~0.049,牡丹组野生种间遗传距离在0.000~0.014,最大为紫斑牡丹与凤丹、稷山牡丹、卵叶牡丹、四川牡丹和杨山牡丹之间,遗传距离为0.014;其次为狭叶牡丹与凤丹、稷山牡丹、卵叶牡丹及四川牡丹之间,遗传距离为0.011;其余牡丹野生种之间遗传距离都较小,说明matK未能将牡丹组大部分种的亲缘关系表现出来。除外类群外,紫斑牡丹、狭叶牡丹与各牡丹种及其栽培品种之间遗传距离较大,表明紫斑牡丹及狭叶牡丹与其它牡丹种的亲缘较远;且紫斑牡丹与其栽培品种之间的遗传距离也较大,这点和ITS序列分析的遗传距离不同(表5)。
基于matK序列的NJ进化树(图2)显示,A组包括凤丹、稷山牡丹和牡丹栽培品种“蝶恋花”、“包龙图”、“粉燕尾”、“大粽紫”、“火炼金丹”、“姚黄”及来自GeneBank的P.suffruticosa。B组包括杨山牡丹与栽培品种“粉妆楼”。C组卵叶牡丹和“粉荷”为一个进化枝。D组四川牡丹单独为一支,说明四川牡丹在系统进化过程中,与其它牡丹亲缘关系较远。E组包括大花黄牡丹、黄牡丹、滇牡丹、狭叶牡丹和紫斑牡丹以及来自GeneBank的P.delavayi。除了A、B、C组不同外,E组和ITS的NJ树的进化结果一致,matK序列的NJ树未清晰地表明其余牡丹种间的亲缘关系。由于所测序试材matK序列的变异位点比较少,大部分仅在序列的83、322、462和500位点的变异,所以牡丹组内的各进化枝自展支持率较低,不能很好的区分牡丹种间及种内的演化关系,但仍支持大花黄牡丹、黄牡丹、滇牡丹及紫斑牡丹聚为一类,表明大花黄牡丹、黄牡丹、滇牡丹及紫斑牡丹的亲缘关系较近。

表5 基于牡丹组matK序列的遗传距离矩阵
3 讨论
3.1 ITS和matK序列及遗传距离分析
本研究ITS序列长度在750~800 bp,含有86个多态位点,其中在42 bp(A),43 bp(G),62 bp(C),78 bp(A),612 bp(A)处大花黄牡丹、黄牡丹、狭叶牡丹及滇牡丹与其余种之间存在碱基位点的差异;通过分析牡丹组野生种之间的K2P遗传距离得出,杨山牡丹、稷山牡丹、卵叶牡丹、紫斑牡丹与四川牡丹之间遗传距离均在0~0.002,而他们与黄牡丹、大花黄牡丹、狭叶牡丹及滇牡丹之间遗传距离均大于0.200。这说明,ITS序列的位点差异、遗传距离均可以作为后续进化树分析结果的基础,可以验证ITS序列将牡丹组分为两个亚组的可靠性。
在所有物种的matK序列长度也在750~800 bp,只有20个多态位点,序列间的平均距离只有0.005 2,且紫斑牡丹、杨山牡丹、卵叶牡丹、稷山牡丹及四川牡丹之间遗传距离在0~0.014,狭叶牡丹他们之间遗传距离为0.011,而大花黄牡丹、黄牡丹与滇牡丹与其他种间的遗传距离只有0~0.008,明显看出所有牡丹种之间遗传距离存在交叉现象,并不能清楚得到牡丹组分类依据,说明以matK序列作为牡丹亚组分类的依据并不可靠,也不能将所有牡丹种之间的亲缘关系表现出来。在牡丹组植物中,matK序列变异速率较高,序列间简约信息位点少,遗传距离结果不理想,因此对于牡丹组野生种间亲缘关系的分析ITS的效果更理想。
3.2 牡丹组野生种间关系
本研究ITS构建的进化树反应了牡丹组植物间的亲缘关系。在以前长期的研究中,由于使用的材料不齐全,或因所使用的基因变异速率太快,牡丹组种间关系一直存在争议[21~22]。
本实验在避免了材料不足的情况下,对牡丹组各植物进行ITS序列分析,结果显示大花黄牡丹、黄牡丹、滇牡丹与狭叶牡丹聚为一支,而稷山牡丹、卵叶牡丹、杨山牡丹和紫斑牡丹聚为一支,这一结果与王莲英,成仿云等通过形态学研究,牡丹组分为革质花盘亚组和肉质花盘亚组的聚类结果一致[23~24],本研究在他们研究的基础上,利用ITS序列分析将牡丹组9个野生种分为两个亚组,证明前人的结论是可靠的。
前人研究关于革质花盘亚组中牡丹间亲缘关系也不尽相同,在本实验结果中,紫斑牡丹与稷山牡丹和杨山牡丹亲缘关系最近,与卵叶牡丹亲缘关系也较近,这与邹喻苹利用RAPD[7]、林启冰Adh[12]和赵宣GPAT[13]稷山牡丹与紫斑牡丹及卵叶牡丹之间亲缘关系的研究结果一致。本实验结果稷山牡丹同时也与凤丹有较近的亲缘关系,这是因为“凤丹”由杨山牡丹驯化而来的栽培品种,这与孟丽在对部分牡丹野生种及栽培品种进行亲缘关系分析的结果一致[25]。而在本研究中四川牡丹,位于革质花盘亚组的底端,与紫斑牡丹栽培品种存在较近的亲缘关系,这个结果与Sang,Tang,Tank[11,27]及邹喻苹[25]分析的结果一致。单从形态学看,稷山牡丹与杨山牡丹、紫斑牡丹及卵叶牡丹之间亲缘关系非常近,该类群花盘在花期全包心皮,心皮5(~7),密被绒毛;而四川牡丹花盘在花期半包心皮,心皮无毛,且不总是5数,与革质花盘亚组中其它种类区别较明显;肉质花盘亚组则全为肉质而仅包心皮下部的花盘。从这一点上,四川牡丹应是革质花盘亚组中最进化的种,肉质花盘亚组处于更进化的地位。
3.3 牡丹栽培品种与野生种间关系
本实验结果中紫斑牡丹栽培品种“蝶恋花”、“包龙图”、“粉荷”3个个体聚在一起,与杨山牡丹亲缘关系最近,其次和紫斑牡丹野生种聚在一起,与稷山牡丹的亲缘关系不如杨山牡丹近,说明紫斑牡丹作为母本,而杨山牡丹作为父本在紫斑牡丹栽培品种中起到了极大地作用,其基因在紫斑牡丹中产生了很大程度的渗透,而稷山牡丹也存在渗透,可能在不同品种中渗透程度不同,且紫斑牡丹作为杂交亲本自身起源的复杂性及培育代数也会影响其栽培品种的形成,这与成仿云,李嘉珏等之前的研究结果是一致的[27]。另外,从树中可以看出紫斑牡丹栽培品种“粉燕尾”、“粉妆楼”与卵叶牡丹及P.suffruticosa聚为一支,说明卵叶牡丹也在一定程度上参与了紫斑牡丹栽培品种的杂交驯化,这个结果与李嘉珏根据形态学及中国牡丹栽培驯化的研究结果一致[28~30];其次和四川牡丹亲缘关系较近,这点与形态学的分类结果不一致。由于四川牡丹花盘革质,仅半包心皮,心皮无毛且不总为5个,洪德元和潘开玉并未将其归在复合体(包括稷山牡丹、卵叶牡丹、紫斑牡丹)内[3]。然而此ITS序列研究则支持紫斑牡丹与四川牡丹有较近的亲缘关系。而中原牡丹品种“大棕紫”、“姚黄”、“火炼金丹”单独聚为一支,除了紫斑牡丹栽培品种“粉妆楼”与三者遗传距离较大为0.053、0.057、0.069外,他们与其他种之间的遗传距离在0.003~0.026,其中与凤丹、稷山牡丹、卵叶牡丹、四川牡丹、杨山牡丹遗传距离较小,说明在中原牡丹品种中杨山牡丹、稷山牡丹及卵叶牡丹及四川牡丹参与其进化,此结果也验证了中原牡丹多元进化的特征,与孟丽等中原牡丹起源的研究结果一致[25]。
本实验对牡丹组野生种及部分栽培品种与来自GeneBank上的49条序列经过比对分析,能够在牡丹组野生种材料充足的条件下利用ITS序列将其清楚的分类,证明牡丹组分为革质和肉质花盘亚组的可靠性,并对他们的亲缘关系进一步分析。而matK基因由于信息位点不足、遗传距离分化不明显及系统进化树不可信,不能单独作为牡丹组及部分栽培品种之间的分类。
牡丹野生组由于地理位置分布的差异导致它们产生地理隔离,种质很难交流,而处于一个分布区的种质亲缘关系则较近,分布区环境及不同的生物因素会影响牡丹组进化过程中形态的变化[31]。研究人员会根据亚组间形态及生物学特性的不同进行牡丹近缘及远缘杂交选育工作,而亲本的选择极为关键[32],所以,清楚了解牡丹组间及野生种与栽培品种之间的亲缘关系对于杂交育种工作极为重要,何丽霞曾对牡丹野生种之间进行亚组内及组间正反的杂交,选育出50多个杂交新品种,为我国牡丹育种工作提供了重要依据[34]。
本研究利用核基因ITS和叶绿体基因matK进行测序,ITS能够揭示出牡丹组野生种间一定的亲缘关系,而叶绿体基因的进化还是比较保守,对牡丹组间的类群所提供的信息位点不足,原因可能与样本量大小有关,在今后的研究中可适当加大样本量。牡丹是“多地”“多元”“多种”起源,种内种间杂交事件复杂及其渐渗杂交和反复回交,导致其具有丰富的遗传多样性[28]。而在牡丹遗传多样性、鉴定品种和选择杂交亲本的研究偏少,因此,寻找高效的遗传标记及在基因组的层面上解决这些问题,能够为牡丹新品种培育提供一定的理论基础[34~35]。
1.赵宣.芍药属牡丹组(Paeoniasect.Moutan)种间关系及栽培牡丹的起源[D].重庆:西南大学,2007.
Zhao X.Phylogenetic relationship among the speices of sect.Moutanand origin of cultivated tree peony[D].Chongqing:Southwest University,2007.
2.中国牡丹全书编纂委员会.中国牡丹全书[M].北京:中国科学技术出版社,2002:1-310.
The Chinese Peony Encyclopedia Compilation Committee.The Chinese peony encyclopedia[M].Beijing:China Science and Technology Press,2002:1-310.
3.洪德元,潘开玉.芍药属牡丹组的分类历史和分类处理[J].植物分类学报,1999,37(4):351-368.
Hong D Y,Pan K Y.Taxonomical history and revision ofPaeoniasect.Moutan(Paeoniaceae)[J].Acta Phytotaxonomica Sinica,1999,37(4):351-368.
4.于玲,何丽霞.紫斑牡丹小孢子形成过程的细胞遗传学研究[J].西北植物学报,2000,20(3):467-471.Yu L,He L X.Cytogenetic study on microspore formative process ofPaeoniarockii[J].Acta Botanica Boreali-Occidentalia Sinica,2000,20(3):467-471.
5.席以珍.中国芍药属花粉形态及其外壁超微结构的观察[J].植物学报,1984,26(3):241-246.
Xi Y Z.The pollen morphology and exine ultrastructure ofPaeonial.in China[J].Acta Botanica Sinica,1984,26(3):241-246.
6.潘开玉.芍药科分布格局及其形成的分析[J].植物分类学报,1995,33(4):340-349.
Pan K Y.The analysis of distribution pattern in the Paeoniaceae and its formation[J].Acta Phytotaxonomica Sinica,1995,33(4):340-349.
7.邹喻苹,蔡美琳,王子平. 芍药属牡丹组的系统学研究—基于RAPD分析[J].植物分类学报,1999,37(3):220-227.Zou Y P,Cai M L,Wang Z P.Systematic studies onPaeoniasect.MoutanDC.based on RAPD analysis[J].Acta Phytotaxonomica Sinica,1999,37(3):220-227.
8.Sang T,Crawford D J,Stuessy T F.Documentation of reticulate evolution in peonies(Paeonia) using internal transcribed spacer sequences of nuclear ribosomal DNA:implications for biogeography and concerted evolution[J].Proceedings of the National Academy of Sciences of the United States of America,1995,92(15):6813-6817.
9.Sang T,Donoghue M J,Zhang D.Evolution of alcohol dehydrogenase genes in peonies(Paeonia):phylogenetic relationships of putative nonhybrid species[J].Molecular Biology and Evolution,1997,14(10):994-1007.
10.Ferguson D,Sang T.Speciation through homoploid hybridization between allotetraploids in peonies(Paeonia)[J].Proceedings of the National Academy of Sciences of the United States of America,2001,98(7):3915-3919.
11.Tank D C,Sang T.Phylogenetic utility of the glycerol-3-phosphate acyltransferase gene:evolution and implications inPaeonia(Paeoni-aceae)[J].Molecular Phylogenetics and Evolution,2001,19(3):421-429.
12.林启冰,周志钦,赵宣,等.基于Adh基因家族序列的牡丹组(Sect.MoutanDC.)种间关系[J].园艺学报,2004,31(5):627-632.
Lin Q B,Zhou Z Q,Zhao X,et al.Interspecific relationships among the wild species ofPaeoniaSect.MoutanDC.based on DNA sequences of Adh Gene family[J].Acta Horticulturae Sinica,2004,31(5):627-632.
13.赵宣,周志钦,林启冰,等.芍药属牡丹组(Paeoniasect.Moutan)种间关系的分子证据:GPAT基因的PCR-RFLP和序列分析[J].植物分类学报,2004,42(3):236-244.
Zhao X,Zhou Z Q,Lin Q B,et al.Molecular evidence for the interspecific relationships inPaeoniasect.Moutan:PCR_RFLP and sequence analysis of glycerol-3-phosphate acyltransferase(GPAT) gene[J].Acta Phytotaxonomica Sinica,2004,42(3):236-244.
14.张金梅,王建秀,夏涛,等.基于系统发育分析的DNA条形码技术在澄清芍药属牡丹组物种问题中的应用[J].中国科学(C辑:生命科学),2008,38(12):1166-1176.
Zhang J M,Wang J X,Xia T,et al.DNA barcoding:species delimitation in tree peonies[J].Science in China Series C:Life Sciences,2009,52(6):568-578.
15.李学营,彭建营,白瑞霞.基于核rDNA的ITS序列在种子植物系统发育研究中的应用[J].西北植物学报,2005,25(4):829-834.
Li X Y,Peng J Y,Bai R X.Use of ITS sequences based on nuclear rDNA for phylogenetic research of seed plants[J].Acta Botanica Boreali-occidentalia Sinica,2005,25(4):829-834.
16.朱元娣,曹敏格,许正,等.基于ITS和matK序列探讨新疆野苹果与中国苹果的系统演化关系[J].园艺学报,2014,41(2):227-239.
Zhu Y D,Cao M G,Xu Z,et al.Phylogenetic relationship between Xinjiang wild apple(MalussieversiiRoem.) andChineseapple(Malus×domesticasubsp.chinesnsis) based on ITS and matK sequences[J].Acta Horticulturae Sinica,2014,41(2):227-239.
17.赵宣,周志钦,林启冰,等.基于多基因序列和形态性状的牡丹组种间关系(英文)[J].植物分类学报,2008,46(4):563-572.Zhao X,Zhou Z Q,Lin Q B,et al.Phylogenetic analysis ofPaeoniasect.Moutan(Paeoniaceae) based on multiple DNA fragments and morphological data[J].Journal of Systematics and Evolution,2008,46(4):563-572.
18.Yan H F,Hao G,Hu C M,et al.DNA barcoding in closely related species:A case study ofPrimulaL.sect.ProliferaePax(Primulaceae) in China[J].Journal of Systematics and Evolution,2011,49(3):225-236.
19.Fu Y M,Jiang W M,Fu C X.Identification of species withinTetrastigma(Miq.) Planch.(Vitaceae) based on DNA barcoding techniques[J].Journal of Systematics and Evolution,2011,49(3):237-245.
20.任保青,陈之端.植物DNA条形码技术[J].植物学报,2010,45(1):1-12.
Ren B Q,Chen Z R.DNA barcoding plant life[J].Chinese Bulletin of Botany,2010,45(1):1-12.
21.于玲,何丽霞,李嘉珏,等.牡丹野生种间蛋白质谱带的比较研究[J].园艺学报,1998,25(1):99-101.
Yu L,He L X,Li J J,et al.Comparative studies on protein zones of wild tree peony species[J].Acta Horticulturae Sinica,1998,25(1):99-101.
22.邹喻苹,蔡美琳,张志宪,等.矮牡丹的遗传多样性与保护对策[J].自然科学进展,1999,9(5):469-472.
Zou Y P,Cai M L,Zhang Z X,et al.Genetic diversity and protection countermeasure ofPaeoniasuffruticosasubsp.Spontanea[J].Progress in Natural Science,1999,9(5):469-472.
23.袁涛,王莲英.中国栽培牡丹起源的形态分析[J].山东林业科技,2004(6):1-3.
Yuan T,Wang L Y.Morphologic analysis on the origin of Chinese peony cultivars[J].Journal of Shandong Forestry Science and Technology,2004(6):1-3.
24.袁涛,王莲英.我国芍药属牡丹组革质花盘亚组的形态学研究[J].园艺学报,2003,30(2):187-191.
Yuan T,Wang L Y.Morphological studies onPaeoniaSect.Moutansubsect vagiatae in China[J].Acta Horticulturae Sinica,2003,30(2):187-191.
25.孟丽.部分野生、栽培牡丹种质资源亲缘关系的RAPD研究[D].泰安:山东农业大学,2003.
Meng L.Phylogenetic Relationship analyzed using RAPD makers among partial germplasm ofPaeoniasect.Moutan[D].Tai’an:Shandong Agricultural University,2003.
26.Sang T,Zhang D M.Reconstructing hybrid speciation using sequences of low copy nuclear genes:hybrid origins of fivePaeoniaspecies based on ADH gene phylogenies[J].Systematic Botany,1999,24(2):148-163.
27.成仿云,李嘉珏,陈德忠,等.中国紫斑牡丹[M].北京:中国林业出版社,2005.
Cheng F Y,Li J J,Cheng D Z,et al..Chinese flare Mudan[M]. Beijing:China Forestry Publishing,2005.
28.李嘉珏.中国牡丹起源的研究[J].北京林业大学学报,1998,20(2):22-26.
Li J J.Studies on the origin of Chinese Mudan(Tree Peony)[J].Journal of Beijing Forestry University,1998,20(2):22-26.
29.何丽霞,李睿,李嘉珏,等.中国野生牡丹花粉形态的研究[J].兰州大学学报,2005,41(4):43-49.
He L X,Li R,Li J J,et al.Studies on pollen morphology of the wild peony[J].Journal of Lanzhou University,2005,41(4):43-49.
30.洪涛,张家勋,李嘉珏,等.中国野生牡丹研究(一)芍药属牡丹组新分类群[J].植物研究,1992,12(3):223-234.
Hong T,Zhang J X,Li J J,et al.Study on the Chinese wild woody peonies(Ⅰ) new taxa ofPaeoniaL.sect.MoutanDC[J].Bulletin of Botanical Research,1992,12(3):223-234.
31.周仁超,姚崇怀.芍药属牡丹组革质花盘亚组的系统演化探讨[J].植物研究,2002,22(1):72-75.
Zhou R C,Yao C H.Studies on phylogeny ofPaeoniasectionMoutansubsection vaginatae[J].Bulletin of Botanical Research,2002,22(1):72-75.
32.王越岚.牡丹的杂交育种及组间杂种育性的研究[D].北京:北京林业大学,2009.
Wang Y L.Cross-breeding in tree peony and fertility research of intersectional hybrids[D].Beijing:Beijing Forestry University,2009.
33.何丽霞,李睿,张延东,等.牡丹杂交育种试验研究[J].甘肃林业科技,2011,36(3):1-6,20.
He L X,Li R,Zhang Y D,et al.Study on cross breeding new cultivars of tree-peony[J].Journal of Gansu Forestry Science and Technology,2011,36(3):1-6,20.
34.施季森,王占军,陈金慧.木本植物全基因组测序研究进展[J].遗传,2012,34(2):145-156.
Shi J S,Wang Z J,Chen J H.Progress on whole genome sequencing in woody plants[J].Hereditas,2012,34(2):145-156.
35.黎裕,李英慧,杨庆文,等.基于基因组学的作物种质资源研究:现状与展望[J].中国农业科学,2015,48(17):3333-3353.
Li Y,Li Y H,Yang Q W,et al.Genomics-based crop germplasm research:advances and perspectives[J].Scientia Agricultura Sinica,2015,48(17):3333-3353.
WithITSnuclear gene sequence as molecular markers to establish research of Paeonia Rockii cultivars in gansu province standard(GNSW-2014-7);Gansu Province Agricultural and Pastoral Office biological technology special project
introduction:CHOU Huan-Huan(1993—),female,master’s graduate students,mainly engaged in forest tree genetics and breeding research.
date:2017-01-06
InterspecificRelationshipamongtheWildSpeciesofPaeoniaSect.MoutanDCwithITSandmatKSequence
CHOU Huan-Huan TANG Hong*
(College of Forestry in Gansu Agricultural University,Lanzhou 730070)
Revealing the interspecific relationship between wild species ofPaeoniaSect.MoutanDC at molecular evidences would provide theoretical basis for preserving and utilizing wild peony resources. The sequence of ribosomal DNA internal transcribed spacer(ITS) and the coding region of thematKgene sequenced from 9PaeoniaSect.MoutanDC, 5 cultivars ofP.rockii, and 3 accessions ofP.suffriticosa. Sequences ofITSandmatKof oneP.suffruticosa, oneP.delavayiand three outgroups ofP.lactiflora,P.veitchiiandP.obovatawere retrieved from GeneBank. The 44 sequences were obtained and corrected, and the divergences, variable sites, parsim-informative sites, the ratio of transition to transversion(R) and pairwise distances were analyzed. The phylogenetic analysis was conducted by Neighbor-joining(NJ) method. The size ofITSsequences ofPaeoniaSect.MoutanDC wild species and cultivars ranged from 750 to 800 bp containing 86 polymorphic sites and 74 parsim-informative sites with R of 1.2, whereas, theirmatKsequences encompassing 20 parsim-informative sites with R of 1.7. From the phylogenetic trees ofITS,P.jishanensis,P.rockii,P.qiuiandP.ostiifell into a large clade. The similarity coefficients betweenP.ludlowii, and the species ofP.lutea,P.potaniniiandP.delavayiwere clustered in another clade. These two clades corresponded well to Subsect.P.decompositawere at the bottom of Subsect. Vaginatae, which showed the classification ofP.decompositainto Subsect Vaginatae. BymatKsequences analysis, there is closely genetic distance between wild species fromPaeoniaSec.MoutanDC; however, there was no clear genetic relationships between wild species. As a result,ITSsequence can be better used to study interspecific relationship among wild species ofPaeoniaSect.MoutanDC.
PaeoniaSect.MoutanDC;ITS;matK;genetic relationship
以核基因ITS序列为分子标记建立甘肃省紫斑牡丹品种鉴定标准的研究(GNSW-2014-7);省农牧厅生物技术专项
丑欢欢(1993—),女,研究生,主要从事林木遗传育种方面的研究。
* 通信作者:E-mail:gsth@21cn.com
2017-01-06
* Corresponding author:E-mail:gsth@21cn.com
S567.1+5
A
10.7525/j.issn.1673-5102.2017.04.017
