甘薯等10种作物基因的生物信息学分析
2017-11-07杨冬静张成玲孙厚俊赵永强谢逸萍
杨冬静,徐 振,张成玲,孙厚俊,赵永强,谢逸萍
(江苏徐淮地区徐州农业科学研究所/中国农业科学院 农业部甘薯生物学与遗传育种重点实验室,江苏 徐州 221131)
甘薯等10种作物基因的生物信息学分析
杨冬静,徐 振,张成玲,孙厚俊,赵永强,谢逸萍*
(江苏徐淮地区徐州农业科学研究所/中国农业科学院 农业部甘薯生物学与遗传育种重点实验室,江苏 徐州 221131)
采用生物信息学相关软件对甘薯等10种NAC1蛋白的氨基酸序列及其理化性质、磷酸化位点、疏水性或亲水性、二级结构和三级结构、跨膜结构域、信号肽、亚细胞定位以及保守结构域等进行了预测和分析。结果表明:供试10种NAC1蛋白的磷酸化位点主要为丝氨酸、苏氨酸和酪氨酸;这些NAC1蛋白均为亲水性蛋白,属于非分泌蛋白,不具有信号肽,没有明显的跨膜结构域;在它们的二级结构中,无规则卷曲所占比例最高;MtNAC1和CaNAC1可能定位于叶绿体,NtNAC1和GmNAC1可能定位于线粒体,甘薯及其他6种作物的NAC1蛋白可能定位于细胞核;甘薯等NAC1蛋白的三级结构与水稻NAC1蛋白具有一定的相似性;10种NAC1蛋白均含有1个NAM (No apical meristem)功能结构域。氨基酸序列比对和进化树分析显示IbNAC1与CaNAC1的亲缘关系最近。
甘薯;NAC1;生物信息学;功能结构域;亚细胞定位;基因
NAC转录因子是一类结构独特、功能多样的植物特有的转录因子[1]。1996年Souer等首次从矮牵牛中分离得到第一个NAC转录因子,研究表明该转录因子影响矮牵牛顶端分生组织的形成与分化[2];1997年Aida等报道矮牵牛NAM、拟南芥ATAF1/2和CUC2编码蛋白质的N端均含有一个保守的NAC结构域[3],取这3个基因名称的首字母,后来将含NAC结构域的蛋白统称为NAC转录因子。近20年来,科学家们相继在水稻[4]、大豆[5]、杨树[6]、葡萄[7]和烟草[8]等多种作物中克隆到NAC转录因子。氨基酸序列分析表明NAC家族转录因子具有显著的结构特点,其编码蛋白的N端含有高度保守的NAM结构域;而C端具有高度的多样性,但脯氨酸、丝氨酸、苏氨酸、谷氨酸等单个氨基酸重复出现的频率高[9]。研究表明,NAC转录因子通过直接参与或调控参与干旱、低温或高盐应答基因的表达,在植物抗干旱、低温或高盐等非生物逆境胁迫中起重要作用。如Hu等通过过表达OsNAC1基因增强了水稻的抗旱性和耐盐性,并且提高水稻结实率34%以上[10];Tran等过表达AtNAC019、AtNAC055和AtNAC072/RD26不仅显著提高了植株的抗旱性,还引起多个胁迫诱导基因的上调表达,并且发现它们可以与干旱诱导反应基因ERD1 (Early responsive to dehydration )启动子中的CATGTG顺式作用元件结合,从而调控ERD1以及下游基因的表达[11]。Tang等(2012)[12]在拟南芥中过表达TaNAC2,发现转基因拟南芥的抗旱、抗盐和抗冷害能力得到增强;Mao等(2012)[13]在烟草中过量表达TaNAC2a,增强了转基因烟草对干旱的耐受性。除了参与非生物胁迫,近年来的研究还发现NAC转录因子家族基因在植物遭受病原菌等生物胁迫或抗病信号分子如SA、JA等处理条件下,其表达水平发生了显著变化[14-15],表明NAC转录因子在作物抗病反应中也起着至关重要的作用。
我国是世界上最大的甘薯生产国,常年种植面积约为500万hm2。然而,部分甘薯主产区由于甘薯种植年限长、引种不规范以及检验检疫技术不完善,导致南病北移、南北病混发,新发生病害如甘薯病毒病SPVD、甘薯褐斑病、甘薯白绢病等频有报道,病虫害发生呈现逐步加重的趋势,严重制约了甘薯产业的发展[16]。由于甘薯在遗传上的高度杂合性以及种内和种间存在的广泛杂交不亲和性,采用常规育种方法选育抗病甘薯品种周期长、效率低。随着分子生物学技术的快速发展,采用基因工程技术定向改良甘薯的性状已成为现实,这为甘薯抗病基因工程育种提供了一条新途径,而获得优良抗病基因是实现这一目标的前提和基础,因此对甘薯抗病相关基因的克隆、序列分析和功能研究显得尤为重要。本文采用生物信息学分析方法,对甘薯等10种植物NAC1基因的氨基酸序列的组成、理化性质、二级结构和三级结构、蛋白质亚细胞定位以及功能结构域等进行了预测和分析,以期为甘薯NAC1基因(IbNAC1基因)的功能研究及其作用机制以及将IbNAC1应用于甘薯抗病抗逆育种提供理论依据。
1 材料与方法
1.1 NAC1基因序列来源
从NCBI (National Center for Biotechnology Information, http: //www.ncbi.nlm.nih.gov/)的核苷酸及蛋白质数据库中下载了甘薯等10种植物的NAC1基因的全长CDS序列及其对应的氨基酸序列:甘薯(Ipomoeabatatas),登录号ACT55332.1;拟南芥(Arabidopsisthaliana),登录号AF198054_1;蒺藜苜蓿(Medicagotruncatula),登录号AAF68626.1;茄子(Solanummelongena),登录号AHB63601.1;大豆(Glycinemax),登录号AAY46121.1;番茄(Solanumlycopersicum),登录号AGJ52115.1;烟草(Nicotianatabacum),登录号ACF19785.1;水稻(Oryzasativa),登录号AIX03023.1;杨树(Populustrichocarpa),登录号XP_002310688.1;辣椒(Capsicumannuum),登录号AAW48094.1。
1.2 研究方法
利用Blastp、ProtParam、NetPhos 3.1server、Protscale、SOPMA、SWISS-MODEL、TMHMM、Signal 3.0、WoLF PROST等在线生物信息学程序进行NAC1编码的氨基酸组成、理化性质、蛋白磷酸化位点预测,蛋白疏水性或亲水性预测和分析,蛋白二级结构和三级结构预测,跨膜结构域、信号肽、亚细胞定位以及保守结构域预测。采用生物信息学软件DNAMAN进行氨基酸多重序列比对。采用Maga 6.0 软件中的Neighbor-Joining法绘制蛋白系统进化树。
2 结果与分析
2.1 不同植物NAC1蛋白的氨基酸组成和理化性质分析
采用Physicochemical Property ProtParam对甘薯、拟南芥、蒺藜苜蓿、茄子、大豆、番茄、烟草、水稻、杨树、辣椒等10种作物中NAC1蛋白的氨基酸组成及理化性质进行分析,氨基酸残基数目和相对分子量见表1。氨基酸含量分析表明,赖氨酸Lys、脯氨酸Pro和亮氨酸Leu为供分析的10种NAC1蛋白的主要氨基酸;蛋白稳定性预测结果表明,甘薯、拟南芥、蒺藜苜蓿和杨树等4种NAC1蛋白为非稳定蛋白,其他6种NAC1蛋白为稳定蛋白;等电点预测结果表明,甘薯、拟南芥、蒺藜苜蓿和水稻等4种NAC1蛋白有酸性等电点,其余6种NAC1蛋白有碱性等电点。以threshold大于0.5为标准,得到各供试作物NAC蛋白磷酸化位点数量的预测结果,如表2所示,NAC1蛋白磷酸化位点主要为丝氨酸、苏氨酸和酪氨酸,除烟草NAC1蛋白外,其他9种作物NAC1蛋白中丝氨酸磷酸化位点最多,苏氨酸和酪氨酸次之。
2.2 不同植物NAC1的疏水性/亲水性的预测和分析
ProtScale预测结果显示:甘薯IbNAC1多肽链第13和14位谷氨酰胺(Q)最低分值为-3.500,亲水性最强;第52位异亮氨酸(I)最高分值为2.011,疏水性最强(图1);从整体上来看,亲水氨基酸数目均大于疏水氨基酸数目,由此推测IbNAC1为亲水性蛋白。对甘薯之外的其他9种NAC1蛋白的亲/疏水性进行预测分析,也发现了各自的亲水性最强的氨基酸和疏水性最强的氨基酸,但从整体分析都发现亲水氨基酸数目大于疏水氨基酸数目,因此初步推测这10种NAC1蛋白均为亲水性蛋白。
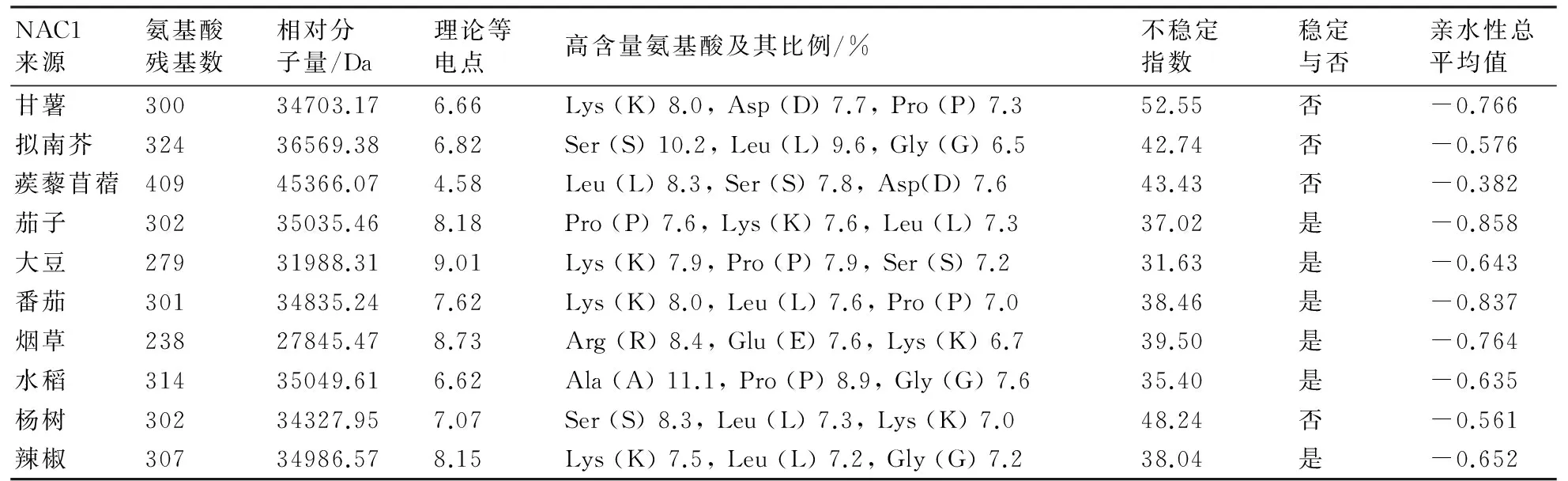
表1 不同作物NAC1蛋白的理化性质预测和分析结果
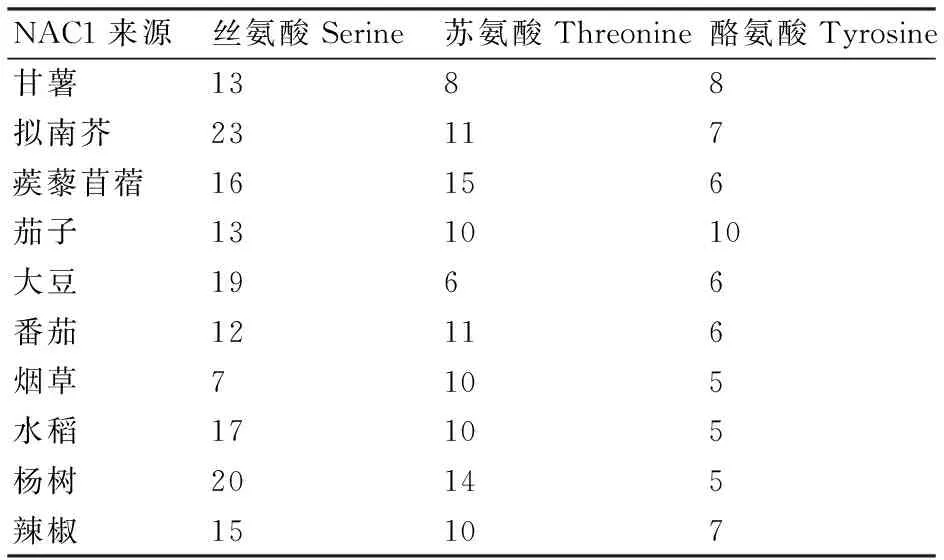
表2 甘薯等10种作物NAC1蛋白磷酸化作用位点的预测
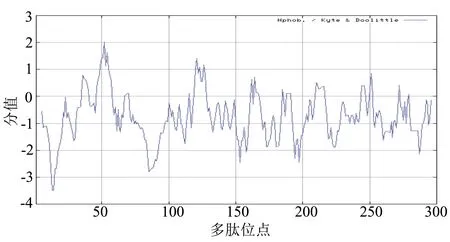
图1 IbNAC1蛋白的亲/疏水性预测
2.3 不同植物NAC1信号肽的预测和分析
采用SignalP 3.0 Server进行信号肽预测,结果表明甘薯NAC1为非分泌蛋白,不含信号肽(图2)。采用同样的方法对其他9种作物中的NAC1蛋白进行信号肽预测,也得到了一致的结果,因此初步推测这10种NAC1蛋白均为非分泌蛋白,不含信号肽。
2.4 不同植物NAC1蛋白的跨膜结构域的预测
利用TMHMM对不同作物NAC1蛋白序列的跨膜结构域进行预测,结果在10种作物的NAC1蛋白中均未发现明显的跨膜结构域(图3)。
2.5 不同植物NAC1的二级结构和亚细胞定位的预测
采用在线软件SOPMA对甘薯等作物NAC1氨基酸序列进行二级结构预测,结果(表3)表明:甘薯NAC1由27.00% α-螺旋、17.00%延伸链、8.33% β-折叠和47.67%无规则卷曲组成;甘薯等10种NAC1蛋白二级结构中无规则卷曲所占比例最高,为38.66%~53.76%;此外,α-螺旋和延伸链也是NAC1蛋白的主要二级元件,其中α-螺旋所占比例为11.83%~32.48%,延伸链所占比例为15.29%~26.52%;β-折叠所占比例最低,为5.41%~10.50%。
图2 IbNAC1蛋白的信号肽预测
图3 IbNAC1蛋白跨膜区的预测
采用WoLF PROST在线软件预测NAC1蛋白的亚细胞定位,预测得分结果如表4所示,由该表可以看出:MtNAC1和CaNAC1定位于叶绿体的可能性较大;NtNAC1和GmNAC1定位于线粒体的可能性较大;甘薯及其他6种作物的NAC1蛋白定位于细胞核的可能性较大。
2.6 不同植物NAC1蛋白的三级结构的预测
以水稻NAC1蛋白3ulx.1.A为模板,进行其他9种NAC1蛋白的同源建模,结果(图4)表明:IbNAC1等其他9种NAC1蛋白的三级结构均与OsNAC1具有一定的相似性;但是α-螺旋和β-折叠的数目和状态以及无规则卷曲的长度等不尽相同,导致其空间结构有所差异。由此推测它们虽然均属同类转录因子,但各自所具有的功能可能不同。
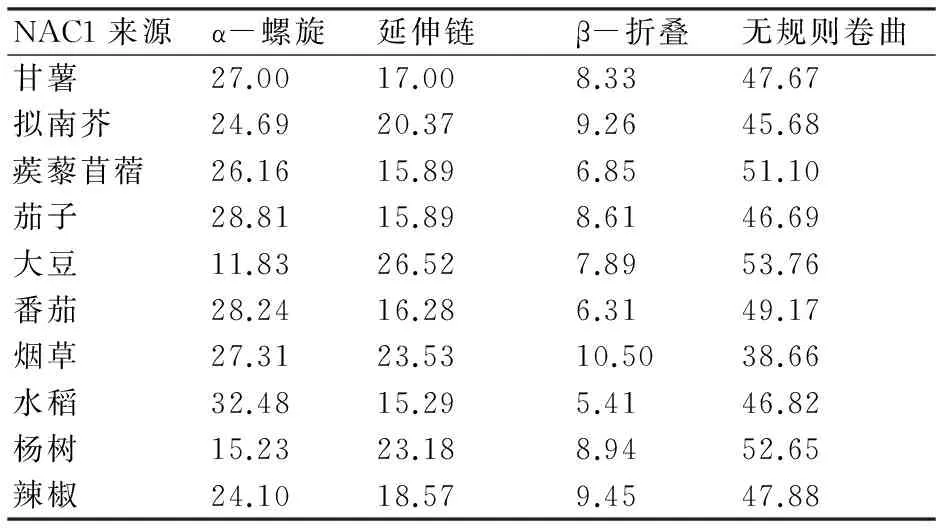
表3 甘薯等10种作物NAC1蛋白二级结构及各元件含量预测 %
2.7 不同植物NAC1蛋白功能结构域的预测和分析
采用NCBI Conserved domain search对NAC1蛋白的功能结构域进行预测,发现:甘薯、辣椒、番茄等8种作物的NAC1蛋白的N-端均含有1个氨基酸残基数目为120多的NAM (No apical meristem)结构域(图5);蒺藜苜蓿NAC1蛋白除含有1个氨基酸残基数目为126的NAM超级家族蛋白功能结构域外,还含有1个氨基酸残基数目为106的Marek-A超级家族蛋白功能结构域(图6);烟草NAC1蛋白含有1个氨基酸残基数目仅为18的NAM超级家族蛋白功能结构域(图7)。
2.8 不同植物NAC1蛋白氨基酸序列比对及系统进化分析
对甘薯、水稻和拟南芥等4种NAC1蛋白氨基酸序列比对结果显示,NAC1蛋白在N-端约150个氨基酸残基具有高度的保守性,而C-端具有很强的多样性,但从图8可以发现,这4种NAC1蛋白C-端仍然有苯丙氨酸(F)、天冬氨酸(D)、脯氨酸(P)等几个较为保守的氨基酸。蛋白系统进化树分析结果如图9所示,10种不同植物的NAC1氨基酸序列聚成3簇,其中烟草和蒺藜苜蓿各自成一簇;甘薯、辣椒、番茄等其他8种作物的NAC1氨基酸序列聚成一簇。这表明IbNAC1与CaNAC1的亲缘关系最近。
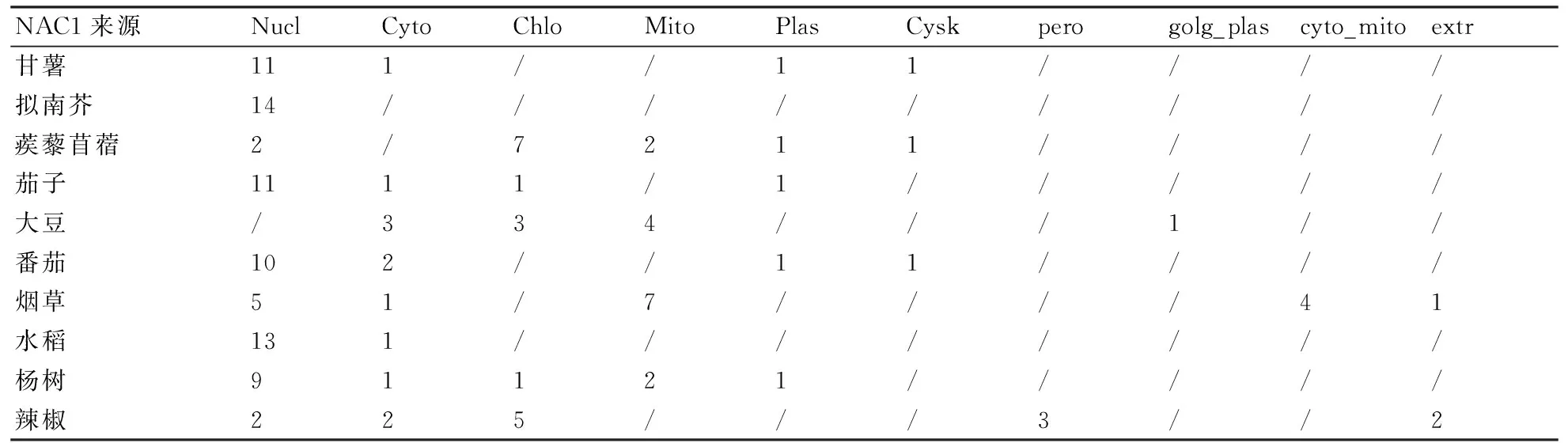
表4 甘薯等10种作物NAC1亚细胞定位预测结果
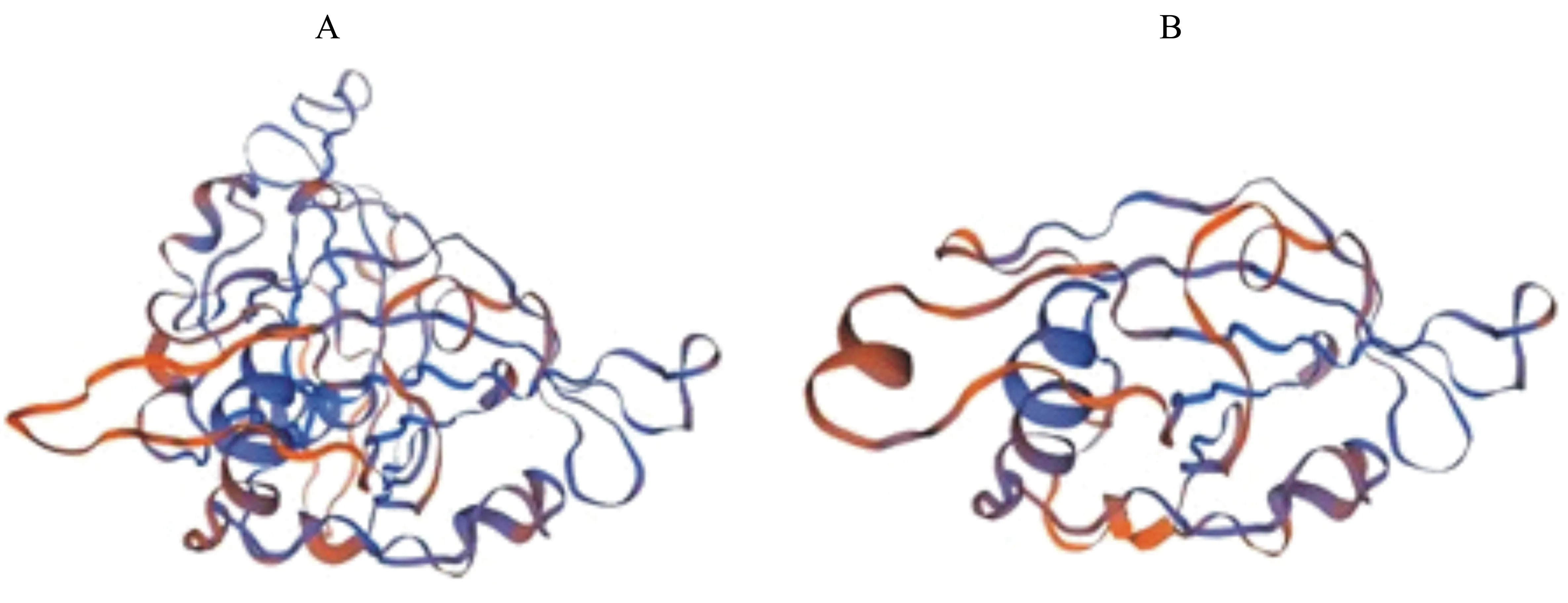
图4 IbNAC1(A)和OsNAC1(B)三维结构模型预测
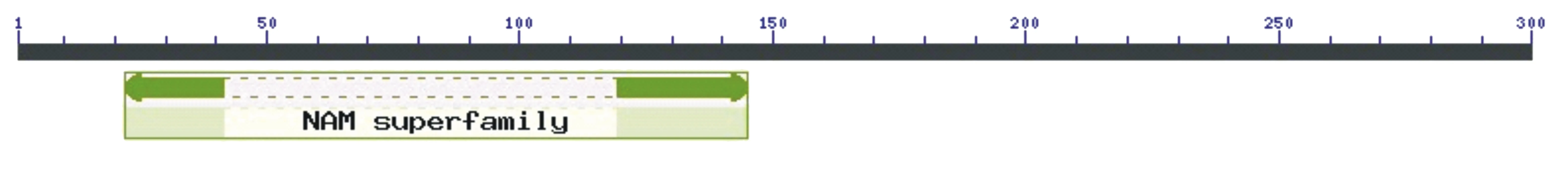
图5 IbNAC1氨基酸序列功能域预测
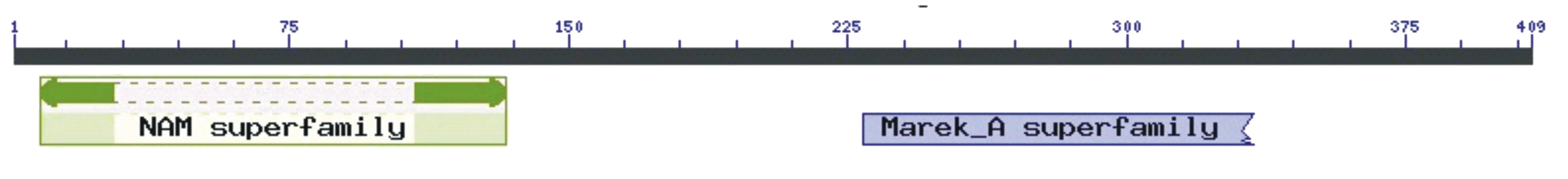
图6 MtNAC1氨基酸序列功能域预测

图7 NtNAC1氨基酸序列功能域预测
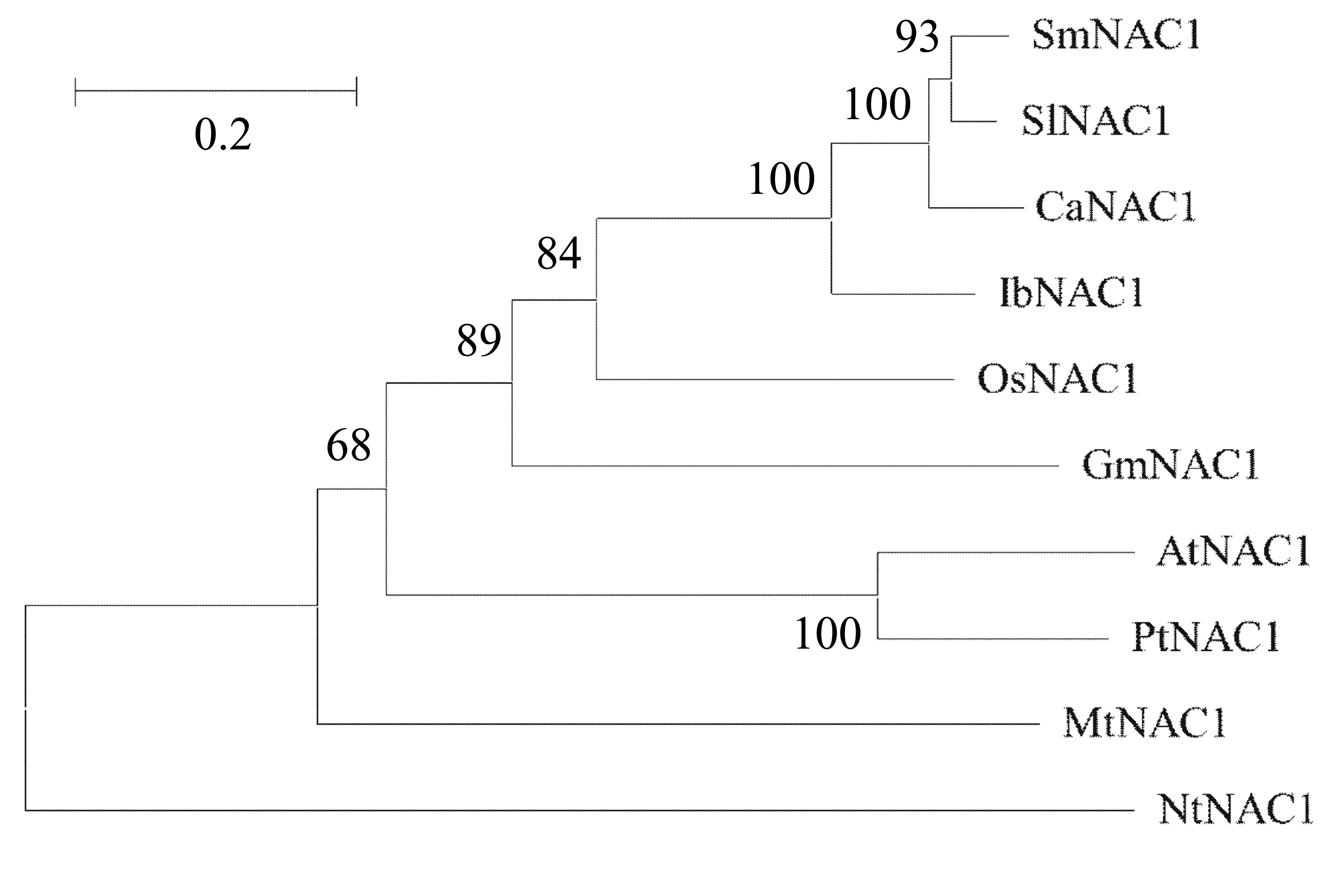
图9 甘薯等10种植物NAC1氨基酸进化树分析
3 讨论和结论
由于甘薯基因组的复杂性,目前关于其抗性基因的研究报道较少。王钰等[17]克隆了甘薯抗病基因的同源序列(RGA),序列分析表明这些RGA属于NBS-LRR类型,与L6、N、RGC1等抗病基因的同源性较高;陈观水等[18]采用反转录PCR和RACE技术,分离得到IbNPR1全长cDNA并构建植物表达载体进行了烟草的遗传转化;柏洁等[19]采用反转录PCR分离到IbSGT1基因的全长序列,定量PCR研究表明该基因在甘薯的根、茎、叶中均有表达;Chen等[20]报道IbNAC1通过结合SWRE基序使得Sporamin基因上调表达从而抵御甘薯的机械损伤和昆虫取食。已有的研究报道表明番茄等NAC1可参与病原菌或病毒侵染后的寄主反应,从而直接或间接调控植株的抗病性[21-22]。目前,我国在甘薯抗病鉴定方法的建立和抗病品种选育等方面取得了较好的成果,但是关于甘薯抗病基因及其分子调控机制方面的研究相对匮乏,IbNAC1是否参与甘薯抗病调控途径值得我们进一步研究。
本研究通过利用生物信息学相关分析软件对甘薯等10种植物NAC1基因的氨基酸序列的组成、理化性质、二级结构、三级结构、亚细胞定位和功能结构域等进行了预测和分析。结果表明:不同植物NAC1基因的氨基酸序列组成和理化性质如稳定性、等电点等有所差异,但都属于亲水性蛋白;供试10种作物NAC1蛋白磷酸化位点均主要为丝氨酸、苏氨酸和酪氨酸;均未发现有明显的跨膜结构域和信号肽;在二级结构中,无规则卷曲所占比例最高,其次是α-螺旋和延伸链,但各自所占比例有所差异;供试的10种NAC1蛋白的三级结构具有一定的相似性,但由于α-螺旋和β-折叠的数目和状态以及无规则卷曲的长度等不尽相同,导致其空间结构不同;甘薯NAC1蛋白可能定位于细胞核;甘薯等10种NAC1蛋白均含有NAM结构域;进化树分析表明IbNAC1和CaNAC1的亲缘关系最近。
对NAC1蛋白氨基酸序列进行预测和分析有助于我们进一步理解其对甘薯的胁迫调控作用及其分子机制,也可为该基因应用于甘薯抗性分子育种奠定理论基础,对于甘薯基础理论研究和实际应用都具有重要意义。
[1] Addie N O, Heidi A E, Leila L L, et al. NAC transcription factors: structurally distinct, functionally diverse [J]. TRENDS in Plant Science, 2005, 2(10): 79-87.
[2] Souer E, Houwelingen V A, Kloos D, et al. The no apical meristem gene of petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordial boundaries [J]. Cell, 1996, 85: 159-170.
[3] Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation inArabidopsis: an analysis of the cup-shaped cotyledon mutant [J]. Plant Cell, 1997(9): 841- 857.
[4] Nuruzzaman M, Manimekalai R, Sharoni A M, et al. Genome-wide analysis of NAC transcription factor family in rice [J]. Gene, 2010, 465(1-2): 30-44.
[5] Le D T, Nishiyama R, Watanabe Y, et al. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress [J]. DNA Research, 2011, 18: 263-276.
[6] Hu R, Qi G, Kong Y, et al. Comprehensive analysis of NAC domain transcription factor gene family inPopulustrichocarpa[J]. BMC Plant Biology, 2010, 10(1): 145.
[7] Wang N, Zheng Y, Xin H, et al. Comprehensive analysis of NAC domain transcription factor gene family inVitisvinifera[J]. Plant Cell Reports, 2013, 32(1): 61-75.
[8] Rushton P J, Bokowiec M T, Han S C, et al. Tobacco transcription factors: novel insights into transcriptional regulation in the Solanaceae [J]. Plant Physiology, 2008, 147(1): 280-295.
[9] Hisako O, Kouji S, Koji D, et al. Comprehensive analysis of NAC family genes inOryzasativaandArabidopsisthaliana[J]. DNA Research, 2003(10): 239-247.
[10] Hu H H, Dai M Q, Yao J L, et al. Overexpressing a NAM, ATAF, and CUC (NAC) transcription factor enhances drought resistance and salt tolerance in rice [J]. Proceedings of the National Academy of Sciences, USA, 2006, 103(35): 12987-12992.
[11] Tran L S, Nakashima K, Sakuma Y, et al. Isolation and functional analysis ofArabidopsisstress inducible NAC transcription factors that bind to a drought responsive cis-element in the early responsive to dehydration stress 1 promoter [J]. Plant Cell, 2004, 16(9): 2481-2498.
[12] Tang Y M, Liu M Y, Gao S Q, et al. Molecular characterization of novelTaNACgenes in wheat and overexpression ofTaNAC2aconfers drought tolerance in tobacco [J]. Physiologia Plantarum, 2012, 114(3): 210-224.
[13] Mao X G, Zhang H Y, Qian X Y, et al.TaNAC2, a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances inArabidopsis[J]. Journal of Experimental Botany, 2012, 63(8): 2933-2946.
[14] Lin R M, Zhao W S, Meng X B, et al. Rice geneOsNAC19 encodes a novel NAC-domain transcription factor and responds to infection byMagnaporthegrisea[J]. Plant Science, 2007, 172(1): 120-130.
[15] Xia N, Zhang G, Liu X Y, et al. Characterization of a novel wheat NAC transcription factor gene involved in defense response against stripe rust pathogen infection and abiotic stresses [J]. Molecular Biology Reports, 2010, 37(8): 3703-3712.
[16] 马代夫,李强,曹清河,等.中国甘薯产业及产业技术的发展与展望[J].江苏农业学报,2012,28(5):969-973.
[17] 王钰,王荣富,何国浩.甘薯抗病基因同源序列的克隆与分析[J].南京农业大学学报,2008,31(3):81-86.
[18] 陈观水.甘薯抗病相关基因的克隆与分析[D].福州:福建农林大学,2007.
[19] 柏洁.甘薯抗病相关基因SGT1的克隆与表达研究[D].福州:福建农业大学,2014.
[20] Chen S P, Lin I W, Chen X, et al. Sweet potato NAC transcription factor, IbNAC1, upregulates sporamin gene expression by binding the SWRE motif against mechanical wounding and herbivore attack [J]. The Plant Journal, 2016, 86: 234-248.
[21] Selth L A, Dogra S C, Rasheed M S, et al. A NAC domain protein interacts with tomato leaf curl virus replication accessory protein and enhances viral replication [J]. Plant Cell, 2005, 17: 311-325.
[22] Xie Q, Sanz-Burgos A P, Guo H, et al. GRAB proteins, novel members of the NAC domain family, isolated by their interaction with a gemini virus protein [J]. Plant Molecular Biology, 1999, 39: 647-656.
BioinformaticsAnalysisofNAC1GeneinTenCropsIncludingSweetPotato
YANG Dong-jing, XU Zhen, ZHANG Cheng-ling, SUN Hou-jun, ZHAO Yong-qiang, XIE Yi-ping*
(Xuzhou Institute of Agricultural Science in Xu-Huai Region of Jiangsu / Key Laboratory of Biology and Genetic Improvement of Sweet Potato of Agricultural Ministry, Chinese Academy of Agricultural Sciences, Xuzhou 221131, China)
In this study, bioinformatics software was used to predict and analyze the amino acid sequence, physicochemical properties, phosphorylation sites, hydrophobicity (or hydrophilicity), secondary structure and tertiary structure, transmembrane domain, signal peptide, subcellular localization and conserved domains of NAC1 protein in sweet potato and other nine crops. The results showed that: the phosphorylation sites of tested ten kinds of NAC1 proteins were mainly serine, threonine and tyrosine; these NAC1 proteins were hydrophilic protein, and they belonged to non-secretory protein without both signal peptide and obvious transmembrane domain; random coil was the most important element in the secondary structure of these NAC1 proteins; MtNAC1 and CaNAC1 maybe were located in chloroplast, NtNAC1 and GmNAC1 might be located in mitochondria, and other six NAC1 proteins including IbNAC1 might be localized in cell nucleus; the tertiary structure of IbNAC1 protein and other NAC1 proteins was somewhat similar to that of OsNAC1 protein; all tested NAC1 proteins contained a NAM (No apical meristem) functional domain. Amino acid sequence alignment and phylogenetic tree analysis indicated that IbNAC1 had the closest genetic relationship with CaNAC1.
Sweet potato; NAC1; Bioinformatics; Functional domain; Subcellular localization; Gene
2017-07-18
国家现代农业产业技术体系建设专项(CARS-11-B-09);江苏省政府留学奖学金项目(JS-2014-120);教育部农作物生
物灾害综合治理重点实验室/农业部华东作物有害生物综合治理重点实验室开放基金项目。
杨冬静(1983─),女,四川射洪人,助理研究员,硕士,主要从事植物病理学研究。*通讯作者:谢逸萍。
S531
A
1001-8581(2017)11-0007-06
(责任编辑:黄荣华)
