大肠杆菌中dAb一级结构与可溶性表达关联
2017-11-01郭雯雯白仲虎杨艳坤戴晓峰
郭雯雯 , 刘 萌 , 白仲虎 , 杨艳坤 *, 戴晓峰
(1.江南大学 粮食发酵工艺与技术国家工程实验室,江苏 无锡 214122;2.江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122;3.江南大学 糖化学与生物技术教育部重点实验室,江苏 无锡 214122)
大肠杆菌中dAb一级结构与可溶性表达关联
郭雯雯1,2,3, 刘 萌1,2,3, 白仲虎1,2,3, 杨艳坤*1,2,3, 戴晓峰1,2,3
(1.江南大学 粮食发酵工艺与技术国家工程实验室,江苏 无锡 214122;2.江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122;3.江南大学 糖化学与生物技术教育部重点实验室,江苏 无锡 214122)
抗体类药物研发是药物发展的一个重要研究方向,目前已有许多抗体在原核表达体系中成功表达。人们通常采用“试错法”优化表达系统,提高蛋白质产量,但该方法费时费力且成功率低,因此迫切需要探索氨基酸组成与蛋白质可溶性表达水平之间的关联,以指导表达系统的选择或蛋白质分子的定向改造。在单域抗体dAb分子的C端末尾添加一个氨基酸,发现单个氨基酸的改变足以引起蛋白质可溶性表达量发生显著变化。通过层次聚类、Cluster Omega对dAb氨基酸序列与表达水平分别进行分类,发现两者分类一致的比例达到70%,证明了氨基酸序列与蛋白质表达水平间的关联。通过对65个CDR随机突变的dAb分子进行线性建模分析其可溶性表达量,发现对dAb胞外质量分数有显著影响作用的氨基酸组合为S(丝氨酸),R(精氨酸),N(天冬酰胺),D(天冬氨酸),Q(谷氨酰胺),Y(酪氨酸),F(苯丙氨酸),G(甘氨酸),对 dAb 胞内质量分数有显著影响作用的氨基酸组合为 G,R,C(半胱氨酸),N,S,Y,K(赖氨酸),A(丙氨酸),对dAb蛋白质总量有显著影响的组合为 R,S,G,N,Y,C,Q,F,T(苏氨酸)。 其中 R、S和 G 的含量显著影响dAb的可溶性表达量且该影响不受分布位置的影响。蛋白质的极性亦是其可溶性表达水平的显著影响因素。
dAb;氨基酸组成;可溶性表达;聚类分析;线性建模
自从20世纪70年代现代生物技术的起步,大肠杆菌(Escherichia coli)因其培养成本低、生长速度快、易于实现生物反应器内高密度细胞培养、易于工程改造等优点[1],成为表达外源基因重组蛋白质中最广泛使用的宿主细胞[2]。在外源蛋白质重组表达研究中,得到一定量的蛋白质纯品是首要解决的问题[3]。然而大部分真核来源的蛋白质在大肠杆菌中的可溶性表达水平很低[4],例如,超过90%具有药用价值的蛋白质因在大肠杆菌中的表达水平过低而使其应用停留在了临床开发的初级阶段[5]。
提高外源蛋白质可溶性表达水平的方法有很多,包括使用弱启动子、降低诱导温度[6]、优化培养基[7]、与分子伴侣共表达[8-9]以及融合表达[10]等。但是由于表达系统的组件与蛋白质本身的关系暂未研究清楚,我们往往只能通过“试错法”不断尝试各种组合,耗时较长并且不易得到很好的效果[11]。
自20世纪90年代以来,生物信息学的学者发现蛋白质的一级结构对于该蛋白质在大肠杆菌中的过表达有重大影响[12-13],并且发表了一系列利用数据库中的信息结合数学分析方法、基于蛋白质的一级结构来预测蛋白质的表达水平的文章,成功构建了一些准确率较高的预测模型。例如Wilkinson-Harrison预测模型[14],多重线性拟合模型[15],基于SI的 模型[16],基于 SVM 的 模 型[17-18],PROSO 模 型[19],SOLpro模型[20]以及PROSOII模型[21]等。这些预测模型可以显著减少表达系统优化过程中提高外源蛋白质可溶性表达水平的“试错法”的步骤,然而由于上述研究中采用的数据均来自不同研究机构,数据的均一性及有效性差,在具体实施过程中的预测准确率低,截至目前应用并不广泛[18]。因此,选择合适的分子模型并保证所得数据的有效性是构建预测模型过程中需要注意的两大重要因素。
单域抗体(domain antibody,dAb)分子作为抗原抗体结合的最小单位,其相对分子质量小,无糖基化修饰,易于在原核体系中高效表达[22];保守区氨基酸比例高,可变区内某些氨基酸的变化不影响其与抗原的结合能力,因此是研究蛋白质一级结构与其表达水平关联的理想素材。表达系统中影响蛋白质可溶性表达水平的因素有很多[23],且各个影响因素之间存在不同程度的交互影响,是一个典型的多变元生物过程。因此作者以dAb作为分子模型,采用逐步线性回归分析的方法分析各个因素的影响程度,找出关键影响因子,从而指导蛋白质分子的定向改造以提高其可溶性表达水平,并在分子生物学层面上就其机理研究提供理论指导方向。
1 材料与方法
1.1 材料
1.1.1 宿主菌和质粒 大肠杆菌BL21(DE3):购自北京全式金生物技术有限公司;质粒pBW:作者所在实验室保存。
1.1.2 主要试剂和药品 分子生物学相关试剂:ThermoFisher公司;胰蛋白胨,酵母提取物:OXOID公司;异丙基硫代半乳糖苷(IPTG):Sigma-Aldrich(中国)公司;HRP-Protein A:武汉博士德生物工程有限公司;四环素(tetracycline),蛋白质定量试剂盒:生工生物工程(上海)股份有限公司;ELISA相关试剂:郑州博赛生物技术股份有限公司;其他药品均为国产分析纯。
1.1.3 培养基配制 LB培养基(g/L):酵母提取物5,蛋白胨 10,NaCl 10;pH 7.0,加入 1.5 g/L 琼脂糖即为固体培养基;TB/SB摇瓶培养基(g/L):酵母提取 物 24, 蛋 白 胨 12,KH2PO42.31,K2HPO4·3H2O 12.55,甘油 10;pH 7.0。
1.1.4 主要耗材和仪器 PCR仪:A500,杭州朗基科学仪器有限公司;酶标仪:EZ Read 800,英国Biochrom公司;洗板机:ST-96W,南京科华生物技术有限公司;冷冻离心机:ST16R,美国ThermoFisher公司;高通量细胞破碎仪:Precellys 24,法国Bertin公司;深孔板:48孔,常州英德生物科技有限公司。
1.2 方法
1.2.1 菌株构建
1)C端添加单个氨基酸重组菌株的构建:所用引物委托苏州金维智生物科技有限公司合成,序列见表1。目的片段经PCR扩增并用NdeI/XhoI双酶切后,连接到表达载体pBW上,再导入表达宿主E.coli BL21(DE3)。从转化平板上挑选单克隆进行测序验证,验证正确的菌株保存为甘油菌。

表1 dAb C端添加单个氨基酸所用引物序列Table 1 Primer sequences used in adding an amino acid to the C-terminal of dAb
2)CDR区随机突变菌株的构建:在dAb的三个互补决定区 (complementarity-determining region,CDR,亦称为超级可变区)各随机挑选5个氨基酸(总计15个氨基酸)进行随机突变,突变时排除稀有密码子突变,最终得到含约107个突变序列的文库,从中随机挑选65个菌株作为本实验的随机突变株。
1.2.2 平行发酵方法 取无菌48孔深孔板,加入1 mL含15 μg/mL四环素的LB培养基。从-80℃冰箱中取出各株菌的甘油菌,各吸取10 μL加入相应的培养孔中,置于30℃摇床,230 r/min培养16 h,此为种子液。取10 μL种子液转接至含15 μg/mL四环素的1 mL TB/SB中,于30℃培养7 h后,加入IPTG至终浓度为0.1 mmol/L,于23℃培养。16 h后将菌液转移至1.5 mL离心管,10 000 g离心 10 min,分离上清液及菌体沉淀并保存。
1.2.3 菌体处理方法 上述菌体重悬于1.0 mL PBS中,全部转移至装有0.5 cm3玻璃珠的破碎管,通过Precellys 24高通量细胞破碎仪进行破碎。破碎程序为:6 000 r/min连续振荡20 s后,间隔2 min,重复此操作4次。振荡完成后,10 000 g离心10 min,分离上清液留存待测。
1.2.4 直接ELISA法检测目的产物质量分数 以作者所在实验室保存的scFv纯品为标准品,按一定比例稀释至不同质量浓度后,各取50 μL加入酶标板(Corning,USA)。另将样品稀释至合适的倍数后,以PBS为空白对照,TB/SB为阴性对照 (样品为菌体裂解液时,以对照菌体裂解液为阴性对照),各取50 μL加入酶标板,37℃温育1 h后用洗板机洗涤五遍,每孔加入300 μL 5%脱脂牛奶,温育及洗涤同上。每孔加入 50 μL稀释 1 500倍的 HRPProtein A,温育及洗涤同上。加入100 μL显色液,37℃避光 15 min后,每孔加入 100 μL 2 mol/L H2SO4终止反应,用酶标仪读取A450/620。以标准品质量浓度为横坐标,以相应读数为纵坐标作出标准曲线后,代入稀释样品读数计算质量分数。
前期实验中所有菌株均未检测到明显包涵体,因此本研究主要关注dAb的可溶性表达量。本研究中发酵上清液中dAb质量分数为胞外可溶性蛋白质量分数,菌体破碎后上清液中dAb质量分数为胞内可溶性蛋白质量分数,两者之和为dAb的可溶性表达量。
1.2.5 Bradford法测定蛋白质总量 参照试剂盒说明书制作BSA蛋白质标准曲线,测定各样品的A595值后代入标准曲线计算蛋白质质量分数,用于计算每微克总蛋白质中dAb的纳克数。
1.2.6 数据分析 探索在dAb的C端添加一个氨基酸对其可溶性表达量影响的研究,采用t检验分析表达量变化的显著性(以P<0.05为显著)。
我们研究蛋白质两方面性质对其可溶性表达量的影响:1)每种氨基酸的含量;2)蛋白质带电性、极性、憎水性、酸性、碱性。按照Vector NTI软件的分类,带电氨基酸包括 R、K、D、C、H(组氨酸)、Y(酪氨酸)及E。酸性氨基酸包括D和E(谷氨酸)。碱性氨基酸包括 K 和 R。 极性氨基酸为 N、T、C、G、Q、S和 Y。 疏水氨基酸 A、V、L(亮氨酸)、I(异亮氨酸)、F和W。我们采用R package从以下两个层面进行分析(显著性以P<0.05作为筛选标准)。第一个层面:采用层次聚类法将蛋白质表达数据分成高表达和低表达两类,然后在这两类数据之间进行t检验,方差分析以找出显著影响蛋白质可溶性表达量的影响因素。第二个层面:分别建立线性模型,然后从全模型开始逐一减掉一个影响因素,最终根据AIC值(越小越好)选择最佳的影响因素组合。
另外,通过 Cluster Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/),根据氨基酸序列信息对这66株菌进行了分类,并将该结果与蛋白质可溶性表达分类结果进行比对分析,计算其一致性比例。该比例越高代表蛋白质可溶性表达与其氨基酸组成的关联越紧密。
2 结果与分析
2.1 dAb C端添加单个氨基酸重组菌株的构建
有文献表明,少量氨基酸的改变便足以影响蛋白质的可溶性表达水平发生显著变化[5]。为探索单个氨基酸对本研究所用dAb可溶性表达水平的影响,我们设计进行此部分实验。因dAb N端的酶切位点NdeI经酶切后具有ATG起始位点,因此我们在dAb的C端进行改造。采用表1中的特异性引物,以原始dAb分子为模板,经PCR扩增出C端增加一个氨基酸约438 bp的条带,见图1。经NdeI/XhoI双酶切连接至pBW载体并导入BL21(DE3)宿主后,单克隆测序正确。

图1 dAb C端添加一个氨基酸的PCR扩增结果Fig.1 PCR amplifications of dAb gene after adding an amino acid at the end of C-terminal
2.2 单个氨基酸的变化对dAb可溶性表达水平具有规律性影响
由于本研究中随机突变的dAb分子是在原始dAb的CDR进行改造,造成各突变分子对互补抗原的结合能力不同,因此常用的用抗原包被酶标板捕获抗体分子的间接ELISA在本研究中并不适用。针对于本研究的样品,我们采用直接ELISA法,用包含dAb分子的样品直接包被酶标板,而蛋白质与酶标板之间的吸附性无特异性,因此消除了因与包被抗原结合能力不同而造成的信号差异。为保持测定方法一致,采用直接ELISA测定dAb可溶性表达量。
将直接ELISA的A450/620读数带入标准曲线公式计算,得出样品中的dAb质量分数,并以各样品的总蛋白质量为标准进行均一化,所得数据见图2。
由图2可知,添加一个氨基酸便足以引起dAb可溶性表达水平的显著变化。其中添加G、P(脯氨酸)、C及Q造成的变化最为显著,其表达量分别为4.53±0.70、4.12±0.32、3.81±0.32、3.78±0.22 ng/μg,均显著 (P<0.01)高于原始分子的表达量1.17±0.02 ng/μg;添加 L、F、M(甲硫氨酸)、W(色氨酸)、Y、I及H的dAb的可溶性表达量虽然提高的幅度不大,但其表达量的变化也具有统计学意义(P<0.02)。
2.3 氨基酸组成与dAb可溶性表达水平显著相关
由于ELISA法中所用的酶标抗原HRP-Protein A是与dAb的恒定区结合,为不影响两者之间的结合并使突变分子之间具有足够大的差异,我们选择在dAb分子的三个CDR进行随机突变。
2.3.1 聚类分析证明氨基酸组成与可溶性表达水平具有很大关联 层次聚类分析显示dAb株菌的高水平表达与低水平表达显著不同,根据氨基酸序列信息对66株菌进行分类,同样得到具有显著差异的两个组。对比这两组分类结果发现,分类一致的菌株有46株,一致性可达70%,说明氨基酸组成与蛋白质的可溶性表达水平间确实存在着关联,见表2。
2.3.2 线性建模揭示显著影响dAb可溶性表达水平的氨基酸组成 为研究20种氨基酸的组成对dAb可溶性表达水平的影响,我们以每种氨基酸的数目为变量进行逐步回归分析,结果见表3。
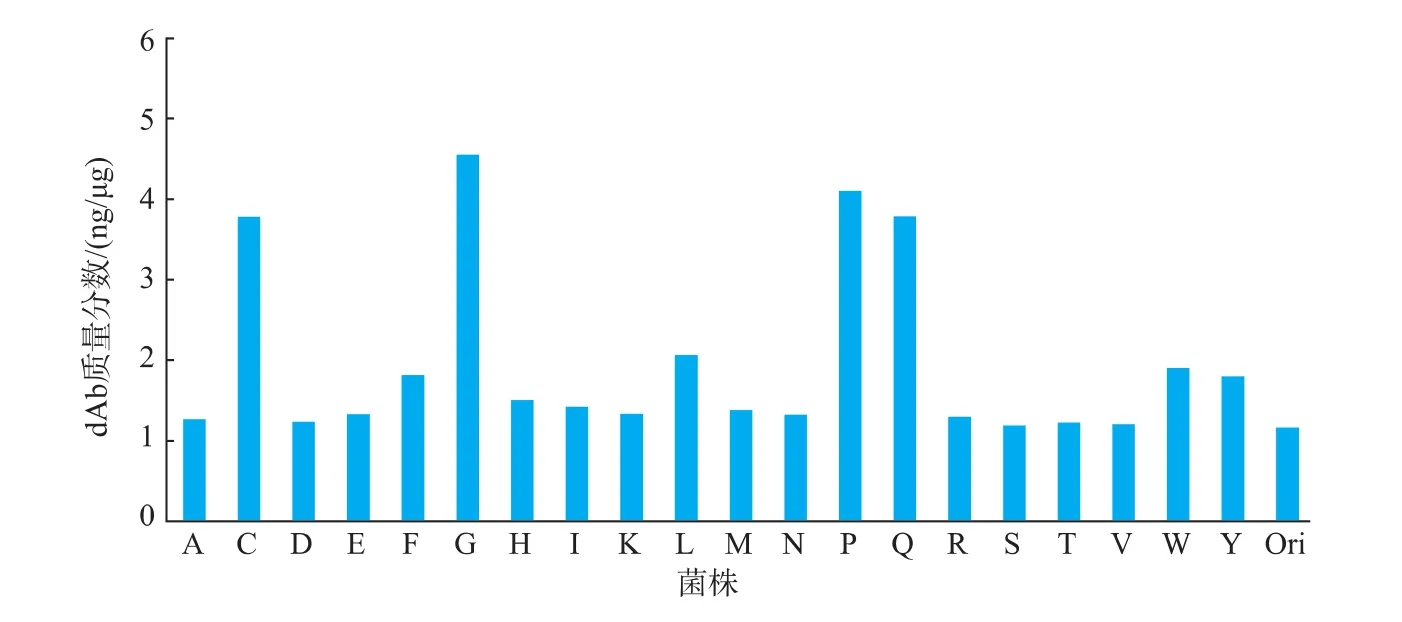
图2 dAb C端添加不同氨基酸后的可溶性表达量Fig.2 Soluble expression amount of dAb after adding different amino acid to the C-terminal
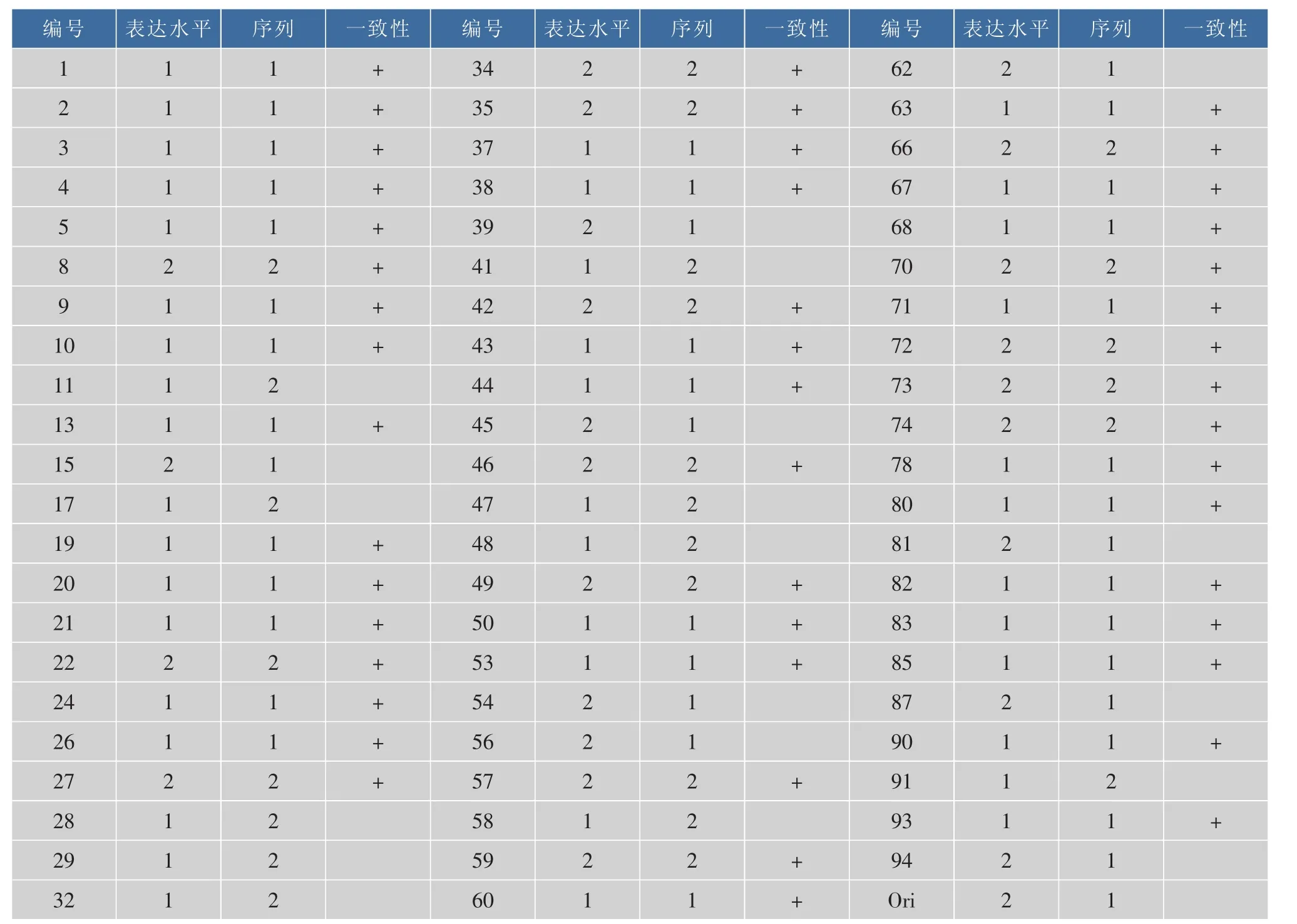
表2 氨基酸序列与可溶性表达水平分类结果Table 2 Clustering results based on amino acid sequences and soluble expression amounts
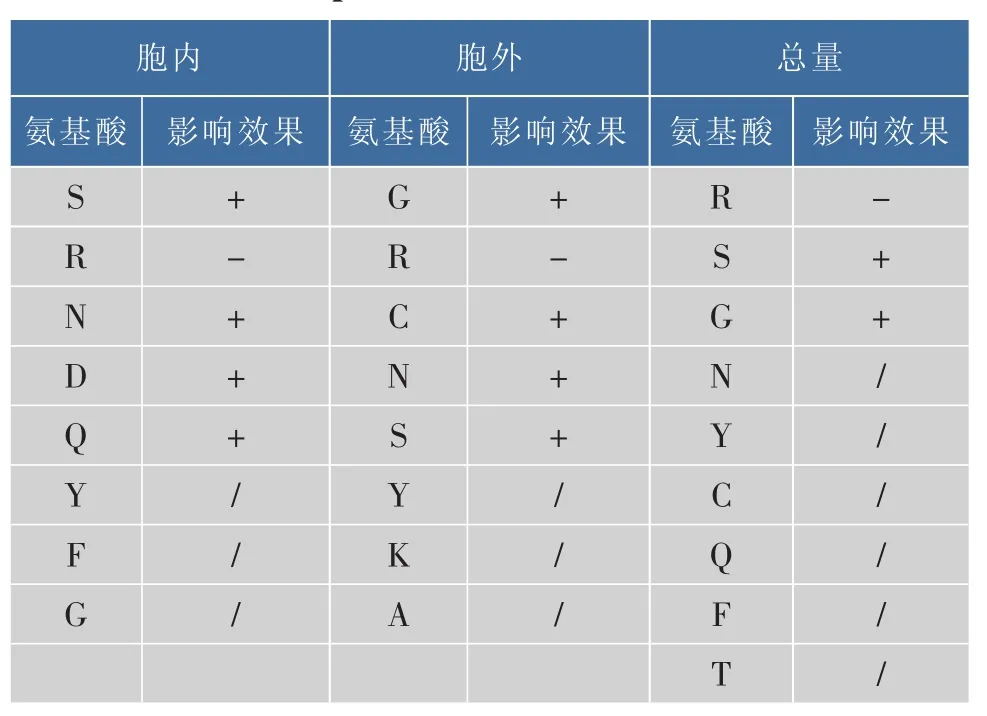
表3 显著影响dAb可溶性表达量的氨基酸组合及其影响效果Table 3 Amino acids that have significantly affection on soluble expression levels of dAbs
我们发现,显著影响dAb可溶性胞外质量分数的变量组合为 S、R、N、D、Q、Y、F 和 G 的含量 (p=0.001 683),其中单个氨基酸影响显著的有S(正相关,p=0.000 623)、R(负相关,p=0.001 229)、N(正相关,p=0.018 052)、D(正相关,p=0.030 956)和 Q(正相关,p=0.046 071);显著影响dAb胞内质量分数的变量组合为 G、R、C、N、S、Y、K 和 A 的含量(p=0.001 851),其中单个氨基酸影响显著的有G(正相关,p=0.012 2)、R(负相关,p=0.015 1)、C(正相关,p=0.019 5)、N(正相关,p=0.029 6)和 S(正相关,p=0.034 9);显著影响dAb可溶性表达水平的变量为R、S、G、N、Y、C、Q、F 和 T 的含量(p=0.000 718 6),其中单个氨基酸影响显著的有 R(负相关,p=0.000 793)、S(正相关,p=0.006 407)和 G(正相关,p=0.027 663)。 我们对影响胞外、胞内和蛋白质总量的氨基酸进行了比较,发现R、S、G、N和Y的含量对蛋白质可溶性表达的影响是不受检测位置变化所影响的,见图3,其中R、S和G具有显著影响(p=0.000 806)。
在对蛋白质带电性、极性、疏水性、酸性、碱性的分析中我们发现,极性显著影响dAb的可溶性表达水平(p=0.021 42)。
3 结语
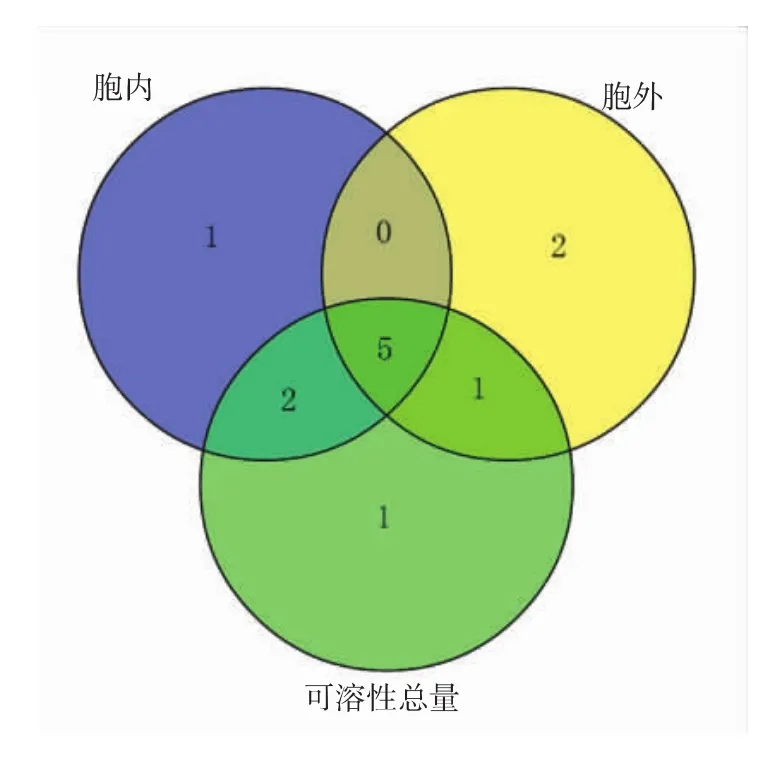
图3 影响dAb胞内、胞外及可溶性表达量的因素的文氏图Fig.3 Venny diagram of factors affecting dAb soluble expression
蛋白质氨基酸组成与其可溶性表达水平的关联研究有很多,但是各研究的结论并不太一致。例如Science上发表的一篇文章通过研究81种不同的蛋白质,提出提高平均电荷量、降低形成转角的氨基酸数目及C的数目等可减少包涵体的形成[14];也有针对G耦联受体蛋白质的研究指出,提高正电荷含量可提高包涵体表达量[15]。再如一项从TargetDB数据库中挑选27 267个蛋白质并研究其性质与高通量分析限速步骤的研究得出,蛋白质疏水性越强越不利于蛋白质的表达[24];而另外一项探索C.elegans基因组10 167个ORF表达情况的研究发现,蛋白质疏水性强并不一定代表蛋白质的表达量低,但是疏水性弱的蛋白质,其可溶性表达水平较高[25]。以上可归因于各研究采用的研究对象、数据来源及分析方法等的不一致,而本研究通过构建dAb随机突变库,在固定的大肠杆菌表达体系中进行高通量平行发酵与检测将有效避免这些问题。
通过对66个dAb分子氨基酸组成和可溶性表达量的分析发现,极性与蛋白质的可溶性表达量呈显著正相关, 即 N、S、C、G、T、Q 和 Y 数目的总和与蛋白质可溶性表达量呈正相关,可能是由于蛋白质折叠后极性氨基酸更容易暴露于溶剂中[26],增加了蛋白质与溶剂之间的相互作用,提高了可溶性,从而间接提高了蛋白质的可溶性表达。
我们发现R的数目与蛋白质可溶性表达量显著负相关,与翻译过程中正电荷氨基酸可降低大肠杆菌蛋白质翻译速率的报道一致[12]。在dAb分子C端添加G与对该分子进行随机突变的结果均显示,G的数目与dAb可溶性表达量呈显著正相关,我们认为这可能是由于G相对分子质量小且具有极性。极性分子S对dAb的可溶性表达量的显著正相关,与我们发现极性显著影响蛋白质可溶性表达的结论相吻合。在7个极性氨基酸分子当中,C含量的增加易于在蛋白质内部形成二硫键,而二硫键在大肠杆菌的周质空间难以正确形成,从而提高了蛋白质不正确折叠的风险,理论上不利于蛋白质可溶性表达;T、Q和Y各项物理、化学性质比较稳定,加上G、S对可溶性表达的正向作用以及R的负影响,因此,在通过定向改造氨基酸而提高蛋白质表达水平方面,我们建议尝试提高G、S、T、Q和Y中个别氨基酸或氨基酸组合的含量,并减少R的含量。
本研究实现了根据蛋白质的氨基酸组成,指导提高蛋白质可溶性表达水平的定向改造,可大大提高工作效率,降低获得一定量蛋白质纯品的难度,从而推进蛋白质的结构研究以及实际应用。
[1]MAKINO T,SKRETAS G,GEORGIOU G:Strain engineering for improved expression of recombinant proteins in bacteria[J].Microbial Cell Factories,2011,10:32.
[2]GRASLUND S,NORDLUND P,WEIGELT J,et al:Protein production and purification[J].Nature Methods,2008,5(2):135-146.
[3]PETI W,PAGE R.Strategies to maximize heterologous protein expression in Escherichia coli with minimal cost[J].Protein Expression and Purification,2007,51(1):1-10.
[4]PELEG Y,UNGER T.Resolving bottlenecks for recombinant protein expression in E.coli[J].Methods in Molecular Biology,2012,800:173-186.
[5]TREVINO S R,SCHOLTZ J M,PACE C N.Amino acid contribution to protein solubility:Asp,Glu,and Ser contribute more favorably than the other hydrophilic amino acids in RNase Sa[J].Journal of Molecular Biology,2007,366(2):449-460.
[6]MAKRIDES S C.Strategies for archieving high-level expression of genes in Escherichia coli[J].Microbiology Review,1996,60(3):512-538.
[7]STUDIER F W.Protein production by auto-induction in high-density shaking cultures[J].Protein Expression and Purification,2005,41(1):207-234.
[8]SONODA H,KUMADA Y,KATSUDA T,et al.Effects of cytoplasmic and periplasmic chaperones on secretory production of single-chain Fv antibody in Escherichia coli[J].Journal of Bioscience and Bioengineering,2011,111(4):465-470.
[9]ENTZMINGER K C,CHANG C,MYHRE RO,et al.The Skp chaperone helps fold soluble proteins in vitro by inhibiting aggregation[J].Biochemistry,2012,51(24):4822-4834.
[10]SUN W,XIE J,LIN H,et al.A combined strategy improves the solubility of aggregation-prone single-chain variable fragment antibodies[J].Protein Expression and Purification,2012,83(1):21-29.
[11]CHANG C C,SONG J,TEY B T,et al.Bioinformatics approaches for improved recombinant protein production in Escherichia coli:protein solubility prediction[J].Briefings in Bioinformatics,2014,15(6):953-962.
[12]PRICE W N,HANDELMAN S K,EVERETT J K,et al.Large-scale experimental studies show unexpected amino acid effects on protein expression and solubility in vivo in E.coli[J].Microbial Informatics and Experimentation,2011,1(1):6.
[13]TIANY,DEUTSCHC,KRISHNAMOORTHYB.Scoringfunctiontopredictsolubilitymutagenesis[J].Algorithms for Molecular Biology,2010,5:33.
[14]WILKINSON D L,HARRISON R G.Predicting the solubility of recombinant proteins in Escherichia coli[J].Biotechnology(N Y),1991,9(5):443-448.
[15]KIEFER H,VOGEL R,MAIER K.Bacterial expression of G-protein-coupled receptors:prediction of expression levels from sequence[J].Receptors and Channels,2000,7(2):109-119.
[16]IDICULA T S,BALAJI P V.Understanding the relationship between the primary structure of proteins and its propensity to be soluble on overexpression in Escherichia coli[J].Protein Science,2005,14(3):582-592.
[17]IDICULA T S,KULKARNI A J,KULKARNI B D,et al.A support vector machine-based method for predicting the propensity of a protein to be soluble or to form inclusion body on overexpression in Escherichia coli[J].Bioinformatics,2006,22(3):278-284.
[18]CHAN W C,LIANG P H,SHIH Y P,et al.Learning to predict expression efficacy of vectors in recombinant protein production[J].BMC Bioinformatics,2010,11 Suppl 1:S21.
[19]SMIALOWSKI P,MARTIN G A J,MIKOLAJKA A,et al.Protein solubility:sequence based prediction and experimental verification[J].Bioinformatics,2007,23(19):2536-2542.
[20]MAGNAN C N,RANDALL A,BALDI P.SOLpro:accurate sequence-based prediction of protein solubility[J].Bioinformatics,2009,25(17):2200-2207.
[21]SMIALOWSKI P,DOOSE G,TORKLER P,et al.PROSO II--a new method for protein solubility prediction[J].FEBS Journal,2012,279(12):2192-2200.
[22]HOLT L J,HERRING C,JESPERS L S,et al.Domain antibodies:proteins for therapy[J].Trends in Biotechnology,2003,21(11):484-490.
[23]GUSTAFSSON C,MINSHULL J,GOVINDARAJAN S,et al.Engineering genes for predictable protein expression[J].Protein Expression and Purification,2012,83(1):37-46.
[24]GOH C S,LAN N,DOUGLAS S M,et al.Mining the structural genomics pipeline:identification of protein properties that affect high-throughput experimental analysis[J].Journal of Molecular Biology,2004,336(1):115-130.
[25]LUAN C H,QIU S H,FINLEY J B,et al.High-throughput expression of C.elegans proteins[J].Genome Research,2004,14(1):2102-2110.
[26]LESSER G J,ROSE G D.Hydrophobicity of amino acid subgroups in proteins[J].Proteins,1990,8(1):6-13.
会议名称(中文):国际生物化学学会第六届年会
所属学科:化学生物学,生物技术与生物工程
开始日期:2017-10-17
结束日期:2017-10-20
所在城市:上海市 黄浦区
具体地点:华东理工大学
主办单位:华东理工大学、生物反应器工程国家重点实验室
E-MAIL:icbs2017@chemical-biology.org
会议网站:http://icbs2017.ecust.edu.cn/
会议背景介绍:ICBS2017年会旨在全球范围内召集生物化学领域的各前沿领导者、技术推动者、领域新星、学员以及做出贡献的研究者们,分享激动人心的研究突破和最新技术、讨论目前存在的挑战及未来发展方向、以期提高生物化学的社会影响力。本次会议将提供面对面的交流互动、技术探讨,以促进商业联系和未来的合作;邀请具有重要贡献的学科建设者、青年科学家和技术先驱者代表,并为其提供一个交互平台;且将从所有提交的演讲摘要中选择部分进行报告交流。
为促进生物化学研究者的职业发展,我们将遵照历届ICBS的传统,在会议上选出“国际生物化学学会青年生物化学学者”的获奖者。此外,ICBS2017的预会议部分将由年轻的研究人员和学生共同组织,预会议将动员下一代年轻科学者,提出创新的方法来迎接科学的重大挑战。
Correlation Between Protein Primary Sequence Structure and Soluble Expression Level of dAbs in Escherichia coli
GUO Wenwen1,2,3, LIU Meng1,2,3, BAI Zhonghu1,2,3, YANG Yankun*1,2,3, DAI Xiaofeng1,2,3
(1.National Engineering Laboratory for Cereal Fermentation Technology,Jiangnan University,Wuxi 214122,China;2.The Key Laboratory of Industrial Biotechnology,Ministry of Education,School of Biotechnology,Jiangnan University,Wuxi 214122,China;3.The Key Laboratory of Carbohydrate Chemistry and Biotechnology,Ministry of Education,School of Biotechnology,Jiangnan University,Wuxi 214122,China)
Lots of antibody drug have been successfully expressed in the prokaryotic system.Lacking knowledge on the process mechanism of recombinant protein expression has forcedresearchers to use"trial and error"to improve soluble production,which is time and labor consuming with low success rate.Adding different amino acids to the C-terminal of a dAb often results in significant variation of soluble expression levels.The amino acid sequence of dAbs highly concords with their soluble expression levels,with a consistency being 70%.Through analyzing the soluble expression and sequence data of 65 dAbs using clustering and linear modeling,we show that certain amino acids panel could significantly affect the soluble expression of dAbs,with the specific amino acids composition in these panels being(S,R,N,D,Q),(G,R,C,N,S) and(R,S,G),respectively,in the supernatant,pellet and total amount.In addition,polar was found a vital factor affecting the soluble production of dAb.These results may be helpful in orthomutation.
dAb,amino acid panel,soluble expression,clustering analysis,linear modeling
Q 815
A
1673—1689(2017)09—0951—08
2015-03-27
国家863计划项目(2015AA020802);国家973计划项目(2013CB733602);中央高校基本科研业务费专项资金项目(JUSRP51401A)。
*通信作者:杨艳坤(1978—),男,河北石家庄人,理学博士,副教授,硕士研究生导师,主要从事微生物学、基因工程、分子遗传、蛋白质的分离纯化及特性等方面的研究。E-mail:yangyankun@jiangnan.edu.cn
郭雯雯,刘萌,白仲虎,等.大肠杆菌中dAb一级结构与可溶性表达关联[J].食品与生物技术学报,2017,36(09):951-958.
