钙离子调控微丝切割蛋白中A6亚基解折叠的单分子力谱研究∗
2017-10-23李鹏飞曹毅秦猛王炜
李鹏飞 曹毅 秦猛 王炜
(南京大学物理学院,固体微结构国家实验室,南京 201193)
钙离子调控微丝切割蛋白中A6亚基解折叠的单分子力谱研究∗
李鹏飞 曹毅 秦猛†王炜
(南京大学物理学院,固体微结构国家实验室,南京 201193)
在生命活动中,金属离子扮演了非常重要的角色.微丝切割蛋白(adseverin)需要钙离子的活化才能行使其切割肌动蛋白微丝的功能.本文通过基于原子力显微镜的单分子力谱研究了微丝切割蛋白C端末的A6亚基在结合钙离子前后的力学解折叠机理.实验结果显示:在未结合钙离子时,A6的解折叠表现为两态过程;在结合钙离子后A6力学稳定性显著提高;同时,钙离子的结合使得A6解折叠过程中出现稳定的中间态.通过对中间态的链长的分析,我们推测了中间态对应着A6的N端部分解折叠.而这一部分的解折叠可以使得掩藏在该结构后的A5亚基中肌动蛋白微丝结合位点暴露,从而促使微丝切割蛋白执行功能.我们的实验结果为理解微丝切割蛋白的工作原理提供了新的实验证据.
微丝切割蛋白,单分子力谱,解折叠,中间态
1 引 言
金属离子在生命活动中扮演这非常重要的角色,例如钾离子使神经纤维产生内外电位差并维持其稳定,使得神经电信号实现传导,镁离子能够激活多种酶的活性,钠离子能够维持生物体的内环境的稳定.金属离子与蛋白质的结合是很多蛋白质完成其生物功能的重要保证.铁离子与血红蛋白的结合使得氧运输得以实现,铜离子与铜伴侣蛋白的结合使生物体能够正常利用铜离子,促进铁的吸收与利用[1].在生物体中,钙离子同样执行着非常重要的功能.钙离子是骨骼构成的重要物质,同时,钙离子对于维持肌肉正常的收缩-舒张也有至关重要的意义.钙离子与凝溶胶蛋白的结合使其结构发生变化,暴露其肌动蛋白的结合位点从而被激活行使生物功能[2−4].同时,钙离子与蛋白质的结合也可能使蛋白质本身的力学性质发生变化.有研究表明,一些蛋白质与钙离子结合后,其本身的力学稳定性得到增强[5].本文利用基于原子力显微镜的单分子力谱方法,研究了受钙离子调控的微丝切割蛋白的C端末端结构域(A6)的受力解折叠性质随钙离子结合的变化.
原子力显微镜单分子力谱技术是新兴的能够很好地研究分子本身力学性质以及分子间相互作用强度的手段[6−8],在高分子材料[9−11]、细胞生物学[12]、力化学等[13,14]方面也有广阔的应用前景.其在生物大分子的研究方面,例如蛋白质的解折叠研究[15,16]、DNA和RNA性质研究[17−19]、生物分子间的相互作用研究[20,21]都有着很好的应用.通过单分子力谱,我们可以直接获得所研究体系的力分布,同时,通过对所得力分布进行分析,根据Kramers方程,可以得到体系的寿命,解离常数(K0)以及转变态长度(∆x),进而通过Bell模型,得到体系的自由能分布[22−24].
微丝切割蛋白通过本身存在的肌动蛋白结合位点与微丝结合,执行对微丝切割、加帽活性以及成核的功能[25,26].在未结合钙离子时,其结构无法与肌动蛋白契合,因此不能与微丝结合.结合钙离子后,其结构发生变化,A4与A6两个亚基能够形成一个与肌动蛋白结构符合的面,进而执行其功能(图1)[4].在其功能执行过程中,会受到微丝的牵引而受力,因此其力学稳定性对于其功能的实现具有重要的意义.微丝切割蛋白与肌动蛋白结合后,位于A5上的纤维形肌动蛋白结合及成核位点是被包埋住的[27],我们发现A6蛋白质在结合钙离子后,其解折叠过程可能会出现稳定的中间态,从而在不影响A6与肌动蛋白结合的情况下,暴露出位于A5上的结合位点.

图1 (网刊彩色)微丝切割蛋白中结合钙离子后A4—A6结合域的三维结构图(PDB ID:3FG6) 图中黑色球体代表钙离子;绿色、橙色以及红色结构分别对应A4—A6;紫红色部分为位于A5上的肌动蛋白结合及成核位点;紫色部分对应A6蛋白质解折叠的中间态打开部分;棕色位置为A4—A6的N端(甘氨酸396);蓝色位置为其C端(色氨酸715)Fig.1.(color online)3D-structure of Ca-bound A4–A6 domain of adseverin(PDBID:3FG6). Ca2+ions are shown as black spheres;the amaranthine part in A5(orange)depicts the actin binding and nucleating module and the purple part corresponds to the unfolding segment in intermediate state of A6(red);the N-terminus of A4–A6 is shown in brown;while the C-terminus is shown in blue.
2 实验方法
2.1 蛋白质载体构建及表达
A6与GB1融合蛋白的基因通过基因工程的方法构建.A6基因和GB1基因分别来自于Robert Robinson教授和Hongbin Li的实验室.多聚蛋白基因通过限制性核酸内切酶BamHI及BglII产生的相同黏性末端连接来实现,在pUC19载体里完成构建.最终多聚蛋白基因通过测序确认,并转入pQE80L载体,通过大肠杆菌BL-21菌株进行表达.由于表达载体上目的基因前端有6个组氨酸编码,因此所得目的蛋白质N端会有六个组氨酸标记,能够特异性地与钴离子结合,纯化过程使用钴离子亲和树脂完成.之后,纯化所得蛋白质溶液加入1 mmol EGTA去除钙离子和其他金属离子后在Tris缓冲液(25 mmol Tris,72 mmol氯化钠,pH=7.4)中进行透析,4°C保存.
2.2 单分子力谱实验
实验通过原子力显微镜(ForceRobot 300,JPK,柏林,德国)实现,在Tris缓冲液中进行,结合钙离子的A6解折叠实验在缓冲液中加入1 mmol氯化钙.实验时,将150µL蛋白质溶液滴加到玻璃基板上,吸附20 min后使用Tris缓冲液冲洗掉多余的蛋白质,之后加入缓冲液即可进行实验.实验所用探针(Biolever-RC-150VB-70,Olympus)的劲度系数通过能量均分定理确定,其特征值为7 pN/nm.实验所用拉伸速度为400 nm/s.
3 实验结果与数据分析
我们构建了(GB1-A6)4(图2(a))多聚蛋白进行单分子力谱.GB1与A6交叠连接,可以防止A6蛋白之间相互影响[28].蛋白质通过物理吸附的方式吸附在玻璃基板上,实验时,原子力显微镜探针下压到基板上时,有与蛋白质结合的概率,这样抬起探针的过程中,蛋白质被拉伸,通过探测探针受蛋白质牵引的偏转即可得到蛋白质解折叠过程的力响应.我们分别在无钙离子以及含有1 mmol的溶液中对(GB1-A6)4进行了单分子力谱实验.基于原子力显微镜的单分子力谱所得多聚蛋白质解折叠图像为锯齿状峰,其形成原因为蛋白质本身是有结构的氨基酸残基多聚分子,当受力解折叠后,转变为被拉直的多肽链,释放一段长度,其中GB1解折叠事件为间隔18 nm的峰,峰高约为150 pN.我们使用蠕虫链模型拟合蛋白质分子链受力与拉伸距离的关系.在同一条力谱曲线上,如果观察到GB1的特征峰,那么由于A6也在同一个多聚蛋白上,应观察到其解折叠事件理应被观察到.因此,在力谱图中,GB1解折叠事件之前,间隔35 nm的峰即代表A6的解折叠.由于A6蛋白质拥有101个氨基酸残基(微丝切割蛋白中614—715残基),每个氨基酸残基延伸长度为0.365—0.4 nm,A6折叠状态N端到C端长度为3.5 nm,因此完全打开A6蛋白应该获得33.4—36.9 nm的长度延伸,这与所得实验结果是符合的.统计A6的解折叠事件,即可得到其解折叠力分布直方图.结果显示,在A6没有结合钙离子的情况下,其解折叠力平均值为25.4 pN±6.0 pN(平均值±标准差,n=99),在A6结合钙离子的情况下,其解折叠力上升至44.7 pN±6.8 pN(n=164).这说明钙离子的结合提高了A6蛋白质的力学稳定性.值得注意的是,探针吸附多聚蛋白的位置是随机的,这使得所得数据并非一定包含所有结构域的解折叠事件[29].
在对A6解折叠事件进行分析的过程中,我们发现A6蛋白质在结合钙离子情况下解折叠有时会存在中间态(图3(a)).这意味着在结合钙离子的情况下,A6蛋白质受力拉伸时,有分步完成解折叠的概率.该中间态的长度均为7.2 nm.同时对比中间态出现后的残余A6部分的力学强度,我们认为解折叠中间态出现后,钙离子并未从A6上脱落.根据单个氨基酸的长度0.365 nm估算,该中间态对应着长度约20个氨基酸的结构被打开,且剩余结构仍然有较高的力学强度.钙离子通过644位的天冬氨酸以及666位的谷氨酸与A6结合.根据实验结果分析其结构,我们发现只有从A6的N端(614位脯胺酸)到643位天冬氨酸打开符合所有条件(图3(e)).同时对比A4—A6部分与肌动蛋白结合的情况,正是该部分结构对A5上的肌动蛋白结合位点的覆盖使其被隔离,因此我们猜想当微丝切割蛋白与微丝结合,微丝切割蛋白受微丝牵引导致部分解折叠,暴露A5上的结合位点,从而使其激活了肌动蛋白成核功能.
对所得力分布使用Dudko-Hummer-Szabo方程,可以得到对应力条件下体系的寿命τ:


图2 (网刊彩色)(GB1-A6)4的单分子力谱实验结果 (a)(GB1-A6)4单分子力谱实验示意图,图中红色方块代表GB1,蓝色圆代表A6;(b)无钙离子结合状态下(GB1-A6)4的力谱图,图中蓝色曲线为A6解折叠事件的蠕虫链模型拟合曲线,红色曲线为GB1解折叠的拟合曲线;(c)无钙离子结合状态下的A6解折叠力分布直方图,蓝色曲线为该分布的高斯拟合;(d)在1 mmol钙离子缓冲液中(GB1-A6)4的力谱曲线图;(e)1 mmol钙离子缓冲液中A6解折叠力分布直方图Fig.2.(color online)Mechanical properties of A6 probed by single-molecule AFM:(a)A schematic of polyprotein(GB1-A6)4used for single-molecule force spectroscopy experiment,red squares and blue circles represent GB1 and A6 domains,respectively;(b)typical unfolding trace for Ca-free(GB1-A6)4,worm-like chain fi tting curves for A6 and GB1 are shown in blue and red,respectively;(c)unfolding force histogram of Ca-free A6;(d)typical unfolding trace for Ca-bound(1 mmol Ca2+)(GB1-A6)4;(e)unfolding force histogram for Ca-bound A6.
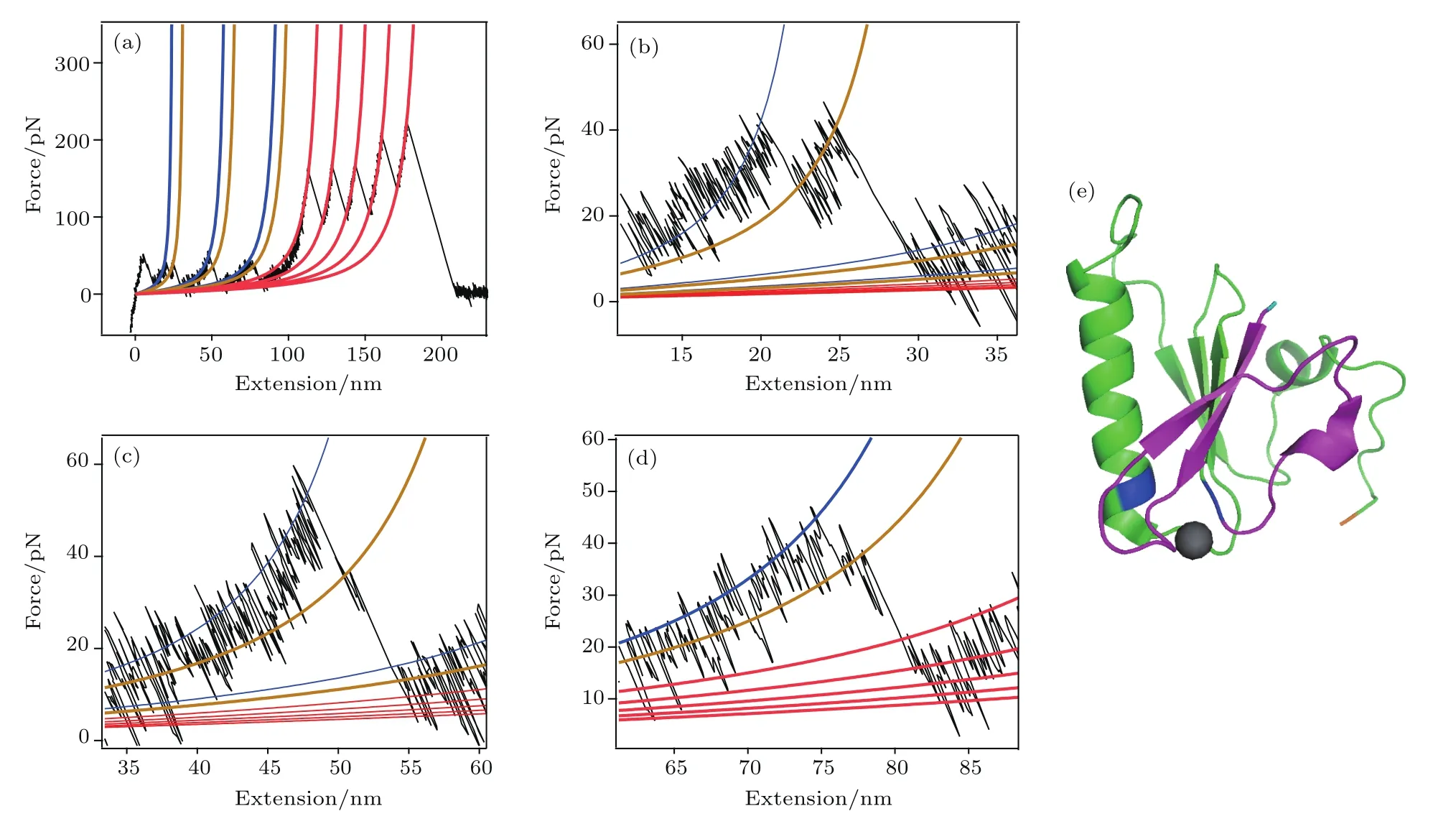
图3 (网刊彩色)结合钙离子情况下A6解折叠的中间态 (a)存在解折叠中间态的(GB1-A6)4单分子力谱曲线,蓝色及棕色曲线分别为A6蛋白质两步解折叠的蠕虫链模型拟合曲线,红色曲线为GB1解折叠的拟合曲线;(b)—(d)为(a)中A6解折叠力谱图的放大图;(e)A6的结构图,紫色部分为猜想的解折叠中间态打开部分(614—643位氨基酸),蓝色标注为A6上的钙离子结合位点,分别为644位天冬氨酸以及666位谷氨酸,浅蓝色位置为A6的N端(脯胺酸614),橙色位置为A6的C端(色氨酸725)Fig.3.(color online)The unfolding intermediate state of Ca-bound A6:(a)The unfolding trace for(GB1-A6)4with intermediate states appeared,blue and brown curves are the worm-like chain fi tting for two steps of A6 unfolding,respectively;(b)–(d)details of unfolding intermediate states of A6 in Fig.(a);(e)structure of A6,the proposed unfolded segment(residues 614–643)in intermediate state is shown in purple,and the Ca2+binding sites(Asp644 and Glu666)are shown in blue,N-terminus of A6 is shown in light blue(Pro614)and C-terminus is shown in orange(Trp715).
方程中F0为直方图中力的开始点,N为直方总数,∆F为每个直方的宽度,hk为第k个直方占总的比例,i与k为直方的编号,˙F为力的加载速率.
将所得体系的寿命-力关系(图3(a))代入Krammers方程并联立Bell模型进行拟合,即可得到体系在不受力情况下的寿命τ0以及转变态长度∆x:

kB为玻尔兹曼常数,T为绝对温度.
经拟合得到无钙离子结合的情况下,A6蛋白质在不受力的情况下寿命τ0为0.5 ms,解折叠转变态长度∆x1为0.82 nm,在结合钙离子的情况下,其寿命τ0提高至13.4 ms,解折叠转变态长度∆x不变.在有解折叠中间态的情况下,第一步解折叠的τ0及∆x不变,第二步解折叠的τ0为4.9 ms,∆x3为1.1 nm.
寿命τ0的倒数即为解离常数k0,由阿伦尼乌斯方程:

方程中∆G为反应的自由能势垒,A为表征自由能形面的指前因子,在本体系中,A取106s−1,经计算得无钙离子结合时,A6蛋白质自发解折叠的自由能势垒高度为6.2kBT,结合钙离子情况下,A6蛋白质自发解折叠的自由能势垒提高到9.5kBT,解折叠中间态第一步势垒与其相同,第二步势垒高度为8.5kBT.由计算结果可知,该中间态可相对稳定存在.由此可得到体系的自由能曲线,如图4(b)所示.

图4 (网刊彩色)A6蛋白质在有无钙离子结合情况下的寿命-力分布及自由能曲线 (a)蓝色三角形代表无钙离子结合情况下A6的寿命-力关系,红色圆形代表结合钙离子情况下的寿命-力分布,紫色方块代表A6解折叠中间态第二步的寿命-力关系,图中蓝色、红色以及紫色曲线分别为三种情况下的Krammers方程拟合;(b)图中蓝色曲线为无钙离子结合情况下的A6蛋白质自由能曲线,红色曲线则对应于结合钙离子情况下无解折叠中间态的自由能分布,紫色曲线对应于结合钙离子情况下有解折叠中间态的自由能分布Fig.4.(color online)The lifetime-force plot and unfolding free-energy landscape for A6 under Ca-free and Ca-bound conditions:(a)Blue,red and purple markers represent the lifetime-force relationships for Cafree,Ca-bound and second step for unfolding intermediate state of A6,and the corresponding colour curves represent the Krammers-analysis;(b)blue,red and purple curves represent the free-energy landscape for Ca-free,Ca-bound and condition of unfolding with intermediate state of A6.
4 结 论
本文通过单分子力谱结合蛋白质工程技术研究了微丝切割蛋白的A6亚基的力学解折叠.结果表明,A6蛋白在结合钙离子之前,其稳定性较差.但结合钙离子后,A6蛋白的力学强度及稳定性有明显的提高.这使得微丝切割蛋白能够稳定地执行其生物功能.同时A6蛋白质在结合钙离子情况下受力可进入稳定的中间态,此中间态可能导致A5上的结合位点暴露,使其功能被激活.具体的验证有待进一步的研究.
[1]Lee J,Pena M M,Nose Y,Thiele D J 2002J.Biol.Chem.277 4380
[2]Nag S,Larsson M,Robinson R C,Burtnick L D 2013Cytoskeleton70 360
[3]Silacci P,Mazzolai L,Gauci C,Stergiopulos N,Yin H L,Hayoz D 2004Cell Mol.Life Sci.61 2614
[4]Chumnarnsilpa S,Lee W L,Nag S,Kannan B,Larsson M,Burtnick L D,Robinson R C 2009Proc.Natl.Acad.Sci.USA106 13719
[5]Lü C,Gao X,Li W,Xue B,Qin M,Burtnick L D,Zhou H,Cao Y,Robinson R C,Wang W 2014Nat.Commun.5 4623
[6]Qian H,Chen H,Yan J 2016Acta Phys.Sin.65 188706(in Chinese)[钱辉,陈虎,严洁2016物理学报 65 188706]
[7]Zhang W K,Wang C,Zhang X 2003Chin.Sci.Bull.48 7(in Chinese)[张文科,王驰,张希 2003科学通报 48 7]
[8]Cui S X 2016Acta Polymerica Sinica2016(9)1160
[9]Feng W,Wang Z,Zhang W 2017Langmuir33 1826
[10]Zhang X,Zhang W K,Li H B,Shen J C 2000Prog.Nat.Sci:Nat.Key Lab.Newsletter10 385(in Chinese)[张希,张文科,李宏斌,沈家骢 2000自然科学进展:国家重点实验室通讯10 385]
[11]Pang X C,Cheng B,Cui S X 2016Chinese Journal of Polymer Science34 578
[12]Yu X T,Yang Z B,Wang X Y,Tang M J,Wang Z Z,Wang H B 2016Prog.Biochem.Biophys.43 28(in Chinese)[于小婷,杨忠波,王鑫艳,汤明杰,王占忠,王化斌2016生物化学与生物物理进展43 28]
[13]Xue Y,Li X,Li H,Zhang W 2014Nat.Commun.5 4348
[14]Cheng B,Cui S X 2015Polymer Mechanochemistry369 97
[15]Yuan G,Le S,Yao M,Qian H,Zhou X,Yan J,Chen H 2017Angew.Chem.Int.Ed.Engl.56 5490
[16]Gao X,Qin M,Yin P,Liang J,Wang J,Cao Y,Wang W 2012Biophys.J.102 2149
[17]Feng W,Wang Z,Zhang W 2017Langmuir33 1826
[18]Luo Z,Cheng B,Cui S 2015Langmuir31 6107
[19]Yang Z J,Yuan G H,Zhai W L,Yan J,Chen H 2016Science China-Physics Mechanics Astronomy59 680013
[20]Schoeler C,Malinowska K H,Bernardi R C,Milles L F,Jobst M A,Durner E,Ott W,Fried D B,Bayer E A,Schulten K,Gaub H E,Nash M A 2014Nat.Commun.5 5635
[21]Pfreundschuh M,Alsteens D,Wieneke R,Zhang C,Coughlin S R,Tampe R,Kobilka B K,Muller D J 2015Nat.Commun.6 8857
[22]Dudko O K,Hummer G,Szabo A 2006Phys.Rev.Lett.96 108101
[23]Dudko O K,Hummer G,Szabo A 2008Proc.Natl.Acad.Sci.USA105 15755
[24]Bell G I 1978Science200 618
[25]Rodriguez Del Castillo A,Lemaire S,Tchakarov L,Jeyapragasan M,Doucet J P,Vitale M L,Trifaro J M 1990EMBO J.9 43
[26]Maekawa S,Sakai H 1990J.Biol.Chem.265 10940
[27]Marcu M G,Zhang L,Elzagallaai A,Trifaro J M 1998J.Biol.Chem.273 3661
[28]Cao Y,Lam C,Wang M,Li H 2006Angew Chem.Int.Ed.Engl.45 642
[29]Rief M,Gautel M,Oesterhelt F,Fernandez J M,Gaub H E 1997Science276 1109
Single molecule force spectroscopy study of calcium regulated mechanical unfolding of the A6 domain of adseverin∗
Li Peng-FeiCao YiQin Meng†Wang Wei
(National Laboratory of Solid Structure,Department of Physics,Nanjing University,Nanjing 201193,China)
Adseverin is a member of calcium-regulated gelsolin superfamily existing in secretory cells,which functions as an actin severing and capping protein.Adseverin is comprised of six independently folded domains(A1–A6),sharing high sequence identity(60%)with that of gelsolin(G1–G6).Calcium binding can convert both adserverin and gelsolin from a globular structure into a necklace structure and expose the actin binding sites.However,compared with gelsolin,adseverin lacks a C-terminal extension.Our previous single molecule force spectroscopy studies indicated that the C-terminal helix is critical to the force regulated calcium activation of gelsolin.It remains largely unexplored how the calcium binding to adseverin is regulated by force.
Here,using atomic force microscopy based single molecule force spectroscopy,we demonstrate that the mechanical unfolding of the sixth domain of adseverin(A6)can be signi fi cantly a ff ected by calcium binding.In order to identify the unfolding events of A6 unambiguously,we construct a hetero-polyprotein(GB1-A6)4,in which A6 is spliced alternatively with well-characterized protein domain GB1.Therefore,in the force-extension traces,GB1 unfolding events can serve as a fi ngerprint to identify the unfolding signature of A6.
In the absence of calcium,the unfolding traces for(GB1-A6)4show two distinct categories of events.The higher force events with unfolding forces of~180 pN and contour length increments of~18 nm correspond to the unfolding of GB1.The other category of events with lower unfolding forces of~25 pN and contour length increments of~35 nm are attributed to the mechanical unfolding of A6.The unfolding force for A6 is similar to that for the structural homological protein,G6.
However,in the presence of calcium ion,the unfolding force of A6 is dramatically increased to~45 pN,indicating that the structure of A6 can be mechanically stabilized by calcium ion-binding.Moreover,we observe a clear mechanical unfolding intermediate state for the unfolding of calcium bound A6(holo A6).Upon stretching,holo A6 is fi rst partially unfolded to an intermediate state with a contour length increment of~7.2 nm.Then,the intermediate state is unfolded to release a contour length of~27.8 nm.The total contour length change is the same as that for the calcium free A6(apo A6).Because each amino acid in the unfolded structure corresponds to a contour length increment of 0.365 nm,according to the contour length change,we infer that in the unfolding intermediate state of A6,its N-terminal regions is partially unfolded.This leads to the exposure of the cryptic actin binding site on A5,which is otherwise buried in the folded structure of A6.The force regulated activation mechanism for A6 is similar to that for G6,except that they use di ff erent sequences from those in the force-sensitive region.In G6 the C-terminal helix serves as the force-responsive tail to regulate actin binding,while in A6 the N-terminal sequences are unstructured upon stretching to promote the actin binding for adseverin.
Therefore,we infer that force may be an important regulator for the actin-binding of all members in the gelsolin family proteins,including adseverin and gelsolin.Our study represents an important step towards the understanding of the function of adseverin at a molecular level.
adseverin,single molecule force spectroscopy,unfolding,intermediate state
8 May 2017;revised manuscript
30 June 2017)
(2017年5月8日收到;2017年6月30日收到修改稿)
10.7498/aps.66.196201
∗国家自然科学基金(批准号:21522402,11674153,11374148,11334004)和国家重点基础研究发展计划(批准号:2013CB834100)资助的课题.
†通信作者.E-mail:qinmeng@nju.edu.cn
©2017中国物理学会Chinese Physical Society
PACS:62.25.–g,64.60.Q–,68.37.Ps
10.7498/aps.66.196201
*Project supported by the National Natural Science Foundation of China(Grant Nos.21522402,11674153,11374148,11334004)and the National Basic Research Program of China(Grant No.2013CB834100).
†Corresponding author.E-mail:qinmeng@nju.edu.cn
