银杏中黄酮类化合物生物合成、调控机制及其影响因素的研究进展
2017-10-20董理想倪君
董理想+倪君

[摘要]银杏叶片提取物对多种疾病具有治疗效果,并已被广泛应用于临床实践。黄酮类化合物是银杏叶片提取物的主要活性成分之一,所以研究银杏黄酮的合成途径具有非常重要的意义。该综述首先回顾了植物黄酮生物合成途径的总体研究概况,并介绍了目前已经发现的银杏黄酮合成相关基因;其次,介绍了高通量测序技术在银杏研究中的几个实际应用。另外,作为银杏的重要研究方向之一,该综述讨论了外界条件对银杏细胞、整株及采摘后叶片的黄酮含量及相关基因表达的影响,并着重介绍了笔者实验室的最新实验结果。最后,该综述对银杏黄酮领域未来的研究方向做了展望。
[关键词]银杏; 黄酮; 基因
Research progress of flavonoid biosynthesis, regulation mechanism and
influence factors in Ginkgo biloba
DONG Lixiang, NI Jun*
(Key Laboratory of Hangzhou City for Quality and Safety of Agricultural Products, College of Life and
Environmental Sciences, Hangzhou Normal University, Hangzhou 310018, China)
[Abstract]The extract of ginkgo leaves showed positive effects on treatments of many diseases, and has been used clinically worldwide Considering the fact that flavonoids are the main bioactive components in the extract of ginkgo leaves, it is very important to investigate the flavonoid biosynthesis in ginkgo leaves In this paper, we first reviewed the research progress of flavonoid biosynthesis in different plants, and introduced the flavonoid biosynthesis related genes discovered in ginkgo Then, several cases of ginkgo researches using high throughput sequencing technology were described in detail In addition, as an important research area in ginkgo, the changes of flavonoid content and the expression of corresponding genes were discussed Specifically, our latest results were described At last, we prospected the development of research area in flavonoid biosynthesis in ginkgo.
[Key words]Ginkgo biloba; flavonoid; gene
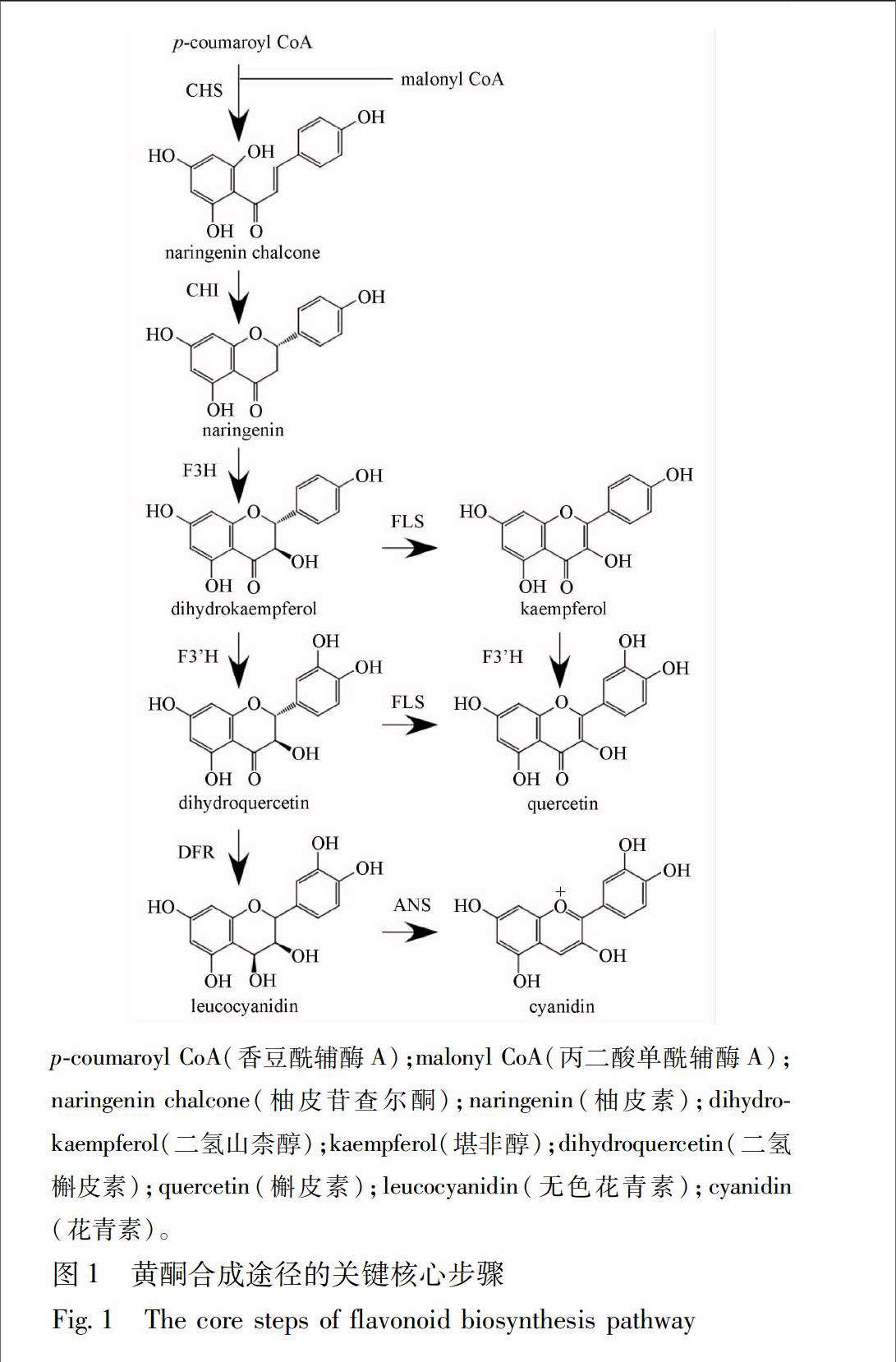
黃酮类化合物(flavonoids)是绝大多数植物共有的一大类次生代谢产物。比如色彩斑斓的花朵,秋天各种颜色的树叶、果实和植物种子,都是由属于黄酮类的色素造成的。目前已知的黄酮类化合物已经超过了6 000种,而且这个数字还在持续上升中[1]。黄酮类化合物的生物合成途径在不同植物中非常保守,并且是植物学中研究最深入的领域之一[2]。它们的合成起始于苯基丙酸类合成途径(phenylpropanoid pathway),查尔酮合成酶(chalcone synthase)被认为是第一个参与黄酮合成的酶,产生的查尔酮则是所有各种黄酮类化合物的源头。从查尔酮开始,在后续各种酶的催化下,形成了不同大类的黄酮类化合物[3]。最后,通过各种基团转移酶的修饰,在黄酮类化合物的骨架上引入糖基化、甲基化或酰基化修饰,形成了数量极其庞大的黄酮类化合物[1,4]。
各种黄酮类化合物不仅广泛参与植物的各项生命活动,而且某些种类对人类很多疾病都具有一定的疗效,因此具有广阔的发展潜力[5]。在已有的各种植物黄酮类制品中,银杏叶提取物EGB761是最著名的产品之一[6]。银杏这一古老物种曾经一度被认为已经灭绝,直到后来在中国被重新发现,因此它又有活化石的美誉。银杏的药用价值很早就在中国被发现,并最早记录在元朝的《食物本草》和《日用本草》中[7],然而直到1965年才由德国药物生理学家真正将银杏叶提取物用于临床治疗,并最终发展成商品化的EGB761[6]。
近年来,随着分子生物学的发展,尤其是高通量测序技术的普及,银杏黄酮合成的研究进入了一个新阶段。本综述回顾了银杏黄酮研究的发展历史,并结合最新的研究成果,展望了该领域未来发展的方向。
1黄酮生物合成途径的总体研究概况
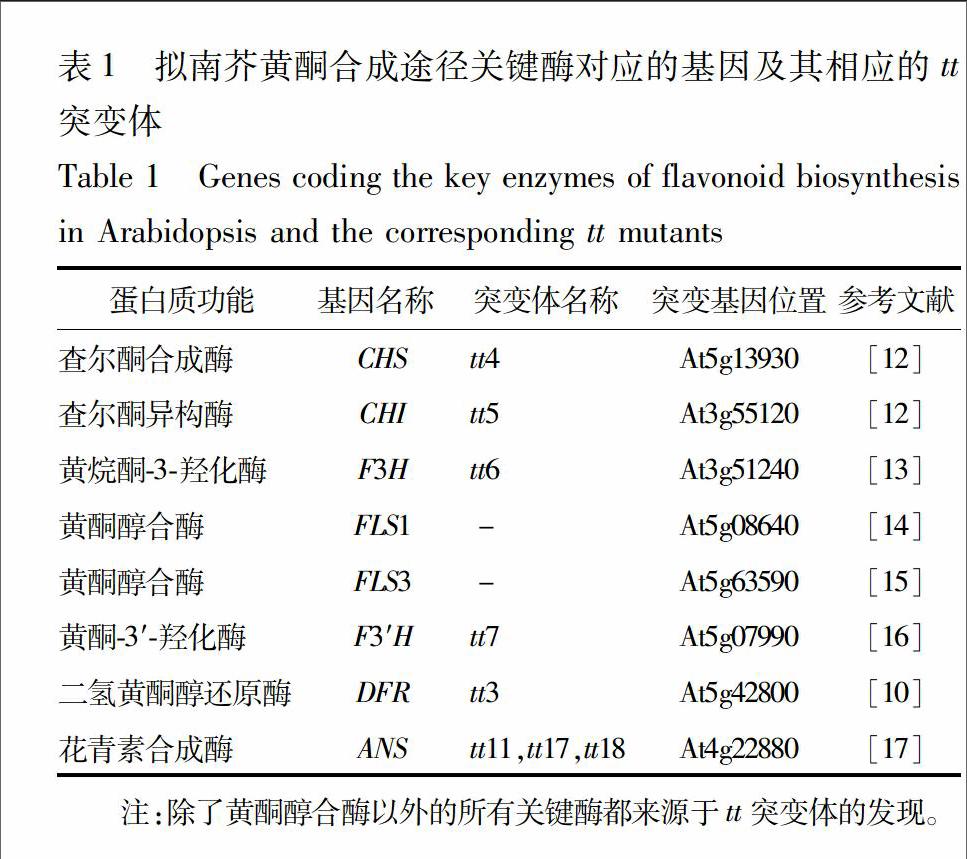
11拟南芥黄酮合成途径相关基因的研究人们很早就在拟南芥中发现了一类由于种皮缺乏色素沉积而造成透明种皮表型的突变体tt(transparent testa),并将它们的表型用于拟南芥早期遗传连锁图谱的构建[8]。随着分子生物学的发展,人们逐渐认识到,造成这些突变表型的原因是编码黄酮合成相关酶的基因发生了突变。在最早发现的3个基因中,编码查尔酮合成酶(chalcone synthase, CHS)的基因突变造成了tt4表型;查尔酮异构酶(chalcone isomerase, CHI)突变造成了tt5表型;二氢黄酮醇4还原酶(dihydroflavonol 4reductase, DFR)突变造成了tt3表型[910]。利用这些信息,人们第一次得到了黄酮保护植物免受紫外线伤害的直接证据:编码黄酮合成相关酶的基因突变后,黄酮含量降低,突变体对紫外敏感[11]。利用这些tt突变体,编码黄酮合成的许多关键酶基因被克隆出来(表1,图1)。endprint
往往没有明显的表型,所以研究起来就相对困难得多。例如在最近1个研究案例中,作者研究了81个拟南芥品种的黄酮苷含量,发现某些品种含有1种黄酮苷(flavonol 3Ogentiobioside 7Orhamnoside, F3GG7R),而另一些品种没有。随后作者挑选2个代表品种构建重组自交系,最后利用图位克隆的方法将这个基因定位出来[18]。由此可见,鉴定这类基因不仅需要更多的工作量,还需要有一定的运气。如果这个黄酮苷在各个品种中都有,那就不能用这种构建重组自交系的方法来克隆了。由于糖基转移酶研究工作困难重重,所以到目前为止也才鉴定出少数几个[19]。这显然与植物体内大量的黄酮苷种类数目形成了巨大的反差。说明在黄酮合成这个巨大的网络中还有很多工作需要做。
可能由于鉴定糖基转移酶的工作量很大而意义却不大,所以目前拟南芥黄酮相关的工作已经逐渐由黄酮合成转向黄酮功能的研究。由于黄酮的抗氧化活性,人们自然想到黄酮可能在植物抵御生物或非生物胁迫中具有一定的功能,因为这些逆境胁迫往往伴随着植物体内活性氧浓度的上升。事实上也确实如此,目前已有大量的研究证明,黄酮在植物抵御各种逆境胁迫中发挥了重要作用[11,2022]。有意思的是,黄酮还在拟南芥其他生命活动,比如在生长素运输中发挥了重要的调控作用[23],但由于这些都不在主题范围内,故不做深入讨论。
12其他物种的黄酮合成途径研究在植物由水生转向陆生的进化过程中,要面对来自陆地的干旱、氧化和紫外线等各种环境胁迫。因此,最原始的陆生植物——苔藓,也普遍含有黄酮类化合物来应对来自陆地的各种威胁[24]。可是由于苔藓类植物的经济价值尚未被充分开发,再加上它在植物进化过程中的特殊地位,所以关于苔藓黄酮合成途经的研究基本都集中在分子进化比较上[25]。另一方面,由于黄酮含量和很多农产品的品质密切相关,所以经济作物的黄酮合成途径被大量研究。
不同于拟南芥和苔藓等模式植物,经济作物的黄酮合成途径研究一般都是以应用导向型为主。人们通过研究它们的黄酮合成途径,期望改善农产品的色泽,口感,营养价值等指标,从而获得更好的经济效益。葡萄酒因富含黄酮类化合物而对人体具有重要的生理保健功效,所以葡萄的黄酮合成研究相对比较深入。在葡萄中,不仅编码各种黄酮合成酶的基因已经被鉴定并克隆出来,而且调控这些基因的转录因子家族也已被发现,它们共同组成了一个调控葡萄黄酮合成途径的网络[26]。目前,很多葡萄黄酮合成途径的研究集中于不同环境因子对黄酮合成基因的调控及对后续黄酮含量的影响[2728]。研究者期望揭示各种环境因子影响葡萄黄酮含量的内在机理,并最终改善葡萄酒的品质。苹果是世界上最重要的水果之一,它被广泛种植并大量消费。临床研究表明,食用苹果能有效降低某些癌症,心血管疾病,糖尿病等多种疾病,而黄酮类化合物则是苹果的重要有效成分之一[29]。已有研究表明,不同品种苹果的黄酮成分及含量差异很大,而且还随着不同的栽培条件而有所变化[3031]。另外,苹果的黄酮合成途径研究也已经较为深入,不仅合成途径中的关键基因被克隆出来,而且这些基因的表达情况已经被用于解释苹果不同的生理现象 [3233]。
2銀杏黄酮合成相关基因的克隆鉴定
由于银杏黄酮明确的药用价值,其合成途径的研究具有重要的意义,也取得了较大的进展。2004年,第一个银杏黄酮合成相关的基因,查尔酮合成酶基因(GbCHS)被鉴定出来。它编码的蛋白质氨基酸序列和其他物种的CHS序列非常相似,说明它们可能具有类似的功能。与它最接近的是裸子植物的CHS序列,说明了它们在进化上的亲缘关系[34]。进一步研究发现,该基因在银杏中可能有多个拷贝,因为用GbCHS基因序列作为探针做的Southern杂交出现了多个条带[35]。果然,利用cDNA末端快速扩增技术(RACE),有研究者克隆了第二个查尔酮合成酶基因,并命名为GbCHS2。这2个基因编码的蛋白质氨基酸序列具有高度的同源性,暗示它们在功能上可能具有一定的冗余[36]。作为黄酮合成过程中的重要基因,它的表达方式自然也是人们感兴趣的对象。为了更好地研究这一点,GbCHS基因的启动子序列被克隆出来,分段接上报告基因(GUS)转入烟草中。通过对转基因烟草的GUS染色,GbCHS基因启动子中的一段重要序列被发现,它对于GbCHS基因响应各种外界刺激具有重要作用[37]。
查尔酮异构酶(CHI)也是黄酮合成途径中的一个关键酶。有研究者利用简并引物成功克隆出银杏中编码查尔酮异构酶的基因,GbCHI。该基因的表达受紫外线和多种激素的诱导,在大肠杆菌中表达的重组蛋白具有查尔酮异构酶的活性,即能够催化6′羟基查尔酮(6′hydroxychalcone)向柚皮素(naringenin)的转化。在银杏叶片中该基因的表达强度与查尔酮异构酶的活性,及叶片的黄酮含量呈现正相关的关系。这些结果都说明克隆到的基因确实是银杏中编码查尔酮异构酶的基因[38]。利用类似的方法,更多的黄酮合成相关基因被鉴定出来(表2)。不过这些研究基本都仅限于同源克隆,体外酶活分析,基因表达和黄酮含量的协同分析等阶段。研究深度较浅,都还没有用拟南芥突变体互补实验来证明它们在体内的真正功能。
目前,除了上述编码黄酮合成相关酶的基因外,还有一个编码调控银杏黄酮合成的转录因子被鉴定出来。GbMYBF2在银杏各处都有表达,且在根部表达量最高(根部的黄酮含量最低)。在叶片的生长过程中,GbMYBF2的表达量和叶片的黄酮含量呈现负相关。这些结果暗示GbMYBF2可能编码银杏黄酮合成过程中的一个负调控因子。为了进一步证实这个猜测,该基因被接上过量表达的启动子,并转入拟南芥。果然,在转基因拟南芥中,不仅黄酮合成相关基因的表达下降,体内的黄酮含量也降低了。这说明GbMYBF2确实在银杏黄酮合成途径中担任负调控因子的角色[43]。
虽然到目前为止很多银杏黄酮合成相关的基因已经被克隆,但是针对它们的研究深度都比较浅,所以还没有发现银杏中的新机制。endprint
3高通量测序浪潮下的银杏基因研究
随着高通量测序的成本越来越低,很多物种的研究都采用了转录组测序的手段,银杏也不例外。最早的一批转录组数据来自银杏的无菌苗。这次的转录组测序得到了39 941个具有蛋白编码序列的特异转录片段,其中的24 645个片段在以前的数据库中有记录。另外,有50个片段编码的蛋白可能参与了银杏黄酮合成途径。有意思的是,这次测序还找到一个编码查尔酮异构酶的基因,且这个基因和先前报道的那个GbCHI不同,所以将其命名为GbCHI1[44]。同年,另一个课题组发表了银杏果转录组的成果。他们利用高通量测序研究银杏果在发育过程中的转录组变化。这次研究一共找到了68 547个基因,其中3 869个基因的表达量在银杏果的发育过程中发生明显变化。可惜的是,虽然这个研究详细讨论了很多基因的注释和表达情况,但对于黄酮合成相关基因却没有涉及[45]。由于银杏雌雄异株,所以有课题组试图通过研究雌芽,雄芽,雌性大孢子叶球和雄性小孢子叶球之间的转录组差异来寻找决定银杏性别的关键基因。虽然最终并没有找到那个基因,但是在研究過程中一共得到了108 307个基因,其中51 953个基因在公共数据库中找到了注释。可惜的是,这次的研究和上述一样,并没有涉及黄酮合成相关的基因[46]。
与此同时,银杏全基因组测序工作也开始启动。经过竞争,最终来自浙江大学、中国科学院植物所和深圳华大基因研究院的联合团队率先发表了银杏全基因组的测序结果[47]。该结果显示,银杏具有1061 Gb的基因组,包含了41 840个基因。长期的积累(银杏是一个古老的物种,被称为活化石)和2次全基因组倍增(whole genome duplication, WGD)造成了银杏巨大的基因组,并含有大量的长末端重复序列的反转录转座子(long terminal repeat retrotransposons, LTRRTs)。另外,该研究还发现了银杏黄酮合成途径中的四类基因发生了基因倍增事件,它们是CHS,F3H,FLS和DFR。这些倍增的基因可能部分解释了银杏体内丰富的黄酮含量和较强的抗病性[47]。
4外界条件对银杏黄酮含量及相关基因表达的影响
相比较于银杏复杂的黄酮合成途径,人们更关心如何才能提高银杏体内的黄酮含量,所以大量的工作集中到提高银杏黄酮含量的方法研究上。本实验室早期的研究关注于外界刺激对银杏细胞系黄酮含量的影响。水杨酸(salicylic acid, SA)和茉莉酸(jasmonic acid, JA)信号在真菌激发子(fungal elicitor)诱导的黄酮合成中起了中介作用,且它们的功能部分互补[48]。另外,臭氧能够诱导银杏细胞的黄酮合成,且该过程依赖于硝酸还原酶介导的NO信号[49]。
由于细胞系的结果并不一定能如实反映银杏体内真实的变化,所以更多的工作建立在银杏树的生理研究上。银杏叶片的采摘时间在制药行业中有一定的规范,有研究者对不同时期采摘的银杏叶片进行黄酮含量分析。利用高效液相色谱(HPLC),该研究者发现5月份采摘的银杏叶片黄酮含量最高,以后逐月递减,深秋掉落的叶片黄酮含量最低。另外,该研究还发现雄树叶片的黄酮含量一般比来自雌树的高一些[50]。有意思的是,另一项来自印度的却研究表明,银杏叶片的黄酮含量和采摘的季节并没有显著的关联,7月份和11月份采摘叶片的黄酮含量差不多[51]。造成这个问题的原因可能是不同的气候条件造成的。印度地处热带,常年高温,没有分明的四季,所以导致了不同时期采摘的叶片具有差不多的黄酮含量。所以不同地区因地制宜地研究银杏最佳采摘时间还是有必要的。光照强度也会对银杏叶片的黄酮含量产生影响。有研究者利用玻璃温室的阳光遮挡,制造出了25%~100%不同的阳光强度,结果发现,在100%阳光强度下,银杏叶片的黄酮含量最高,同时银杏黄酮合成相关基因(GbPAL, GbCHS, GbF3H和GbFLS)的表达量也达到最高。可是在黄酮产量方面,100%的光照强度却不是最合适的,而是在76%光照强度下达到最高。因为在这个光照强度下,银杏生物量的增加是最大的。此时虽然黄酮的含量不是最高,但单位面积的产量却达到最高[52]。还有研究者在人工气候室内采用土培盆栽的方法探索了不同温度和土壤水分对银杏叶片黄酮含量的影响。他们发现虽然低温能够提高叶片的黄酮含量,但是由于高温下银杏的生物量最大,所以高温下银杏单株总黄酮含量却是最高。另外,土壤水分对黄酮含量影响不大[53]。有意思的是,另一项研究却发现了水分和黄酮含量之间的关系。根系分区灌溉技术(partial rootzoom irrigation)是香港中文大学的张建华教授发明并推广的一项新型节水灌溉技术,在农业生产方面具有巨大的应用价值[54]。 有研究者将此项技术推广到银杏黄酮合成领域,并取得了很好的效果。他们发现,银杏在根系分区灌溉技术处理下,叶片的黄酮含量显著升高[55]。另外,昆虫的啃食也能提高银杏黄酮的含量。有研究表明,昆虫的啃食会导致银杏叶片细胞的去极化,并伴随胞内钙离子和双氧水浓度的升高,同时银杏黄酮的含量也出现了显著升高。有意思的是,并不是所有黄酮合成相关基因的表达都是上调的,比如编码黄酮醇合酶的基因GbFLS表达就出现了下调。目前,造成这种基因表达和终产物浓度情况不一致的具体原因还不清楚[56]。综上所述,虽然目前已经发现了多种方法都能提高银杏树叶片的黄酮含量,但是由于它们较少涉及到基因层面,所以研究深度不够,很难从机理上进行改进而进一步提高黄酮的含量。
另一方面,由于采摘后的银杏叶片细胞仍具有活性,所以适当处理采摘后的叶片也可能提高其黄酮含量。这种方法易于进行大规模操作,具有可快速应用于制药工业的可能性,所以本实验室的一个研究方向就是采摘后处理银杏叶片以提高其黄酮含量的研究。采摘后的叶片经过紫外(UVB)照射处理能够显著增加叶片的黄酮含量。基因表达检测显示,银杏黄酮合成相关的基因都能够被紫外照射上调,且黄酮合成的负调控因子GbMYBF2的表达下调[57]。这个结果符合预期,因为有类似的实验得出了相同的结果[58]。另外,本实验室还研究了其他环境因子对叶片黄酮含量的影响,最后发现,NaCl处理同样能够增加采摘后叶片的黄酮含量。可是当检测相关基因的表达时,却发现黄酮合成相关的基因却是下调的,并且GbMYBF2表达上调。这个结果和预期完全相反。有意思的是,当直接用NaCl处理银杏苗时,其黄酮含量升高,合成相关的基因表达也升高,GbMYBF2表达降低,这个结果又和原先的预期一致。综上所述,叶片采摘后的状态可能和自然生长的状态有差别,研究者不能简单地套用过去的植物生理实验结果来预测采摘后银杏叶片的生理反应 [57]。endprint
5展望
目前擬南芥黄酮合成途径的研究已经比较清楚,剩下的只有最后一步负责各种修饰的基团转移酶编码基因的鉴定[19]。但是银杏黄酮合成基因的研究目前还停留在传统的拟南芥同源基因的克隆层面。这种方法虽然有一定的效果,但是很难找全所有的基因。已经有越来越多的证据表明,不同于拟南芥的单拷贝基因,在银杏黄酮合成途径中,每一步的反应都可能由一个基因家族控制。在NaCl处理采摘后银杏叶片的实验中,虽然黄酮含量显著升高,但是黄酮合成基因的表达却下降。因此,必然存在另一条还不知道的途径,而这条途径则伴随着黄酮合成的新基因[57]。所以彻底研究银杏黄酮合成途径每个步骤的所有基因成了下一步要完成的目标。最近银杏的全基因组测序已经完成,结果也已经公布[47]。虽然目前还看不到基因注释的详细结果,但是不久以后,一张更加详细的全新银杏黄酮合成调控网络将出现在大家面前。
目前银杏黄酮的利用还局限于叶片的总黄酮提取物,对其中某些成分进行分离提取的需求还比较少。银杏叶片目前已经被鉴定出含有70多种黄酮,其中的30多种已经被分离出来[59]。而在这么多种类的银杏黄酮中,并不是所有的成分都是具有药用价值的。已经有研究表明,不同种类的银杏黄酮对细胞具有不同的作用[60]。所以未来的银杏黄酮研究将朝着精细化的方向发展,人们将关注某些银杏黄酮的变化而不是总黄酮。所以在未来的实验中,用HPLC技术鉴定每种银杏黄酮对各种处理的反应将逐步取代酸化水解后的总黄酮测定。
[参考文献]
[1]Ferrer J L, Austin M B, Stewart C J, et al. Structure and function of enzymes involved in the biosynthesis of phenylpropanoids[J]. Plant Physiol Biochem, 2008, 46(3):356.
[2]WinkelShirley B It takes a garden How work on diverse plant species has contributed to an understanding of flavonoid metabolism[J]. Plant Physiol, 2001, 127(4):1399.
[3]Martens S, Preuss A, Matern U Multifunctional flavonoid dioxygenases:flavonol and anthocyanin biosynthesis in Arabidopsis thaliana L.[J]. Phytochemistry, 2010, 71(10):1040.
[4]Bowles D, Isayenkova J, Lim E K, et al. Glycosyltransferases:managers of small molecules[J]. Curr Opin Plant Biol, 2005, 8(3):254.
[5]Lu M F, Xiao Z T, Zhang H Y. Where do health benefits of flavonoids come from? Insights from flavonoid targets and their evolutionary history[J]. Biochem Biophys Res Commun, 2013, 434(4):701.
[6]De Feudis F V A brief history of EGb 761 and its therapeutic uses[J]. Pharmacopsychiatry, 2003, 36(Suppl 1):S2.
[7]Tredici P D Ginkgos and peoplea thousand years of interaction[J]. Arnoldia(Boston), 1991, 51(2):2.
[8]Koornneef M, van Eden J, Hanhart C J, et al. Linkage map of Arabidopsis thaliana[J]. J Hered, 1983, 74:265.
[9]Chang C, Bowman J L, DeJohn A W, et al. Restriction fragment length polymorphism linkage map for Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 1988, 85(18):6856.
[10]Shirley B W, Hanley S, Goodman H M Effects of ionizing radiation on a plant genome:analysis of two Arabidopsis transparent testa mutations[J]. Plant Cell, 1992, 4(3):333.
[11]Li J, OuLee T M, Raba R, et al. Arabidopsis flavonoid mutants are hypersensitive to UVB irradiation[J]. Plant Cell, 1993, 5(2):171.
[12]Shirley B W, Kubasek W L, Storz G, et al. Analysis of Arabidopsis mutants deficient in flavonoid biosynthesis[J]. Plant J, 1995, 8(5):659.endprint
[13]Pelletier M K, Shirley B W Analysis of flavanone 3hydroxylase in Arabidopsis seedlings(Coordinate regulation with chalcone synthase and chalcone isomerase)[J]. Plant Physiol, 1996, 111(1):339.
[14]Pelletier M K, Murrell J R, Shirley B W, Characterization of flavonol synthase and leucoanthocyanidin dioxygenase genes in Arabidopsis (further evidence for differential regulation of "early" and "late" genes)[J]. Plant Physiol, 1997, 113(4):1437.
[15]Owens D K, Alerding A B, Crosby K C, et al Functional analysis of a predicted flavonol synthase gene family in Arabidopsis[J]. Plant Physiol, 2008, 147(3):1046.
[16]Schoenbohm C, Martens S, Eder C, et al. Identification of the Arabidopsis thaliana flavonoid 3′hydroxylase gene and functional expression of the encoded P450 enzyme[J]. Biol Chem, 2000, 381(8):749.
[17]Devic M, Guilleminot J, Debeaujon I, et al The BANYULS gene encodes a DFRlike protein and is a marker of early seed coat development[J]. Plant J, 1999, 19(4):387.
[18]Ishihara H, Tohge T, Viehover P, et al Natural variation in flavonol accumulation in Arabidopsis is determined by the flavonol glucosyltransferase BGLU6[J]. J Exp Bot, 2016, 67(5):1505.
[19]Schffner A R Flavonoid biosynthesis and Arabidopsis genetics:more good music[J]. J Exp Bot, 2016, 67(5):1203.
[20]Keilig K, LudwigMüller J Effect of flavonoids on heavy metal tolerance in Arabidopsis thaliana seedlings[J]. Bot Stud, 2009, 50(3):311.
[21]Nakabayashi R, YonekuraSakakibara K, Urano K, et al. Enhancement of oxidative and drought tolerance in Arabidopsis by overaccumulation of antioxidant flavonoids[J]. Plant J, 2014, 77(3):367.
[22]Schulz E, Tohge T, Zuther E, et al. Flavonoids are determinants of freezing tolerance and cold acclimation in Arabidopsis thaliana[J]. Sci Rep, 2016, 6:34027.
[23]Kuhn B M, Geisler M, Bigler L, et al. Flavonols accumulate asymmetrically and affect auxin transport in Arabidopsis[J]. Plant Physiol, 2011, 156(2):585.
[24]Markham K R The flavonoids[M] Berlin:Springer, 1988:427.
[25]Liu S, Ju J, Xia G Identification of the flavonoid 3′hydroxylase and flavonoid 3′, 5′hydroxylase genes from Antarctic moss and their regulation during abiotic stress[J] Gene, 2014, 543(1):145.
[26]Czemmel S, Heppel S C, Bogs J R2R3 MYB transcription factors:key regulators of the flavonoid biosynthetic pathway in grapevine[J] Protoplasma, 2012, 249(2):109.
[27]MartínezLüscher J, SánchezDíaz M, Delrot S, et al. UltravioletB radiation and water deficit interact to alter flavonol and anthocyanin profiles in grapevine berries through transcriptomic regulation[J] Plant Cell Physiol, 2014, 55(11):1925.endprint
[28]Downey M O, Dokoozlian N K, Krstic M P Cultural practice and environmental impacts on the flavonoid composition of grapes and wine:a review of recent research[J] Am J Enol Viticult, 2006, 57(3):257.
[29]Boyer J, Liu R H Apple phytochemicals and their health benefits[J] J Nutr, 2004, 3(1):5.
[30]Vinson J A, Su X, Zubik L, et al. Phenol antioxidant quantity and quality in foods:fruits[J] J Agric Food Chem, 2001, 49(11):5315.
[31]Vrhovsek U, Rigo A, Tonon D, et al. Quantitation of polyphenols in different apple varieties[J] J Agric Food Chem, 2004, 52(21):6532.
[32]Honda C, Kotoda N, Wada M, et al. Anthocyanin biosynthetic genes are coordinately expressed during red coloration in apple skin[J] Plant Physiol Bioch, 2002, 40(11):955.
[33]HenryKirk R A, McGhie T K, Andre C M, et al. Transcriptional analysis of apple fruit proanthocyanidin biosynthesis[J] J Exp Bot, 2012, 63(15):5437.
[34]Pang Y, Shen G A, Liu C, et al. Molecular cloning and sequence analysis of a novel chalcone synthase cDNA from Ginkgo biloba[J]. DNA Seq, 2004, 15(4):283.
[35]Pang Y, Shen G, Wu W, et al. Characterization and expression of chalcone synthase gene from Ginkgo biloba[J]. Plant Sci, 2005, 168(6):1525.
[36]Xu F, Cheng S Y, Cheng S H, et al. Time course of expression of chalcone synthase gene in Ginkgo biloba[J]. J Plant Physiol Mol Biol, 2007, 33(4):309.
[37]Li L, Cheng H, Yuan H, et al. Functional characterization of the Ginkgo biloba chalcone synthase gene promoter in transgenic tobacco[J]. Genet Mol Res, 2014, 13:3446.
[38]Cheng H, Li L, Cheng S, et al. Molecular cloning and function assay of a chalcone isomerase gene(GbCHI)from Ginkgo biloba[J]. Plant Cell Rep, 2010, 30(1):49.
[39]Shen G, Pang Y, Wu W, et al. Cloning and characterization of a flavanone 3hydroxylase gene from Ginkgo biloba[J]. Biosci Rep, 2006, 26(1):19.
[40]Xu F, Li L, Zhang W, et al. Isolation, characterization, and function analysis of a flavonol synthase gene from Ginkgo biloba[J]. Mol Biol Rep, 2011, 39(3):2285.
[41]Hua C, Linling L, Shuiyuan C, et al. Molecular cloning and characterization of three genes encoding dihydroflavonol4reductase from Ginkgo biloba in anthocyanin biosynthetic pathway[J]. PLoS ONE, 2013, 8(8):e72017.
[42]Xu F, Cheng H, Cai R, et al. Molecular cloning and function analysis of an anthocyanidin synthase gene from Ginkgo biloba, and its expression in abiotic stress responses[J]. Mol Cells, 2008, 26(6):536.endprint
[43]Xu F, Ning Y, Zhang W, et al. An R2R3MYB transcription factor as a negative regulator of the flavonoid biosynthesis pathway in Ginkgo biloba[J]. Funct Integr Genomics, 2014, 14(1):177.
[44]Han S, Wu Z, Jin Y, et al. RNASeq analysis for transcriptome assembly, gene identification, and SSR mining in ginkgo(Ginkgo biloba L)[J]. Tree Genet Genomes, 2015, 11(3):37.
[45]He B, Gu Y, Xu M, et al. Transcriptome analysis of Ginkgo biloba kernels[J]. Front Plant Sci, 2015, 6:819.
[46]Du S, Sang Y, Liu X, et al. Transcriptome profile analysis from different sex types of Ginkgo biloba L[J]. Front Plant Sci, 2016, 7:871.
[47]Guan R, Zhao Y, Zhang H, et al Draft genome of the living fossil Ginkgo biloba[J]Gigascience, 2016, 5(1):49.
[48]Xu M, Dong J, Wang H, et al. Complementary action of jasmonic acid on salicylic acid in mediating fungal elicitorinduced flavonol glycoside accumulation of Ginkgo biloba cells[J]. Plant Cell Environ, 2009, 32(8):960.
[49]Xu M, Zhu Y, Dong J, et al. Ozone induces flavonol production of Ginkgo biloba cells dependently on nitrate reductasemediated nitric oxide signaling[J]. Environ Exp Bot, 2012, 75:114.
[50]Fan Y, Wang Y, Tan R, et al. Seasonal and sexual variety of Ginkgo flavonol glycosides in the leaves of Ginkgo biloba L[J]. China J Chin Mater Med, 1998, 23(5):267.
[51]Kaur P, Chaudhary A, Singh R D, et al. Spatial and temporal variation of secondary metabolite profiles in Ginkgo biloba leaves[J]. Chem Biodivers, 2012, 9(2):409.
[52]Xu Y, Wang G, Cao F, et al. Light intensity affects the growth and flavonol biosynthesis of Ginkgo(Ginkgo biloba L)[J]. New Forest, 2014, 45(6):765.
[53]汪贵斌,郭旭琴,常丽,等. 温度和土壤水分对银杏叶黄酮类化合物积累的影响[J]. 应用生态学报, 2013, 24(11):3077.
[54]Marris E Agronomy:five crop researchers who could change the world[J]. Nature, 2008, 456(7222):563.
[55]Wang L, Shi H, Wu J, et al. Alternative partial rootzone irrigation enhances leaf flavonoid accumulation and water use efficiency of Ginkgo biloba[J]. New Forest, 2016, 47(3):377.
[56]Mohanta T K, Occhipinti A, Zebelo S A, et al. Ginkgo biloba responds to herbivory by activating early signaling and direct defenses[J] PLoS ONE, 2012, 7(3):e32822.
[57]Ni J, Hao J, Jiang Z, et al. NaCl induces flavonoid biosynthesis through a putative novel pathway in postharvest Ginkgo leaves[J]. Front Plant Sci, 2017, 8:920.
[58]Sun M, Gu X, Fu H, et al. Change of secondary metabolites in leaves of Ginkgo biloba L in response to UVB induction[J]. Innov Food Sci Emerg, 2010, 11(4):672.
[59]Liu X G, Wu S Q, Li P, et al. Advancement in the chemical analysis and quality control of flavonoid in Ginkgo biloba[J]. J Pharm Biomed Anal, 2015, 113:212.
[60]Xie H, Wang J R, Yau L F, et al. Quantitative analysis of the flavonoid glycosides and terpene trilactones in the extract of Ginkgo biloba and evaluation of their inhibitory activity towards fibril formation of betaamyloid peptide[J]. Molecules, 2014, 19(4):4466.
[責任编辑丁广治]endprint
