桃钙调磷酸酶B类似蛋白基因家族鉴定与分析
2017-10-20张杰伟魏建华
张杰伟,吴 婷,任 飞,谢 华**,魏建华**
(1. 北京市农林科学院 农业基因资源与生物技术北京市重点实验室,北京 100097;2. 中国科学院大学,北京 100101)
桃钙调磷酸酶B类似蛋白基因家族鉴定与分析
张杰伟1,吴 婷2*,任 飞1,谢 华1**,魏建华1**
(1. 北京市农林科学院 农业基因资源与生物技术北京市重点实验室,北京 100097;2. 中国科学院大学,北京 100101)
为了研究桃钙调磷酸酶B类似蛋白(CBLs)基因家族在桃中如何发挥作用。利用桃基因组数据库,通过生物信息学手段,鉴定桃CBL家族基因的基因结构、染色体定位和编码蛋白,通过序列比对进行进化和分类分析。结果表明,桃基因组中含有8 个CBL家族基因,分布于桃的5 条染色体上。MEME和Pfam保守结构域分析显示,桃CBL蛋白均含有4个保守的EF结构域。进化树分析表明CBLs可分为4个亚家族。TMHMM 2.0Server分析显示仅PpCBL1含有1个跨膜区。PlantsPFeatureScan分析显示PpCBL2、3和5均含有1个N-豆蔻酰化位点。NetPhos 2. 0 Server结果显示PpCBLs存在着大量的丝氨酸(Ser)、苏氨酸(Thr)及酪氨酸(Tyr)潜在磷酸化位点。以上结果将为今后揭示桃CBL的功能提供重要的理论基础。
钙调磷酸酶B类似蛋白;桃;基因家族;进化分析;序列特征
钙调磷酸酶B类似蛋白(calcineurin B-like,CBL)是植物所特有的一类钙信号感受器,其通过结合Ca2+被激活后以磷酸化的方式激活下游的钙调磷酸酶B类似蛋白互作蛋白激酶(CBL-interacting protein kinases, CIPKs),激活的CIPKs 迅速通过磷酸化下游信号分子传递钙离子信号[1]。研究表明:植物CBL-CIPK途径不仅参与病原体与防御反应、非生物逆境胁迫等过程,而且参与了营养元素吸收与平衡、激素应答等植物生长和发育过程[1-3]。植物CBL基因最早在拟南芥中发现,目前已从玉米、杨树、棉花、豆类、水稻等作物中成功分离[4]。植物CBL 蛋白的结构较为保守,每个CBL 蛋白都包含4 个结合Ca2+的螺旋-环-螺旋的EF 手型(elongation factor hand) 结构域,但不同CBL 之间EF 手型结构域存在不同程度的变异[5]。植物中研究最早同时功能解析比较完整的应答逆境胁迫的信号途径是拟南芥CBL4-CIPK24-SOS1 通路,也被称为SOS途径[6-7]。研究表明:高盐胁迫时,拟南芥AtCBL4 (SOS3)及AtCBL10结合Ca2+后与AtCIPK24 (SOS2)蛋白激酶形成蛋白复合体,在地上及地下部分分别调控相应靶蛋白的表达,从而使植物能抵御高盐胁以减少植株的伤害[8]。此外,拟南芥AtCBL4 (SOS3)还可通过介导肌动蛋白重排,从而提高植株对盐胁迫的耐受[9]。最近的研究表明,CBL除典型的依赖CIPK信号途径以外还存在不依赖CIPK的信号途径,拟南芥AtCBL10在感受到钙信号的变化之后,可直接与AtAKT1互作,削弱其介导的钾离子内流,同时AtCBL10可以与AtCIPK23竞争性结合AtAKT1调节植物细胞内的K+平衡[10]。桃是全球人民都喜欢的水果,截至2012 年,我国桃总面积和总产量约占全国果品总面积和总产量的8.65 %和6.63 %,在果品生产中居第四位。2012 年中国桃产量占世界总产量的57.05 %。因其是二倍体(2n=16),并且基因组相对较小(225.7 Mb)已成为蔷薇科研究中模式植物[11]。2013 年桃基因组草图测序完成[11],为后续通过生物信息学挖掘、鉴定和分析桃基因组奠定了相关基础。本研究从桃基因组数据库出发,重点分析桃CBL家族基因的基因结构、染色体定位和蛋白进化关系及编码蛋白保守结构域,为克隆桃CBL基因提供有价值的信息,以期为进一步研究PpCBL 蛋白的生物学功能奠定相关理论基础。
1 材料与方法
1.1 材料
拟南芥CBL基因及蛋白序列下载自TAIR数据库(http://www.arabidopsis.org),桃的CBL基因及蛋白序列均下载自JGI数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Ppersica)。
1.2 方法
1.2.1 桃CBL家族基因基因结构分析 对鉴定出的桃CBLs 基因根据其预测编码区序列和基因组序列进行结构分析,利用GSDS(http://gsds.cbi.pku.edu.cn/) 在线进行[12]。
1.2.2 CBL家族基因系统进化树的构建及编码蛋白保守结构分析 通过Clustal X(2.0)软件对拟南芥、杨树和桃的CBL蛋白进行多序列比对分析,将序列比对结果使用MEGA 7.0软件,采用Neighborjoining法(Bootstrap值设为1000)生成CBL家族基因的系统进化树。应用在线软件MEME(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)和Pfam 数据库(http://pfam.sanger.ac.uk/search)对拟南芥和桃的CBL蛋白保守结构进行分析[13-14]。
1.2.3 桃CBL家族基因蛋白序列分析 利用在线软件PlantsPFeatureScan(http://plantsp.genomics.purdue.edu/index.html)对PpCBL蛋白序列进行N-豆蔻酰化位点分析。利用在线软件NetPhos 2. 0 Server(http://www.cbs.dtu.dk)对PpCBL蛋白质序列进行磷酸化位点预测。利用在线软件TMHMM 2.0 Server(http://www.cbs.dtu.dk/services/TMHMM/)对PpCBL蛋白序列进行跨膜区分析。
2 结果与分析
2.1 桃CBL家族基因的鉴定和命名
根据拟南芥中已经鉴别出来的10个CBL蛋白序列,分别利用phytozome 数据库提供的Blast 程序中进行BlastP,得到68个桃候选CBL基因。利用保守域在线预测软件Pfam 和MEME进行验证EF手型(PF13833、PF13499、PF13202、PF00036)结构域的存在,初步确定了8个桃CBL基因。其中桃CBL基因分布在第1、2、3、5、6染色体上,根据其在染色体上的定位分别命名为PpCBL1-8,每个成员的基因长度、氨基酸长度、染色体定位、外显子个数等详细信息见表1。桃CBL基因家族成员中PpCBL1和4含有9个外显子,其余成员均含有8个外显子(图1)。

表1 桃CBL家族基因的基本特征

图1 桃CBL家族基因结构分析Fig.1 Gene structure of the peach CBL gene family
2.2 桃CBL基因家族保守结构域分析
使用在线软件Pfam和MEME对PpCBL 蛋白保守结构域进行分析,桃CBL 蛋白包含4个保守EF手型结构域。其中EF1 和EF2 之间有23 个氨基酸,EF2 和EF3 之间有25 个氨基酸外,EF3 和EF4 之间除PpCBL4中间有36个氨基酸外,其余的成员之间有32 个氨基酸(图2)。

图2 桃CBL家族基因编码蛋白氨基酸序列的比对Fig.2 Alignment of the deduced amino acid sequences of peach CBL proteins
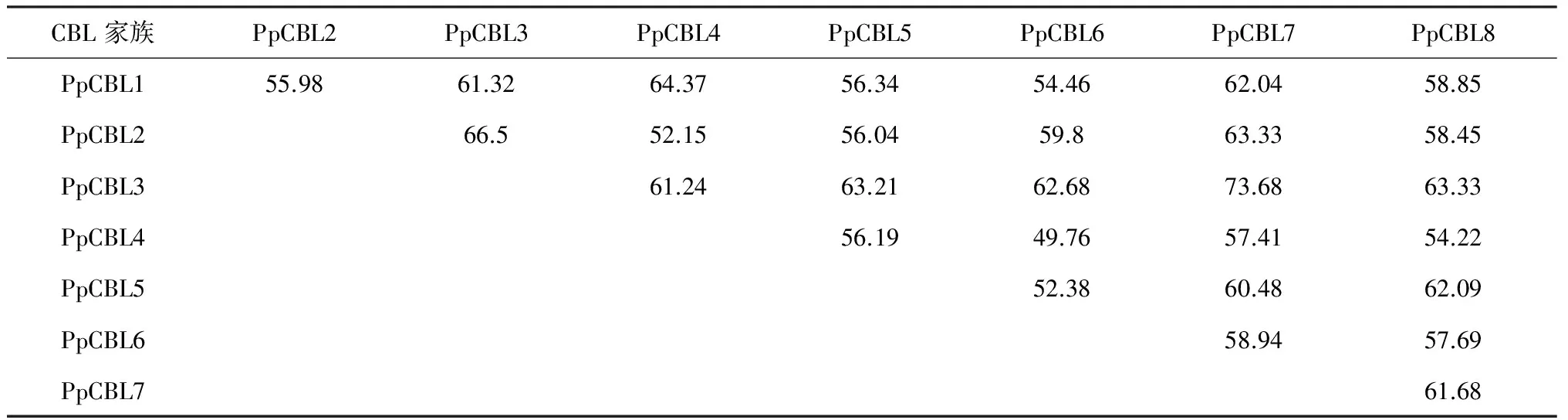
表2 桃基因组中CBL蛋白氨基酸相似性分析
2.3 桃CBL家族蛋白相似性和系统进化分析
利用进化树分析软件MEGA 7.0 对10个拟南芥CBL和8个桃CBL全蛋白序列进行进化分析。结果显示:这18个CBL分为4个亚家族,分别含有7、3、3和5个成员(图3)。8个桃CBL蛋白的氨基酸序列相似性在49.76 % ~ 73.68 %,其中PpCBL4 和PpCBL6氨基酸序列的相似性最低,仅为49.76 %,亚家族II中的成员PpCBL3 和PpCBL7氨基酸序列的相似性最高达到73.68 %(表2)。进化树分析结果显示,桃PpCBL2、PpCBL3、PpCBL6、PpCBL7聚类到第Ⅰ亚家族,PpCBL5聚类到第Ⅱ亚家族;PpCBL1、4聚类到第Ⅲ亚家族;PpCBL8聚类到第Ⅳ亚家族(图3)。
2.4 桃CBL基因家族蛋白序列分析
利用PlantsPFeatureScan对桃CBL蛋白进行N-豆蔻酰化位点预测,结果显示:PpCBL2、3、5均含有1个N-豆蔻酰化位点。利用NetPhos 2. 0 Server软件对PpCBL蛋白进行磷酸化位点预测,结果显示PpCBL 存在着大量的丝氨酸(Ser)、苏氨酸(Thr)及酪氨酸(Tyr)潜在磷酸化位点,这也是大多数Ser /Thr /Tyr 蛋白激酶所具有的特征,其中Ser位点为5~11个,Thr位点为1~4个,Tyr为点位0~4个,每个基因的磷酸化位点的数目和组成也不尽相同,最少的潜在磷酸化位点为6个,最多的为16个。利用TMHMM 2.0Server对PpCBL蛋白进行预测,结果显示:PpCBL1在13~35 aa处可能存在有一个跨膜区,其余PpCBL均未发现跨膜区(表3)。
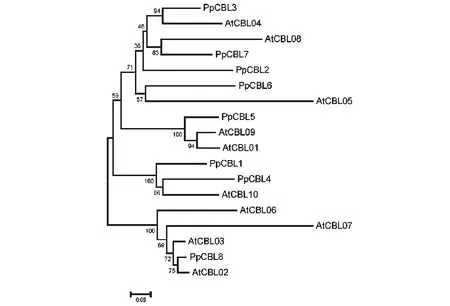
图3 拟南芥和桃的CBL蛋白进化树Fig.3 Phylogenetic tree for Arabidopsis and peach CBL proteins
3 讨 论
随着分子生物学技术的发展,CBL基因家族已经在拟南芥、水稻、杨树等中鉴定[15-16],但桃CBL基因家族尚未鉴定。本研究鉴定桃CBL基因家族含有8个成员,根据其在染色体上的定位分别命名为PpCBL1-8。已鉴定的CBL家族成员的数量在拟南芥、水稻、梨、桃、番茄、甜瓜、大白菜、高粱和苹果中存在差异。桃PpCBLs在基因组内呈现不均匀分布,这与在其他物种中的报道相一致[4]。桃PpCBLs 编码212~270 个氨基酸,但由于内含子和非翻译区差异导致PpCBLs 在桃基因组上的差异较大,在2.1~5.4 kb。EF手型结构域是CBL 蛋白的典型结构域。Kolukisaoglu等研究表明不同的CBL 蛋白的EF手型结构发生不同程度的变异,但在所有已知的CBL蛋白中,EF手型区域的个数以及它们之间的距离是保持不变的[5]。结构域分析表明,8个桃PpCBLs 都含有4个EF-hand结构域,其中EF3 和EF4 之间除PpCBL4中间有36个氨基酸外,其他结构域之间的距离非常保守,研究各个EF 手型结构发现,除PpCBL7含有13个氨基酸外,其他EF结构域均含有12 个氨基酸,随着PpCBL4和PpCBL7的分离以及生物学功能的进一步解析可能进一步的丰富人们对植物CBL生物学功能的认识。

表3 桃基因组中CBL 蛋白蛋白序列分析
系统进化分析表明,桃与拟南芥CBL蛋白在个亚家族之间均有成员分布。AtCBL01、AtCBL09和PpCBL5同源关系较近,拟南芥AtCBL01 能被高盐、干旱、低温和伤害等非生物胁迫强烈诱导,但不受外源ABA 胁迫的影响[2],而拟南芥AtCBL09 则能被ABA胁迫诱导表达[17];推测PpCBL5可能响应高盐、干旱、低温等胁迫诱导,但其具体响应何种胁迫还需要进一步研究其在植物体内功能。AtCBL10、PpCBL1和PpCBL4同源性较高,拟南芥AtCBL10 主要受盐胁迫诱导,且主要集中在地上部表达[18]。据此推测PpCBL1和PpCBL4可能响应高盐胁迫,但各自具体的生物学功能也需要进一步分析。
目前,植物CBLs的研究主要集中于拟南芥、水稻等少数几种模式植物响应高盐、低钾、低温、干旱等少数的几条途径[19]。Cuéllar等研究发现,葡萄VvCBL02-VvCIPK03和VvCBL01-VvCIPK04复合体通过激活其体内向的钾离子通道VvK1.2, 导致细胞内钾离子的积累促进了葡萄的果实成熟[20]。拟南芥AtCBL02和AtCBL03参与调控植株生长和种子大小[21]。这些结果表明植物CBLs具有促进果实成熟和调控种子大小的作用。运用反向遗传学及特殊突变体图位克隆技术,更加系统和深入的解析各PpCBL的结构及生物学功能,以期完善对植物CBL蛋白的结构与生物学功能的理解。
4 结 论
桃基因组中鉴定出8 个CBL家族基因,分布于桃的5 条染色体上。桃PpCBLs均含有4个保守的EF结构域,可分为4 个亚家族。PpCBLs 存在着大量的丝氨酸、苏氨酸及酪氨酸潜在磷酸化位点;PpCBL2、3和5均含有1个N-豆蔻酰化位点;PpCBL1含有1个跨膜区。
[1]Luan S. The CBL-CIPK network in plant calcium signaling[J]. Trends Plant Sci, 2009, 14(1): 37-42.
[2]Cheong Y, Kim K, Pandey G, et al. CBL1, a calcium sensor that differentially regulates salt, drought and cold responses inArabidopsis[J]. Plant Cell, 2003,15(8):1833-1845.
[3]Huang C, Ding S, Zhang H, et al. CIPK7 is involved in cold response by interacting with CBL1 inArabidopsisthaliana[J]. Plant Sci, 2011, 181(1): 57-64.
[4]许园园, 蔺 经, 李晓刚,等. 梨CBL基因家族全基因组序列的鉴定及非生物胁迫下的表达分析[J]. 中国农业科学, 2015, 48(4):735-747.
[5]Kolukisaoglu U, Weinl S, Blazevic D, et al. Calcium sensors and their interacting protein kinases: genomics of theArabidopsisand rice CBL-CIPK signaling networks[J]. Plant Physiol, 2004, 134(1):43-58.
[6]Halfter U, Ishitani M, Zhu J K. TheArabidopsisSOS2 protein kinase physically interacts with and is activated by the calcium-binding protein SOS3[J]. Proc Natl Acad Sci USA, 2000, 97(7):3735-3740.
[7]Kudla J, Batistic O, Hashimoto K. Calcium signals: the lead currency of plant information processing[J]. Plant Cell, 2010, 22(3):541-563.
[8]Ji H, Pardo J M, Batelli G, et al. The salt overly sensitive (SOS) pathway: established and emerging roles[J]. Mol Plant, 2013, 6(2): 275-286.
[9]Ye J, Zhang W, Guo Y.ArabidopsisSOS3 plays an important role in salt tolerance by mediating calcium-dependent microfilament reorganization[J]. Plant Cell Rep, 2013, 32(1): 139-148.
[10]Ren X,Qi G,Feng H,et al. Calcineurin B-like protein CBL10 directly interacts with AKT1 and modulates K+homeostasis inArabidopsis[J]. Plant J, 2013, 74(2): 258-266.
[11]Verde I, Abbott G A, Scalabrin S, et al. The high-quality draft genome of peach (Prunuspersica) identifies unique patterns of genetic diversity, domestication and genome evolution[J]. Nature Genet, 2013, 45: 487-494.
[12]郭安源, 朱其慧, 陈 新,等. GSDS:基因结构显示系统[J]. 遗传, 2007, 29(8):1023-1026.
[13]Bailey T, Boden M, Buske F, et al. MEME Suite: Tools for motif discovery and searching[J]. Nucleic Acids Res, 2009, 37(Suppl 2): 202-208.
[14]Finn R D, Coggill P, Eberhardt R Y, et al. The Pfam protein families database: towards a more sustainable future[J]. Nucleic Acids Res, 2016, 44(d1):D279-D285.
[15]Yu Y, Xia X, Yin W, et al. Comparative genomic analysis of CBL gene family inArabidopsisandPopulus[J]. Plant Growth Regul, 2007, 52(2):101-110.
[16]Gu Z, Ma B, Jiang Y, et al. Expression analysis of the calcineurin B-like gene family in rice (OryzasativaL.) under environmental stresses[J]. Gene, 2008, 415(1-2):1-12.
[17]Pandey G K, Cheong Y H, Kim KNGrant J J, et al. The calcium sensor calcineurin B-like 9 modulates abscisic acid sensitivity and biosynthesis inArabidopsis[J]. Plant Cell, 2004, 16(7):1912-1924.
[18]Quan R, Lin H, Mendoza I, et al. SCABP8/CBL10, a putative calcium sensor, interacts with the protein kinase SOS2 to protectArabidopsisshoots from salt stress[J]. Plant Cell, 2007, 19(4):1415-1431.
[19]汤湖斌, 闵康康, 徐玲玲,等. CBL-CIPKs信号系统的研究进展[J]. 中国细胞生物学学报, 2015, 37(1):100-105.
[20]Cuéllar T, Azeem F, Andrianteranagna M, et al. Potassium transport in developing fleshy fruits: the grapevine inward K+channel VvK1.2 is activated by CIPK-CBL complexes and induced in ripening berry flesh cells[J]. Plant J, 2013, 73(6):1006-1018.
[21]Eckert C, Offenborn J N, Heinz T, et al. The vacuolar calcium sensors CBL2 and CBL3 affect seed size and embryonic development inArabidopsisthaliana[J]. Plant J, 2014, 78(1):146-156.
(责任编辑 李 洁)
Genome-wideAnalysisandIdentificationofCBLGeneFamilyinPeach(PrunuspersicaL.)
ZHANG Jie-wei1, WU Ting2*, REN Fei1, XIE Hua1 **, WEI Jian-hua1 **
(1. Beijing Key Laboratory of Agricultural Genetic Resources and Biotechnology, Beijing Academy of Agriculture and Forestry Sciences, Beijing 100097, China; 2. University of the Chinese Academy of Sciences, Beijing 100101, China)
To study the function of peach calcineurin B-like proteins (CBLs) in Peach,on the basis of peach genome database and bioinformatics method, we obtained peach CBL family genes structure and their positions on chromosome and duplication information. CBL proteins were classified according to their phylogenetic relationship. The results showed that 8PpCBLgenes existed in the peach genome and located on 5 chromosomes. Multiple alignment and motif display results indicated that all PpCBL proteins contain four conserved domains (elongation factor hand). TMHMM 2.0Server showed that there is only one deduced transmembrane region in PpCBL1. PlantsPFeatureScan showed that there is only one N-terminal myristoylation in PpCBL2,3 and 5. NetPhos 2. 0 Server showed that there are many potential phosphorylation sites in PpCBLs. Phylogenetic analysis revealed that the PpCBL family could be divided into four groups. These results would provide the theoretical basis for functional studies to reveal the role of peach CBL gene family.
CBL;Prunuspersica; Gene family; Phylogeny analysis; Sequence character
1001-4829(2017)3-0511-06
10.16213/j.cnki.scjas.2017.3.005
S662.1
A
2016-04-29
北京市农林科学院青年科研基金项目(QNJJ2 01416);北京市科技计划(Z151100001015005);北京市农林科学院科技创新能力建设专项(KJCX20140202)
张杰伟(1982-),男,副研究员,研究方向为林木分子生物学,E-mail:jwzhang919@163.com,*为并列第一作者,吴婷(1988-),女,硕士研究生,研究方向为生物信息学,E-mail:wuting_work@163.com,**为通讯作者。
