食品香辛料和调味品中克罗诺杆菌的分离与鉴定
2017-10-19,,,,,
, , , ,,
(徐州医科大学 公共卫生学院,江苏徐州 221004)
食品香辛料和调味品中克罗诺杆菌的分离与鉴定
李远宏,姜华,焦阳,徐湾,邹小倩,裴尚飞
(徐州医科大学 公共卫生学院,江苏徐州 221004)
利用生理生化鉴定方法和分子生物学方法对从湖南、湖北、江苏和上海等地的8个地区采集的64份干制食品香辛料和调味品样品进行了克罗诺杆菌分离与鉴定,结果从19份样品中检出了克罗诺杆菌,污染率为29.7%,其中检出率较高的样品为白胡椒粉、花椒粉和红辣椒粉,污染率分别为62.5%、50.0%和33.3%。本研究从采样的7个地区的样品中检出了克罗诺杆菌,其中检出率最高的地区为湖南永州,其次为上海市和湖北赤壁,检出率分别为50.0%、37.5%和33.3%。利用基于fusA基因序列分析的方法对克罗诺杆菌分离株进行了种的水平的鉴定,结果将本研究分离得到的克罗诺杆菌鉴定为5个种,其中9株(47.4%)被鉴定为阪崎克罗诺杆菌(C.sakazakii),4株(21.0%)为苏黎世克罗诺杆菌(C.turicensis),3株(15.8%)为都柏林克罗诺杆菌(C.dublinensis),2株(10.5%)为丙二酸盐阳性克罗诺杆菌(C.malonaticus),1株(5.3%)为尤尼沃斯克罗诺杆菌(C.universalis)。研究结果表明市售干制食品香辛料和调味品中存在克罗诺杆菌的污染,应该加强对该类食品中克罗诺杆菌的流行病学监测。
克罗诺杆菌,食品香辛料和调味品,分离,鉴定
克罗诺杆菌(Cronobacterspp.),原称阪崎肠杆菌(Enterobactersakazakii),是一种重要的食源性条件致病菌,能引起新生儿及婴幼儿脑膜炎、菌血症和坏死性小肠结肠炎等疾病,死亡率高达20%~50%[1]。目前,该菌已经被划分为7个种,包括C.sakazakii、C.malonaticus、C.dublinensis、C.turicensis、C.universalis、C.muytjensii(穆汀斯克罗诺杆菌)和C.condiment(康迪蒙提克罗诺杆菌)[2-3]。研究发现能够导致婴幼儿感染的克罗诺杆菌主要为C.sakazakii、C.malonaticus和C.turicensis,尤其是C.sakazakii序列型ST4与婴幼儿与新生儿感染密切相关[4-5]。
长期以来,食品微生物学者对克罗诺杆菌监测的主要对象是婴幼儿配方食品,尤其是婴幼儿配方奶粉[6-7]。然而研究表明克罗诺杆菌在自然界中分布广泛,该菌不仅存在于婴幼儿配方食品中,而且在水果、蔬菜、谷类食品、水产品、豆类制品和肉制品等食品以及食品加工环境样本中也有检出[8-11]。由于克罗诺杆菌引起的感染具有发病急、致死率高、难以治愈等特点,而且该菌的传播途径和致病机制依然不是十分清楚[12]。因此,关于克罗诺杆菌预防与监控的研究已引起了国内外越来越多的重视,现已成为食品致病微生物研究领域的一个研究热点。
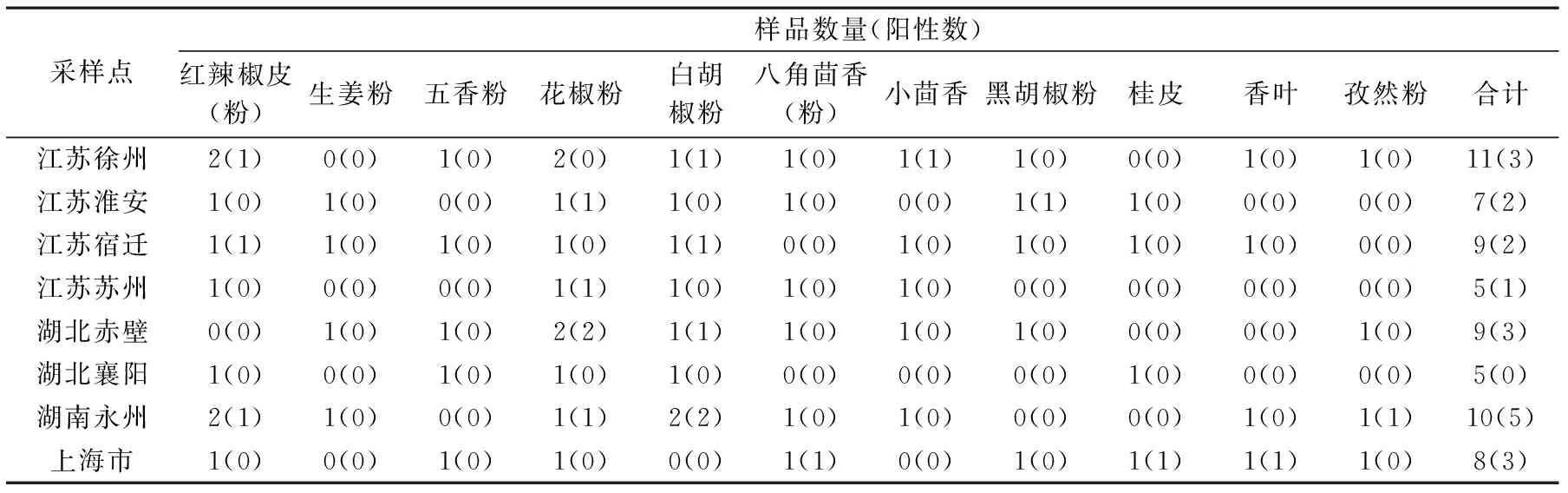
表1 不同地区样品采集情况及克罗诺杆菌检出情况Table 1 Details of sample collection and prevalence of Cronobacter spp. in different regions
作为一种重要的食源性条件致病菌,研究发现克罗诺杆菌不仅能感染新生儿及婴幼儿,而且还能导致成年人,特别是老年人及免疫力低下的人群致病,因此了解各类食品中克罗诺杆菌的污染情况显得十分必要。干制食品香辛料及调味品是食品加工过程中使用的重要的食品辅料,也在人们日常烹调中经常使用,污染了克罗诺杆菌的干制食品香辛料及调味品将导致巨大的食品安全隐患,威胁人类健康。本研究主要研究了干制食品香辛料和调味品中克罗诺杆菌的污染情况,并利用生理生化鉴定、特异性PCR鉴定和基于保守基因fusA序列分析的方法对克罗诺杆菌分离株进行了分类与鉴定,将有助于人们进一步阐明该菌在食品中的分布情况、流行特征,为该菌的监控、暴发流行的检测及污染源追溯提供科学依据。
1 材料与方法
1.1材料与仪器
克罗诺杆菌参考菌株C.sakazakiiCICC 21560和C.muytjensiiCICC 21563 由南京农业大学食品科技学院酶工程实验室馈赠。缓冲蛋白胨水肉汤(buffered peptone water,BPW)、改良月桂基硫酸盐胰蛋白胨肉汤(modified lauryl sulfate tryptose broth,mLST)、万古霉素溶液(vancomycin,Vm)和胰蛋白胨大豆琼脂(trypticase soy agar,TSA) 北京陆桥技术有限责任公司;克罗诺杆菌显色培养基HiCromeTMCronobacterspp. agar(配方:酪蛋白15.0 g/L,大豆蛋白胨5.0 g/L,显色混合物10.17 g/L,氯化钠5.0 g/L,琼脂15.0 g/L) sigma公司;克罗诺杆菌生化鉴定管 杭州微生物试剂有限公司;细菌总基因组DNA提取试剂盒 美国Omega公司。2×Taq Master Mix 南京诺唯赞生物科技有限公司。DL2000 DNA Marker 大连Takara公司。引物 由上海生工生物工程有限公司合成。
SW-CJ-2FD型超净工作台 苏州安泰;DNP9162型电热恒温培养箱 上海精宏;5418型小型高速离心机 德国Eppendorf;Veriti 96 PCR仪 美国ABI;DYY-8C型凝胶电泳仪 北京六一;GelDoc XR Biorad型凝胶成像系统 美国BIO-RAD。
1.2实验方法
1.2.1 样品采集 从2014年9月至2015年6月,本次实验从湖南、湖北、上海和江苏等地的8个地区的超市采集了64份干制食品香辛料和调味品样品,包括花椒粉10份,红辣椒粉(皮)9份,白胡椒粉8份,八角茴香(粉)6份,五香粉、小茴香和黑胡椒粉各5份,桂皮、香叶、生姜粉和孜然粉各4份(表1)。样品运回实验室后立即进行检测。
1.2.2 克罗诺杆菌分离与鉴定 参考GB 4789.40-2010《食品卫生微生物学检验 阪崎肠杆菌检验》对克罗诺杆菌进行分离鉴定[13]。具体操作方法如下:在无菌操作条件下称取10.0 g干制食品香辛料或调味品样品加入装有90 mL BPW的锥形瓶中,置于37 ℃恒温培养箱中静置培养18 h。然后移取1 mL培养液转接于10 mL MLST-vm肉汤,44 ℃培养24 h。混匀mLST-Vm肉汤培养物,分别取增菌培养液划线接种于克罗诺杆菌显色培养平板,37 ℃培养24 h。挑取1~3个蓝绿色疑似菌落划线接种于TSA平板上,于25 ℃培养 48 h。将在显色平板上呈蓝绿色且能够在TSA平板上产生黄色素的疑似菌株初步鉴定为疑似克罗诺杆菌分离株。
1.2.3 生理生化实验 利用克罗诺杆菌生化鉴定管对疑似克罗诺杆菌分离株进行生化鉴定。主要的鉴定指标有:黄色素产生、氧化酶实验、L-赖氨酸脱羧酶、L-鸟氨酸脱羧酶、L-精氨酸双水解酶、柠檬酸水解和糖发酵实验(D-山梨醇、L-鼠李糖、D-蔗糖、D-蜜二糖、苦杏仁甙)。
1.2.4 基因组DNA提取 将经分离纯化的疑似菌株接种于LB培养基,采用基因组DNA提取试剂盒(Omega,USA)提取细菌基因组总DNA,具体操作流程按操作说明进行。
1.2.5 特异性PCR鉴定 以克罗诺杆菌分离株总DNA为模板,利用正向引物ESSF:5′-GGA TTTAACCGTGAACTTTTCC-3′;反向引物:ESSR 5′-CGCCAGCGATGTTAGAAGA-3′进行PCR扩增OmpA基因特异性片段[14],PCR产物长度为469 bp。PCR反应体系:2×Taq Master Mix 25 μL,引物ESSF和ESSR各2 μL,DNA模板1 μL,加ddH2O补足至50 μL。PCR反应条件:95 ℃预变性5 min;95 ℃变性 30 s,55 ℃退火45 s,72 ℃延伸50 s,35个循环;72 ℃ 延伸7 min。取反应产物5 μL上样,1%琼脂糖凝胶电泳,于凝胶成像系统下观察并拍照记录结果。
1.2.6fusA基因扩增及测序 以克罗诺杆菌分离株的总DNA为模板,利用引物fusA-SF:5′-GAAACCGTATGGCGTCAG-3′和fusA-SR:5′-AGAACCGAAGTGCAGACG-3′进行PCR扩增,获得长度为2114 bp的DNA片段[15]。PCR反应体系:2×Taq Master Mix 25 μL,引物fusA-SF和fusA-SR各2 μL,DNA模板1 μL,加ddH2O补足至50 μL。PCR反应条件:96 ℃预变性1 min;96 ℃变性 1 min,58 ℃退火1 min,72 ℃延伸1 min,30个循环;72 ℃ 延伸5 min。fusA基因的测序由南京金思瑞生物科技有限公司完成。测序引物序列为fusA-sf:5′-GCTGG ATGCGGTAATTGA-3′,fusA-sr:5′-CCCATACCAG CGATGATG-3′[15]。测序后获得fusA等位基因片段长度为438 bp。
1.2.7fusA基因序列分析及系统发育分析 将fusA基因序列在NCBI中进行Blast比对分析,对疑似菌株分离株进行初步鉴定。同时利用克罗诺杆菌MLST分型数据库(http://pubmlst.org/cronobacter/)分析分离株的fusA序列获得其等位基因编号,然后再利用基于fusA基因的系统发育分析方法对克罗诺杆菌分离株进行聚类分析[16]。方法如下:在MLST分型数据库中查询克罗诺杆菌参考菌株C.sakazakiiATCC 29544、C.malonaticusLMG23826、C.dublinensisLMG23823、C.turicensisLMG23827、C.muytjensiiATCC 51329、C.universalisNCTC9529和C.condimentiLMG26250的fusA基因的等位基因号,然后依据等位基因号下载参考菌株对应的fusA基因序列,再通过 Clustalx 2.0 软件对参考菌株及分离株的fusA基因序列进行多重比较,利用MEGA 4.0 软件以邻近法(Neighbour-joining)构建系统发育树分析克罗诺杆菌的种属关系。
2 结果与分析
2.1克罗诺杆菌检出情况
依据GB 4789.40-2010《食品卫生微生物学检验 阪崎肠杆菌检验》对干制食品香辛料和调味品中克罗诺杆菌进行分离。将分离株在显色平板上呈蓝绿色且在TSA平板上培养48 h后产生黄色素的菌落初步确定为疑似克罗诺杆菌。利用克罗诺杆菌生化鉴定管对可疑分离株进行了生理生化鉴定,结果表明本研究分离的克罗诺杆菌分离株的生理生化反应特征均符合国家标准GB 4789.40-2010中对阪崎肠杆菌的描述(表2)。研究结果表明本研究从19份干制香辛料和调味品样品中检出了克罗诺杆菌,总检出率为29.7%,其中污染率较高的样品为白胡椒粉、花椒粉和红辣椒皮,污染率分别达到62.5%、50.0%和33.3%,而生姜粉和五香粉样品中未检出克罗诺杆菌(表3)。在研究采样的8地区中,除了来自湖北襄阳的样本未检出克罗诺杆菌之外,其他地区的样品均检出了克罗诺杆菌,其中污染率最高的地区为湖南永州(5/10,50%),其次为上海市(3/8,37.5%)和湖北赤壁(3/9,33.3%)(表1)。

表2 克罗诺杆菌分离株生理生化反应特征Table 2 Physiological and biochemicalcharacteristics of Cronobacter isolates

表3 不同干制食品香辛料和调味品中克罗诺杆菌的污染率Table 3 Prevalence of Cronobacter isolates recoveredfrom dried spices and condiments
注:a,基于fusA基因等位基因号及系统发育分析结果;b,ND:未检出。
2.2可疑克罗诺杆菌分离株PCR验证
利用克罗诺杆菌属特异性引物分别对19株疑似分离株进行PCR检测。经琼脂糖凝胶电泳检测后,结果显示19株分离株均在500 bp附近扩增出了与预期片段大小(469 bp)相符的目的条带,因而证实了PCR检测结果均呈阳性,进一步证实了全部分离株均为Cronobacterspp.(图1)。
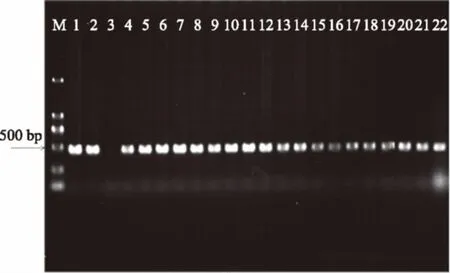
图1 疑似阳性菌株特异性 PCR 检测结果Fig.1 Detection of Cronobacter isolates from dried spicesand condiments by genus-specific PCR注:M为DL2000 DNA ladder marker;1为阳性对照(C. sakazakii CICC 21560);2为阳性对照(C. muytjensii CICC 21563);3为阴性对照;4~22:疑似克罗诺杆菌分离株。
2.3fusA基因序列分析
将测序获得的fusA基因序列在NCBI数据库中进行BLAST同源性比对分析,结果表明19株可疑分离株均为Cronobacterspp.,其中9株分离株(XZCRO002、XZCRO003、XZCRO004、XZCRO006、XZCRO007、XZCRO012、XZCRO039、XZCRO040和XZCRO042)被初步鉴定为C.sakazakii;4株分离株(XZCRO008、XZCRO009、XZCRO010和XZCRO0015)被初步鉴定为C.turicensis;3株分离株(XZCRO001、XZCRO005和XZCRO013)被初步鉴定为C.dublinensis;2株(XZCRO011和XZCRO041)被初步鉴定为C.malonaticus;1株(XZCRO0014)被初步鉴定为C.universalis(表4)。
2.4分离株系统发育分析
注:+,阳性。
利用克罗诺杆菌MLST数据库对克罗诺杆菌分离株的fusA基因序列进行比对分析,获得了克罗诺杆菌分离株fusA基因的等位基因号(图2),结果表明将9株(47.4%)分离株鉴定为C.sakazakii,4株(21.0%)为C.turicensis,3株(15.8%)为C.dublinensis,2株(10.5%)为C.malonaticus,1株(5.3%)为C.universalis。利用MEGA 4.0软件对19株克罗诺杆菌分离株和7株参考菌株的fusA基因序列进行系统发育分析,其系统发育树如图2所示。由图2可知,19株克罗诺杆菌分离株的fusA基因被分为5个簇,其中XZCRO002、XZCRO003、XZCRO004、XZCRO006、XZCRO007、XZCRO012、XZCRO039、XZCRO040和XZCRO042与参考株C.sakazakiiATCC29544位于一簇,被鉴定为C.sakazakii。同理,XZCRO011和XZCRO041被鉴定为C.malonaticus;XZCRO008、XZCRO009、XZCRO010和XZCRO0015被鉴定为C.turicensis;XZCRO0014被鉴定为C.universalis;XZCRO001、XZCRO005、XZCRO013被鉴定为C.dublinensis。分离株系统发育分析结果与Genbank数据库中BLAST比对分析结果一致。

图2 基于fusA基因序列的克罗诺杆菌分离株的系统发育树Fig.2 Phylogenetic tree inferred by Neighbor-joining method based on the fusA gene sequences
3 结论与讨论
本研究通过生理生化实验、特异性PCR鉴定实验、fusA序列分析及系统发育分析等方法对食品香辛料和调味品中克罗诺杆菌的污染情况进行了调查分析,结果从19份样品中分离出了可疑克罗诺杆菌分离株,总检出率为29.7%。除了生姜粉和五香粉样品中未检出克罗诺杆菌之外,其他样品均在不同程度上检出了克罗诺杆菌,污染率约为16.7%~62.5%。不同样品间克罗诺杆菌污染状况的差异可能是由于这些样品的生产加工环境中的卫生状况差异所导致的。Reich和Lou等[17-18]的研究结果表明食品加工环境中存在的克罗诺杆菌能够转移至食品中,是导致食品污染克罗诺杆菌的重要原因之一。在本研究采样的8个地区中有7个地区的样品检出了克罗诺杆菌污染,其中克罗诺杆菌污染率最高的地区为湖南永州(50%),其次为上海市(37.5%)和湖北赤壁(33.3%)。不同地区样品中的克罗诺杆菌的污染状况有所差异,其原因可能是由于本研究采集的样品数量不足而导致的误差或其加工环境中卫生状况差异等因素造成的。
将分离株鉴定到种的水平有助于人们尽快找出污染源、切断传播途径,从而制定更加迅速有效的控制措施以预防病原菌的扩散。基于保守基因序列分析的方法是一种常规的细菌分类学鉴定方法,目前已经建立一些基于16S rDNA、OmpA、rpoB和fusA基因的序列分析方法,然而由于克罗诺杆菌属内不同种间菌株亲缘关系非常接近,研究表明基于16S rDNA、OmpA和rpoB的序列分析方法无法准确将其区分到种的水平[19-21]。相比较而言,基于fusA基因的序列分析方法更加准确可靠,已被用于克罗诺杆菌不同菌株的种鉴定[16,22-24]。通过基于fusA序列分析及系统发育分析方法进一步证实19株可疑分离株鉴定为Cronobacterspp.,其中9株被鉴定为C.sakazakii,4株为C.turicensis,3株为C.dublinensis,2株为C.malonaticus,1株为C.universalis。研究结果表明市售食品香辛料和调味品中存在克罗诺杆菌污染,C.sakazakii是其优势菌种,该结论与国内外其他食品中克罗诺杆菌污染情况一致[22,25-26]。本研究结果表明干制食品香辛料和调味品是克罗诺杆菌的一个重要污染源,应该加强对该类食品中克罗诺杆菌污染状况的调查和流行病学监测以进一步了解克罗诺杆菌在食品及环境中的存在状况。
[1]Bowen B,Braden R. InvasiveEnterobactersakazakiidisease in infants[J]. Emerging Infectious Diseases,2006,12(8):1185-1189.
[2]Iversen C,Lenhner A,Munllane N,et al. The taxonomy ofEnterobactersakazakii:proposal of a new genusCronobactergen. nov and descriptions ofCronobactersakazakiicomb. novCronobactersakazakiisubsp sakazakii,comb. nov.,Cronobactersakazakiisubsp malonaticus subsp nov.,Cronobacterturicensissp nov.,Cronobactermuytjensiisp nov.,Cronobacterdublinensissp nov andCronobactergenomospeciesI[J]. BMC Evolutionary Biology,2007,7:64.
[3]Joseph S,Cetinkaya E,Drahovska H,et al.Cronobactercondimentisp.nov. isolated from spiced meat andCronobacteruniversalissp. nov. a novel species designation forCronobctersp. genomospecies 1,recovered from a leg infection,water and food ingredients[J]. International Journal of Systematic and Evolutionary Microbiology,2012,62:1277-1283.
[4]Joseph S,Forsythe J. Predominance ofCronobactersakazakiisequence type 4 in neonatal infections[J]. Emerging Infectious Diseases,2011,17:1713-1715.
[5]Alsonosi A,Hariri S,Kajsík M,et al. The speciation and genotyping ofCronobacterisolates from hospitalised patients[J]. European Journal of Clinical Microbiology & Infectious Diseases,2015,34(10):1-10.
[6]Caubilla-Barron J,Hurrell E,Townsend S,et al. Genotypic and phenotypic analysis ofEnterobactersakazakiistrains from an outbreak resulting in fatalities in a neonatal intensive care unit in France[J]. Journal of Clinical Microbiology,2007,45(12):3979-3985.
[7]Lai KK.Enterobactersakazakiiinfections among neonates,infants,children,and adults:Case reports and a review of the literature[J]. Medicine,2001,80(2):113-122.
[8]徐湾,姜华,张逸飞,等. 我国克罗诺杆菌污染现状与预防控制措施研究进展[J].中国病原生物学杂志,2015,10(9):848-851.
[9]李远宏,张逸飞,张庆成,等. 谷类食品中克罗诺杆菌的分离与鉴定[J].食品工业科技,2016,37(15):154-158,164.
[10]张德福,赵禹宗,张明,等. 添加扩增内标的PCR方法快速检测食品中的阪崎克罗诺杆菌[J].食品工业科技,2017,38(2):49-52,58.
[11]Sonbol H,Joseph S,Mcauley CM,et al. Multilocus sequence typing ofCronobacterspp. from powdered infant formula and milk powder production factories[J]. International Dairy Journal,2013,30(1):1-7.
[12]曾彪,综述,马晓晨,等. 克罗诺杆菌病的流行特征和防控策略研究[J].卫生研究,2015,44(6):1032-1035.
[13]GB 4789.40-2010.食品微生物学检验 阪崎肠杆菌检验[S]. 北京:中国标准出版社,2010.
[14]Nair M,Venkitanarayanan S. Cloning and sequencing of the ompA gene ofEnterobactersakazakiiand development of an ompA-targeted PCR for rapid detection ofEnterobactersakazakiiin infant formula[J].Applied and Environmental Microbiology,2006,72:2539-2546.
[15]Baldwin A,Loughin M,Caubilla-Barron J,et al. Multilocus sequence typing ofCronobactersakazakiiandCronobactermalonaticusreveals s
Table clonal structures with clinical significance which do not correlate with biotypes[J]. BMC microbiology,2009,9:223.
[16]Fei P,Man C,Lou B,et al. Genotyping and Source Tracking ofCronobactersakazakiiandC.malonaticusIsolates from powdered infant formula and an infant formula production factory in China[J]. Applied and Environmental Microbiology,2015,81(16):5430-5439.
[17]Reich F,König R,von Wiese W,et al. Prevalence ofCronobacterspp. in a powdered infant formula processing environment[J]. International Journal of Food Microbiology,2010,15:140(2-3):214-217.
[18]Lou X,Si G,Yu H,et al. Possible reservoir and routes of transmission ofCronobacter(Enterobacter sakazakii)via wheat flour[J]. Food control,2014,43:258-262.
[19]迟涛,方景泉,满朝新,等.克罗诺杆菌分子鉴定方法的比较[J].中国乳品工业,2015,43(7):10-12.
[20]黄启红,蔡大川,张志军,等.食品中克罗诺杆菌的分离和鉴定[J].食品研究和开发,2015,37(9):192-194.
[21]Li Y H,Cao L,Zhao J F,et al. Use of rpoB gene sequence analysis for phylogenetic identification ofCronobacterspecies[J]. Journal of Microbiological Methods,2012,88:31 6-318.
[22]Cui J,Du X,Wei R,et al. Multilocus sequence typing analysis ofCronobacterspp. isolated from China[J]. Arch Microbiology,2015,197:665-672.
[23]张丽丽.婴幼儿配方食品中克罗诺杆菌的分离鉴定及分型[J].食品研究和开发,2016,37(12):166-168.
[24]Brandão ML,Umeda NS,Jackson,et al. Isolation,molecular and phenotypic characterization,and antibiotic susceptibility ofCronobacterspp. from Brazilian retail foods[J]. Food Microbiology,2017,63:129-138.
[25]Vojkovska H,Karpiskova R,Orieskova M,et al. Characterization ofCronobacterspp. isolated from food of plant origin and environmental samples collected from farms and from supermarkets in the Czech Republic[J]. International Journal of Food Microbiology,2016,217:130-136.

IsolationandidentificationofCronobacterspp.fromdriedspicesandcondiments
LIYuan-hong,JIANGHua,JIAOYang,XUWan,ZOUXiao-qian,PEIShang-fei
(School of Public Health,Xuzhou Medical University,Xuzhou 221004,China)
A total of sixty-four dried spices and condiments,collected from retail markets in 8 cities of Hunan,Hubei and Jiangsu province,as well as Shanghai,were analyzed for the presence ofCronobacterspp. In total,19(29.7%)of 64 dried spices and condiments were positive forCronobacterspp.,with the highest percentage(62.5%)coming from white pepper powder,followed by Chinese prickly ash powder(50.0%)and red chilli powder(33.3%). Dried spices and condiments samples from 7 cities were found to be contaminated withCronobacterspp.,with highest detection rate for Yongzhou,Hunan(50%),followed by Shanghai(37.5%)and Chibi,Hubei(33.3%). Based on the fusA gene sequence analysis,Cronobacterisolates were divided into 5Cronobacterspecies. Most of theCronobacterisolates were identified as C. sakazakii(9/19,47.4%),followed byC.turicensis(4/19,21.0%),C.dublinensis(3/19,15.8%)andC.malonaticus(2/19,10.5%),while the remaining isolate(1/19,5.3%)was identified asC.universalis. The results indicated that commercially available dried spices and condiments were possible reservoir ofCronobacterspp.,and therefore the epidemiological surveillance ofCronobacterspp. should be strengthened in dried spices and condiments.
Cronobacterspp.;spices and condiments;isolation;identification
TS207.4
A
1002-0306(2017)19-0125-06
10.13386/j.issn1002-0306.2017.19.024
2017-03-06
李远宏(1984-),男,博士,讲师,研究方向:食品微生物与生物技术,E-mail:lyh@xhzmu.edu.cn。
国家自然科学基金青年科学基金项目(31401595);徐州医学院校级科研课题资助项目(D2014002,2014KJ01)。
