可产木聚糖酶的蕙兰根内生细菌的筛选与鉴定
2017-10-19马春玲李思含赵东芳李广硕马睿忆孙磊
马春玲,李思含,赵东芳,李广硕,马睿忆,孙磊
(河北大学 生命科学学院 河北省微生物多样性研究与应用重点实验室,河北 保定 071002)
可产木聚糖酶的蕙兰根内生细菌的筛选与鉴定
马春玲,李思含,赵东芳,李广硕,马睿忆,孙磊
(河北大学 生命科学学院 河北省微生物多样性研究与应用重点实验室,河北 保定 071002)
为了获得可产木聚糖酶的功能菌,通过刚果红染色法对592株蕙兰(Cymbidiumfaberi)根内生细菌进行初筛,DNS法对初筛获得的菌株进行复筛,并通过形态学、生理生化特性、(G+C)的物质的量分数及16S rRNA基因序列分析对菌株进行初步鉴定.结果筛选出31株可产木聚糖酶的菌株,占筛选总菌株数量的5.23%,其中7株产酶活性较强;复筛结果显示菌株6hRe76产木聚糖酶活力最高,其发酵液木聚糖酶酶活为57.15 U/mL,初步鉴定该菌株为克里布所类芽胞杆菌(Paenibacilluskribbensis).该研究为木聚糖酶的生产提供了潜在的新资源.
木聚糖酶;类芽孢杆菌;内生细菌
木聚糖是半纤维素的主要成分,也是自然界中仅次于纤维素的第2大类可再生的生物物质,可以被降解为有用的产物[1].木聚糖主要存在于植物细胞的细胞壁和中胶层[2],其基本分子结构是D-木糖残基通过β-1,4-糖苷键相连构成线性分子主链,主链上带有乙酰基、阿拉伯糖残基[3].木聚糖完全水解需要多种酶相互结合才能完成,起主要作用的包括内切-β-1,4-木聚糖酶(EC3.2.1.8)和β-木糖苷酶(EC 3.2.1.37)等[4].木聚糖酶随机水解β-1,4-糖苷键产生低聚木糖和少量木糖,然后β-木糖苷酶进一步水解低聚木糖释放木糖残基[5].从半纤维素中获取低聚木糖、木糖的有效途径就是通过木聚糖酶的水解,木聚糖酶已经在饲料、造纸及食品[6]等行业得到了广泛应用.因此对木聚糖酶的研究对于可再生资源的有效利用具有重要意义.
许多微生物包括细菌、真菌和酵母都能产生不同类型的木聚糖酶.植物内生细菌指能在健康植物组织内栖居而对植物无实质性危害的细菌,其中一部分还能使植物受益,并与植物建立和谐联合关系的微生物,植物内生细菌具有多种重要的功能,如促生、生防、促进植物修复等[7],同时内生细菌还能分泌抗菌、抗病毒、抗癌及酶等多种活性物质[8].植物内生细菌作为活性物质的重要来源日益受到人们的重视.
本研究主要以植物内生细菌为材料筛选获得可产木聚糖酶的功能菌株,以期获得可产木聚糖酶的新资源.
1 材料和方法
1.1 材料
1.1.1 内生细菌
分离自蕙兰的592株根内生细菌.
1.1.2 培养基
1)菌种纯化培养基(LB培养基):蛋白胨10.0 g,酵母粉5.0 g,NaCl 10.0 g,琼脂15.0 g,蒸馏水1 000 mL,pH 7.0,121 ℃灭菌30 min.
2)初筛培养基[9]:玉米芯木聚糖10.0 g,酵母粉0.3 g,(NH4)2SO45.0 g,K2HPO42.0 g,MgSO4·7H2O 0.3 g,NaCl 5.0 g,琼脂15.0 g,蒸馏水1 000 mL,pH 7.4,121 ℃灭菌30 min.
3)产酶培养基[10]:麸皮40.0 g,蛋白胨5.0 g,K2HPO45.0 g,蒸馏水1 000 mL,pH 7.0,121 ℃灭菌30 min.
1.2 实验方法
1.2.1 菌种的初筛
将实验室保藏的592株蕙兰根内生细菌活化后点接至以玉米芯木聚糖为碳源的初筛培养基平板上,30 ℃培养2 d,然后用刚果红对初筛平板染色,观察并记录透明圈的大小[11].
1.2.2 菌种的复筛
将初筛得到的菌株接种于LB培养基中培养24 h获得种子液,再以10%的接种量接种于液体产酶培养基中,30 ℃,180 r/min,摇床振荡培养24 h,然后吸取发酵液, 4 000 r/min离心10 min,其上清液即为粗酶液,用于测定木聚糖酶活力.
1.2.3 木聚糖酶活力测定
木聚糖酶活力测定采用DNS法[12].木聚糖酶活力单位:以木糖为标准,每min产生1 μmoL木糖所需酶量定义为1个酶活力单位(U).计算公式为U=N×G/(V×t×m),式中N为酶液稀释倍数,G为酶解溶液木糖含量,V为加酶量,t为酶解时间,m为木聚糖质量分数.
1.2.4 菌株的鉴定
1)产酶菌株的16S rRNA基因序列分析采用细菌16S rRNA基因通用引物27f[13](5-AGAGTTTGATCCTGGCTCAG-3)和1492r[14](5-GGTTACCTTGTTACG ACTT-3)对其进行扩增.反应条件:94 ℃预变性5 min;94 ℃变性1 min,54 ℃退火45 s,72 ℃延伸45 s,30个循环;72 ℃延伸7 min.PCR产物经切胶回收后由北京宝锐通生物技术有限公司测序,测序结果提交EzTaxon数据库(http://www.ezbiocloud.net/eztaxon/identify)进行序列比对,选取同源性较高的序列用邻接法(neighbor-joining method)[15]进行分析,采用Mega7.0构建系统发育树.
2)菌落形态观察和生理生化鉴定:在平板培养基上观察菌落形态、颜色,对培养24 h的菌株进行革兰氏染色和鞭毛染色,并对该菌进行接触酶实验、氧化酶实验、VP实验及甲基红实验[16],同时进行Biolog实验.
3)产酶菌株(G+C)的物质的量分数测定:采用HPLC法[17]测定菌株基因组(G+C)的物质的量分数.
2 结果
2.1 初筛结果
利用刚果红对592株蕙兰根内生细菌染色,比较透明圈大小,进行木聚糖酶产生菌的筛选,结果见表1.共筛选出可产木聚糖酶的功能菌31株,占筛选总菌株数量的5.23%;其中7株为功能较强菌株,占功能菌株数量的22.6%.根据初筛结果,选定菌株6hRe76、6hRe1、N58-1、T58-1、ahR29、hRa12、ahR12进行复筛.

表1 部分木聚糖酶产生菌株的水解圈直径与菌落直径的比值Tab.1 Hydrolysis circle diameter to colony diameter ratio of xylanase-producing strains
2.2 复筛结果
利用DNS法对选定的7株细菌进行发酵产酶测定,实验结果如表2所示.其中木聚糖酶活力最高的是培养1 d后的菌株6hRe76的发酵液,其酶活性为57.15 U/mL.此菌株为筛选到的可产木聚糖酶的功能菌株,对其进行鉴定.
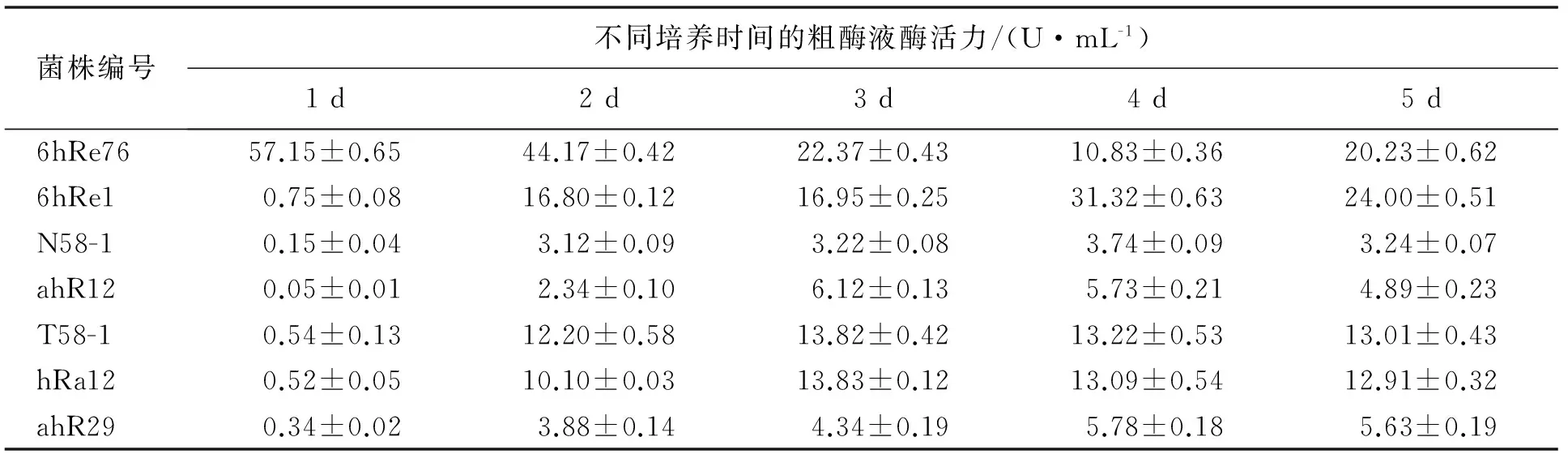
表2 复筛菌株木聚糖酶活力测定结果Tab.2 Xylanase activity determination results of secondary screening strains
2.3 菌株鉴定结果
2.3.1 菌株6hRe76的16S rRNA基因序列分析
将菌株6hRe76的16S rRNA基因序列提交GenBank,序列登录号为(MF359544).在 EZTaxon 数据库中作比对分析,并构建系统发育树(图1).如图1所示,菌株6hRe76与克里布所类芽胞杆菌(Paenibacilluskribbensis)(AF391123)在同一个分支中,序列相似性为99%.根据此结果,菌株6hRe76可初步鉴定归属于类芽孢杆菌属.

括号内数字为GenBank 登录号;分支数表示1 000次Bootstrap 重抽样分析的支持百分比;图例0.005为遗传距离.图1 邻接法构建菌株6hRe76及其相关菌株16S rRNA基因的系统发育树Fig.1 Phylogenetic tree based on the 16S rRNA gene sequences of strain 6hRe76 and related strains by neighbor-joining method
2.3.2 菌落形态特征观察
菌株6hRe76在LB培养基上培养2 d,菌落表面光滑,半透明,微黄色,边缘不规则.革兰氏染色可变(图2),周生鞭毛(图3).
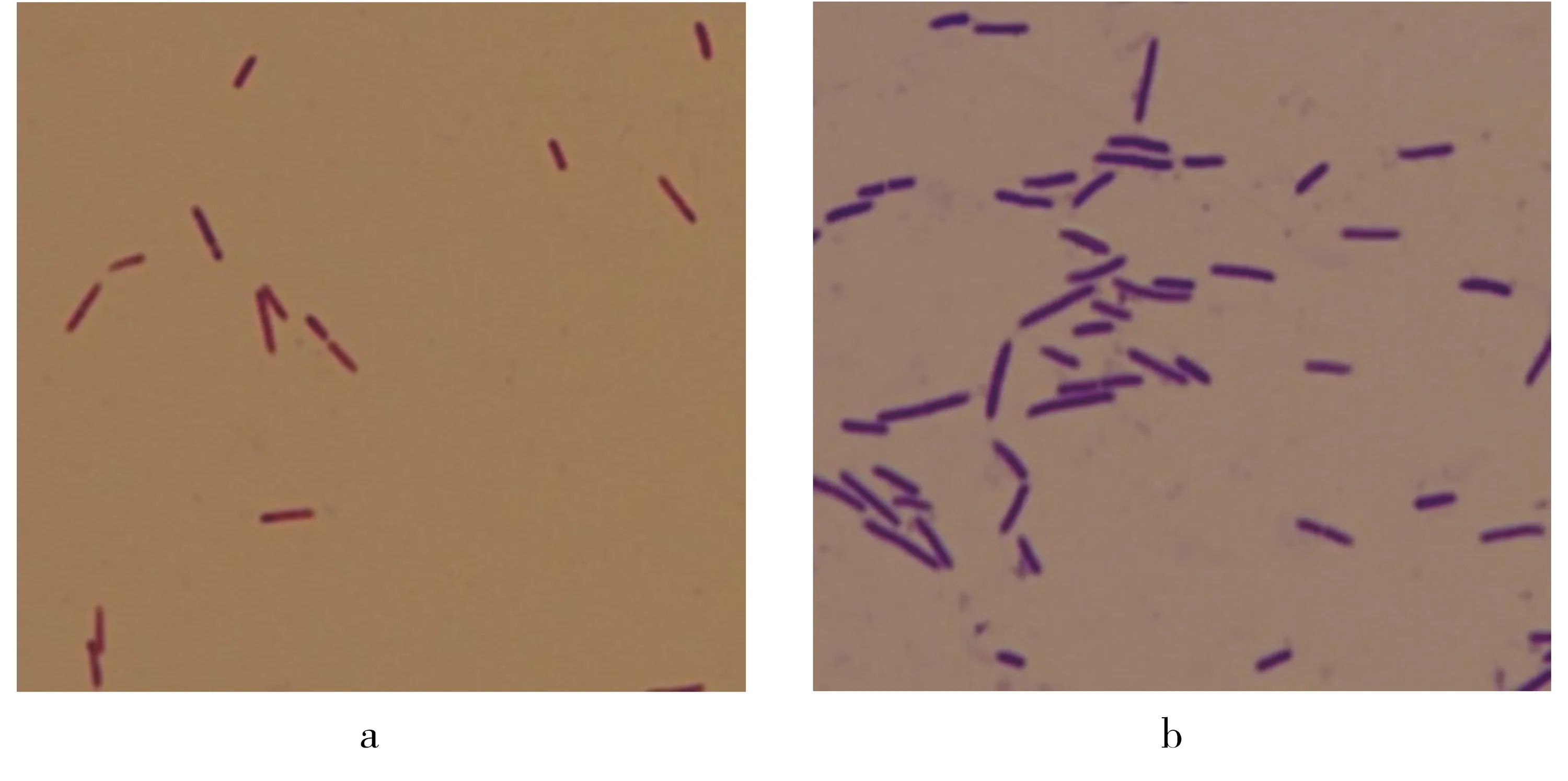
a.革兰氏阴性(培养2 d的染色结果);b.革兰氏阳性(培养7 d的染色结果).图2 革兰氏染色结果(1 000×)Fig.2 Result of Gram staining(1 000×)

图3 鞭毛染色结果(1 000×)Fig.3 Result of flagellum staining(1 000×)
2.3.3 菌株生理生化特征测定
菌株6hRe76呈氧化酶阴性,接触酶阳性,甲基红反应阴性,VP实验结果阳性.该菌与克里布所类芽孢杆菌的菌落形态特征、革兰氏染色特征及接触酶等生理生化特征一致.
Biolog测定结果显示,菌株6hRe76可利用的糖类包括D-麦芽糖、D-海藻糖、D-纤维二糖等,氨基酸的种类包括D-天冬氨酸、L-天冬氨酸、L-丝氨酸,羧酸及酯和脂肪酸包括氨基乙酰-L-脯氨酸、D-半乳糖醛酸等.在pH5、pH6、10 g/L NaCl条件下可生长,能够利用乳酸钠、盐酸胍、亚碲酸钾.
2.3.4 菌株6hRe76的(G+C)的物质的量分数
通过HPLC法测得菌株6hRe76的(G+C)的物质的量分数为47.10%,类芽孢杆菌属的(G+C)的物质的量分数为45%~54%.
根据形态特征、生理生化特征、(G+C)的物质的量分数及16S rRNA基因系统发育分析,菌株6hRe76被鉴定为克里布所类芽胞杆菌(Paenibacilluskribbensis).
3 讨论
木聚糖酶在自然界广泛存在,现已发现几十个属的100多个种的微生物可产木聚糖酶.目前报道较多的高产木聚糖酶的是真菌[18]和芽孢杆菌属的细菌[19].真菌所产木聚糖酶的活性高于细菌,但是细菌所产木聚糖酶的耐碱性及耐热性要优于真菌[20].类芽孢杆菌属目前至少包含202个种(LPSN,http://www.bacterio.net/),类芽孢杆菌属的细菌具有多种生物活性,也已发现多种类芽胞杆菌具有产木聚糖酶的能力,如Paenibacilluscurdlanolyticus[21]、Paenibacillusterrae[22]、Paenibacilluscampinasensis[23]、Paenibacillusfavisporus[24]等.本研究从592株植物内生细菌中筛选到1株可产木聚糖酶的菌株6hRe76,经鉴定为克里布所类芽胞杆菌,为木聚糖酶产生菌提供了新资源.
[1] COLLINS T,GERDAY C,FELLER G.Xylanases,xylanases families and extremophilic xylanases[J].FEMS Microbiol Rev,2005,29: 3-23.DOI:10.1016/j.femsre.2004.06.005.
[2] SALEH A M,RASHAD R A.Partial purification and characterization of xylanases fromAspergillusawamoriandAspergillusphoenicis[J].African Journal of Microbiology Research,2012,6(23):5025-5034.
[3] SQUINA F M,MORT A J,DECKER S R,et al.Xylan decomposition byAspergillusclavatusendo-xylanase[J].Protein Expres Purif,2009,68(1): 65-71.DOI: 10.1016/j.pep.2009.06.014.
[4] FANG T J,LIAO B C,LEE S C.Enhanced production of xylanase byAspergilluscarneusM34 in solid-state fermentation with agricultural waste using statistical approach[J].New Biotechnol,2010,27(1): 25-32.DOI:10.1016/j.nbt.2009.09.008.
[5] FANG H Y,CHANG S M,HSIEH M C,et al.Production,optimization growth conditions and properties of the xylanase fromAspergilluscarneusM34[J].J Mol Catal B-Enzyme,2007,49(16): 36-42.DOI:10.1016/j.molcatb.2007.08.002.
[6] 柏文琴, 王钦宏,马延和,等.嗜热和嗜碱木聚糖酶研究进展[J].生物工程学报,2014,30(6): 828-837.DOI: 10.13345/j.cjb.140172.
BAI W Q,WANG Q H,MA Y H,et al.Progress in the thermophilic and alkalophilic xylanases[J].Chin J Biotech,2014,30(6): 828-837.DOI: 10.13345/j.cjb.140172.
[7] RYAN R P,GERMAINE K,FRANKS A,et al.Bacterial endophytes: recent developments and applications[J].FEMS Microbiol Lett,2008,278:1-9.DOI:10.1111/j.1574-6968.2007.00918.x.
[8] STAIEK A,WOERDENBAG H J,KAYSER O.Endophytes: exploiting biodiversity for the improvement of natural product-based drug discovery[J].J Plant Interact,2008,3:75-93.DOI: 10.1080/17429140801886293.
[9] 王雪鹏.高产木聚糖酶芽孢杆菌的选育及产酶的研究[D].青岛:中国海洋大学,2004.
WANG X P.Studies on the breeding of xylanase overproduction strain and the conditions of xylanase production fromBacillussp.[D]Qingdao:Ocean University of China,2004.
[10] 包怡红,李雪龙,杨传平.类芽孢杆菌木聚糖酶产生菌株的筛选及其产酶条件优化[J].东北林业大学学报,2008,36(9): 70-73.DOI:10.13759/j.cnki.dlxb.2008.09.006.
BAO Y H,LI X L,YANG C P.Screening experiment of xylanase-producingPanibacillussp.and optimization of production culture conditions[J].Journal of Northeast Forestry University,2008,36(9):70-73.DOI:10.13759/j.cnki.dlxb.2008.09.006.
[11] GOWDHAMAN D,MANASWINI V S,JAYANTHI V,et al.A comparison of plate assay methods for detecting extracellular cellulase and xylanase activity[J].International Journal of Biological Macromolecules,2014,64: 90-98.
[12] BAILEY M J,BIELY P,POUTANEN K.Interlaboratory testing of methods for assay of xylanase activity[J].J Biotechnol,1992,23(3): 257-270.
[13] EDWARDS U,ROGALL T,BLACKER H,et al.Isolation and direct complete nucleotide determination of entire genes: characterization of a gene coding for 16S ribosomal RNA[J].Nucleic Acids Research,1989,17:7843-7853.DOI:10.1093/nar/17.19.7843.
[14] LANE D J.Nucleic acid techniques in bacterial systematics[M].New York:John Wiley &Sons,1991:115-175.
[15] SAITOU N,NEI M.The neighbor-joining method: a new method for reconstructing phylogenetic trees[J].Molecular Biology Evolution,1987,4: 406-425.
[16] 东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社.2001:62-63,349-398.
[17] MESBAH M,PREMACHANDRAN U,WHITMAN W B.Precise measurement of G+C content of deoxyribonucleic acid by high performance liquid chromatography[J].International Journal of Systematic and Evolutionary Microbiology,1989,39: 159-167.DOI:10.1099/00207713-39-2-159.
[18] POLIZELIM,RIZZATTIS,MONTI R,et al.Xylanases from fungi: properties and industrial applications[J].Appl Microbiol Biotechnol,2005,67: 577-591.DOI: 10.1007/s00253-005-1904-7.
[19] 刘巍,范树田,李心治.地衣芽抱杆菌H-1的鉴定及其产木聚糖酶性质的研究[J].工业微生物,1996,26(4): 11-15.
[20] 孙晓霞,谢响明,吴玉英,等.白色链霉菌产木聚糖酶规律及其耐热耐碱性的初步研究[J].北京林业大学学报,2005,27(3):72-75.
SUN X X,XIE X M,WU Y Y,et al.Introduction of xylanase fromStrepoymycesalbusand analysis of hermostability and alkali-tolerance[J].Journal of Beijing Forestry University,2005,27(3):72-75.
[21] SERMSATHANASWADI J,BARAMEE S,TACHAAPAIKOON C,et al.The family 22 carbohydrate -binding module of bifunctional xylanase/β-glucanase Xyn10E fromPaenibacilluscurdlanolyticusB-6 has an important role in lignocellulose degradation[J].Enzyme Microb Technol,2017,96: 75-84.DOI: 10.1016/j.enzmictec.
[22] KIM D R,LIM H K,LEE K I,et al.Identification of a novel cellulose-binding domain within the endo-β-1,4-xylanase KRICT PX-3 fromPaenibacillusterraeHPL-003[J].Enzyme Microb Technol,2016,11 (93-94):166-173.
[23] LIU Y,HUANG L,LI W,et al.Studies on properties of the xylanbinding domain and linker sequence of xylanase XynG11 fromPaenibacilluscampinasensisG11[J].J Ind Microbiol Biotechnol,2015,42(12):1591-1599.DOI: 10.1007/s10295-015-1698-2.
[24] PADILHA I Q,VALENZUELA S V,GRISI T C,et al.A glucuronoxylan-specific xylanase from a newPaenibacillusfavisporusstrain isolated from tropical soil of Brazil[J].Int Microbiol,2014,17(3):175-184.DOI: 10.2436/20.1501.01.220.
(责任编辑:赵藏赏)
Screeningandidentificationofxylanase-producingendophyticbacteriaofCymbidiumfaberiroots
MAChunling,LISihan,ZHAODongfang,LIGuangshuo,MARuiyi,SUNLei
(1.Key Laboratory of Microbial Diversity Research and Application of Hebei Province,College of Life Sciences,Hebei University,Baoding 071002,China)
In order to obtain the xylanase-producing functional strains,592 endophytic bacteria fromCymbidiumfaberiroots were screened by using the method of Kongo red dye according to the size of hydrolyzed circle.The xylanase activity of strains screened was re-screened by DNS.The xylanase-producing strain was identified based on its morphological,physiological and biochemical characteristics,mole fraction of (G+C)and 16S rRNA gene sequence analysis.The results showed that 31 xylanase-producing strains were obtained,which account for 5.23% of the total strains.Seven strains with high xylanase yields were screened through DNS method.Strain 6hRe76 with the highest enzyme activity was obtained,of which the activity of xylanase was 57.15 U/ mL.The strain 6hRe76 was identified asPaenibacilluskribbensis.The research provided the new source for xylanase production.
xylanase;Paenibacillus;endophytic bacteria
Q939.13
A
1000-1565(2017)05-0518-05
10.3969/j.issn.1000-1565.2017.05.011
2017-06-22
国家自然科学基金资助项目(31100002);河北省生物工程重点学科经费资助项目;河北大学大学生创新创业项目(2016063)
马春玲(1992—),女,河北邢台人,河北大学硕士研究生,主要从事微生物资源开发研究.E-mail: 799169587@qq.com
孙磊(1971—),女,河北唐山人,河北大学教授,博士,主要从事环境微生物学研究.E-mail: sunlei1018@126.com
