R基团搜索技术用于硼酸三肽蛋白酶体 抑制剂的分子设计
2017-10-18仝建波李园园江国艳李康楠
仝建波, 李园园, 江国艳, 李康楠
(陕西科技大学化学与化工学院,陕西西安 710021)
近年研究显示,蛋白酶体抑制剂可以通过调控细胞周期,抑制核转录活化因子,从而达到抗肿瘤的目的[1 - 2]。硼酸肽类化合物是一种临床上常用的蛋白酶体抑制剂,用于治疗血液系统恶性肿瘤细胞。该类抑制剂的主要代表为治疗多发性骨髓瘤等多种肿瘤的药物硼替佐米(Velcade)[3]。作为第二代共价类蛋白酶体抑制剂的硼酸肽类,其代谢稳定,且具较高的抑制活性和选择特异性,毒副作用相对较低[4]。
三维定量构效关系(3D-QSAR)是用统计分析工具和理论计算研究一系列分子三维结构与其生物活性之间的关系,是药物研究中的重要理论和常用手段[5]。考虑到配体与受体作用时的空间取向,在不了解受体结构的情况下,将分子周围的力场分布与活性定量地联系起来,明确给出对现有分子进行修饰的建议[6]。3D-QSAR中具代表性的方法比较分子场分析方法(CoMFA)[7]与比较分子相似性指数分析(CoMSIA)[8]已被人们用于硼酸肽类的构效关系研究[9]。本文采用Topomer CoMFA[10]对硼酸三肽蛋白酶体抑制剂进行QSAR研究,在此基础上进行基于R基团的Topomer search[11]研究,这为硼酸肽蛋白酶体抑制剂分子的设计及新型抗肿瘤药物的研发提供了参考。
1 原理与方法
1.1 数据集准备
本文所选的44个Tyropeptin硼酸三肽类蛋白酶体抑制剂来自文献[2],分为训练集(25个化合物)和测试集(19个化合物,用*标注)。活性标度为pIc50,其分子结构及活性数据见表1。
1.2 Topomer CoMFA

表1 44个Tyropeptin硼酸三肽蛋白酶抑制剂分子骨架及活性Table 1 The struetures and biological activities data of 44 boronic acid derivatives of tyropetin inhibitors
*:test set.
采用SYBYL-X2.0软件包构建分子的三维结构,加载Gasetiger-Hcukle电荷,采用Powell共轭梯度法和Tripos力场,优化次数设定为1 000次,其余参数设置均由SYBYL-X2.0软件包默认。以23号分子为模板,将分子切成一个核心基团和三个R基团,分割后运行Topomer CoMFA方法计算,切割方式如图1所示。
1.3 Topomer Search
Topomer Search与Topomer CoMFA连用对化合物库进行筛选,可高效地用于完整分子的搜索以及先导化合物的优化等。Topomer Search是采用构象的独立性及Topomer的相似性进行完整分子、侧链或骨架的筛选[19]。Topomer Search结果中主要包含两项:Topomer距离和R基团的活性贡献值[20],其可从数据库中筛选并测出相应R基团贡献值。通常在分子相似程度允许范围内,贡献值越大的基团越有考虑价值。本文以建立的Topomer CoMFA模型进行Topomer search研究,搜索ZINC(2012)[21]数据库中的Fragment-like类,包含34 292个分子。设置Topomer距离为185,其余参数均选取SYBYL默认。用活性最高3c号样本对选中分子进行过滤,选取比模板分子R1、R2和R3贡献值大的R片段。
2 结果与讨论
2.1 Topomer CoMFA模型评估

图2 Tyropeptin硼酸三肽蛋白酶抑制剂分子训练集和预测集样本活性的线性回归图Fig 2 Linear regressions between observed and predicted pIc50 of boronic acid derivatives of tyropetin inhibitors

以34号分子为模板绘制各R基团静电场和立体场的Topomer CoMFA模型等势图如图3所示。由图3(a)可看出,在R1残基周围存在大量绿色色块图,表示在此区域宜引入大体积的基团。化合物8(pIc50=7.229),其R1基团整体相对34(pIc50=6.824)号化合物较大,活性也比后者高。R1远端三氟甲基附近有黄色色块图,比较化合物34(pIc50=6.824)、31(pIc50=7.022)、26(pIc50=7.056),将三氟甲基依次替换成溴和氯时,随着取代基体积变小活性升高。由图3(b)可以看出,三氟甲基邻位附近有蓝色体积图,比较34(pIc50=6.824)和35(pIc50=6.959),强负电性基团氢原子用正电性基团三氟甲基代替之后,化合物活性明显提高。3(c)中,甲氧基周围有黄色色块,对比化合物5(pIc50=7.658)和3b(pIc50=8.357),甲氧基被氢替换,体积变小,化合物活性提高了很多。由图3(d)可以知,在切割点远端的甲氧基周围出现了红色等势图,甲氧基附近有蓝色色块图。化合物2(pIc50=7.721)和3b(pIc50=8.357),苯基增加了甲氧基,负电性增加,活性升高。由图3(f)可知静电场对化合物的活性影响较小。观察图3(e)可知,取代基残基周围有大片黄色区域,表明此处替换的取代基残基体积变小,就有利于化合物活性升高。对比化合物15(pIc50=6.638)与53(pIc50=6.959),其活性有明显提高。但是此处的基团体积也不是越小就越好,比较化合物52(pIc50=6.585)和化合物53(pIc50=6.959),在其他基团相同的情况下,取代基残基体积减小,化合物活性反而出现了降低。

图3 Topomer CoMFA模型Fig.3 Contour map of Topomer CoMFA model
2.2 基于Topomer Search的分子设计
通过Topomer search对ZINC数据库中的分子式筛选,得到高贡献值的R1、R2、R3基团分别为1 544、813和417个。综合Topomer距离和取代基贡献值,以3c号分子为模板进行过滤,分别选取较高贡献值的R1和R3基团7个和5个,由于未筛选到高于0.19的R2基团,所以本文保持R2基团不变。依次替换R1和R3,设计得到35个新分子,对其进行优化,优化方式同44个样本分子。其Topomer距离、R1及R3贡献值列于表2。其中,20个化合物的pIc50均大于模板分子3c(pIc50=8.59)。由表可以看出,随着R1体积变大,活性也呈现出升高的规律。比较化合物22(pIc50=8.81)和23(pIc50=8.75),随着R3取代基体积增大化合物活性减小。但是观察后5个化合物,其活性低于模板分子,可能是由于R1集团的体积与模板分子体积相较小所致,但所设计的所有分子均与前面所分析的Topomer CoMFA等势图分析一致。

表2 新化合物的预测活性以及R基团的Topomer距离及其贡献值Table 2 Structures of new designed molecules,Topomer distances,contribution values of R-groups and predicted pIc50 by Topomer CoMFA
(续表2)

No.StructureTopomer distanceR1 controbution valueR3 controbution valueR1R3Topomer CoMFATopomer searchTopomer CoMFATopomer searchPredicted pIc50by Topomer CoMFA06146341.071.030-0.05-0.0408.68107146341.071.030-0.11-0.0408.625081461021.071.030-0.10-0.1008.622091461191.071.030-0.04-0.1108.696101461511.071.030-0.04-0.0508.69011143340.351.020-0.04-0.0407.98112143340.351.020-0.04-0.0407.986131431020.351.020-0.10-0.1007.912141431190.351.020-0.11-0.107.918151431510.351.020-0.05-0.0507.972161691511.241.180-0.05-0.0508.855
(续表2)
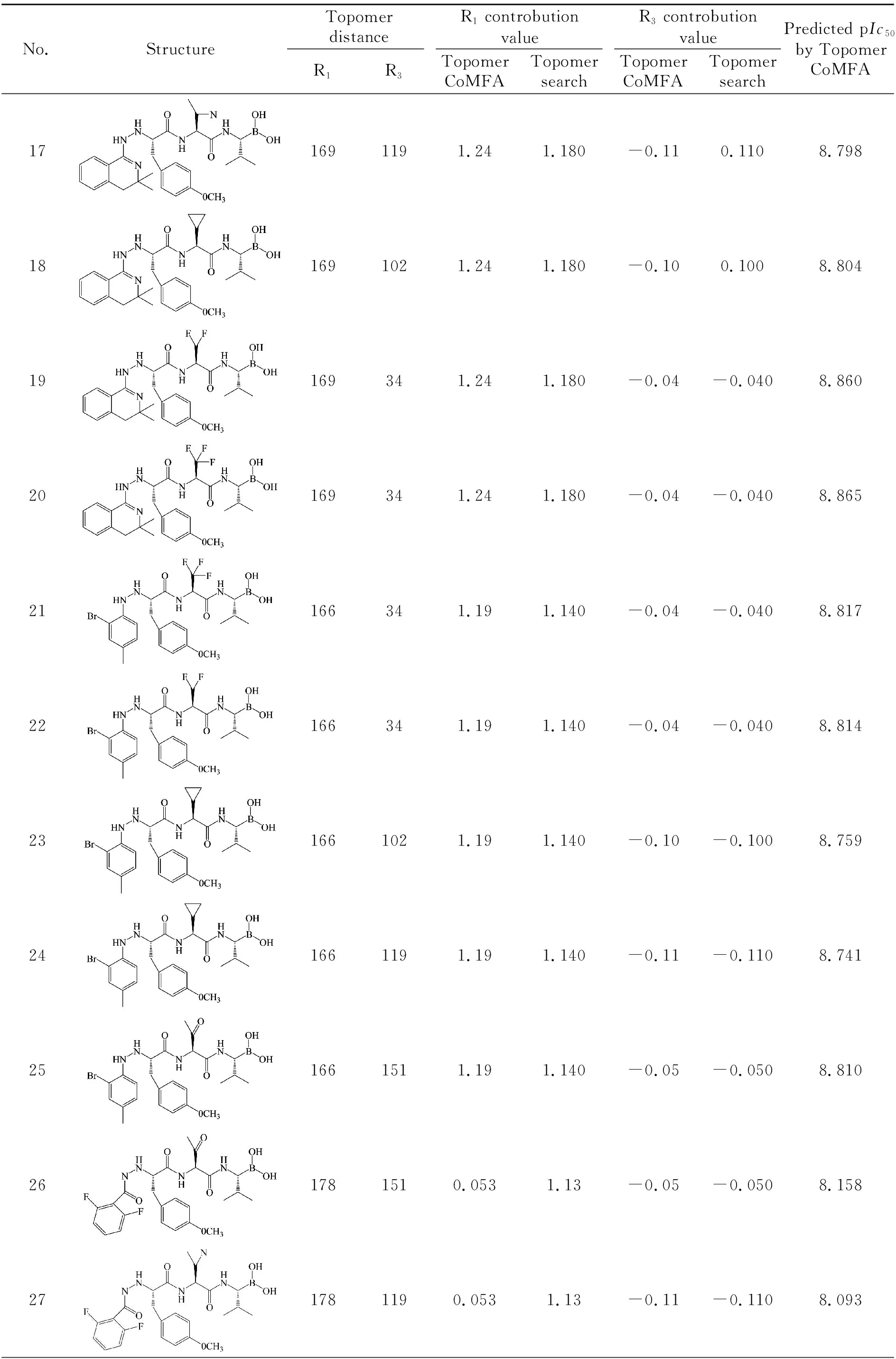
No.StructureTopomer distanceR1 controbution valueR3 controbution valueR1R3Topomer CoMFATopomer searchTopomer CoMFATopomer searchPredicted pIc50by Topomer CoMFA171691191.241.180-0.110.1108.798181691021.241.180-0.100.1008.80419169341.241.180-0.04-0.0408.86020169341.241.180-0.04-0.0408.86521166341.191.140-0.04-0.0408.81722166341.191.140-0.04-0.0408.814231661021.191.140-0.10-0.1008.759241661191.191.140-0.11-0.1108.741251661511.191.140-0.05-0.0508.810261781510.0531.13-0.05-0.0508.158271781190.0531.13-0.11-0.1108.093
(续表2)

No.StructureTopomer distanceR1 controbution valueR3 controbution valueR1R3Topomer CoMFATopomer searchTopomer CoMFATopomer searchPredicted pIc50by Topomer CoMFA281781020.0531.13-0.10-0.1008.09429178340.0531.13-0.04-0.0408.16430178340.0531.13-0.04-0.0408.16631179340.691.12-0.04-0.0408.31332179340.691.12-0.04-0.0408.317331791020.691.12-0.10-0.1008.254341791190.691.12-0.11-0.1108.243351791510.691.12-0.05-0.0508.316
3 结论
本文采用Topomer CoMFA对44个Tyropeptin硼酸三肽类蛋白酶体抑制剂进行了三维定量构效关系分析,并利用所建模型对19个测试集抑制剂进行了活性预测。采用Topomer Search技术在ZINC数据库进行R基团的筛选,对分子中的两个R基团进行替换,所设计出的20个分子理论抑制活性值比模板分子有较大提高,Topomer CoMFA等势图分析结果与模板分子一致,结果表明采用基于Topomer CoMFA模型的R基团搜索技术进行抑制剂的分子设计为抗肿瘤新药设计提供新的候选物。
