大豆主要过敏原β-伴大豆球蛋白β亚基的抗原表位分析及分段克隆
2017-10-16,,,
, ,,
(河南工业大学,粮油食品学院,河南郑州 450001)
大豆主要过敏原β-伴大豆球蛋白β亚基的抗原表位分析及分段克隆
皮江一,席俊*,贺梦雪,李爽
(河南工业大学,粮油食品学院,河南郑州450001)
利用生物信息学软件SWISS-MODEL对大豆主要过敏原β-伴大豆球蛋白β亚基建立出三级结构模型,并根据DiscoTope 2.0服务器预测出该模型的构象表位相关区段,采用分段克隆方式扩增目的基因的3个片段区域,利用PCR技术扩增目的基因全长及3个互相重叠的片段(A、B、C)将其连接至pMD18-T载体,并转化到大肠杆菌感受态JM109。经蓝白斑筛选,以及对重组质粒进行PCR和双酶切验证,测序结果与设计的基因序列一致,成功得到了目的基因片段的阳性克隆子,为β-伴大豆球蛋白β亚基分段表达及抗原区域位置的筛选与鉴定提供参考。
β-伴大豆球蛋白,β亚基,生物信息学,抗原位点,克隆
Abstract:The model of tertiary ofβ-conglycinin beta-subunit was constructed by the bioinformation tool SWISS-MODEL,and the conformational epitopes were predicted by DiscoTope 2.0 server. The overlapping fragments ofβ-conglycinin beta-subunit genes were amplified by PCR and inserted into pMD-18T vector,then preserved into competent cell ofEscherichiacoliJM109. The recombinant plasmids were evaluated by blue-white selection,PCR and double enzyme digestion,and DNA sequence analysis showed that the cloned sequence was the very fragment designed. The successful cloning of the genes can provide a reference for the study of the expresstion ofβ-conglycinin beta-subunit and screening of antigenic epitopes.
Keywords:β-conglycinin;beta-subunit;bioinformation;antigenic epitopes;clone
大豆素有“田中之肉”、“绿色牛乳”之称,是优质蛋白的重要来源。但同时也是八大食物过敏原之一[1-2],调查显示世界上约有4%的人口对食物过敏,其中就有25%的人口对大豆过敏[3]。β-伴大豆球蛋白是7S球蛋白的主要成分,约占7S组分的85%,是重要的大豆贮藏蛋白,占大豆总球蛋白含量的30%以上[4]。且β-伴大豆球蛋白比大豆球蛋白表现出更强的抗原性[5]。β-伴大豆球蛋白分子质量约180 kDa,是由α′(72 kDa)、α(68 kDa)和β(52 kDa)三种亚基组成三聚体复合物,其含量分别为45%、35%和20%[6]。其中β亚基由416个氨基酸组成[7],不含人体必需氨基酸,且经研究表明β亚基同样具有免疫学活性[8],是大豆蛋白的主要过敏原之一。
导致食物过敏的是能引起免疫反应的食物抗原分子。按照抗原蛋白质内部结构的不同可将其分为线型和构象型表位。目前,对抗原表位的研究多为B细胞构象型表位,由于蛋白质空间结构的复杂性,所以预测存在一定的局限。许多学者对此采取了多种研究方法进行探讨,包括酶解产生多肽片段进行筛选方法、根据氨基酸序列设计重叠多肽并用单克隆抗体进行筛选的方法、噬菌体展示库肽技术以及利用NMR分析和X射线等手段对抗原位点进行分析与研究等等[9-10]。但是这些方法工作量大,存在操作繁琐及效率低等弊端。β-伴大豆球蛋白β亚基作为重要的大豆过敏原,目前国内研究主要集中在β-伴大豆球蛋白检测分析、致敏性鉴定及脱敏技术研究等方面。Maruyama等[11]研究分析了重组大豆和天然大豆β-伴大豆球蛋白β亚基的晶体结构,卫会君等[12]通过筛选289份具有多样种子外观性状的大豆种质,发掘出具有β亚基缺失性状的大豆。对于大豆过敏原表位的定位研究主要采用的是重叠多肽技术。而噬菌体展示技术具有操作简单、成本低等优势。杨廷亚等[13]应用噬菌体展示技术筛选出血症病毒抗原模拟表位。但是应用噬菌体展示技术对大豆过敏原表位定位的研究还未见报道。生物信息学与噬菌体展示技术作为一种新的过敏原表位预测和定位技术,具有操作简单、成本相对较低等优势。在分析食物过敏抗原表位方面具有广阔的应用前景[14]。

表1 β-伴大豆球蛋白β亚基基因分段引物Table 1 Primer for β-conglycinin beta-subunit gene segments
本研究利用生物信息学技术分析方法,运用SWISS-MODEL服务器及DiscoTope 2.0服务器构建β-伴大豆球蛋白β亚基蛋白三维晶体结构以及对抗原表位进行预测。利用PCR技术分别扩增β亚基基因片段,构建克隆载体,为研究利用噬菌体展示β-伴大豆球蛋白β亚基的分段表达及抗原表位的筛选提供理论依据。
1 材料与方法
1.1 材料与仪器
β-伴大豆球蛋白β亚基基因(1320 bp) 由上海生工生物工程股份有限公司合成并保存于PUC57载体质粒中;大肠杆菌JM109感受态细胞、pMD18-T克隆质粒及限制性内切酶EcoRⅠ、HindⅢ 大连宝生物公司;质粒小提试剂盒及琼脂糖凝胶DNA回收试剂盒 康为世纪生物公司;X-Gal、IPTG及Ex-Taq DNA酶、基因分段引物 上海生工生物工程股份有限公司。
PCR仪 Eppendorf公司;琼脂糖凝胶电泳仪、凝胶成像系统 上海天能科技有限公司。
1.2 实验方法
1.2.1β-伴大豆球蛋白β亚基氨基酸序列 通过NCBI GenBank数据库检索到β-伴大豆球蛋白β亚基(LOC547465)的氨基酸序列:

1.2.2β-伴大豆球蛋白β亚基三级结构构建 登录PDB(Protein Data Bank,蛋白质资料库)数据库检索β-伴大豆球蛋白β亚基蛋白质,作为三级结构模板。登录SWISS-MODEL服务器,选择Alignment Mode模式,分别上传β-伴大豆球蛋白β亚基的氨基酸序列,对β-伴大豆球蛋白β亚基的三级结构进行构建。
1.2.3β-伴大豆球蛋白β亚基的B细胞构象表位预测 根据β-伴大豆球蛋白β亚基三级结构模型,在DiscoTope 2.0服务器中预测其构象表位,并找出预测抗原区域主要位置。
1.2.4 引物设计 根据蛋白质三级结构及B细胞构象表位预测区段位置,本实验将β-伴大豆球蛋白β亚基基因分为3段:A(73~600 bp),B(478~930 bp),C(808~1317 bp)。相邻片段之间设置约120 bp重叠区域。引物设计利用primer 5.0软件,依据结构:5′-保护碱基+酶切位点+引物配对区-3′,上游酶切位点设计为EcoRⅠ,下游为HindⅢ,且引物溶解温度设计为58 ℃左右。具体见表1。
1.2.5β-伴大豆球蛋白β亚基基因的克隆及载体构建
1.2.5.1β-伴大豆球蛋白β亚基全长及片段A,B,C的扩增 使用PCR仪扩增目的基因片段,反应体系见表2。
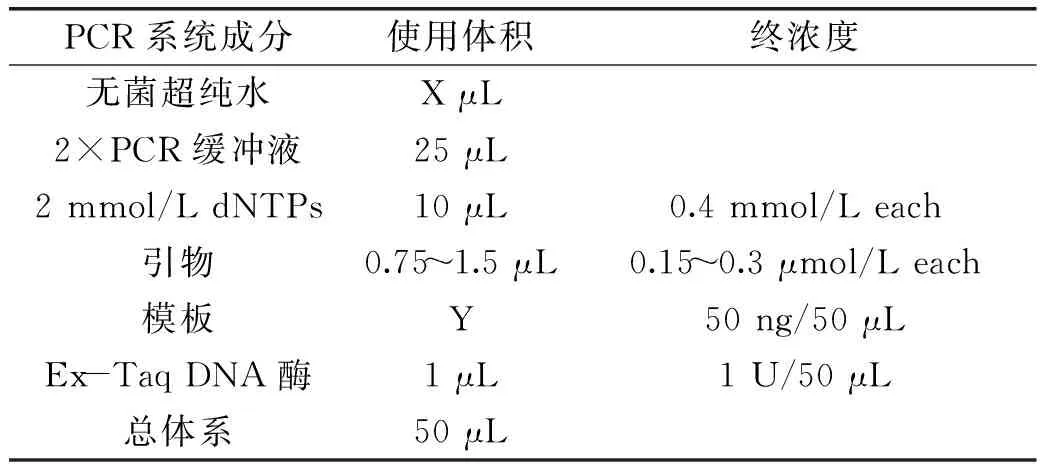
表2 PCR反应体系Table 2 Reaction system for PCR
反应参数:94 ℃预变性5 min;94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸90 s,共30个循环;72 ℃延伸10 min。PCR产物以2%琼脂糖电泳检查,将扩增片段分别命名为β亚基、A、B、C。
1.2.5.2 克隆载体的构建 DNA回收试剂盒将β-伴大豆球蛋白β亚基全长及片段A、B、C基因回收,具体参照试剂盒操作手册。将片段与pMD18-T载体相连,16 ℃水浴过夜,反应体系为:pMD18-T vector 2.0 μL;DNA 3.0 μL;ligation 5.0 μL。用预冷枪头吸取10 μL连接产物转移至50 μL感受态细胞中,轻轻摇匀,置于冰上30 min,然后42 ℃水浴60~90 s,随后在冰中冷却5 min,最后加入900 μL 37 ℃预热LB培养基,37 ℃振荡培养1 h。此时将50 μL x-gal(20 mg/mL)和13 μL IPTG(50 mg/mL)涂布含50 μg/mL氨苄青霉素(Amp+)的LB琼脂平板上,37 ℃预热0.5 h。吸取100 μL培养液吹打在预热平板上,37 ℃恒温培养16~18 h。挑选白色菌落扩大培养,用质粒小提试剂盒提取质粒。将重组质粒分别命名为pTβ、pTA、pTB、pTC。
1.2.5.3 重组质粒的鉴定 对重组质粒进行PCR和双酶切鉴定,酶切体系为:重组质粒1.0 μL,10×M Buffer 5.0 mol/L,EcoRⅠ 1.0 μL,HindⅢ 1.0 μL,无菌超纯水39.0 μL,总体系50.0 μL,混匀后37 ℃水浴4 h。PCR反应体系参照表2。回收产物用2%琼脂糖凝胶分析初步鉴定后,送至TaKaRa测序。
2 结果与分析
2.1 β-伴大豆球蛋白β亚基蛋白的三级结构构建
通过PDB数据库搜索到蛋白质三级结构模型(登录号:1ipk.1.A),氨基酸序列相似度达到99.52%,模型即为蛋白质亚基结构。从得到的蛋白质三级结构图(图1)可以看出,有部分α-螺旋和β-转角及无规则卷曲结构在蛋白质外侧,绝大部分β-折叠在蛋白质中心区域。构象表位一般位于蛋白质表面的无规则卷曲等优势区域,一般认为α-螺旋、β-折叠常位于蛋白质内部化学键能较高处,其形态结构比较稳定且受外界物理化学等因素影响较小。而β-转角和无规则卷曲多位于蛋白质表面,容易与抗体嵌合,因而成为抗原表位的可能性大[15]。
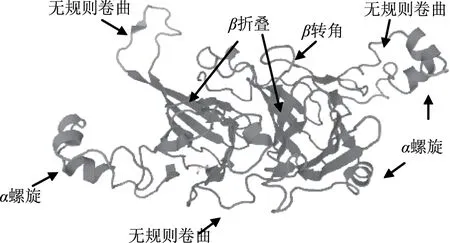
图1 β-伴大豆球蛋白β亚基蛋白的三级结构模型Fig.1 Model of tertiary structure of β-conglycinin beta-subunit
2.2 β-伴大豆球蛋白β亚基B细胞构象表位的预测
DiscoTope 2.0服务器根据建立的β-伴大豆球蛋白β亚基三级结构模型预测出了91个B细胞构象表位的氨基酸残基。这91个氨基酸位于30个区段,分别为31、33、39~40、48~50、58、60~68、70、81~82、111~112、117、137~141、154~157、167、190、200~204、207、212、214~220、222~227、229~238、245~246、261~263、265、268~269、312~324、326、366~367、398、402、408。如图2所示,预测的抗原表位绝大部分位于蛋白质模型的β转角和无规则卷曲处。
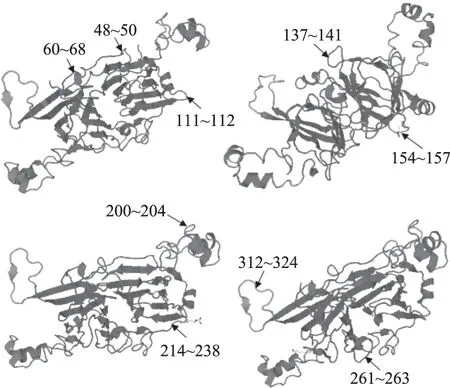
图2 β-伴大豆球蛋白β亚基部分抗原表位示意图Fig.2 Sketch map of epitopes of β-conglycinin beta-subunit注:以上四组图片为β亚基模型的不同视角。
2.3 基因的分段克隆及重组质粒构建
2.3.1 目的基因片段的克隆 将载体质粒PUC57中的目的基因经PCR扩增,得到全片段β亚基及分段片段A、B、C。扩增片段经琼脂糖电泳显示,可观察到条带大小与实验预先设计的片段A、B、C长度相符,表明已正确扩增出目的片段,见图3。

图3 β亚基全片段及A,B,C基因扩增电泳图Fig.3 Amplified electrophoresis of β-subunit and A,B,C bypass PCR注:M:DL1500Marker;1~4分别为β亚基全片段及片段A、B、C。
2.3.2 重组质粒构建及PCR、双酶切鉴定 PCR扩增目的基因片段经切胶回收与pMD18-T载体相连,并转化到JM109感受态细胞,挑取3个白色菌落进行扩大培养,经PCR和双酶切鉴定,如图4、图5的1、2、3、4泳道分别出现与β亚基全片段及片段A、B、C长度大小相符的条带,表明重组质粒构建成功[16]。
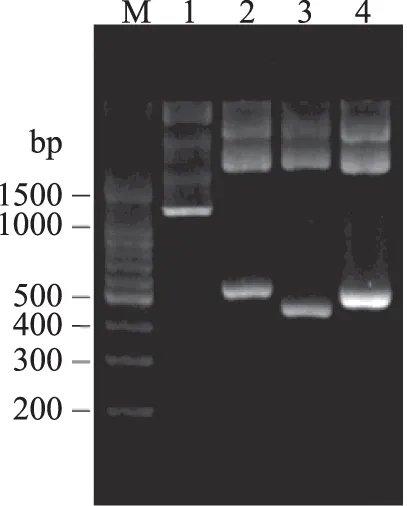
图4 重组质粒pTβ、pTA、pTB、pTC的PCR电泳图Fig.4 Amplified electrophoresis of pTβ,pTA,pTB,PTC by PCR注:M:DL1500Marker;1~4分别为重组质粒pTβ、pTA、pTB、pTC。

图5 重组质粒pTβ、pTA、pTB、pTC双酶切电泳图Fig.5 Electrophoresis of pTβ,pTA,pTB,pTC by double enzyme digestion注:M:DL1500Marker;1~4分别为重组质粒pTβ、pTA、pTB、pTC。
3 讨论与结论
本研究利用生物信息学方法,对β-伴大豆球蛋白β亚基进行分段,克隆出β-伴大豆球蛋白β亚基基因及分段基因,并成功构建克隆载体。下一步,将利用T7噬菌体展示系统分别表达β亚基及片段,结合分子生物学、免疫学、生物信息学对β-伴大豆球蛋白β亚基过敏表位进行定位研究。对探明β-伴大豆球蛋白β亚基致敏性的分子机制、开展无过敏和低过敏的食品研究、防止食物过敏疾病的发生以及过敏性疾病的诊断提供了理论和实践依据。
[1]ALOK V,SANDEEP K,MUKUL D,et al. A comprehensive review of legume allergy[J]. Clinical Reviews in Allergy and Immunology,2013,45(1):30-46.
[2]HE L,HAN M,QIAO S,et al. Soybean Antigen Proteins and their Intestinal Sensitization Activities[J]. Current Protein and Peptide Science,2015,16(7):613-621.
[3]SUN X,SHAN X,YAN Z,et al. Prediction andcharacterization of the linear IgE epitopes for the major soybeanallergenβ-conglycinin using immunoinformatics tools[J]. Food and Chemical Toxicology,2013,56(12):254-260.
[4]Maruyama N,Adachi M,Takahashi K,et al. Crystal structures of recombinant and native soybean beta-conglycinin beta homotrimers[J]. European Journal of Biochemistry,2001,268(12):3595-3604.
[5]MEINLSCHMIDT P,SCHWEIGGERT-WEISZU,BRODE V,et al. Enzyme assisted degradation of potential soy protein allergens with special emphasis on the technofunctionality and the avoidance of a bitter taste formation[J].LWT-Food Science and Technology,2016,68:707-716.
[6]朱婷伟,陈复生,布冠好,等. 大豆蛋白过敏原结构与功能的研究进展[J]. 食品工业科技,2013,34(21):377-380.
[7]Coates JB,Medeiros JS,Thanh VH,et al. Characterization of the subunits of beta-conglycinin[J]. Archives of Biochemistry & Biophysics,1985,243(1):184.
[8]郑树贵,曹松屹,孙泽威,等.β-伴大豆球蛋白β亚基的纯化及免疫活性鉴定[J]. 大豆科学,2009,28(2):301-304.
[9]闫慧丽. 大豆主要过敏原Gly m Bd 28K IgG结合表位的定位研究[D]. 郑州:河南工业大学,2016.
[10]Larralde O G,Martinez R,Camacho F,et al. Identification of hepatitis a virus mimotopes by phage display,antigenicity and immunogenicity[J]. Journal of Virological Methods,2007,140(1-2):49-58.
[11]Maruyama N,Adachi M,Takahashi K,et al. Crystal structures of recombinant and native soybeanβ-conglycininβhomotrimers[J]. Eur J Biochem,2001,268(12):3595-3604.
[12]卫会君,司晓霞,田怀东.β-伴大豆球蛋白β亚基缺失的大豆遗传资源的发掘[J]. 科技与创新,2014(4):159.
[13]杨廷亚,王芳,姜平,等. 应用噬菌体展示技术筛选兔出血症病毒抗原模拟表位[J]. 畜牧兽医学报,2012,43(8):1281-1286.
[14]袁水林,李欣,陈红兵,等.噬菌体展示技术定位主要食物过敏原表位的研究进展[J].食品工业科技,2015,36(3):379-384.
[15]Wang W,Han J X,Wu Y J,et al. Simultaneous detection of eight food allergens using optical thin-film biosensor chips[J]. J Agric Food Chem,2011,59(13):6889-6894.
[16]张丹,鲁会军,金扩世,等. 猪乙型脑炎病毒E蛋白基因的分段克隆及原核表达[C]. 中国畜牧兽医学会2010年学术年会——中国兽医临床大会,2010:303-305.
Analysisofantigenicepitopesandcloningofsoybeanmajorallergenβ-conglycininbeta-subunit
PIJiang-yi,XIJun*,HEMeng-xue,LIShuang
(College of Food Science and Technology,Henan University of Technology,Zhengzhou 450001,China)
TS201.6
A
1002-0306(2017)18-0090-04
2017-02-23
皮江一(1993-),男,硕士研究生,研究方向:食品免疫学,E-mail:577129671@qq.com。
*通讯作者:席俊(1977-),女,博士,副教授,研究方向:食品免疫学,E-mail:Xijunhnu@163.com。
国家自然科学基金项目(31671778;31301409);河南省高等学校重点科研项目(16A550001)。
10.13386/j.issn1002-0306.2017.18.018
