真核生物中支架蛋白家族(IQGAPfamily)的分子进化分析
2017-10-13热孜亚吾买尔曹立环
热孜亚·吾买尔,曹立环,余 龙
(1. 复旦大学 生命科学学院,上海 200438; 2. 复旦大学 遗传工程国家重点实验室,上海 200438)
真核生物中支架蛋白家族(IQGAPfamily)的分子进化分析
热孜亚·吾买尔1,2,曹立环1,2,余 龙1,2
(1. 复旦大学 生命科学学院,上海 200438; 2. 复旦大学 遗传工程国家重点实验室,上海 200438)
支架蛋白家族(IQGAP family)广泛存在于真核生物中,在细胞的信号转导、骨架运动、细胞分裂的过程中都有着重要功能.研究分析IQGAP蛋白家族的分子进化,有助于深入全面地了解整个IQGAP蛋白家族在不同物种中功能.通过使用相关数据库,在35个真核生物物种中检索到了70个支架蛋白家族成员的序列信息,并对其进行了系统的分子进化研究.我们发现: 支架蛋白家族基因广泛存在于后生动物(Metazoan)、真菌(Fungi)、变形虫界(Amoebozoa)以及其他一些真核生物中,提示支架蛋白存在于最后的真核生物的共同祖先(Last Eukaryotic Common Ancestor, LECA).系统发育分析表明脊椎动物的IQGAP蛋白和非脊椎动物中代表性IQGAP基因共享一个共同的祖先基因,单个的IQGAP祖先基因在早期脊椎动物中(在四足总纲和硬骨鱼纲的分离之前),发生了两次基因复制事件,导致现存的脊椎动物中3组IQGAP基因(IQGAP1,IQGAP2,IQGAP3)产生.
真核生物; 支架蛋白家族; 分子进化分析
基因的分子进化研究能通过序列同源性的比较,系统发育树的构建等方法研究基因在不同物种中的起源,复制,突变,丢失的信息,这有助于人们了解在不同生物物种中基因结构变化及其功能衍化的规律,使人们更深入全面地了解基因的功能.
支架蛋白家族(IQGAP family)是一种进化上保守的多结构域蛋白质,IQGAP蛋白在酵母,鱼类,非洲爪蟾和哺乳动物中,都有广泛表达[1].在真菌中,酿酒酵母(Saccharomycescerevisiae)IQGAP同源蛋白iqg1和裂殖酵母(Schizosaccharomycespombe)IQGAP同源蛋白RNG2都在胞质分裂过程中发挥重要作用,两者都定位于细胞收缩环和负责招募其他参与细胞分裂的蛋白[2-3].变形虫界的盘基网柄菌(Dictyosteliumdiscoideum)的两种IQGAP同源蛋白RGAA和GAPA也都被发现在胞质分裂时发挥关键作用[4-5].人拥有3个IQGAP家族的成员(IQGAP1,IQGAP2和IQGAP3),研究发现,IQGAP家族的成员在各组织中有不同表达模式.人IQGAP1蛋白在人体的组织中广泛表达,而人IQGAP2仅表达于肝脏和睾丸,人IQGAP3主要在脑和肺中表达[6].当前,人们对IQGAP蛋白的研究主要集中在IQGAP1蛋白.IQGAPl分子大小190kD,位于染色体15q26.IQGAPl有钙调理蛋白同源结构域(Calponin Homology Domain, CHD),IQ结构域(IQ domain),聚脯氨酸结合区(polyproline binding region, Ww),RasGAP结构域(RasGap domain),以及在蛋白C末端的PfamRasGAP_C结构域[7].其中,IQGAPl的钙调理蛋白同源结构域能结合肌动蛋白(Actin),聚脯氨酸结合区能结合ERK蛋白,IQGAP1上的RasGap结构域能结合Cdc42蛋白和Rac1蛋白,PfamRasGAP_C 结构域能结合钙黏附蛋白E(E-cadherin)和β-链蛋白(β-catenin),IQGAP1被认为是一个介导多个信号通路的枢纽,参与细胞生长、分化和分裂[8].人IQGAP1和IQGAP3在HeLa细胞的胞质分裂中发挥重要作用,RNA干扰IQGAP1或IQGAP3地表达能导致胞质分裂受阻,多核细胞产生[9].
最近,关于人IQGAP家族在肿瘤发生发展中的作用也引起了人们的注意.在肝癌细胞中,人IQGAP1蛋白表达水平显著上升,而人IQGAP2蛋白表达水平却下降[10].同时,肝癌病人中的人IQGAP1,IQGAP2的表达水平与病人的愈后相关[11].
尽管IQGAP蛋白家族分子被发现和研究已经20多年了,但人们对整个IQGAP家族在多种生物中的功能研究依然十分有限,对IQGAP蛋白家族的分子进化历史的了解也不明确.当前,随着越来越多生物的基因组被完全测序,使我们有机会在相对广泛的真核生物物种中探讨IQGAP基因的分子进化历史,了解不同物种间的IQGAP分子以及同一物种间不同的IQGAP分子之间的进化关系,帮助我们进一步深入地理解IQGAP蛋白的功能.
1 材料与方法
1.1真核生物中IQGAP蛋白的检索
我们下载了SMART蛋白结构域数据库[12](http:∥smart.embl-heidelberg.de/)中RasGAP结构域(SM00323)的种子序列(alignment Consensus sequences)的序列,利用HMMER程序在非冗余蛋白序列数据库进行HMM搜索(http:∥hmmer.janelia.org/search/hmmsearch)[13],所得到的序列在计算机上用BLAST程序对人蛋白序列数据库进行BLAST搜索[14],仅保留其中BEST Hit为人IQGAP蛋白的序列,而其他的BEST Hit为人RASA1, RASL3等蛋白则被丢弃.对于每一个潜在的IQGAP家族基因,我们最后只收录蛋白序列最长的剪接本,其他剪接本或重复提交序列都没有被最后收录.最后,我们选择了35个物种中的IQGAP蛋白进行研究,35个物种中包含16个后生生物物种:Homosapiens(人);Musmusculus(小鼠);Rattusnorvegicus(大鼠);Bostaurus(牛);Gallusgallus(原鸡);Xenopuslaevis(非洲爪蟾);Xenopustropicalis(热带爪蟾);Daniorerio(斑马鱼);Tetraodonnigroviridis(绿河豚);Cionaintestinalis(玻璃海鞘);Cionasavignyi;Branchiostomafloridae(文昌鱼);Caenorhabditiselegans(线虫);Nematostellavectensis(海葵);Trichoplaxadhaerens(丝盘虫);Amphimedonqueenslandica(海绵).3个和后生动物(Metazoa)亲缘关系很近的原生生物物种:Monosigabrevicollis;Salpingoecarosetta;Capsasporaowczarzaki.8个真菌(Fungi)物种:Allomycesmacrogynus;Candidaalbicans(白念珠菌);Coccidioidesimmitis(粗球孢子菌);Coprinopsiscinerea(灰盖鬼伞);Neurosporacrassa(粗糙链孢霉);Saccharomycescerevisiae(酿酒酵母);Schizosaccharomycespombe(裂殖酵母);Spizellomycespunctatus.5个归属于变形虫界(Amoebozoa)的物种:Acanthamoebacastellanii(卡氏棘阿米巴);Dictyosteliumdiscoideum(盘基网柄菌);Dictyosteliumpurpureum;Entamoebahistolytica(溶组织内阿米巴);Polysphondyliumpallidum; 以及其他的3个物种:Naegleriagruberi(尾刺耐格里原虫);Thecamonastrahens;Spongosporasubterranean.
1.2蛋白结构域组成分析
利用SMART蛋白结构域数据库[12](http:∥smart.embl-heidelberg.de/)和Pfam蛋白结构域数据库[15](http:∥pfam.xfam.org/)对我们所收集的70个蛋白进行结构域组成分析.
1.3蛋白多序列比对和系统发育分析
我们利用Mafft程序[16],参数设定为(L-INS-I),进行蛋白序列的多序列比对.我们选择多序列比对中的最保守区段(IQGAP蛋白中的RasGap结构域部分)进行进一步的系统发育分析,我们先用ProtTest2.4程序[17],利用Akaike Information Criterion(AIC)标准计算了相应的最优蛋白替代模型,然后用最大似然法(Maximum Likelihood, ML)和贝叶斯推断法(Bayesian Inference, BI)构建进化树.我们基于CIPRES Science Gateway V. 3.1[18]利用RAxML 7.2.8程序[19]构建ML树,bootstrap值设定为1000;用PHYLOBAYES3.3程序[20]构建贝叶斯进化树,停止蒙特卡罗马尔可夫链计算的参数设定为Maxdiff<0.1,并使用Dendroscope程序[21]显示所有进化树.
2 结 果
2.1IQGAP蛋白的检索
我们最后检索到35个真核物种中的70个IQGAP蛋白的序列信息,所有IQGAP蛋白合乎下列3个条件: (1)含有RasGap结构域(SM00323)或pfamRasGap结构域(PF00616);(2)这些蛋白对人蛋白序列数据库进行BLAST搜索,Best hit为人的IQGAP1,IQGAP2或IQGAP3蛋白;(3)所有蛋白含有PfamRasGap_C(PF03836)结构.我们发现这些蛋白分布在后生动物、真菌、变形虫界和其他的原生生物中,具体结果见表1.本文中蛋白名称均依据Uniprot蛋白数据库.

表1 代表性真核生物物种中的IQGAP蛋白
注: 此表的数据,在汇集本文鉴定的IQGAP家族蛋白的同时,部分整合了本文图2中系统发育分析的数据,其中脊椎动物IQGAP被成功地归类于3个小组(IQGAP1,IQGAP2,IQGAP3),而其他物种的IQGAP蛋白未能进行这三个小组的分类.用黑体标注的蛋白被认为是脊椎动物IQGAP的直系同源基因(参见正文).
2.2各物种中IQGAP蛋白的蛋白结构域组成
首先,我们研究了这些蛋白的蛋白结构域的组成,我们发现,大部分IQGAP蛋白(图1,蛋白4~13)都含有钙调理蛋白同源结构域,IQ结构域,RasGAP结构域,以及在C末端的PfamRasGAP_C结构域.但在相当多的真菌和变形虫来源的IQGAP蛋白,例如来源于变形虫界的L8GLC0_ACACA、L8HI44_ACACA、Q54I64_DICDI、RGAA_DICDI、GAPA_DICDI等蛋白,以及来源于真菌的0A0L0T9F0_ALLMA、J3KKA8_COCIM、A8NUP6_COPC7、Q7S5G6_NEUCR、AP1_SCHPO等蛋白,缺失钙调理蛋白同源结构域和IQ结构域,但这些蛋白中的RasGAP结构域和C末端的PfamRasGAP_C结构域依然存在,限于篇幅,图1中仅显示了3个此类代表性蛋白(蛋白1~3),这显示RasGAPs和PfamRasGAP_C这两个结构域是IQGAP家族最为核心的区域.另外,我们只在部分IQGAP蛋白(图1,蛋白7,8,11)中检测到聚脯氨酸结合区(Ww)蛋白结构域.
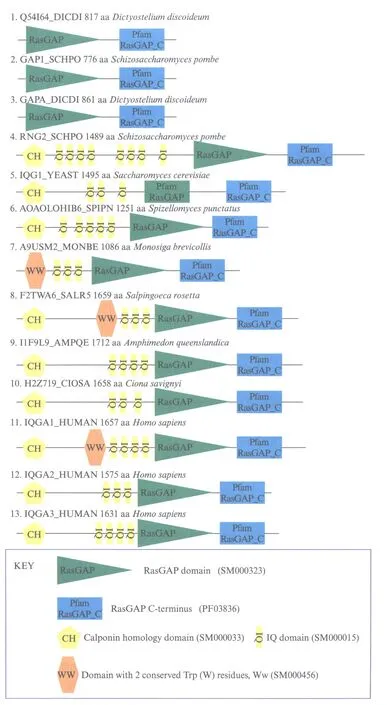
图1 真核生物中代表性IQGAP蛋白的结构域组成Fig.1 Domain organization of representative IQGAP proteins in Eukaryotes真核生物中的代表性IQGAP蛋白,依次标示了蛋白名称、序列长度、物种名称和结构域组成.蛋白名称依据Uniprot蛋白数据库,结构域组成依据SMART蛋白结构域数据库和Pfam蛋白结构域数据库的结果.
2.3真核生物中IQGAP蛋白的系统发育分析结果
因为来源于线虫的O17772_CAEEL蛋白,酿酒酵母的IQG1_YEAST蛋白,裂殖酵母的RNG2_SCHPO蛋白,白念珠菌的IQG1_CANAL蛋白尽管是IQGAP的同源蛋白,但这4个蛋白与其他的IQGAP蛋白序列差异较大,会干扰我们整个系统发育分析的可信度.为了获得有效的系统发育分析结果,我们的分析中没有涵盖上述的4个蛋白.这样,我们对70个蛋白中余下的66个蛋白进行了系统发育分析.由于来源于动物、真菌、变形虫界等的IQGAP蛋白相互之间的进化距离较远,故这些IQGAP蛋白序列比对中某些区域的序列相似性也较差.为获得可信的系统发育分析结果,我们选择IQGAP蛋白中的最保守蛋白序列区段(IQGAP蛋白中的RasGap domain部分)进行多序列比对和进化分析.
我们对上述66个IQGAP家族的基因的蛋白序列用最大似然法(ML)和贝叶斯推断法(BI)进行了系统发育分析,来自人的RASA1_HUMAN, RASL3_HUMAN和SYGP1_HUMAN蛋白设定为我们进化分析的外类群(outgroup),结果显示最大似然法(ML)和贝叶斯推断法(BI)产生了几乎相同的拓扑结构(图2).在图2中所有的IQGAP蛋白家族也总结于表1.
图2显示,所有的来自脊椎动物(人,小鼠,大鼠,牛,原鸡,非洲爪蟾,热带爪蟾,斑马鱼,绿河豚)的IQGAP蛋白都可以很好地归类到3个IQGAP组,分别为IQGAP1Group,IQGAP2Group,IQGAP3Group.同时,我们还发现,来自海葵的A7SXE3_NEMVE,丝盘虫的B3RR54_TRIAD,海绵的I1F9L9_AMPQE蛋白和脊椎动物中的IQGAP蛋白很好的归类到一起(图中标示为IQGAPsinAnimalia),这一结果提示这些基因应共享一个共同的祖先基因.事实上,从图中可以看到,一些来源于后生动物进化关系最近的单细胞生物的IQGAP蛋白(如A9USM2_MONBE,F2TWA6_SALR5),一些来源于真菌的IQGAP蛋白(图中标示为IQGAPsinFungi)和一些来源于变形虫界的IQGAP蛋白(图中标示为IQGAPsinAmoebozoa),都可被认为是脊椎动物中的IQGAP蛋白直系同源基因.这提示最后的真核生物的共同祖先(Last Eukaryotic Common Ancestor, LECA)已拥有支架蛋白家族基因这一古老的基因.
尽管脊椎动物IQGAP蛋白与真菌的IQGAP蛋白,变形虫界的IQGAP蛋白共享一个共同的祖先基因,但脊椎动物中3组IQGAP基因(IQGAP1,IQGAP2,IQGAP3)的形成却发生于相对晚近的时期.我们的分析结果显示在早期脊椎动物(在四足总纲和硬骨鱼纲的分离之前)一个IQGAP的祖先基因,发生了两次基因复制事件,其中第一次复制,形成IQGAP1/2的祖先基因和IQGAP3基因,然后,所产生的IQGAP1/2的祖先基因再次发生基因复制,产生相应的IQGAP1和IQGAP2基因,这两次基因复制事件导致脊椎动物中3组IQGAP基因(IQGAP1,IQGAP2,IQGAP3)形成(图3,见第380页).
在进一步对多种脊椎动物和非脊椎动物中的IQGAP蛋白进行多序列比对时,我们发现人,小鼠,大鼠,牛的IQGAP2基因的蛋白序列相对于它们对应的IQGAP1和IQGAP3发生了长度为84个氨基酸的片段缺失(图4,见第380页),其缺失的片断位于IQGAP蛋白的钙调理蛋白同源结构域和IQ结构域之间.因为脊椎动物中热带爪蟾和斑马鱼中IQGAP2基因,以及非脊椎动物中相应直系同源基因(如海鞘的H2Z719_CIOSA,海绵的I1F9L9_AMPQE基因)未见此类型的蛋白片段缺失.提示部分脊椎动物中发生此类片断缺失发生在相对晚近的时期.但此类发生在部分脊椎动物中的IQGAP2基因片断缺失对IQGAP蛋白的功能影响还未见报道.
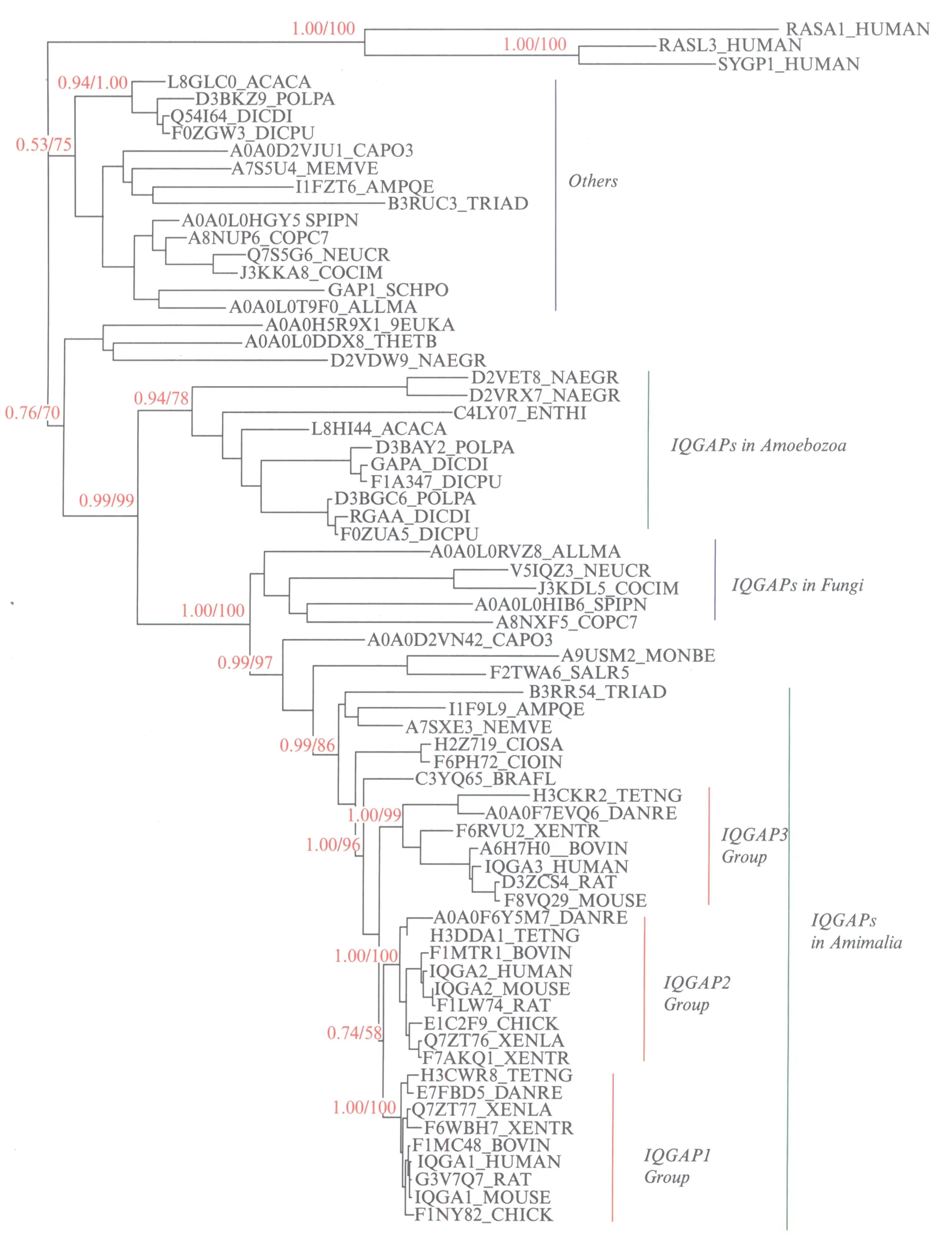
图2 真核生物中IQGAP蛋白的系统发育分析Fig.2 Phylogenetic analysis of IQGAP proteins in Eukaryotic lineages对真核生物中IQGAP家族的的蛋白序列用最大似然法(ML)和贝叶斯推断法(BI)进行了系统发育分析,结果显示最大似然法(ML)和贝叶斯推断法(BI)产生了几乎相同的拓扑结构.进化树分支中的第一个数字表明贝叶斯后验概率(只有这些关键分支被标记),进化树分支中的第二个数字显示最大似然法中最大似然比.来自人的RASA1_HUMAN, RASL3_HUMAN,和SYGP1_HUMAN蛋白设定为我们进化分析的外类群(outgroup).蛋白名称依据Uniprot蛋白数据库.

图3 IQGAP基因在早期脊椎动物中复制的示意图Fig.3 Summaries of the gene duplication events for IQGAP genes in early vertebrataIQGAP的祖先基因在早期脊椎动物(在四足总纲和硬骨鱼纲的分离之前)发生了两次基因复制事件,导致脊椎动物中3组IQGAP基因(IQGAP1,IQGAP2,IQGAP3)的形成.

图4 脊椎动物和非脊椎动物IQGAP蛋白的多序列比对Fig.4 Multiple sequence alignment of IQGAP proteins from vertebrate and invertebrate lineages来自人、小鼠、牛、斑马鱼、热带爪蟾、海鞘、海绵等的IQGAP蛋白进行多序列比对(仅显示与IQGAP2蛋白序列片断缺失相近区域部份).蛋白名称依据Uniprot蛋白数据库.
3 讨 论
我们检索了整个真核生物蛋白数据库中的IQGAP家族蛋白,发现IQGAP蛋白在植物中缺失;然后,我们选择了35个真核生物物种作为我们的研究范围,对这些物种的选择,首先考虑了物种广泛性,35个真核生物物种包含了16种动物物种,3种和后生动物亲缘关系很近的单细胞物种,8种真菌物种,5种变形虫界和3种其他的物种.这些物种中包含了在动物进化历史上有重要意义的代表性的生物.例如: 扁盘动物门(Placozoa)中丝盘虫(T.adhaerens)是一种原始的后生动物,被认为是目前发现的最简单的真后生动物(Eumetazoan),虫体由上下两层细胞构成,无体腔,无消化腔,无神经系统[22].刺胞动物门(Cnidaria)的海葵(N.vectensis)是一个非两侧对称动物[23].领鞭毛虫类(Choanoflagellate)的M.brevicollis被认为是与后生动物进化关系最近的单细胞生物,在研究后生动物的起源和多样化有重要的价值[24].盘基网柄菌(D.discoideum)作为一种土壤变形虫,具有特殊的能力,能以单细胞形式和多细胞形式交替的方式存在.在生物的进化谱系上,盘基网柄菌和后生生物的分离,早于酵母和后生生物的分离,但晚于植物和后生生物的分离[25].
在研究方法上,在同源蛋白搜索中,我们结合了HMMer方法[13]和BLAST方法[14],以保证方法的灵敏性和可靠性.多序列比对所用的Mafft程序[16],系统发育树构建所用的最大似然法的RAxML 7.2.8程序[17]和贝叶斯法的PHYLOBAYES3.3程序[18]都是当前国际上进行系统发育研究的主流方法和程序,这也保证了我们结果的可靠性.
我们的研究结果表明,IQGAP基因存在于后生动物、真菌、变形虫界等真核生物中,在Excavata类的真核生物尾刺耐格里原虫(Naegleriagruberi),在有孔虫界(Rhizaria)的Spongosporasubterranean中也发现了IQGAP的同源基因(表1),这充分显示了IQGAP基因广泛分布于真核生物中,跟据上述物种在真核生物中的进化关系[26].我们认为支架蛋白存在于LECA,即在后生动物,真菌,变形虫界和有孔虫界等真核生物的物种分离之前,IQGAP基因就已经存在.但是,变形虫界IQGAP基因一般仅拥有RasGAP结构域,C末端的PfamRasGAP_C结构域.而后生动物,真菌中的IQGAP基因除了含有RasGAP结构域,C末端的PfamRasGAP_C结构域外,往往还含有钙调理蛋白同源结构域,IQ结构域(图1).关于各物种IQGAP基因的结构域组成变化的进化历史,以及结构域组成对IQGAP基因功能的影响将会是进一步研究的对象.
但人的3个IQGAP基因是在早期脊椎动物中的一个IQGAP的祖先基因通过发生了两次基因复制事件后形成的.其中第一次基因复制,形成IQGAP1/2的祖先基因和IQGAP3基因,然后,所产生的IQGAP1/2的祖先基因再次发生基因复制,产生相应的IQGAP1和IQGAP2基因(图3).这一结果与人IQGAP1,IQGAP2,IQGAP3基因相互之间较高的序列相似性相吻合.在人的3个IQGAP基因中,IQGAP1基因表达于人体绝大多数组织中,并受到广泛的研究,而IQGAP2基因和IQGAP3研究则相对较少[6,27].人的3个IQGAP基因,除了在人体各组织的表达谱不同以外,功能也有所不同[27].人IQGAP1基因被认为是一个癌基因(oncogene),在肝细胞癌、肺癌、胃癌、乳腺癌表达上升[28],而人IQGAP2基因在肝细胞癌,前列腺中表达下降[29-30].实验研究还表明敲除IQGAP2基因的小鼠易发生肝癌,但敲除IQGAP1和IQGAP2双基因的小鼠则能正常生存,提示IQGAP1基因和IQGAP2基因发生某种程度的功能拮抗[31].而IQGAP3功能与人IQGAP1有相似性,两者都能促进肝细胞和乳腺上皮细胞的增殖[32-33].这样,IQGAP2相对IQGAP1和IQGAP3基因,有其功能的特殊性.有趣的是我们发现人,小鼠,大鼠,牛的IQGAP2基因的蛋白序列相对于它们对应的IQGAP1和IQGAP3发生了长度为84个氨基酸的片段缺失(见图4).现在还不清楚是否是IQGAP2上84个氨基酸的片段缺失造成了IQGAP2基因和IQGAP1基因的功能拮抗,进一步研究IQGAP2基因上的84个氨基酸的片段缺失对IQGAP功能的影响将给我们以新的提示.
我们的研究明确揭示了IQGAP家族在早期脊椎动物中的进化历史,即在早期脊椎动物中发生了两次基因复制事件,导致现存的脊椎动物中3组IQGAP基因(IQGAP1,IQGAP2,IQGAP3)产生.人,小鼠,大鼠,牛的IQGAP2基因的蛋白序列相对于它们对应的IQGAP1和IQGAP3发生了长度为84个氨基酸的片段缺失.这些结果将有助于我们进一步研究人和多种其他生物中IQGAP蛋白的功能.
[1] SHANNON K. IQGAP family members in yeast, dictyostelium, and mammalian cells [J].IntJCellBiol, 2012, 894817.
[2] EPP A, CHANT J. An IQGAP-related protein controls actin-ring formation and cytokinesis in yeast [J].CurrBiol, 1997,7: 921-929.
[3] ENG K, NAQVI N, WONG K,etal. Rng2p, a protein required for cytokinesis in fission yeast, is a component of the actomyosin ring and the spindle pole body [J].CurrBiol, 1998,8(11): 611-621.
[4] LEE S, ESCALANTE R, FIRTEL R A. A Ras GAP is essential for cytokinesis and spatial patterning in Dictyostelium [J].Development, 1997,124: 983-996.
[5] ADACHI H, TAKAHASHI Y, HASEBE T,etal. Dictyostelium IQGAP-related protein specifically involved in the completion of cytokinesis [J].JCellBiol, 1997,137: 891-898.
[6] WANG S, WATANABE T, NORITAKE J. IQGAP3, a novel effector of Rac1 and Cdc42, regulates neurite outgrowth [J].JCellSci, 2007,120(4): 567-577.
[7] HEDMAN A C, SMITH J M, SACKS D B. The biology of IQGAP proteins: Beyond the cytoskeleton [J].EMBORep, 2015,16, 427-446.
[8] WHITE C D, ERDEMIR H H, SACKS D B. IQGAP1 and its binding proteins control diverse biological functions [J].CellSignal, 2012,24,826-834.
[9] ADACHI M, KAWASAKI A, NOJIMA H,etal. Involvement of IQGAP family proteins in the regulation of mammalian cell cytokinesis [J].GenesCells, 2014,19(11): 803-820.
[10] WHITE C D, KHURANA H, GNATENKO D V,etal. IQGAP1 and IQGAP2 are reciprocally altered in hepatocellular carcinoma [J].BMCGastroenterol, 2010,10: 125.
[11] XIA F D, WANG Z L, CHEN H X,etal. Differential expression of IQGAP1/2 inHepatocellularcarcinomaand its relationship with clinical outcomes [J].AsianPacJCancerPrev, 2014,15(12): 4951-4956.
[12] LETUNIC I, DOERKS T, BORK P. SMART: Recent updates, new developments and status in 2015 [J]NucleicAcidsRes, 2015,43(Database issue): 257-260.
[13] FINN R D, CLEMENTS J, EDDY S R. HMMER Web Server: Interactive sequence similarity searching [J].NucleicAcidsRes, 2011,39: 29-37.
[14] ALTSCHUL S F, GISH W, MILLER W,etal. Basic local alignment search tool [J].JMolBiol, 1990,215: 403-410.
[15] FINN R D, COGGILL P, EBERHARDT R Y,etal. The Pfam protein families database: Towards a more sustainable future [J].NucleicAcidsRes, 2016,44(D1): 279-285.
[16] KATOH K, KUMA K, TOH H, MIYATA T. MAFFT version 5: Improvement in accuracy of multiple sequence alignment[J].NucleicAcidsRes, 2005,33(2): 511-518.
[17] DARRIBA D, TABOADA G L, DOALLO R,etal. ProtTest 3: Fast selection of best-fit models of protein evolution [J].Bioinformatics, 2011,27(8): 1164-1165.
[18] STAMATAKIS A. RAxML version 8: A tool for phylogenetic analysis and post-analysis of large phylogenies [J].Bioinformatics, 2014,30(9): 1312-1313.
[19] MILLER M A, PFEIFFER W, SCHWARTZ T. Creating the CIPRES science gateway for inference of large phylogenetic trees [C]∥Proceedings of the Gateway Computing Environments Workshop(GCE). New Orleans, LA: ACM. 2010: 1-8.
[20] LARTILLOT N, LEPAGE T, BLANQUART S. PhyloBayes 3: A Bayesian software package for phylogenetic reconstruction and molecular dating [J].Bioinformatics, 2009,25(17): 2286-2288.
[21] HUSON D H, RICHTER D C, RAUSCH C,etal. Dendroscope: An interactive viewer for large phylogenetic trees [J].BMCBioinforma, 2007,8: 460.
[22] SRIVASTAVA M, BEGOVIC E, CHAPMAN J,etal. The Trichoplax genome and the nature of placozoans [J].Nature, 2008,454(7207): 955-960.
[23] PUTNAM N H, SRIVASTAVA M, HELLSTEN U,etal. Sea anemone genome reveals ancestral eumetazoan gene repertoire and genomic organization [J].Science, 2007,317(5834): 86-94.
[24] KING N, WESTBROOK M J, YOUNG S L,etal. The genome of the choanoflagellateMonosigabrevicollisand the origin of metazoans [J].Nature, 2008,451(7180): 783-788.
[25] EICHINGER L, PACHEBAT J A, GLOCKNER G,etal. The genome of the social amoebaDictyosteliumdiscoideum[J].Nature, 2005,435(7038): 43-57.
[26] BURKI F. The eukaryotic tree of life from a global phylogenomic perspective [J].ColdSpringHarbPerspectBiol, 2014,6(5): a016147.
[27] SMITH J M, HEDMAN A C, SACKS D B. IQGAPs choreograph cellular signaling from the membrane to the nucleus [J].TrendsCellBiol, 2015,25(3): 171-184.
[28] JOHNSON M, SHARMA M, HENDERSON B R. IQGAP1 regulation and roles in cancer [J].CellSignal, 2009,21(10): 1471-1478.
[29] GNATENKO D V, XU X, ZHU W,etal. Transcript profiling identifies IQGAP2(-/-) mouse as a model for advanced human hepatocellular carcinoma [J].PLoSOne, 2013,8(8): e71826.
[30] XIE Y, YAN J, CUTZ J C,etal. IQGAP2, A candidate tumour suppressor of prostate tumorigenesis [J].BiochimBiophysActa, 2012,1822(6): 875-884.
[31] SCHMIDT V A, CHIARIELLO C S, CAPILLA E,etal. Development of hepatocellular carcinoma in Iqgap2-deficient mice is IQGAP1 dependent [J].MolCellBiol, 2008,28(5): 1489-1502.
[32] KUNIMOTO K, NOJIMA H, YAMAZAKI Y,etal. Involvement of IQGAP3, a regulator of Ras/ERK-related cascade, in hepatocyte proliferation in mouse liver regeneration and development [J].JCellPhysiol, 2009,220(3): 621-631.
[33] NOJIMA H, ADACHI M, MATSUI T,etal. IQGAP3 regulates cell proliferation through the Ras/ERK signalling cascade [J].NatCellBiol, 2008,10(8): 971-978.
Abstract: IQGAP family proteins present widely in eukaryotic lineages, and play important roles in the process of cell signal transduction, cell skeletal movement and cell division. Analysis of the molecular evolution of the IQGAP protein family is helpful in understanding the function of the whole IQGAP protein family in different species. Here we retrieved 70 members of the IQGAP family in 35 eukaryotic species, and carried a comprehensive phylogenetic analysis. We found that IQGAP family proteins exist in Metazoan, Fungi, Amoebozoa, as well as some other eukaryotes, suggesting that the common ancestor of IQGAP family present in the Last Eukaryotic Common the Ancestor (LECA). Phylogenetic analysis also showed that vertebrate IQGAP genes shared a common ancestral gene with several representative non vertebrate IQGAP genes, and a single ancestral IQGAP gene duplicated two times in early vertebrates (before tetrapoda and teleostei divergence), resulting in three groups of IQGAP genes (IQGAP1, IQGAP2, IQGAP3) formed in vertebrates.
Keywords: Eukaryota;IQGAP family; Phylogenetic analysis
PhylogeneticAnalysisofIQGAPProteinsinEukaryoticLineages
REZIYA Wumaier1,2, CAO Lihuan1,2, YU Long1,2
(1.StateKeyLaboratoryofGeneticEngineering,InstituteofGenetics,Shanghai200438,China; >2.SchoolofLifeSciences,FudanUniversity,Shanghai200438,China)
Q51
A
0427-7104(2017)03-0374-09
2016-09-09
国家自然科学基金(31071186)
热孜亚·吾买尔(1990—),女,硕士研究生;余 龙,男,教授,通信联系人,E-mail: longyu@fudan.edu.cn;曹立环,男,副教授,通信联系人,E-mail: lihuancao@fudan.edu.cn.
