肠道菌群及其代谢产物在结直肠癌中的研究进展
2017-09-29孔程高仁元黄林生张鹏秦环龙
孔程 高仁元 黄林生 张鹏 秦环龙
肠道菌群及其代谢产物在结直肠癌中的研究进展
孔程 高仁元 黄林生 张鹏 秦环龙
理解肠道微生态和代谢的改变,鉴定可靠的标志物对结直肠癌早期筛查和早期诊断至关重要。近期的研究发现,肠道菌群及其代谢产物形成的多样化和复杂的肠道生态环境深刻地影响肠内稳态和疾病状态。本文主要介绍了结直肠癌患者肠道菌群及其代谢产物的研究,并集中评述了它们用于癌症早期诊断的最新进展。
结直肠肿瘤; 代谢; 肠道菌群; 研究进展
结直肠癌(colorectal cancer,CRC)在男性和女性中都是第三大常见癌症。结直肠癌有许多危险因素,包括吸烟、饮酒、高脂肪低纤维饮食、加工红肉或酒精的微生物代谢物介导的肠道菌群的改变等,特别是西方化生活方式引起的普遍肥胖和体能活动不足,增加了结直肠癌的发病率[1-5]。
一、结直肠癌的危险因素
目前研究发现酒精通过两个途径增加CRC风险:(1)醇代谢途径:乙醇氧化成乙醛,导致细胞暴露于潜在致癌代谢物乙醛中[6];(2)叶酸代谢途径:酒精作为叶酸拮抗剂导致叶酸缺乏,进而导致DNA低甲基化和异常DNA合成[7-8]。相反,高叶酸摄入或低酒精消费可降低CRC风险[9]。而饮食可以调节肠道微生物的组成,例如长期高蛋白高脂饮食可增加拟杆菌门等胆汁耐受性微生物的丰度,并减少产丁酸盐的厚壁菌门丰度[10],而碳水化合物饮食可增加普雷沃氏菌属丰度[11]。饮食依赖的微生物定植还可改变宿主表观遗传:西方化的饮食模式抑制了多糖饮食相关微生物对宿主染色质的作用,抑制了微生物短链脂肪酸的产生,而补充短链脂肪酸的无菌小鼠可以重新观察到与肠菌定植相关的表观遗传表型[12]。此外,高红肉饮食促进产硫化氢的硫酸盐还原细菌的生长,硫化氢是一种遗传毒性剂[13]。还发现红肉消费和癌症富集细菌(另支菌属、嗜胆菌属、拟杆菌属)丰度呈正相关[14],这可能与长期高红肉饮食增加了氨基酸利用细菌的丰度有关[14]。研究发现,结直肠癌的早期检测有助于提高患者的生存率,其中一期患者的5年生存率为93%,而四期患者仅为8%[15]。
二、结直肠癌肠道菌群改变
人类肠道含有约100万亿个微生物,包括约500~1 000个不同种群[16]。所有这些微生物的基因组被定义为微生物组,据估计其包含的基因数量是人类基因组基因数量的150倍。这些肠道微生物群与其宿主存在共生关系,在宿主代谢、免疫系统发育和抗病原体定植中发挥重要作用[17]。虽然微生物可以为宿主提供有益的营养性代谢信号,但它也可能成为疾病的风险因素,这取决于环境和宿主的易感性[18]。肠道微生物群及其形成的多样化和复杂的生态群落深刻地影响肠内稳态和疾病状态。结直肠癌患者肠道微生物群的细菌组成变化已经在许多研究中报道,目前已经被确认在结直肠癌发生中起作用的菌种包括牛链球菌、幽门螺杆菌、脆弱拟杆菌、粪肠球菌、败血梭状芽胞杆菌、具核梭杆菌和大肠杆菌[19]。通过比较CRC和非CRC患者或相应动物模型的肠道微生物群,发现肠道微生物主要由四个细菌门(厚壁菌门、拟杆菌门、变形菌门和放线菌门)组成[2]。Ahn等学者发现CRC患者肠道细菌多样性减少,拟杆菌门(如阿托波菌属、促炎症卟啉单胞菌属)和梭杆菌门(梭形杆菌属)富集,其可诱发结肠炎并增加CRC风险,而厚壁菌门(包括瘤胃球菌属、梭菌属)丰度减少[20]。类似的,Sinha等[21]人通过比较结直肠癌患者和健康成人粪便菌群多样性的变化,也发现结直肠癌患者粪便中拟杆菌门(卟啉单胞菌属)和梭杆菌门(梭杆菌属)丰度较高,而厚壁菌门(梭菌属)、毛螺菌属丰度较低。综合类似的研究,结直肠癌或腺瘤患者肠道微生物群中拟杆菌门(阿托波菌属、卟啉单胞菌属)、梭杆菌门(梭形杆菌属、消化链球菌、Mogibacterium菌)、微小微单胞菌、大肠杆菌和脱硫弧菌占优势,而健康成人肠道内厚壁菌门(双歧杆菌属、瘤胃球菌属、Blautia菌、梭菌属)、毛螺菌属、奇异菌属、韦荣球菌属丰度相对较高[22-27]。但Zackular等[28-29]研究结果与以上研究相反,而与Liang等学者的研究发现一致,其发现厚壁菌门(瘤胃球菌属、梭菌属)为结直肠癌患者肠道中的优势菌种,而拟杆菌属丰度则较低。另外,Ohigashi等[30]学者发现肠杆菌科水平在结直肠癌患者粪便中显著降低,虽然结直肠癌组中短链脂肪酸浓度显著降低,但是肠道pH增加,因此推测结直肠癌患者肠道微生物群的改变,短链脂肪酸的降低和pH的升高可能与结直肠癌发病有关。结直肠癌受致病微生物的丰度、多样性、代谢和免疫启动等机制的影响,但仍需更多的研究来阐明这些机制[31]。研究人员发现高丰度的梭杆菌属通过激活NF-kB信号通路诱导结直肠癌的发生[22-23]。这些研究阐明了结直肠癌和微生物群变化之间的可能机制,是解决形成结直肠癌与肠道菌群之间复杂相互作用的第一步,为肠道菌群在肿瘤个体化治疗中的应用提供了理论依据。
三、结直肠癌代谢改变
结直肠癌的诊断和治疗进展相对缓慢的原因之一是这种恶性疾病的生物学机制非常复杂。目前的实验研究多集中在癌症相关的基因表达,而较少致力于确定这种表达改变如何引起结直肠癌下游代谢产物的异常。代谢组学通过定量收集代谢产生的低分子量化合物,如蛋白质、脂质、小肽、维生素和其它蛋白质辅助因子[32]。代谢产物是细胞代谢的最终产物,其浓度反映了组织的功能状态,且与被观察对象的表型密切相关。当有机体处于异常状态时,会出现生物学通路和代谢物浓度的改变。了解结直肠癌发生和进展中代谢表型的改变可以改善诊断试验,发现新的治疗和预防结直肠癌的途径。大量证据表明,肠道微生物群不仅通过特定病原体致癌,而且通过细菌的代谢物影响而致癌[16]。通过鉴定癌症相关细菌,了解细菌代谢在健康和疾病中贡献的差异,微生物及其代谢标志物将可用于肠道疾病的预测[33]。本节总结了结直肠癌患者粪便、血液代谢物和肠道细菌代谢产物的研究(表1)。
(一)粪便代谢物变化
由于粪便与结直肠的直接接触和短暂停留,其携带有大量关于结肠和直肠的健康/疾病状态的有用信息,而且粪水或粪便代谢物的提取是一种廉价的、可重复的和有效的检测结直肠癌标志物的方法。通过综合基于粪便的代谢组学分析结果,我们将会对结直肠癌有更深入的理解。
磷脂酰胆碱、胆碱和肉碱等膳食营养物质被肠道微生物群特异性加工并产生三甲胺(trimethylamine,TMA),TMA在肠道中被吸收并在肝黄素单加氧酶(favin-containing monooxygenase,FMO)作用下在肝脏中转化为氧化三甲胺(trimethylamine oxide,TMAO)[34]。人类食用肉类和鸡蛋等食物会增加TMAO水平,而TMAO水平又与冠状动脉心脏病患者心血管事件发生风险呈正相关,这些发现进一步支持肠道菌群参与TMAO的产生[34]。Xu等[35]学者研究饮食、微生物组代谢和疾病表观遗传学之间的相互作用,发现TMAO相关基因与CRC相关基因在免疫系统、细胞周期、癌症途径和Wnt信号通路中有着共同的基因通路,TMAO可能是连接高蛋白高脂肪饮食、肠道微生物群代谢和CRC风险的一个重要的中间标志物。
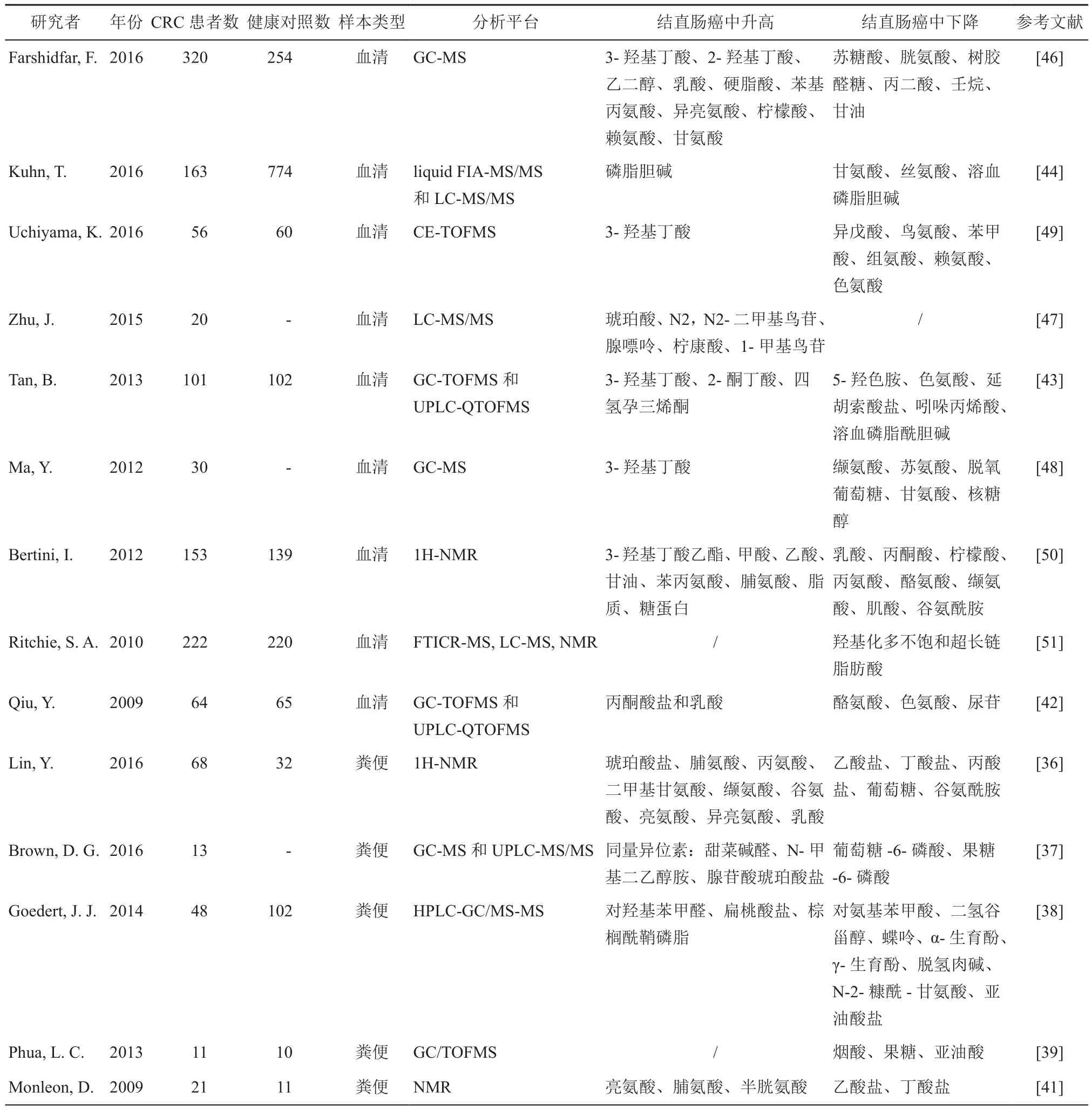
表1 结直肠癌患者血液和粪便代谢物改变
综合不同研究的结直肠癌粪便代谢物分析结果,确定的粪便代谢物大多属于氨基酸、无机酸、醛、脂肪酸、脂肪酸酯和多元醇等,这些粪便代谢物的改变可能与正常菌群的破坏、营养物质吸收不良、糖酵解和谷氨酰胺分解增加有关[36]。结直肠癌患者粪便中谷氨酸、脯氨酸、半胱氨酸、肌氨酸、亮氨酸、异亮氨酸、缬氨酸、琥珀酸盐、对羟基苯甲醛、棕榈酰鞘磷脂和N-甲基二乙醇胺等代谢物含量明显增加,而乙酸盐、丙酸盐、丁酸盐、葡萄糖、果糖、肉碱、谷氨酰胺、烟酸和亚油酸水平显著减少[36-41]。其中脯氨酸和半胱氨酸作为大多数结肠上皮粘液糖蛋白的主要组成部分,其浓度在结直肠癌患者粪便样本中显著升高[41];棕榈酰鞘磷脂、共轭亚油酸和对氨基苯甲酸作为梭形杆菌属和卟啉单胞菌属的代谢产物,通过影响细胞脱落、炎症和先天免疫参与结直肠癌进展[21];谷氨酸和琥珀酸盐是葡萄糖分解的中间产物,两者含量的升高可能与癌症糖酵解上调有关[36];总之,上述研究中总结的各种不同的代谢产物提供了大量关于结直肠癌代谢途径改变的重要信息。
(二)血液代谢物变化
血液标本的收集创伤小、患者易于接受。血清代谢谱在结直肠癌的早期筛选中有很大的潜力,且有助于了解结直肠癌发生发展的潜在机制。
在血清或血浆代谢组学研究中观察到三羧酸循环、尿素循环、糖酵解、精氨酸和脯氨酸代谢、脂肪酸代谢和油酰胺代谢的改变[42-43]。综合不同的研究结果,3-羟基丁酸、甲酸、乙酸、苯丙氨酸、脯氨酸、琥珀酸、1-甲基鸟苷、脂质和糖蛋白含量在结直肠癌患者血液样本中升高,而柠檬酸、丙酮酸、丙氨酸、酪氨酸、缬氨酸、肌酸、谷氨酰胺、苏氨酸、脱氧葡萄糖、核糖醇、鸟氨酸、组氨酸、丝氨酸、色氨酸、乳酸、羟基化多不饱和超长链脂肪酸、溶血磷脂酰胆碱含量减少[42-49]。其中3-羟基丁酸、甲酸、乙酸等短链脂肪酸,溶血磷脂酰胆碱等脂质,酪氨酸、缬氨酸、色氨酸和苯丙氨酸等氨基酸为多数研究中的共性代谢产物。
乳酸、丙酮酸、丙氨酸和谷氨酰胺水平的降低可能与肝脏葡萄糖摄取的增加和经肝糖异生增加有关[50]。多不饱和超长链脂肪酸的减少和长链脂肪酸的增加与结直肠癌患者脂肪酸去饱和酶的变化相关[51]。绝大多数研究发现,结直肠癌患者血液样本中3-羟基丁酸含量显著升高[43,46,48-50],该分子由脂肪酸氧化形成,其高水平由肝糖异生增加时增高的能量需求所致[50];脂肪酸水平的升高可能与溶血磷脂酰胆碱(LysoPCs)的降解增加有关[43]。脯氨酸、琥珀酸盐、丙酮酸、谷氨酰胺和葡萄糖等肠道细菌代谢物,其在结直肠癌患者血液和粪便样本中有一致的改变。
四、结直肠癌的早期筛查
除了粪便免疫化学检测(fecal immunochemical test,FIT)和结肠镜检查外,对粪菌代谢产物和菌群改变的检测用于CRC发生发展越来越受重视和关注。
(一)结直肠癌代谢产物的诊断价值
有学者对结直肠癌患者粪便、血清/血浆代谢物及特征性肠道菌群代谢产物进行鉴定,并对其诊断试验的灵敏度、特异度、准确度和受试者工作特征曲线下面积(AUC)进行评估,作为结直肠癌早期筛查的方法。Zhu等[47]建立了一组基于五种代谢物(琥珀酸、N2,N2-二甲基鸟苷、腺嘌呤、柠康酸和1-甲基鸟苷)的偏最小二乘判别分析(PLS-DA)模型,该模型与传统的结直肠癌癌胚抗原监测标记相比,灵敏度分别为94%和75%,特异性分别为94%和76%,AUC分别为0.91和0.80,有着优异的诊断效能;Ritchie等[45]应用FTICR-MS技术在三个独立的样品组中筛选出羟基化多不饱和超长链脂肪酸作为代谢生物标志物,并使用三重四极杆多反应监测靶向测定(TQ-MRM)成功验证了FTICR-MS的结果,AUC在0.85至0.98(0.91±0.04)的范围内;Farshidfar等[46]从320个结直肠癌患者和254个匹配对照的血清样本中筛选出一组存在显著差异的代谢物诊断模型并进行验证,该诊断模型的灵敏度、特异度、精确度(阳性预测值)和 AUC分别为85%、86%、89%和0.91(95%CI:0.87~0.96);Uchiyama等使用苯甲酸,组氨酸,辛酸和癸酸诊断模型来区分结直肠癌患者与健康对照。在这些代谢物中,苯甲酸显示出优异的诊断效能,其灵敏度、特异度和AUC分别为89%、82%和0.89[49];因此,代谢产物作为结直肠癌早期筛查的手段具有较好的应用价值。
(二)结直肠癌菌群的诊断价值
另有学者尝试将基于肠道菌群构建的诊断模型作为诊断标准。Baxter等[52]通过16S rRNA基因测序分析了490例结直肠癌患者、腺瘤及正常人的粪便样本,建立基于卟啉单胞菌、具核梭杆菌、微小微单胞菌、消化链球菌、兼性双球菌属和未分类的普雷沃氏菌等34个微生物群的随机森林模型并进行验证。该模型的诊断效能优异,AUC为0.847,而FIT检测的AUC为0.929,显著优于诊断模型(P=0.005)。同课题组通过对404名结直肠癌患者粪便进行16S rRNA基因测序,建立了基于具核梭杆菌、不溶性卟啉单胞菌、消化链球菌和微小微单胞菌等32个OTUs的诊断模型,该模型的AUC为0.853[26]。Zeller等[24]使用宏基因组测序技术对156名参与者的粪便样品进行检测,建立基于两种具核梭杆菌亚种(vincentii,animalis)、不溶性卟啉单胞菌和消化链球菌的诊断模型,并在来自不同国家的独立患者和对照群体(n=335)中验证,该诊断模型的AUC为0.84。若将诊断模型与粪便隐血试验组合测试,得到的AUC为0.87。因此,基于菌群的大肠癌诊断模型也具有较好的诊断效果。此外,CRC患者结肠灌洗样本的微生物多样性和均匀度与结肠活检样本相似,且没有样本大小的限制,因此结肠灌洗样本的菌群组成可代表结肠活检样本的菌群组成[53]。
五、目前的研究局限
尽管近年来结直肠癌肠道菌群及其代谢产物研究获得大量的成果,但是要将这些研究结果用于临床仍需要克服一些困难:(1)研究的样本量:目前针对结直肠癌患者肠道菌群及相关代谢组学研究的样本量普遍较少,不同的研究结果存在较大的偏移,因此进行多中心、多人群、大样本的研究将提升研究的可重复性和可靠性,从而更好的将肠道微生态与代谢组学运用于结直肠癌诊治;(2)研究的技术:目前肠道菌群和相关代谢产物检测手段仍缺乏公认的标准,各个研究和平台间存在较大的个体化差异,在质量控制和学术交流上也存在难题。同时,由于现有的分析技术平台并不能覆盖所有种类代谢物,各研究又大多使用单一分析技术平台,使代谢物种类的覆盖面更小;(3)统计学的方法:各研究因样本在体内外受多种混杂因素的影响,应用统计学方法时显然存在多种偏倚,而目前的结直肠癌研究并没有很好地去除这些混杂因素,从而影响了研究结果的可靠性。利用多重机器算法开展早期诊断模型的构建,也是目前研究的重要组成部分;(4)诊断试验的局限性:大多数研究缺乏诊断试验的敏感性和特异性数据,一些诊断试验筛选的生物标志物在不同的研究中表现出相反的变化趋势。
六、未来的研究方向
目前肠道微生态和代谢组学已成为研究热点,在各个领域的应用也越来越广泛。然而,对结直肠癌肠道微生态和代谢组学的研究还处在发展阶段,迫切需要更大样本量以及多民族,地域人群的综合性队列研究;代谢物组数据库还需完善,其分析技术手段有限,迫切需要多种技术同时分析所有的代谢产物,运用整合大数据的研究方法;增加对高危人群的筛查,进行早期干预性研究对于改善结直肠癌的预后有着重要作用,肠道微生态领域的研究也必将成为未来第二生命科学发展的趋势。
[ 1 ] Svensson T, Yamaji T, Budhathoki S, et al. Alcohol consumption,genetic variants in the alcohol- and folate metabolic pathways and colorectal cancer risk: the JPHC Study [J]. Sci Rep, 2016, 6: 36607.
[ 2 ] Tuan J, Chen YX. Dietary and Lifestyle Factors Associated with Colorectal Cancer Risk and Interactions with Microbiota: Fiber, Red or Processed Meat and Alcoholic Drinks [J]. Gastrointest Tumors,2016, 3(1): 17-24.
[ 3 ] Yang C, Wang X, Huang CH, et al. Passive Smoking and Risk of Colorectal Cancer: A Meta-analysis of Observational Studies [J].Asia Pac J Public Health, 2016, 28(5): 394-403.
[ 4 ] Goss PE, Strasser-Weippl K, Lee-Bychkovsky BL, et al.Challenges to effective cancer control in China, India, and Russia [J]. Lancet Oncol, 2014, 15(5): 489-538.
[ 5 ] Varghese C and Shin HR, Strengthening cancer control in China [J].Lancet Oncol, 2014, 15(5): 484-485.
[ 6 ] Seitz HK, Stickel F. Molecular mechanisms of alcohol-mediated carcinogenesis [J]. Nat Rev Cancer, 2007, 7(8): 599-612.
[ 7 ] Halsted CH, Villanueva JA, Devlin AM, et al., Metabolic interactions of alcohol and folate [J]. J Nutr, 2002, 132(8 Suppl): 2367S-2372S.
[ 8 ] Duthie SJ. Folic acid deficiency and cancer: mechanisms of DNA instability [J]. Br Med Bull, 1999, 55(3): 578-592.
[ 9 ] Freudenheim JL, Graham S, Marshall JR, et al. Folate intake and carcinogenesis of the colon and rectum [J]. Int J Epidemiol, 1991,20(2): 368-374.
[ 10 ] David LA, Maurice CF, Carmody RN, et al. Diet rapidly and reproducibly alters the human gut microbiome [J]. Nature, 2014,505(7484): 559-563.
[ 11 ] Wu GD, Chen J, Hoffmann C, et al. Linking long-term dietary patterns with gut microbial enterotypes [J]. Science, 2011,334(6052): 105-108.
[ 12 ] Krautkramer KA, Kreznar JH, Romano KA, et al. Diet-Microbiota Interactions Mediate Global Epigenetic Programming in Multiple Host Tissues [J]. Mol Cell, 2016, 64(5): 982-992.
[ 13 ] Albenberg LG, Wu GD. Diet and the intestinal microbiome:associations, functions, and implications for health and disease [J].Gastroenterology, 2014, 146(6): 1564-1572.
[ 14 ] Feng Q, Liang S, Jia H, et al. Gut microbiome development along the colorectal adenoma-carcinoma sequence [J]. Nat Commun, 2015,6: 6528.
[ 15 ] Sung JJ, Lau JY, Goh KL, et al. Increasing incidence of colorectal cancer in Asia: implications for screening [J]. Lancet Oncol, 2005,6(11): 871-876.
[ 16 ] Ohtani N. Microbiome and cancer [J]. Semin Immunopathol, 2015,37(1): 65-72.
[ 17 ] Zeng MY, Inohara N, and Nunez G.Mechanisms of infammationdriven bacterial dysbiosis in the gut [J]. Mucosal Immunol, 2017,10(1): 18-26.
[ 18 ] Shanahan F. The colonic microbiota and colonic disease [J]. Curr Gastroenterol Rep, 2012, 14(5): 446-452.
[ 19 ] Gagniere J, Raisch J, Veziant J, et al. Gut microbiota imbalance and colorectal cancer [J]. World J Gastroenterol, 2016, 22(2): 501-518.
[ 20 ] Ahn J, Sinha R, Pei Z, et al. Human gut microbiome and risk for colorectal cancer [J]. J Natl Cancer Inst, 2013, 105(24): 1907-1911.
[ 21 ] Sinha R, Ahn J, Sampson JN, et al. Fecal Microbiota, Fecal Metabolome, and Colorectal Cancer Interrelations [J]. PLoS One,2016, 11(3): e0152126.
[ 22 ] Kostic AD, Chun E, Robertson L, et al. Fusobacterium nucleatum potentiates intestinal tumorigenesis and modulates the tumor-immune microenvironment [J]. Cell Host Microbe, 2013, 14(2): 207-215.
[ 23 ] McCoy AN, Araujo-Perez F, Azcarate-Peril A, et al.Fusobacterium is associated with colorectal adenomas [J]. PLoS One, 2013, 8(1):e53653.
[ 24 ] Zeller G, Tap J, Voigt AY, et al. Potential of fecal microbiota for early-stage detection of colorectal cancer [J]. Mol Syst Biol, 2014,10: 766.
[ 25 ] Kohoutova D, Smajs D, Moravkova P, et al. Escherichia coli strains of phylogenetic group B2 and D and bacteriocin production are associated with advanced colorectal neoplasia [J]. BMC Infect Dis,2014, 14: 733.
[ 26 ] Baxter NT, Koumpouras CC, Rogers MA, et al. DNA from fecal immunochemical test can replace stool for detection of colonic lesions using a microbiota-based model [J]. Microbiome, 2016, 4(1): 59.
[ 27 ] Hale VL, Chen J, Johnson S, et al. Shifts in the fecal microbiota associated with adenomatous polyps [J]. Cancer Epidemiol Biomarkers Prev, 2016.
[ 28 ] Zackular JP, Rogers MA, Ruffin MTt, et al. The human gut microbiome as a screening tool for colorectal cancer [J]. Cancer Prev Res (Phila), 2014, 7(11): 1112-1121.
[ 29 ] Liang JQ, Chiu J, Chen Y, et al. Fecal Bacteria Act as Novel Biomarkers for Non-Invasive Diagnosis of Colorectal Cancer [J].Clin Cancer Res, 2017, 26(1): 85-94.
[ 30 ] Ohigashi S, Sudo K, Kobayashi D, et al. Changes of the intestinal microbiota, short chain fatty acids, and fecal pH in patients with colorectal cancer [J]. Dig Dis Sci, 2013, 58(6): 1717-1726.
[ 31 ] Kelly DL, Lyon DE, Yoon SL, et al. The Microbiome and Cancer:Implications for Oncology Nursing Science [J]. Cancer Nurs, 2016,39(3): E56-62.
[ 32 ] Claudino WM, Quattrone A, Biganzoli L, et al. Metabolomics:available results, current research projects in breast cancer, and future applications [J]. J Clin Oncol, 2007, 25(19): 2840-2846.
[ 33 ] Belcheva A, Irrazabal T, and Martin A. Gut microbial metabolism and colon cancer: can manipulations of the microbiota be useful in the management of gastrointestinal health? [J]. Bioessays, 2015,37(4): 403-412.
[ 34 ] Herbert Tilg MD. A Gut Feeling about Thrombosis [J]. N Engl J Med, 2016, 23;374(25): 2494-2496.
[ 35 ] Xu R, Wang Q, and Li L. A genome-wide systems analysis reveals strong link between colorectal cancer and trimethylamine N-oxide(TMAO), a gut microbial metabolite of dietary meat and fat [J].BMC Genomics, 2015, 16 (Suppl 7): S4.
[ 36 ] Lin Y, Ma C, Liu C, et al.NMR-based fecal metabolomics fingerprinting as predictors of earlier diagnosis in patients with colorectal cancer [J]. Oncotarget, 2016, 7(20): 29454-29464.
[ 37 ] Brown DG, Rao S, Weir TL, et al. Metabolomics and metabolic pathway networks from human colorectal cancers, adjacent mucosa,and stool [J]. Cancer Metab, 2016, 4: 11.
[ 38 ] Goedert JJ, Sampson JN, Moore SC, et al. Fecal metabolomics:assay performance and association with colorectal cancer [J].Carcinogenesis, 2014, 35(9): 2089-2096.
[ 39 ] Phua LC, Koh PK, Cheah PY, et al. Global gas chromatography/time-of-fight mass spectrometry (GC/TOFMS)-based metabonomic profiling of lyophilized human feces [J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2013, 937: 103-113.
[ 40 ] Metallo CM. Expanding the reach of cancer metabolomics [J].Cancer Prev Res (Phila), 2012, 5(12): 1337-1340.
[ 41 ] Monleon D, Morales JM, Barrasa A, et al. Metabolite profling of fecal water extracts from human colorectal cancer [J]. NMR Biomed,2009, 22(3): 342-348.
[ 42 ] Qiu Y, Cai G, Su M, et al. Serum metabolite profiling of human colorectal cancer using GC-TOFMS and UPLC-QTOFMS [J]. J Proteome Res, 2009, 8(10): 4844-4850.
[ 43 ] Tan B, Qiu Y, Zou X, et al. Metabonomics identifies serum metabolite markers of colorectal cancer [J]. J Proteome Res, 2013,12(6): 3000-3009.
[ 44 ] Kuhn T, Floegel A, Sookthai D, et al. Higher plasma levels of lysophosphatidylcholine 18: 0 are related to a lower risk of common cancers in a prospective metabolomics study [J]. BMC Med, 2016,14: 13.
[ 45 ] Ritchie SA, Ahiahonu PW, Jayasinghe D, et al. Reduced levels of hydroxylated, polyunsaturated ultra long-chain fatty acids in the serum of colorectal cancer patients: implications for early screening and detection [J]. BMC Med, 2010, 8: 13.
[ 46 ] Farshidfar F, Weljie AM, Kopciuk KA, et al. A validated metabolomic signature for colorectal cancer: exploration of the clinical value of metabolomics [J]. Br J Cancer, 2016, 115(7): 848-857.
[ 47 ] Zhu J, Djukovic D, Deng L, et al. Targeted serum metabolite profiling and sequential metabolite ratio analysis for colorectal cancer progression monitoring [J]. Anal Bioanal Chem, 2015,407(26): 7857-7863.
[ 48 ] Ma Y, Zhang P, Wang F, et al.An integrated proteomics and metabolomics approach for defining oncofetal biomarkers in the colorectal cancer [J]. Ann Surg, 2012, 255(4): 720-730.
[ 49 ] Uchiyama K, Yagi N, Mizushima K, et al. Serum metabolomics analysis for early detection of colorectal cancer [J]. J Gastroenterol,2017, 52(6): 677-694.
[ 50 ] Bertini I, Cacciatore S, Jensen BV, et al. Metabolomic NMR fingerprinting to identify and predict survival of patients with metastatic colorectal cancer [J]. Cancer Res, 2012, 72(1): 356-364.
[ 51 ] Ritchie SA, Tonita J, Alvi R, et al. Low-serum GTA-446 anti-inflammatory fatty acid levels as a new risk factor for colon cancer [J]. Int J Cancer, 2013, 132(2): 355-362.
[ 52 ] Baxter NT, Ruffn MTt, Rogers MA, et al. Microbiota-based model improves the sensitivity of fecal immunochemical test for detecting colonic lesions [J]. Genome Med, 2016, 8(1): 37.
[ 53 ] Watt E, Gemmell MR, Berry S, et al.Extending colonic mucosal microbiome analysis-assessment of colonic lavage as a proxy for endoscopic colonic biopsies [J]. Microbiome, 2016, 4(1): 61.
Research progression of gut microbiota and its metabolites in colorectal cancer
Kong Cheng,Gao Renyuan, Huang Linsheng, Zhang Peng, Qin Huanlong.
Department of GI Surgery, Shanghai Tenth People′s Hospital Affliated to Tongji University; Research Institute for Intestinal Diseases of Tongji University School of Medicine, Shanghai 200072, China
Qin Huanlong, Email: qinhuanlong@126.com
Understanding of intestinal micro-ecological and metabolic changes, identifcation of reliable markers of early colorectal cancer screening and early diagnosis is essential. Recent studies have found that gut microbiota and its metabolites formed a variety of complex intestinal ecological environment which profoundly affect the intestinal stability and disease state. This review focuses on the study of gut microbiota and its metabolites in colorectal cancer patients, and reviews the recent progress of then for the early diagnosis of cancer.
Colorectal neoplasms; Metabolism; Gut microbiota; Study progress
2017-01-02)
(本文编辑:赵志勋)
10.3877/cma.j.issn.2095-3224.2017.05.015
国家自然科学基金(No.81230057;No.81472262;No.81302066);上海新兴前沿技术联合攻关项目(No.SHDC 12012106)
200072 上海,同济大学附属第十人民医院普外科;同济大学医学院肠道疾病研究所
秦环龙,Email:qinhuanlong@126.com
孔程, 高仁元, 黄林生, 等. 肠道菌群及其代谢产物在结直肠癌中的研究进展[J/CD]. 中华结直肠疾病电子杂志, 2017,6(5): 421-426.
