基于线粒体COXⅠ基因变异探讨国内外猪种的遗传多样性及系统进化研究
2017-09-03周秀敏杨永江毕英杰任卫合
周秀敏,杨永江,毕英杰,任卫合,张 丽
(西北民族大学 生命科学与工程学院,甘肃 兰州730030)
基于线粒体COXⅠ基因变异探讨国内外猪种的遗传多样性及系统进化研究
周秀敏,杨永江,毕英杰,任卫合,张 丽*
(西北民族大学 生命科学与工程学院,甘肃 兰州730030)
基于mtDNACOXⅠ基因,探讨了6个猪种687个样本(大白猪、长白猪、杜洛克、藏猪、甘肃黑猪和八眉猪)的遗传多样性和各猪种间的亲缘关系。对各猪种样本mtDNACOXⅠ基因构建混合池,并利用直接测序技术获得6个猪种COXⅠ基因的序列,采用MEGA 6.0分析软件基于Kimurk双参数模型应用邻接法构建系统发生树。结果表明,6个猪种mtDNACOXⅠ基因序列共存在21个突变位点,其中8个突变为各群体特有。长白猪与杜洛克猪的多态性较丰富,含有13个相同位点的突变,核苷酸的转换数(si)和颠换数(sv)的比值(R)分别为12和15,序列替换远未达到饱和。而藏猪、黑猪和八眉猪的多态性较贫乏。系统进化树和遗传距离分析显示,6个猪种及野猪先聚为一支而后与同属为偶蹄目的牛、羊聚为一支,3个地方猪种间遗传距离较近。外来猪种引入中国后主要是用作父本以提高生产速度和瘦肉率等,对本地猪种未有母系贡献,线粒体COXⅠ基因可有效区别6个猪种的亲缘关系,在一定程度上为中国地方猪品种的有效保护和合理利用提供理论依据。
猪;线粒体细胞色素C氧化酶Ⅰ亚基;系统地理学
线粒体DNA(mitochondrial DNA,mtDNA)是存在于细胞质中的共价闭合环状双链DNA分子,是独立于细胞核染色体外的基因组,具有自我复制、转录、编码等功能,同时受核DNA控制[1]。与核DNA相比,线粒体DNA具有分子结构简单、以母系遗传方式遗传、核苷酸歧异度大、进化速度快等特点,可以用来研究种群进化关系和物种鉴定[2]。mtDNA拷贝远多于核基因组DNA,核DNA降解时,仍有可能存在mtDNA。因此利用mtDNA成为判断物种亲缘关系远近最为有力的工具。线粒体DNA轻链上编码基因包括ND6和7个tRNA基因,重链上编码基因包括NADH-脱氢酶的7个亚基,细胞色素b(cytb),ATP合成酶的两个亚基,细胞色素C氧化酶的3个亚基(COXⅠ、COXⅡ、COXⅢ),2个rRNA以及位于tRNAPro和tRNAPhe之间的一段不编码任何基因的替代环区(D-loop)组成。
线粒体细胞色素C氧化酶Ⅰ亚基(mitochondrial cytochrome C oxidase subunit 1,COXⅠ)为独立于核基因组以外的线粒体环状DNA编码的呼吸链细胞色素氧化酶,普遍存在于原核生物和真核生物中,它的进化速度快,包含从种内到种间的遗传进化信息,可用于比较生物体进化之间的相互关系。目前关于线粒体COXⅠ的研究多见于寄生虫种间和种内的遗传变异[3-5],而有关大白猪、长白猪、杜洛克猪与青海八眉猪、甘肃黑猪、藏猪的研究还未见报道。本研究以3个地方猪种和3个国外猪种为研究对象,探讨引进猪种与我国地方猪种系统地理学及起源,为我国地方猪品种的有效保护和合理利用提供有价值的理论依据。
1 材料与方法
1.1 试验材料
本研究选用3个引进猪种(大白猪、长白猪、杜洛克)和3个地方猪种(八眉猪、藏猪、甘肃黑猪)共687头作为试验群体(表1)。于仔猪出生后一周内采集耳组织,每头仔猪剪取耳组织3~5 g,置于装有75%乙醇的离心管中,-70 ℃冰箱冷冻保存。
1.2 基因组DNA提取和DNA池的构建
采用常规的苯酚-氯仿抽提法[6]进行耳组织DNA提取,用1.0%琼脂糖凝胶电泳检测DNA 提取效果,经紫外分光光度计检测每个DNA 样品浓度,加双蒸水调整DNA样品浓度至100 ng·μL-1,各取5 μL构建DNA混合池。
1.3 引物设计、PCR扩增与测序
根据GenBank中发布的野猪线粒体COXⅠ基因序列(GenBank No.: EU333163),应用Primer6.0软件设计1对特异性引物,用于扩增3个引进猪种和3个地方猪种线粒体COXⅠ基因序列。其引物序列为:F: 5′-GACATTCACCACGGAACT-3′,R: 5′-GAAAGGGTAAGCCATAGAG-3′,引物由杭州金唯智生物科技有限公司合成,扩增片段为1 790 bp。PCR扩增体系总体积为20 μL:基因组DNA模板0.8 μL,上下游引物各0.4 μL,TaqPCR MasterMix 11.0 μL,ddH2O 7.4 μL。PCR扩增程序为:94 ℃增预变性3 min;94 ℃变性30 s,61 ℃退火40 s,72 ℃延伸30 s,34个循环;72 ℃延伸10 min,4 ℃保存;PCR产物经1%琼脂糖凝胶电泳检测后特异性良好的PCR产物经凝胶回收试剂盒(北京天根)纯化后送至杭州金唯智生物科技有限公司测序。测序结果用DNAStar、MegAlign和Editseq软件比对和拼接获得猪线粒体COXⅠ基因编码区序列。
表1 试验猪群体、样本数量及来源
Table 1 Population, numbers and origin of pig samples
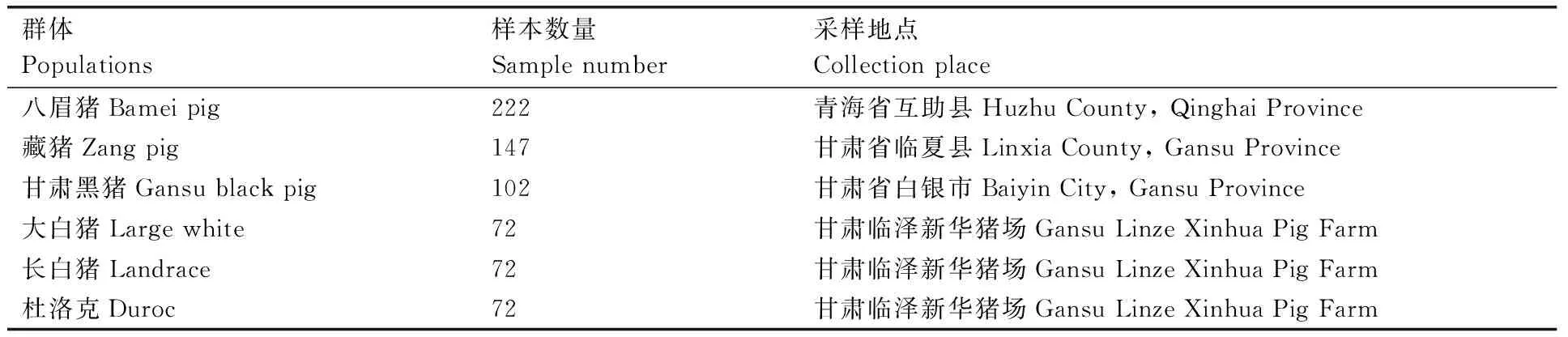
群体Populations样本数量Samplenumber采样地点Collectionplace八眉猪Bameipig222青海省互助县HuzhuCounty,QinghaiProvince藏猪Zangpig147甘肃省临夏县LinxiaCounty,GansuProvince甘肃黑猪Gansublackpig102甘肃省白银市BaiyinCity,GansuProvince大白猪Largewhite72甘肃临泽新华猪场GansuLinzeXinhuaPigFarm长白猪Landrace72甘肃临泽新华猪场GansuLinzeXinhuaPigFarm杜洛克Duroc72甘肃临泽新华猪场GansuLinzeXinhuaPigFarm
1.4 统计分析
应用EditSeq和MegAlign软件进行序列比对突变位点。利用序列图谱分析软件BioEdit和MWSnap来测量各SNPs位点等位基因的峰高,根据公式Fi=hi/(h1+h2)估算等位基因频率,其中Fi表示SNP位点某等位基因频率(i=1,2),h1和h2分别表示测序图上该SNP等位基因1和2的峰高度。用MEGA 6.0分析软件基于Kimurk双参数模型应用邻接法构建系统发生树,分析结果探讨引进猪种与中国地方猪种系统地理学及起源。
2 结果与分析
2.1 猪线粒体COXⅠ基因PCR扩增
线粒体COXⅠ基因扩增后,经1%琼脂糖凝胶(120 V,20 min)电泳检测后均得到一条特异性好、条带清晰、大小为1 500~2 000 bp的目的片段,且与预期的目的片段大小一致(图1)。
2.2 猪线粒体COXⅠ基因测序峰图、突变位点筛查及等位基因频率估算
将大白猪、长白猪、杜洛克、甘肃黑猪、八眉猪和藏猪线粒体COXⅠ基因与GenBank中野猪COXⅠ基因序列进行比对剪切(图2—图7),分别获得6个猪种COXⅠ基因编码区序列1 545 bp,共存在21个突变位点,国外猪种突变位点(16个)明显多于国内猪种(5个),其中c.336A>T、c.433C>T这两个突变位点为大白猪、长白猪、杜洛克所共有(表2、表3)。
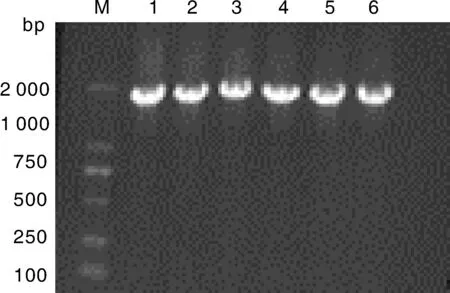
M为Marker2000;1-6泳道依次为大白猪、长白猪、杜洛克、八眉猪、甘肃黑猪、藏猪M represents Marker2000; 1-6 Lane are PCR amplifications of Large white, Landrace, Duroc, Bamei pig, Gansu black pigs and Zang pigs图1 猪线粒体COX Ⅰ基因PCR扩增电泳Fig.1 Detections of PCR products COX Ⅰ gene in pigs
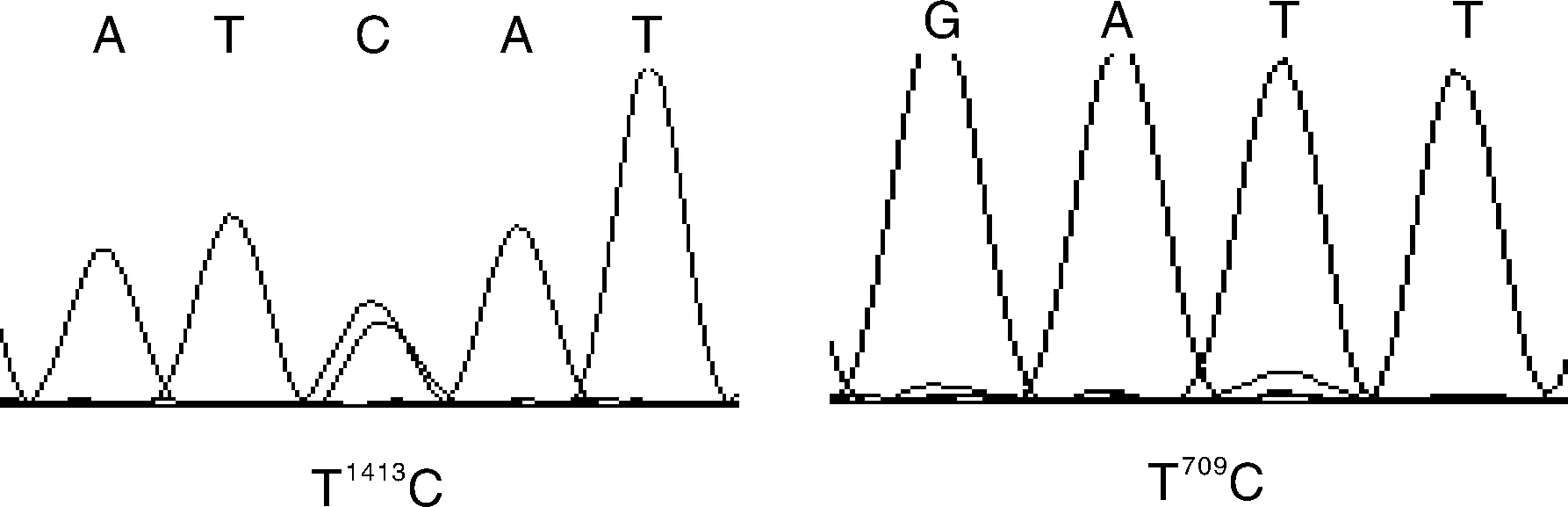
图2 甘肃黑猪COX Ⅰ基因测序峰图Fig.2 COX Ⅰ gene sequence diagram of Gansu black pigs
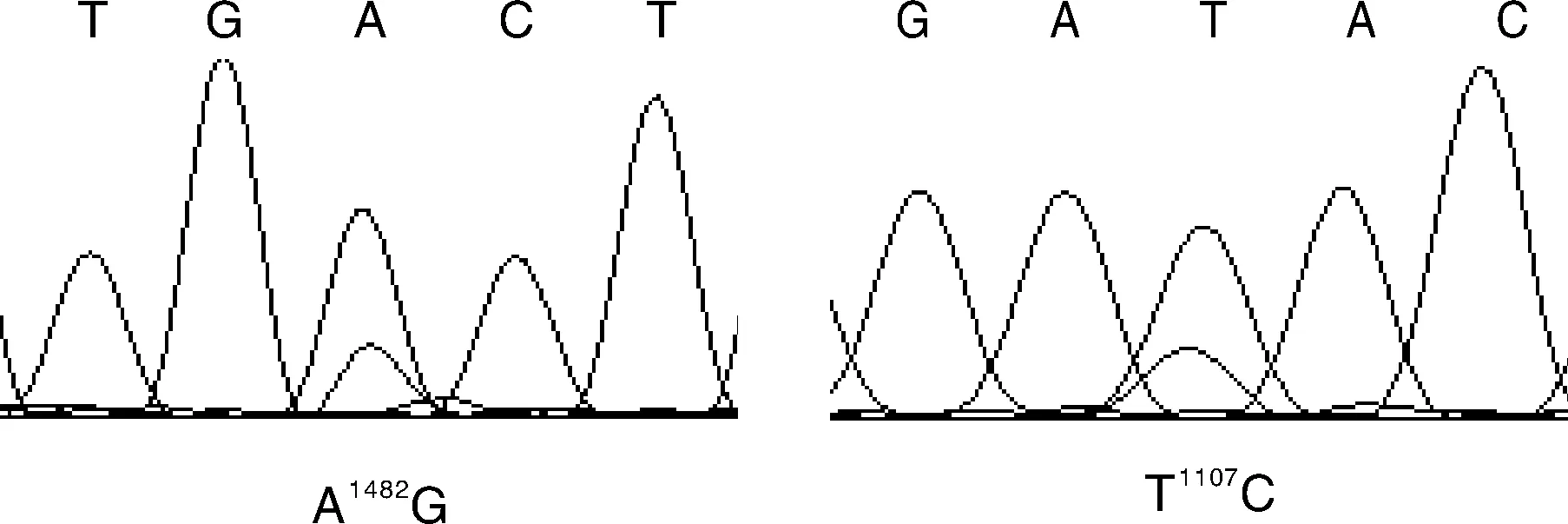
图3 八眉猪COX Ⅰ基因测序峰图Fig.3 COX Ⅰ gene sequence diagram of Bamei pigs
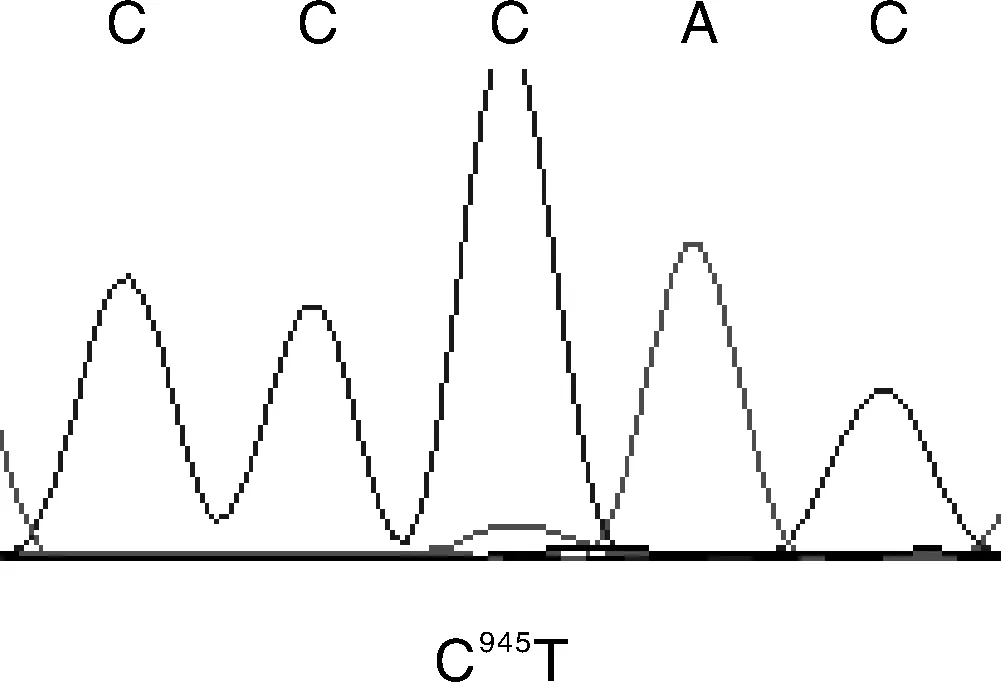
图4 藏猪COX Ⅰ基因测序峰图Fig.4 COX Ⅰ gene sequence diagram of Zang pigs
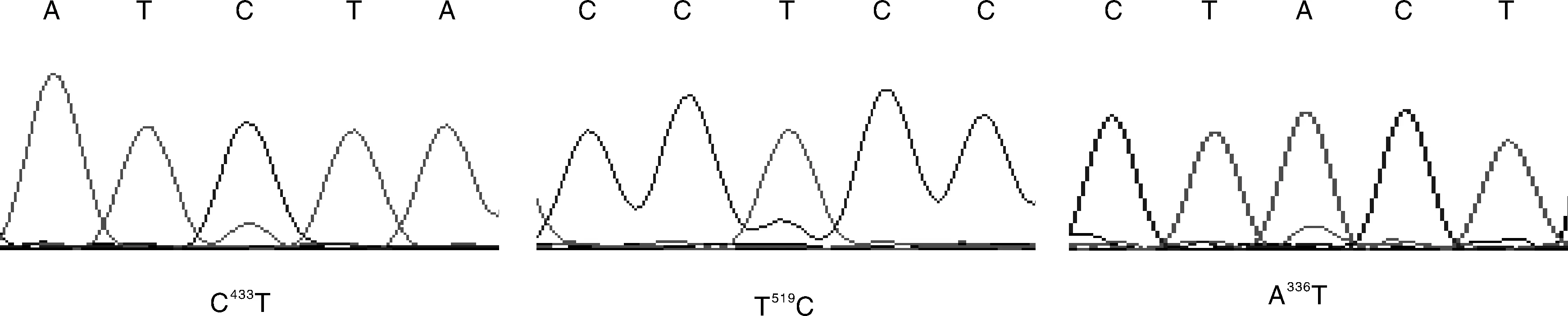
图5 大白猪COX Ⅰ基因测序峰图Fig.5 COX Ⅰ gene sequence diagram of Large white

图6 长白猪COX Ⅰ基因测序峰图Fig.6 COX Ⅰ gene sequence diagram of Landrace
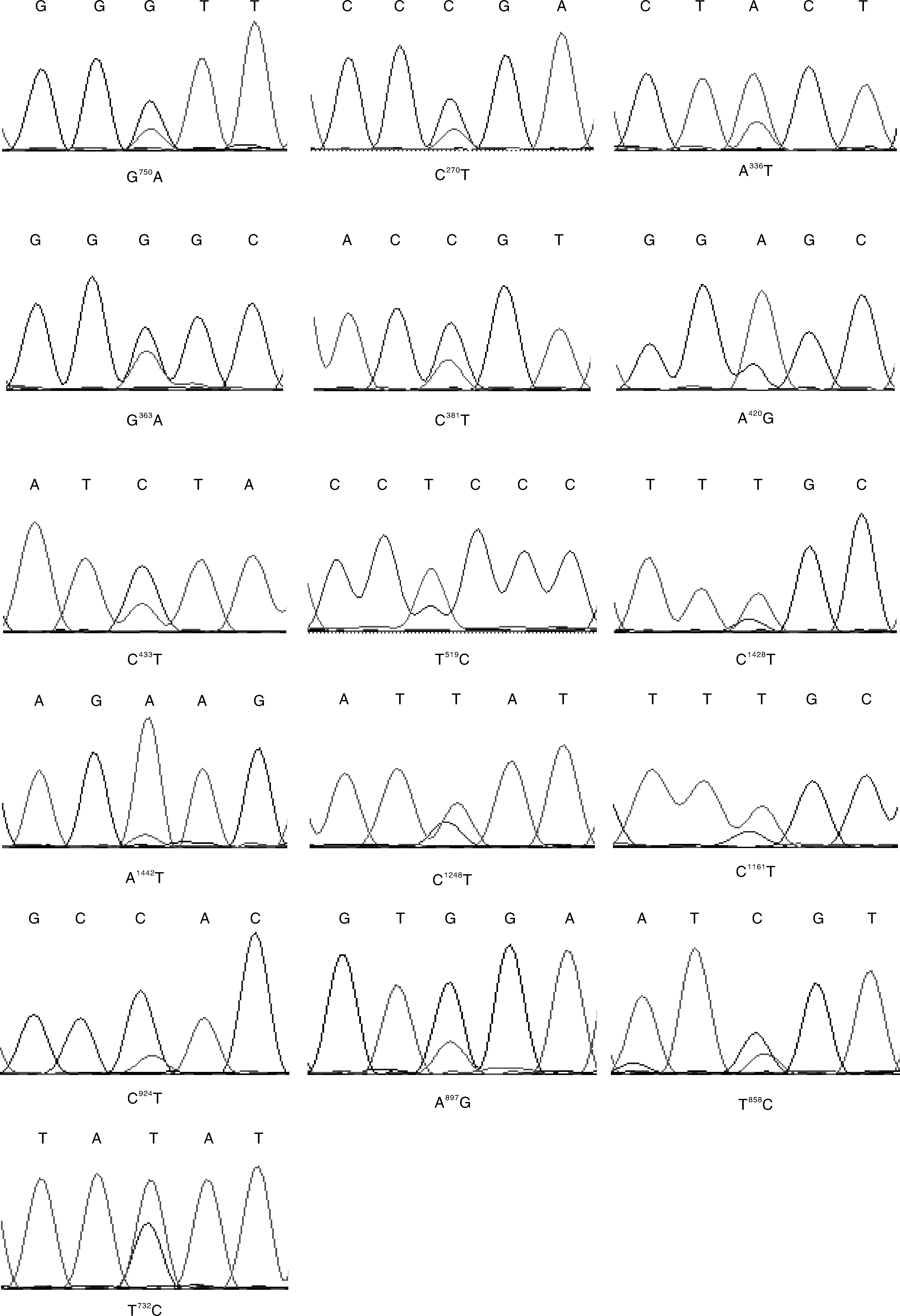
图7 杜洛克COX Ⅰ基因测序峰图Fig.7 COX Ⅰ gene sequence diagram of Duroc
表2 不同品种猪COXⅠ基因突变位点
Table 2 Mutation sites ofCOXⅠ gene in different pig breeds

群体Populations突变位点Mutantsite大白猪Largewhitec.336A>T,c.433C>T,c.519T>C长白猪Landracec.336A>T,c.363G>A,c.381C>T,c.420A>G,c.433C>T,c.732T>C,c.750G>A,c.858T>C,c.897A>G,c.924C>T,c.1161C>T,c.1248C>T,c.1428T>C杜洛克Durocc.270C>T,c.336A>T,c.363G>A,c.381C>T,c.420A>G,c.433C>T,c.519T>C,c.732T>C,c.750G>A,c.858T>C,c.897A>G,c.924C>T,c.1161C>T,c.1248C>T,c.1428T>C,c.1442A>T甘肃黑猪Gansublackpigsc.709T>C,c.1413T>C八眉猪Bameipigsc.1107T>C,c.1482A>G藏猪Zangpigsc.945C>T
表3 等位基因频率估算
Table 3 Estimation of allele frequency
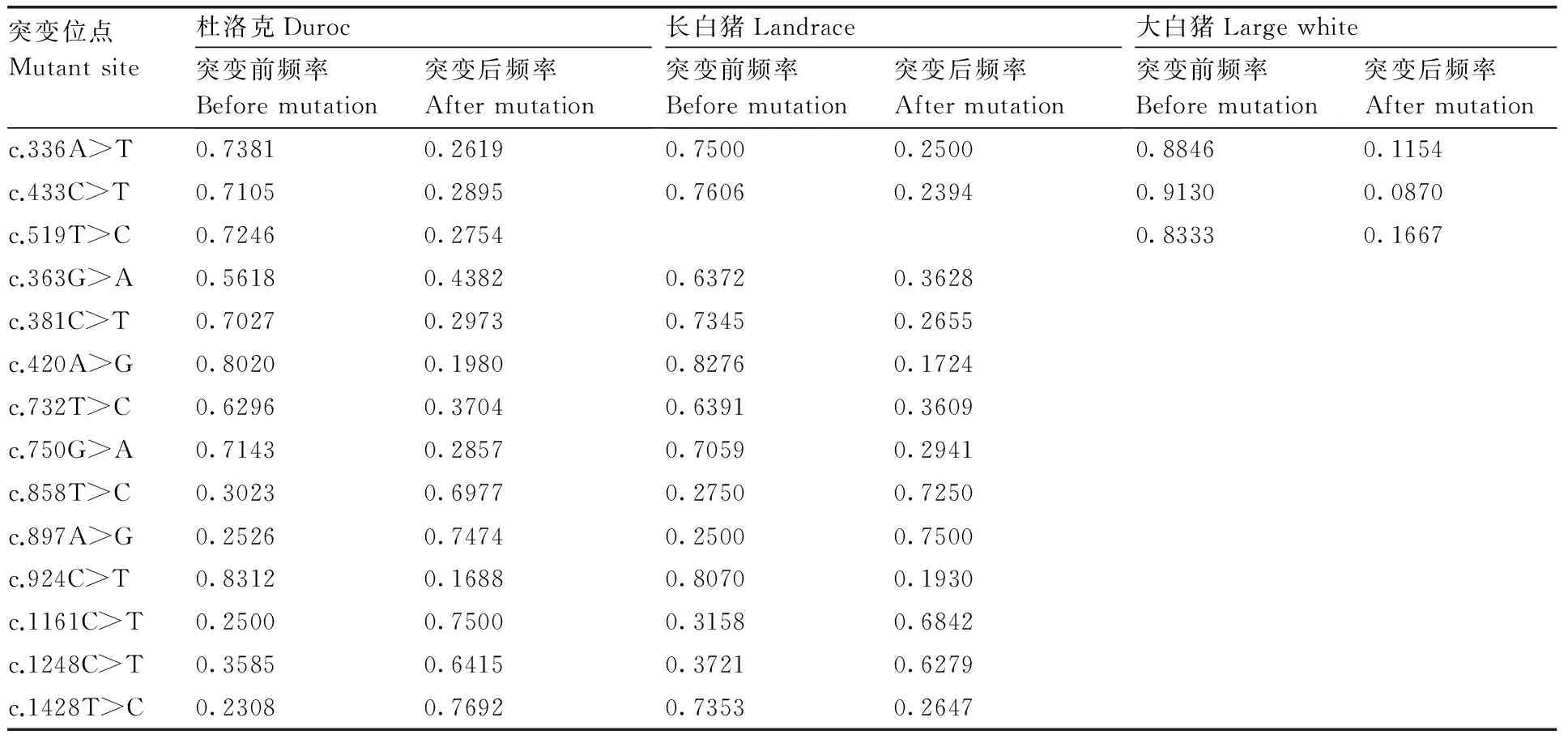
突变位点Mutantsite杜洛克Duroc突变前频率Beforemutation突变后频率Aftermutation长白猪Landrace突变前频率Beforemutation突变后频率Aftermutation大白猪Largewhite突变前频率Beforemutation突变后频率Aftermutationc.336A>T0.73810.26190.75000.25000.88460.1154c.433C>T0.71050.28950.76060.23940.91300.0870c.519T>C0.72460.27540.83330.1667c.363G>A0.56180.43820.63720.3628c.381C>T0.70270.29730.73450.2655c.420A>G0.80200.19800.82760.1724c.732T>C0.62960.37040.63910.3609c.750G>A0.71430.28570.70590.2941c.858T>C0.30230.69770.27500.7250c.897A>G0.25260.74740.25000.7500c.924C>T0.83120.16880.80700.1930c.1161C>T0.25000.75000.31580.6842c.1248C>T0.35850.64150.37210.6279c.1428T>C0.23080.76920.73530.2647
2.4 系统发生树的构建
将从GenBank检索到的人、牛、绵羊、鸡、鼠序列与本试验6条序列一同应用MEGA6.0进行系统发育分析(图8)。系统发生树中长白猪及杜洛克亲缘关系较近,先聚为一支,再与大白猪、八眉猪、藏猪、黑猪及野猪(EU333163.1)聚为一支,而后与同属为偶蹄目的牛(HQ025805.1)、绵羊(KT148968)聚为一支。鼠(AC-000022.2)、人(KC292251.1)、鸡(KP244335.1)各为一支,这与动物分类学基本一致。三个地方猪种间遗传距离较近,藏猪与甘肃黑猪的遗传距离为0.005,八眉猪与藏猪、甘肃黑猪间的遗传距离为0.004(表4)。
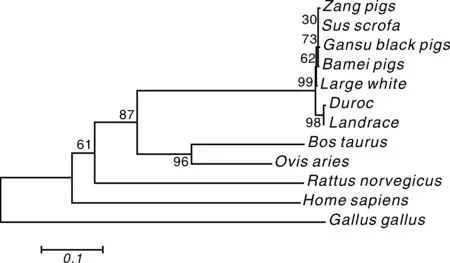
图8 应用邻接法(NJ)构建系统发生树Fig.8 Phylogenetic tree constructed by using neighbor joining method (NJ)
表4 遗传距离
Table 4 Genetic distance

名称Name藏猪Zangpig甘肃黑猪Gansublackpig杜洛克Duroc长白猪Landrace八眉猪Bameipigs大白猪Largewhite鸡Gallusgallus牛Bostaurus人Homosapiens绵羊Ovisaries鼠Rattusnorvegicus甘肃黑猪Gansublackpig0.005杜洛克Duroc0.0220.022长白猪Landrace0.0180.0180.004八眉猪Bameipig0.0040.0040.0200.017大白猪Largewhite0.0060.0060.0160.0140.005鸡Gallusgallus1.0461.0411.0861.0801.0411.052牛Bostaurus0.5750.5730.5880.5840.5730.5720.885人Homosapiens0.7590.7600.7330.7660.7600.7571.0040.688绵羊Ovisaries0.5050.5040.5080.5070.5040.5060.9630.3130.748鼠Rattusnorvegicus0.7100.7050.6980.6990.6980.6890.9700.7060.7580.660野猪Susscrofa0.0020.0020.0190.0160.0010.0041.0410.5730.7570.5040.702
3 讨论
本研究对6个猪种687个样本mtDNACOXⅠ基因构建DNA混合池,并利用直接测序技术获得6个猪种COXⅠ基因的序列。DNA混合池是指将具有某一共同特征群体的部分个体DNA提取后经过定量和稀释成一定浓度后,按比例或等量混合构成一个池。这种混合DNA样品的某些多态性位点经过PCR扩增后,其产物经过电泳、测序等方法绘制等位基因型和估算等位基因频率,确定该位点与性状的相关性[7]。利用DNA混合池和测序技术可以快速对未知SNPs或已知SNPs等位基因频率进行筛查,大幅度缩短了实验周期,显著减少了基因组DNA的消耗。但是利用这种方法进行基因频率估算也存在一定的偏差。DNA 池进行直接测序时所要求的等位基因频率最低10%[8]。利用测序峰高比值估算频率不超过10%的等位基因时,准确度则会相对较低,本研究中仅大白猪c.433C>T突变后频率低于10%(8.70%),数据总体可靠。同时,需保证DNA定量的精确性,并且在测序反应中对背景信号应做适当的校正,存在的背景信号会对等位基因相应峰高的测量产生一定的干扰,影响估算结果[8]。
目前,线粒体DNA分析已成为研究动物起源的有力手段[9-12],即使亲源关系比较近的种属也可以被区分开来[13]。Larson等[10]分析了世界范围内的686个野猪和家猪个体的 D-Loop区序列,认为家猪的起源在欧亚大陆,有多个独立的驯化中心。Jin等[14]分析了主要来自四川和西藏高原的513个猪样本线粒体DNA高变区序列和1 394条亚洲家猪、野猪序列信息,得出分布于长江上游地区的家猪由原地驯化而来。范丽丽等[15]以猪线粒体Cybt基因序列为靶位点设计引物和探针,进行荧光定量PCR扩增,建立猪源性成分快速且准确的检测方法。
脊椎动物线粒体 DNA的不同区域的进化速率不同,D-loop 区的进化速度最快,而COXⅠ、ND4 和Cytb基因进化速度适中。细胞色素C氧化酶(COXⅠ)作为线粒体呼吸链终端的一个成员,其作用是把呼吸底物的电子经过细胞色素系统直接传递给分子态氧。COXⅠ 已被广泛用于分析绦虫、灵长类[16]及鲈形目鱼类[17]等分子进化规律及亲缘关系的系统发育规律。陈咏霞等[18]分析了中国鲷科6属12种鱼类的COXⅠ基因序列,得出中国棘鲷属是一个有共同祖先的单系群,属内存在两个平行进化的分支,且两分支的种间关系具有明显的分化。张顺等[19]分析16 种弹涂鱼的82 个体的COXⅠ基因序列,认为弹涂鱼属出现明显分化大约发生于渐新世末期至中新世早期(23.61~15.65 Ma)。白鹏霞等[20]对采自牦牛体内的捻转血矛线虫线粒体COXⅠ基因部分序列进行分析,在基因和蛋白两个层面间接印证了毛圆科内不同属线虫在形态和生活史方面有较多相似和不同之处。
本研究获得猪线粒体COXⅠ基因编码序列1 545 bp,共存在21个突变位点,有8个突变为各群体特有。杨俊静等[21]发现民猪的COXⅠ基因序列与其他猪种相比,存在18个核苷酸突变。本研究的国内猪种无相同突变位点,国外猪种中杜洛克和长白猪有13个相同突变位点,其中杜洛克和大白猪有3个相同突变位点,大白猪和长白猪有2个相同突变位点。我国引入国外猪种与本地猪种杂交获得杂种优势,改善肉质,使其更加符合市场需求的同时也致使国外猪种相同碱基突变较多。近年来,利用峰高比值估算等位基因频率的方法已被广泛应用,李敬瑞等[22]利用测序图中SNPs等位基因峰高的比值估算可乐猪、白香猪和大约克这3个猪品种等位基因的频率。崔建勋等[23]利用测序图中SNP等位基因峰高的比值估算各鸡品种等位基因的频率,其中大部分位点等位基因频率的估算结果分别被PCR-RFLP/PCR-SSCP所验证。本研究中利用峰高比值估算各等位基因频率,根据碱基突变前后频率可以将国外猪种相同部分突变分为3类。c.858T>C、c.897A>G、c.1161C>T、c.1248C>T为一类,突变后频率高于突变前频率;c.336A>T、c.433C>T、c.519T>C、c.363G>A、c.381C>T、c.420A>G、c.732T>C、c.750G>A、c.924C>T为一类,突变后频率低于突变前频率,其中,杜洛克群体中c.363G>A突变前后频率较接近,突变前频率为0.5618,突变后频率为0.4382;c.1428T>C单为一类,杜洛克群体中突变后频率高于突变前频率,而长白猪群体中突变后频率低于突变前频率。国内猪种核苷酸序列位点变异表现为碱基T→C、C→T、A→G,核苷酸的替代仅为转换;国外猪种14个相同核苷酸序列位点变异表现为碱基A→T、C→T、T→C、G→A、A→G的转变,且核苷酸的替代主要以转换为主。钱建新等[24]对青海牦牛样品的线粒体COXⅠ基因部分序列进行研究,发现11个核苷酸序列位点变异,主要表现为碱基G→T、T→G、A→T 和 T→A 的转变,核苷酸的替代同样主要以转换为主。转换数(si)和颠换数(sv)的比值(R)可以用来估计序列替换的饱和程度。大白猪R值为2,长白猪R值为12,杜洛克R值为15,黄原[25]认为,如果R>2,说明序列替换远未达到饱和,而随着分歧时间的增加,R会降低,说明序列中多重替换数增加。
长白猪原产于丹麦,大白猪原产于英国约克郡,杜洛克原产于英国。藏猪、甘肃黑猪和八眉猪均产于我国西北地区,其生长环境相似,遗传距离近,多态性极其贫乏,作为地方猪种种质资源得到了较好的保护。相比之下,长白猪和杜洛克线粒体COXⅠ基因具有丰富的多态性,可能是因为作为引进猪种,为提高其适应性和生产能力与地方猪种杂交所致,这也导致了长白猪和杜洛克遗传距离近,仅为0.004。Watanabe等[26]分析了亚洲和欧洲猪的mtDNA限制性图谱,表明大白猪同时具有欧亚两大猪群的母性血统起源,本研究中大白猪与中国地方猪种间遗传距离小于杜洛克和长白猪在一定程度上与其符合,该猪场为获得较好的经济利益,根据自身的需要展开选育,使其适应当地环境和市场需求。同时,线粒体DNA的基因结构全部是外显子,没有内含子,不同品种间表现出的碱基突变很可能影响蛋白质的空间结构,从而影响其功能,该基因具有作为遗传标记的潜在可能。本研究表明,线粒体COXⅠ基因可有效区别6个猪种的亲缘关系,在一定程度上为我国地方猪品种的有效保护和合理利用提供理论依据。
[1] 曹宏卿, 顾为望. FMMU白化豚鼠线粒体DNA RFLP分析研究[J].中国实验动物学报,2005, 13(4):242-245. CAO H Q, GU W W. Restriction fragment length polymorphism (RFLP)analysis of mitochondrial DNA from FMMU Albino Guinea Pig[J].ActaLaboratoriumAnimalisScientiaSinica,2005, 13(4):242-245. (in Chinese with English abstract)
[2] 张亚平, 施立明. 动物线粒体DNA多态性的研究概况[J]. 动物学研究, 1992, 13(3):289-298. ZHANG Y P, SHI L M. A survey of mitochondrial DNA polymorphism in animals[J].ZoologicalResearch, 1992, 13(3):289-298. (in Chinese with English abstract)
[3] 梁祥解, 张浩吉, 李高强,等. 犬泡状带绦虫线粒体cox1和nad1基因的扩增与种系发育分析[J]. 佛山科学技术学院学报(自然科学版), 2010, 28(5):72-76. LIANG X J, ZHANG H J, LI G Q, et al. Amplification and phylogenetic analysis of mitochondrialcox1 andnad1 genes forTaeniahydatigenain dog in Guangdong[J].JournalofFoshanUniversity(NaturalScienceEdition) , 2010, 28(5):72-76. (in Chinese with English abstract)
[4] 郝桂英. 猪囊尾蚴西昌分离株线粒体cox1和nad1基因的序列测定与种系发育分析[J]. 中国畜牧兽医, 2015, 42(1):61-66. HAO G Y. Sequence and phylogenetic analysis of mitochondrialcox1 andnad1 genes forCysticercuscellulosaein Xichang[J].ChinaAnimalHusbandry&VeterinaryMedicine, 2015, 42(1):61-66. (in Chinese with English abstract)
[5] 王凝, 古小彬, 汪涛,等. 基于cox1基因对中国青藏高原地区细粒棘球绦虫遗传多态性的研究[J].畜牧兽医学报, 2015, 46 (3) :453-460. WANG N, GU X B, WANG T, et al. Genetic variability ofEchinococcusgranulosusdetermined by the mitochondrial cytochrome c oxidase subunit 1 gene in the Tibet Plateau of China[J].ActaVeterinariaEtZootechnicaSinica, 2015, 46 (3) :453-460. (in Chinese with English abstract)
[6] 何根林, 唐发辉, 周萍,等. 2种车轮虫DNA抽提方法比较[J]. 安徽农业科学, 2009, 37(27):12948-12950. HE G L, TANG F H, ZHOU P, et al. Comparative research of two methods on DNA extraction ofTrichodinidssubtilisaLom[J].JournalofAnhuiAgriculturalSciences, 2009, 37(27):12948-12950. (in Chinese with English abstract)
[7] 黄智伟, 黄琛. DNA检测技术研究现状[J]. 传感器世界, 2001, 7(1):9-14. HUANG Z W, HUANG C. Research status of DNA detection technology[J].SensorWorld, 2001, 7(1) :9-14.(in Chinese)
[8] 李树珍, 万慧荣, 杨光. DNA池结合DHPLC和直接测序技术在江豚SNPs检测中的应用[J]. 兽类学报, 2009, 29(2):185-190. LI S Z, WAN H R,YANG G. Application of DNA pooling in combination with DHPLC and direct sequencing in SNPs detection of the finless porpoise[J].ActaTheriologicaSinica, 2009, 29(2):185-190. (in Chinese with English abstract)
[9] GIUFFRA E, KIJAS J M, AMARGER V, et al. The origin of the domestic pig: independent domestication and subsequent introgression [J].Genetics, 2000, 154(4):1785.
[10] LARSON G, DOBNEY K, ALBARELLA U, et al. Worldwide phylogeography of wild boar reveals multiple centers of pig domestication[J].Science, 2005, 307(5715):1618.
[11] SAVOLAINEN P, ZHANG Y P, LUO J, et al. Genetic evidence for an East Asian origin of domestic dogs.[J].Science, 2002, 298(5598):1610-1613.
[12] JANSEN T, FORSTER P, LEVINE M A, et al. Mitochondrial DNA and the origins of the domestic horse.[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2002, 99(16):10905-10910.
[13] SABETI P C, VARILLY P, FRY B, et al. Genome-wide detection and characterization of positive selection in human populations[J].Nature, 2007, 449(7164):913-918.
[14] JIN L, ZHANG M, MA J, et al. Mitochondrial DNA evidence indicates the local origin of domestic pigs in the upstream region of the Yangtze River[J].PloSOne, 2012, 7(12):e51649.
[15] 范丽丽, 李培, 傅春玲,等. 实时荧光聚合酶链式反应法检测食品中猪源性成分[J]. 食品科学, 2013, 34(8):224-227. FAN L L, LI P, FU C L, et al. Detection for pig-derived components in foods by real-time polymerase chain reaction[J].FoodScience, 2013, 34(8):224-227. (in Chinese with English abstract)
[16] WU W, SCHMIDT T R, GOODMAN M, et al. Molecular evolution of cytochrome c oxidase subunit I in primates: is there coevolution between mitochondrial and nuclear genomes?[J].MolecularPhylogenetics&Evolution, 2000, 17(2):294-304.
[17] THACKER C E. Molecular phylogeny of the gobioid fishes (Teleostei: Perciformes: Gobioidei).[J].MolecularPhylogenetics&Evolution, 2003, 26(3):354-368.
[18] 陈咏霞, 吴仁协, 梁娜,等. 基于线粒体COI基因序列的中国鲷科鱼类系统进化关系[J]. 海洋与湖沼, 2015, 46(3):611-619. CHEN Y X, WU R X, LIANG N, et al. Phylogenetic relationship in family sparidae of China in mitochondrial COI gene sequences[J].OceanologiaEtLimnologiaSinica, 2015, 46(3):611-619. (in Chinese with English abstract)
[19] 张顺, 廖健, 郭昱嵩,等. 基于线粒体cox1基因序列的弹涂鱼类系统进化关系[J]. 广东海洋大学学报, 2017, 37(1):21-27. ZHANG S, LIAO J, GUO Y S, et al. Phylogenetic relationship of coated fish based on mitochondrialcox1 gene sequences[J].JournalofGuangdongOceanUniversity, 2017, 37(1) :21-27. (in Chinese with English abstract)
[20] 白鹏霞, 郝宝成, 陈长江,等. 捻转血矛线虫线粒体COXⅠ基因序列测定及进化分析[J]. 中兽医医药杂志, 2016(5):13-16. BAI P X, HAO B C, CHEN C J, et al. Sequence determination and phylogenetic analysis of mitochondrial COX I gene in the Chinese wild plant[J].JournalofTraditionalChineseVeterinaryMedicine, 2016(5):13-16. (in Chinese with English abstract)
[21] 杨俊静, 别墅, 张冬杰,等. 民猪细胞色素C氧化酶Ⅰ基因的克隆、多态性检测及冷诱导研究[J]. 吉林农业大学学报, 2012, 34(1):99-103. YANG J J, BIE S, ZHANG D J, et al. Study on cloning and polymorphism analysis of COX Ⅰ gene in Min Pig during cold induction[J].JournalofJilinAgriculturalUniversity, 2012, 34(1):99-103. (in Chinese with English abstract)
[22] 李敬瑞, 丁远华, 张玉龙,等. 利用DNA池和测序技术快速筛查SNPs及估算基因频率[J]. 畜牧与兽医, 2011, 43(9):17-20. LI J R, DING Y H, ZHANG Y L, et al. Rapidly screening SNPs and estimation allelic frequencies by DNA pooling and sequencing[J].AnimalHusbandry&VeterinaryMedicine, 2011, 43(9):17-20. (in Chinese with English abstract)
[23] 崔建勋, 杜红丽, 张细权. 利用DNA池和测序技术快速筛查SNPs及估算基因频率[J]. 遗传学报, 2005, 32(4):372-377. CUI J X, DU H L, ZHANG X Q. Rapidly screening SNPs and estimation allelic frequencies by DNA pooling and sequencing[J].ActaGeneticaSinica, 2005, 32(4):372-377. (in Chinese with English abstract)
[24] 钱建新, 钱和平, 徐志伟,等. 基于线粒体cox1和nad1基因对青海牦牛种系发育关系的研究[J]. 中国畜牧兽医, 2013, 40(2):151-154. QIAN J J, QIAN H P, XU Z W, et al. Relationship between mitochondrialcox1 andnad1 genes and their phylogenetic relationships in Qinghai yaks[J].ChinaAnimalHusbandry&VeterinaryMedicine, 2013, 40(2):151-154. (in Chinese with English abstract)
[25] 黄原. 分子系统学:原理、方法及应用[M]. 北京:农业出版社, 1998.
[26] WATANABE T, HAYASHI Y, KIMURA J, et al. Pig mitochondrial DNA: polymorphism, restriction map orientation, and sequence data[J].BiochemicalGenetics, 1986, 24(5):385.
(责任编辑 张 韵)
Genetic diversity and phylogenetic relationship among domestic and foreign pig breeds based on mtDNACOXⅠ gene
ZHOU Xiumin, YANG Yongjiang, BI Yingjie, REN Weihe, ZHANG Li*
(CollegeofLifeScienceandEngineering,NorthwestUniversityforNationalities,Lanzhou730030,China)
Based on the mitochondrial DNACOXⅠ (mtDNACOXⅠ) gene, the genetic diversity and phylogenetic relationships of 6 pig breeds (Large White pig, Landrace pig, Duroc pig, Zang pig, Gansu black pig and Bamei pig) were studied. The mtDNACOXⅠ gene sequences obtained from 6 pig breeds were sequenced and analyzed. Besides, we constructed phylogenetic tree by using MEGA 6.0 analysis software and neighbor joining method based on Kimurk two parameter models. The results showed that there were 21 mutations in sequences ofCOXⅠ (1 545 bp), including 8 group specific mutations. Landrace pig and Duroc pig were abundant in polymorphism (13 same mutation sites), and the ratios (R) of nucleotides’ conversion number (si) and transition number (sv) were 12 and 15, which proved sequence substitutions were far from saturation. While the polymorphisms of Zang pigs, Gansu black pigs and Bamei pigs were poor. The introduced pigs were used as sire, not dam, to improve the growth performance and lean meat percentage of the domestic pig breeds. The mtDNACOXⅠ gene can effectively distinguish the relationship between six pig breeds. This study can provide theoretical basis for the effective protection and rational utilization of pig breeds in our country.
pig; mitochondrial cytochrome C oxidase subunit Ⅰ; phylogeography
10.3969/j.issn.1004-1524.2017.08.07
2017-05-14
西北民族大学国家级大学生创新创业训练计划项目(201710742088)
周秀敏(1996—),女,江苏盐城人,本科生,动物科学专业。E-mail: 1029007641@qq.com
*通信作者,张丽,E-mail: zhangli2008@aliyun.com
S828
A
1004-1524(2017)08-1271-10
周秀敏,杨永江,毕英杰,等. 基于线粒体COXⅠ基因变异探讨国内外猪种的遗传多样性及系统进化研究[J].浙江农业学报,2017,29(8): 1271-1280.
