超高效液相色谱-串联质谱法分析骨髓间充质干细胞衰老过程中的全基因组DNA甲基化水平变化
2017-08-29何国东杨翔宇李晓红余细勇
何国东,杨翔宇,李晓红,潘 宇,余细勇
[1.广州医科大学药学院,广东 广州 511436;2.广东省人民医院(广东省医学科学院)医学研究部,广东 广州 510080]
超高效液相色谱-串联质谱法分析骨髓间充质干细胞衰老过程中的全基因组DNA甲基化水平变化
何国东1,2,杨翔宇2,李晓红2,潘 宇2,余细勇1
[1.广州医科大学药学院,广东 广州 511436;2.广东省人民医院(广东省医学科学院)医学研究部,广东 广州 510080]
目的使用超高效液相色谱-串联质谱测定骨髓间充质干细胞中全基因组DNA甲基化水平,观察其衰老过程中甲基化率的变化。方法从骨髓间充质干细胞中提取DNA,使用酶解法将DNA分解成单个脱氧核苷,采用超高效液相色谱法分离,正离子电喷雾与多反应监测模式进行定量,分析5-甲基脱氧胞嘧啶核苷、脱氧鸟嘌呤核苷的相对含量,计算全基因组DNA 甲基化率。结果对1 μg DNA样本进行1 h酶解反应,2 min内可分析出5.00×10-4水平以上的DNA甲基化率,日内及日间精密度分别在7.12×10-2和0.119以内。随着骨髓间充质干细胞传代扩增培养,干细胞不断衰老,其甲基化率逐渐降低,传至P4~P6代时,干细胞甲基化率最低,随着传代的继续,其甲基化率逐渐上升。结论此方法成功应用于检测研究骨髓间充质干细胞在衰老过程中全基因组DNA甲基化水平的变化,得到了衰老影响骨髓间充质干细胞DNA甲基化的初步数据。经考察,该方法样本前处理方法简便,可操作性强,定量方式快速准确,具有良好的灵敏度与重复性。
超高效液相色谱;质谱;骨髓间充质干细胞;衰老;DNA甲基化;酶解法
基因组DNA甲基化检测的方法众多,如免疫组织化学法、糖基化化学标记法、限制性内切酶与重亚硫酸盐测序法等等[1],但以上方法测定过程繁琐。随着表观遗传学的发展,液相色谱-串联质谱法开始应用于检测DNA中的甲基化水平,其样本处理方式主要分为两种,一种是利用甲酸加热水解DNA[2-3],通过水解物的标准物质配成的溶液与其同位素内标校准进行含量测定,另一种是利用酶解的方式将DNA酶解,同样通过酶解产物的标准物质配成的溶液与其同位素内标校准进行含量测定[4-5]。不论是酸解还是酶解,由于是使用标准溶液进行校准定量,校准曲线样本无法模拟标本DNA提取降解后的生物基质,而降解DNA的过程步骤较为繁琐,需时6~12 h,分析效率不高。本文在以前研究的基础上,改进DNA样本处理方法和定标方式,并应用于分析骨髓间充质干细胞(bone marrow mesenchymal stem cells, BMMSCs)衰老过程中全基因组DNA甲基化水平。该方法样本处理方式简单快速,分析周期短,重现性好,具有良好的灵敏度和精密度,可满足生物样品的全基因组DNA甲基化水平分析。
1 材料与方法
1.1仪器与试剂ACQUILY UPLC超高效液相色谱仪(Waters,美国);Xevo TQ-s三重四极质谱仪(Waters,美国);METTLER XA205DU十万分之一电子分析天平(METTLER,德国);Allegra X-30R台式高速冷冻离心机(贝克曼,美国);SevenEasy Plus pH计(METTLER,德国);MDF-U73V超低温冰箱(SANYO,日本);LABSCALE TFF超纯水机(MILLI-Q,美国);气压式手动移液器(20、100、200、1000 μL)(GILSON,法国);Imagequant las 500生物分子成像仪(GE,美国); MCO-18M三气培养箱(SANYO,日本);NanoDrop2000超微量分光光度计(Merinton,美国);OSE-PRO干式恒温金属浴(TIANGEN,中国);DNA Degradase Plus、5-Methylcytosine DNA Standard Set(Zymo Research,美国);乙腈(HPLC级,Merk,德国);间充质干细胞基础培养基(ScienCell,美国);Trypsin-EDTA Solution(碧云天生物技术,中国);Senescence β-Galactosidase Staining Kit(Cell Signaling Technology,美国);TIANamp Genomic DNA Kit(TIANGEN,中国)。
1.2细胞衰老实验本研究经医学伦理委员会批准,BMMSCs由广东省人民医院血液科的4名骨髓捐赠者的骨髓通过梯度离心法获得原代细胞,使用间充质干细胞基础培养基分别于25 mL培养瓶和35 mm培养皿进行培养,细胞长满培养瓶后,使用Trypsin-EDTA Solution消化传代,留取一半细胞于-80℃冰箱保存,用于提取DNA。35 mm培养皿的细胞作β-半乳糖苷酶染色,使用Senescence β-Galactosidase Staining Kit进行细胞染色,步骤按照试剂说明书进行,验证细胞衰老。分别对第1、3、5、7、9、13代细胞进行收集与染色。
1.3DNA的提取和酶解从-80℃冰箱取出的细胞样本,置于常温1 h后,使用TIANamp Genomic DNA Kit进行DNA提取,步骤按照试剂说明书进行,DNA样本经NanoDrop2000超微量分光光度计检测,测定提取纯度和浓度。取不小于1 μg DNA样本,加入DNA Degradase Plus,其中包含2.5 μL buffer和 1 μL DNA Degradase,加入无核酶水补足反应体积至25 μL,于OSE-PRO干式恒温金属浴中37℃反应1 h,酶解后取2 μL用1.5×10-2琼脂糖凝胶电泳检验酶解效果,加入175 μL 1.0×10-3甲酸水溶液终止酶解反应。
1.4超高效液相色谱串联检测串联质谱条件:采用电喷雾离子源(ESI),正离子模式,多反应监测模式(Multiple Reaction Monitoring, MRM)定量;毛细管电压(Capillary)3.0 kV,雾化温度500℃,干燥气流速650 L·h-1;5-甲基脱氧胞嘧啶核苷(5-methyl-2′-deoxycytidine , 5mdC)的定量离子对为242.03~126.00 m·z-1,锥孔电压4 V,碰撞电压6 V;2’-脱氧鸟苷(2′-deoxyguanosine, dG)的定量离子对为268.08~152.0 m·z-1,锥孔电压2 V,碰撞电压20 V。超高效液相色谱条件:ACQUITY UPLC HSS T3 C18 (1.7 μm,2.1 mm×100 mm);流动相:A为5.0 mmol·L-1乙酸铵、2.0×10-3甲酸水溶液,B为乙腈;流速为0.3 mL·min-1;柱温30℃,进样量5 μL;梯度洗脱程序:0~0.3 min,0.70 A;0.3~0.6 min,0.70 A降至0.05 A,0.6~1.2 min维持0.05 A,1.2~1.5 min升至0.70 A,1.5~2.0 min维持0.70 A。洗针液为1 ∶1甲醇水溶液。
1.5定标曲线及质控样本的配制5-Methylcytosine DNA Standard Set包含有两条序列一致、长度均为897 bp、浓度均为50 mg·L-1的标准DNA,其中一条DNA上的脱氧胞嘧啶全部经过甲基化处理变为5mdC,另一条DNA上的脱氧胞嘧啶均未被修饰过。使用以上两条标准DNA按比例混合,模拟出甲基化水平为(5.00×10-4、1.00×10-3、2.00×10-3、5.00×10-3、1.00×10-2、2.00×10-2、5.00×10-2、1.00×10-1)的标准DNA样本和甲基化水平为(1.00×10-3、5.00×10-3、5.00×10-2)的质控DNA样本。按“1.3”的方法处理标准DNA和质控DNA,同时检测5mdC与dG的色谱峰面积,以5mdC/dG峰面积比值为纵坐标,横坐标为甲基化水平,绘制标准曲线。
2 结果
2.15mdC和dG质谱特征与参数优化以多反应监测模式(MRM)为扫描模式,通过梯度优化毛细管电压、雾化温度、干燥气流速、锥孔电压、碰撞电压等参数后,分别使得5mdC离子对(242.0~126.0 m·z-1)和dG离子对(268.0~152.0 m·z-1)灵敏度最高,优化结果详见实验部分。Fig 1A、1B为5 mdC和dG子离子扫描图。从Fig 1可看出,5mdC对应母离子为242.0 m·z-1,经碰撞后生成的主要子离子为5-甲基胞嘧啶(126.0 m·z-1);dG对应母离子为268.0 m·z-1,经碰撞后生成的主要子离子为鸟嘌呤(152.0 m·z-1)。
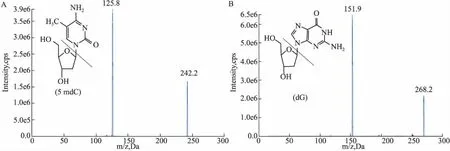
Fig 1 Product ion scanning of 5mdC(A) and dG (B)
2.2液相色谱条件优化Fig 2中A1、A2分别为最低定量下限标准样本中5mdC和dG的色谱图;Fig 2中B1、B2分别为BMMSCs样本中5mdC和dG的色谱图,从图中可以看出,5mdC保留时间为1.06~1.08 min,dG保留时间为1.11 min,两分析物峰型对称,无拖尾现象,无杂峰干扰。Fig 2中C1和C2为连续10针500 μg·L-15mdC和dG混合溶液后的1针空白样本,从图中可以看出,连续高浓度进样后对空白样本并无干扰,该液相方法无残留效应,洗针液也满足分析要求。
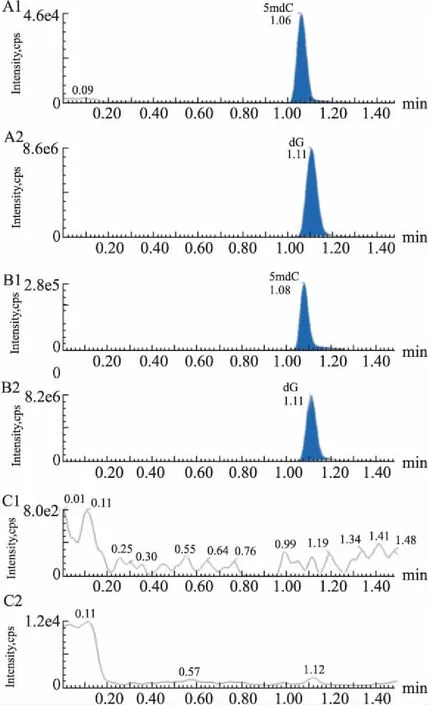
Fig 2 Chromatographic separation of 5mdC and dG
2.3BMMSCs衰老模型的验证Fig 3为P1、P3、P5、P7、P9、P13代细胞染色结果,阳性率结果分别为0.02、0.11、0.35、0.72、0.86、0.94。BMMSCs经过不断传代培养,逐渐出现衰老特征,细胞体积逐代增大,细胞质逐渐铺开,细胞核逐代增大并且颜色不断加深,β-半乳糖苷酶染色阳性率逐渐增加,颜色逐代加深。
2.4样本前处理方法考察使用TIANamp Genomic DNA Kit试剂盒对细胞进行DNA提取,收集到的DNA样本取2 μL用NanoDrop2000超微量分光光度计进行纯度与浓度测定,A260/A280比值在1.8~2.0之间,浓度在50 ~100 mg·L-1,结果显示该方法提取的DNA样本纯度与浓度均满足检测要求。采用2.5 μL Degradase buffer和 1 μL DNA Degradase对1 μg DNA样本进行酶解,降解产物通过琼脂糖凝胶电泳分析(Fig 4),1 h与2 h酶解反应效果一致且酶解效果优异,故酶解反应1 h即可满足分析需要。
2.5方法学考察本方法采用两条标准DNA(5-Methylcytosine DNA Standard Set)作为定标曲线和质控样本进行方法学考察和样本分析定量。按照生物样品测定方法验证指导原则[6],考察日内、日间3个分析的批精密度与准确度。每个分析批包含1条定标曲线、最低定量下限和三水平质控样本,最低定量下限和每水平质控样本平行处理6份。Tab 1为该方法学的精密度、准确度及最低定量下限验证数据。

Tab 1 Validation of accuracy and precision
CV: Coefficient of variance; RE: Relative error.
2.6对BMMSCs样本的分析利用上述方法对来自4个不同个体的P1~P13代BMMSCs的DNA甲基化水平进行检测。Fig 5为检测结果,BMMSCs从P1代开始DNA甲基化水平逐渐下降,至P4~P6代时DNA甲基化水平最低,P7~P13代DNA甲基化水平则逐渐上升。
3 讨论
DNA甲基化已是公认的重要表观遗传学修饰之一,其动态调控对细胞的正常生理功能起着举足轻重的作用[7-8]。DNA甲基化是指在DNA甲基转移酶(DNA methyltransferases, DNMTs) 的作用介导下,S-腺苷甲硫氨酸(S-adenosyl methionine, SAM) 会提供一个甲基基团,把基因组DNA序列中的胞嘧啶(cytosine, C) 甲基化转化为5-甲基化胞嘧啶(5-methylcytosine, 5-mC) 的过程[9]。分析全基因组DNA甲基化水平对于研究干细胞分化过程中基因组DNA甲基化修饰的作用,以及干细胞多分化潜能的维持与定向分化等机制具有重要的作用[10-11]。
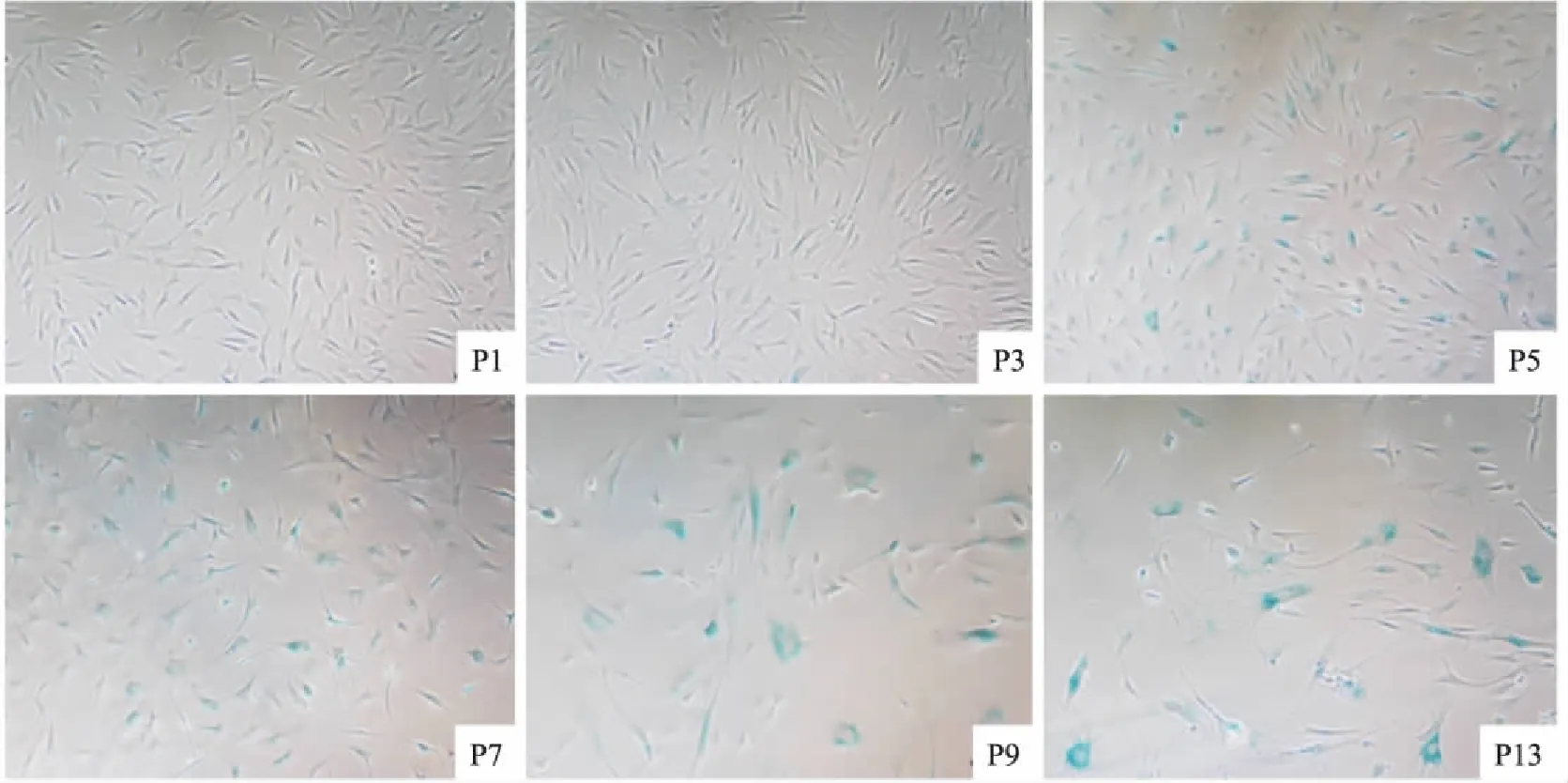
Fig 3 Results of BMMSCs β-galactosidase staining

Fig 4 Agarose gel electrophoresis of DNA degradation products

Fig 5 The DNA methylation level of BMMSCs of P1-P13
由于衰老会对表观遗传学产生异常的改变[12],本次研究采用超高效液相色谱-串联质谱技术对人BMMSCs衰老过程中DNA甲基化率的变化进行观察。
衰老细胞通常体积会增大,在 pH 6.0的环境下,有高酶活性的β-半乳糖苷酶,以X-Gal为底物,在衰老特异性的β-半乳糖苷酶催化下会生成深蓝色产物[13]。由于β-半乳糖苷酶染色结果受pH影响较大[14],为防止染色结果出现假阳性或假阴性,染色过程中严格控制染液pH值范围在5.9~6.1。由β-半乳糖苷酶染色的结果看,BMMSCs随着传代扩增培养,出现不断衰老的现象。
收集各代BMMSCs的DNA后,利用DNA Degr-adase Plus试剂降解DNA,与传统酸法降解或酶法降解相比,无需分多步进行,操作步骤简单,样本前处理时间亦大大缩减,可操作性强。由于5mdC和dG为强极性弱碱化合物,因此,采用适合极性化合物分析专用柱ACQUITY UPLC HSS T3 C18,经过多次流动相调整,以乙腈-甲酸乙酸铵溶液峰型最好,分析单个样本时间只需2 min。之前的实验方法[4-5]多数使用降解产物的标准物质按比例配制的溶液作为定标曲线样本,溶液基质与待测样本DNA酶解后的基质不同,定标曲线无法校正样本前处理过程中带来的误差。本方法使用标准DNA作为定标曲线与质控品样本,与待测样本经过统一的酶解法处理,提高了定量准确性。
一项关于BMMSCs传代扩增培养与干细胞干性的维持关系的研究[15]发现,随着传代BMMSCs的干性逐渐增强,传代至P5~P6代时,干细胞干性最强,随着传代的继续,其干性逐渐下降。而本研究则发现,随着传代BMMSCs不断衰老,甲基化率逐渐降低,传至P4~P6代时细胞甲基化率最低,随着传代的继续,其甲基化率逐渐上升。提示BMMSCs的细胞干性与其DNA甲基化水平存在密切联系。
综上所述,本研究建立了超高效液相色谱-串联质谱测定细胞中全基因组DNA甲基化水平的方法,并应用于观察BMMSCs在衰老过程中DNA甲基化水平的变化,得到了衰老影响BMMSCs的全基因组甲基化率的初步数据。该方法样本前处理方法简便,可操作性强,定量方式快速准确,具有良好的灵敏度与重复性。
(致谢:本实验主要是在广东省人民医院的医学研究部中心实验室完成的,感谢课题组所有成员对实验的指导与帮助。)
[1] Mikeska T, Candiloro I L, Dobrovic A. The implications of heterogeneous DNA methylation for the accurate quantification of methylation[J].Epigenomics, 2010,2(4):561-73.
[2] Friso S, Choi S W, Dolnikowski G G, et al. A method to assess genomic DNA methylation using high-performance liquid chromatography/electrospray ionization mass spectrometry[J].AnalChem, 2002,74(17):4526-31.
[3] Ma H, Zhang W, Hu J, et al. Analysis of global DNA methylation levels in human blood using high-performance liquid chromatography/tandem electrospray ionization mass spectrometry[J].EurJMassSpectrom(Chichester), 2009,15(4):555-61.
[4] Li X, Franke A A. High-throughput and cost-effective global DNA methylation assay by liquid chromatography-mass spectrometry[J].AnalChimActa, 2011,703(1):58-63.
[5] Vandegehuchte M B, Lemière F, Janssen C R. Quantitative DNA-methylation in Daphnia magna and effects of multigeneration Zn exposure[J].CompBiochemPhysiolCToxicolPharmacol, 2009,150(3):343-8.
[6] Booth B, Arnold M E, DeSilva B, et al. Workshop report: Crystal City V-quantitative bioanalytical method validation and implementation: the 2013 revised FDA guidance[J].AAPSJ, 2015,17(2):277-88.
[7] 段 菊, 凌 霜, 甘我挺,等. DNA主动去甲基化酶研究进展及药物开发[J]. 中国药理学通报, 2013,29(10):1345-9.
[7] Duan J, Ling S, Gan W T, et al. DNA demethylases: a new direction for drug development[J].ChinPharmacolBull, 2013,29(10):1345-9.
[8] Zhong J, Agha G, Baccarelli A A. The role of DNA methylation in cardiovascular risk and disease: methodological aspects, study design, and data analysis for epidemiological studies[J].CircRes, 2016,118(1): 119-31.
[9] Mohan K N. Stem cell models to investigate the role of DNA methylation machinery in development of neuropsychiatric disorders[J].StemCellsInt, 2016,2016(3):1-8.
[10] Jones P A, Takai D. The role of DNA methylation in mammalian epigenetics[J].Science, 2001,293(5532):1068-70.
[11] Berdasco M. DNA methylation in stem cell renewal and multipotency[J].StemCellResTher, 2011,2(5):1-9.
[12] 张均田.认知过程中的表观遗传学机制[J]. 中国药理学通报, 2015,31(1): 1-7.
[12] Zhang J T. Epigenetic mechanism in cognitive function[J].ChinPharmacolBull, 2015,31(1):1-7.
[13] Severino J, Allen R G, Balin S, et al. Is beta-galactosidase staining a marker of senescenceinvitroandinvivo[J].ExpCellRes, 2000,257(1):162-71.
[14] Albuquerque T L D, Peirce S, Rueda N, et al. Ion exchange of β-galactosidase: the effect of the immobilization pH on enzyme stability[J].ProcessBiochem, 2016,51(7):875-80.
[15] 李 萍, 金世柱, 张 明,等. 骨髓间充质干细胞传代扩增培养与干细胞干性的维持关系的研究[J]. 国际免疫学杂志, 2015,38(6):507-11.
[15] Li P, Jin S Z, Zhang M, et al. The relationship between the cell culture periods and the stem character of bone marrow mesenehymal stem cells[J].IntJImmunol, 2015,38(6):507-11.
Analysisofmethylationlevelofbonemarrowmesenchymalstemcellsduringsenescencebyultraperformanceliquidchromatography-tandemmassspectrometry
HE Guo-dong1,2, YANG Xiang-yu2, LI Xiao-hong2, PAN Yu2, YU Xi-yong1
[1.SchoolofPharmaceuticalSciences,GuangzhouMedicalUniversity,Guangzhou511436,China;2.DeptofMedicalResearch,GuangdongGeneralHospital(GuangdongAcademyofMedicalSciences),Guangzhou510080,China]
AimTo measure the methylation rate of bone marrow mesenchymal stem cells (BMMSCs) using ultra-performance liquid chromatography-tandem mass spectrometry, and determine the methylation rate in the process of senescence.MethodsThe DNA extracted from BMMSCs would be digested into individual deoxynucleosides using enzymatic hydrolysis. To quantify the global genomic DNA methylation rate,we developed a method using ultra-performance liquid chromatography-tandem mass spectrometry with multiple reaction monitoring to simultaneously measure the levels of 5-methyl deoxycytidine and deoxyguanosine in digested genomic DNA.ResultsThe DNA methylation rate could be analyzed within two minutes after hydrolyzing 1μg DNA for an hour. The detection limit of DNA methylation rate was 5.00×10-4. The coefficient of variance of the intra-day and inter-day precision was within 7.12×10-2and 0.119, respectively. With the subculture of BMMSCs, the methylation rate gradually decreased. The methylation rate of stem cells was the lowest in 4~6 generation, and gradually increased with the subculture.ConclusionsUsing this method, the preliminary data on DNA methylation of BMMSCs in the process of senescence are obtained. The method is simple and convenient for sample preprocessing, and the method is fast and accurate with good sensitivity and repeatability.
ultra performance liquid chromatography; mass spectrometry; bone marrow mesenchymal stem cells; senescence; DNA methylation; enzymatic hydrolysis
时间:2017-8-20 16:47 网络出版地址:http://kns.cnki.net/kcms/detail/34.1086.R.20170820.1647.042.html
2017-05-09,
2017-06-08
国家自然科学基金重点资助项目(No 81330007, U1601227);广东省科技计划资助项目(No 2015B020225006, 2014A050503047)
何国东(1988- ),男,硕士生,研究方向:表观遗传药理学,E-mail: toyakowo@126.com; 余细勇(1962- ),男,博士,教授,博士生导师,研究方向:心血管药理学及干细胞转化医学,通讯作者,E-mail:yuxycn@aliyun.com
10.3969/j.issn.1001-1978.2017.09.021
A
:1001-1978(2017)09-1298-06
R329.24;R339.38;R394.2;R446.1
