模拟鸽子优化过程的蛋白质复合物识别算法*
2017-08-16丁玉连雷秀娟
丁玉连,雷秀娟,代 才
陕西师范大学 计算机科学学院,西安 710062
模拟鸽子优化过程的蛋白质复合物识别算法*
丁玉连,雷秀娟+,代 才
陕西师范大学 计算机科学学院,西安 710062
蛋白质复合物的检测对人类了解细胞组织和疾病预测起着至关重要的作用。然而,当前的蛋白质复合物识别方法的准确率低,对噪音敏感等缺点导致其识别效果并不理想。提出了一种新的蛋白质复合物识别方法PIOC(pigeon-inspired optimization clustering)。该方法根据蛋白质复合物的特性提出了簇的紧密邻接点概念和附件对核心的附着度概念,基于这两个概念,PIOC通过模拟鸽子优化算法中鸽子寻找目的地的过程来识别蛋白质复合物;结合鸽子算法中先全局搜索再局部搜索的特性和蛋白质复合物的核心附件结构,先通过鸽子算法中地图罗盘操作的全局搜索形成蛋白质复合物的核心,再通过鸽子算法地标操作的局部搜索将附件蛋白质聚集到核心簇中形成蛋白质复合物。基于酵母蛋白质相互作用网络DIP上的实验表明,PIOC比当前其他的蛋白质复合物识别算法能更有效地识别蛋白质复合物。
蛋白质相互作用(PPI);鸽子优化算法;蛋白质复合物;聚类
1 引言
在生物体中,蛋白质不是独立存在的,而是相互联系在一起形成蛋白质复合物,从而实现它们的生物功能[1]。因此,正确识别蛋白质复合物在了解细胞功能机制和预测未知蛋白质的功能中起着重要的作用[2]。生物实验的方法识别蛋白质复合物昂贵且耗时,因此通过计算的方式识别蛋白质相互作用(protein-protein interaction,PPI)网络的蛋白质复合物成为人们研究的热点[3-5]。
目前,计算方式识别蛋白质复合物的普遍做法是将蛋白质相互作用信息表示成一个无向图,将蛋白质复合物对应图中的稠密子图,利用各种图聚类分析方法来识别蛋白质复合物。一般通过聚类方法是否考虑到网络的全局特性,蛋白质复合物识别方法可以分为两大类:一类是全局聚类方法;一类是局部聚类方法。
全局聚类方法主要是通过划分整个PPI网络为独立的子网来挖掘蛋白质复合物。例如,G-N(Girvan-Newman)[6]算法主要是通过不断移除具有最高的介数中心性的边来划分网络。马尔科夫聚类(Markov clustering,MCL)[7]算法主要是通过在PPI网络中模仿随机步来检测蛋白质复合物。虽然这些聚类方法考虑到了PPI网络的全局性,但不能识别重叠的蛋白质复合物,而蛋白质复合物之间是高度重叠的[8]。
局部聚类方法主要考虑局部的邻居结点来识别蛋白质复合物。例如,基于最大簇聚类(clusteringbased on maximal cliques,CMC)[9]算法主要是通过找最大簇来识别蛋白质复合物。MCODE(molecular complex detection)[10]算法主要是先给结点加权,然后选择权重高的结点作为种子,扩充种子形成蛋白质复合物。还有一些其他聚类算法如,DPClus(development clustering)[11]、HC-PIN(hierarchical clusteringprotein interaction network)[12]和ClusterONE(clustering with overlapping neighborhood expansion)[13]等,这些方法都忽视了蛋白质复合物的内部核心附件结构。COACH(core-attachment)[14]算法基于蛋白质复合物的核心附件结构先形成稠密子图作为核心,然后将附件蛋白质聚到相应的核心中形成复合物。局部聚类算法虽然能识别重叠蛋白质复合物,但忽视了PPI网络的全局性。
传统的聚类方法识别蛋白质复合物都存在一定的局限性,性能并不是很好。生物仿生算法为复合物识别提供了一个新视角。例如,PMABC-ACE(propagating mechanism of artificial bee colony cluster module based on aggregation coefficient of edge)[15]通过模仿人工蜂群繁殖进行蛋白质复合物识别。基于萤火虫的马尔科夫聚类(fire fly-Markov clustering,FMCL)[16]通过结合MCL算法和萤火虫优化算法来识别蛋白质复合物。这些算法比传统聚类算法表现出了更好的性能。鸽子优化算法[17]作为一种新颖的群智能优化算法,通过地图指南针算子和地标算子模拟鸽子找家的过程。它被证明比其他群智能算法的优化特性更强[18],并被广泛应用,例如无人机目标探测[19]、PID控制器设计[20]等。
本文结合鸽子算法的优化特性和蛋白质复合物的特征提出了一种新的蛋白质复合物识别方法PIOC(pigeon-inspired optimization clustering)。其中蛋白质复合物的特征包括:复合物是由核心和附件构成,复合物内蛋白质之间的最短距离一般不超过2,复合物是个高密度的簇等。PIOC方法先通过鸽子算法中地图罗盘操作的全局搜索形成蛋白质复合物的核心,再通过鸽子算法地标操作的局部搜索将附件蛋白质聚集到核心簇中形成蛋白质复合物。
本文组织结构如下:第2章主要介绍基本鸽子算法和PPI网络的相关基础知识;第3章描述基于鸽子优化算法的聚类算法;第4章展示实验结果,并对结果加以分析;最后对本文进行总结,并指出进一步的研究方向。
2 鸽子算法与PPI网络基础知识
2.1 鸽子算法
信鸽有着天生的归巢能力,它们通过使用磁场、太阳和地标3个导航工具,准确找到远方的家。基于鸽子的导航行为,一种新颖的群智能仿生算法鸽子优化(pigeon-inspired optimization,PIO)算法[17]在2014年被提出。PIO算法主要通过两个操作来模拟鸽子的导航行为,即地图罗盘算子和地标算子。地图罗盘算子的提出主要是基于太阳和磁场,而地标算子的提出主要基于地标。
(1)地图罗盘算子
在地图罗盘算子中,通过计算机随机产生鸽子。用Xi、Vi分别表示鸽子i的位置和速度,并且在一个d维的搜索空间里,每只鸽子的位置和速度在每次迭代中不断更新。第i只鸽子在第t次迭代中新的位置和速度通过以下公式获得:
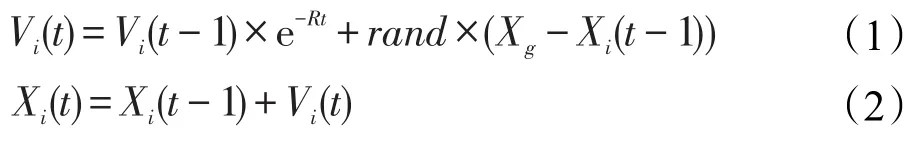
其中,R是地图罗盘算子;rand是一个随机数(一般取0~1之间的数);Xg是全局最优位置。
(2)地标算子
在地标算子中,假设鸽子都还没有到达目的地,并且它们对地标都不熟。所有的鸽子都根据它们的适应度值排序,再通过式(3)减少一半鸽子(Np/2):

然后在第t次迭代时剩下的鸽子里找到中心鸽子的位置(Xc),Xc被认为是最理想的位置,通过式(4)更新,其他鸽子的位置通过式(5)更新:
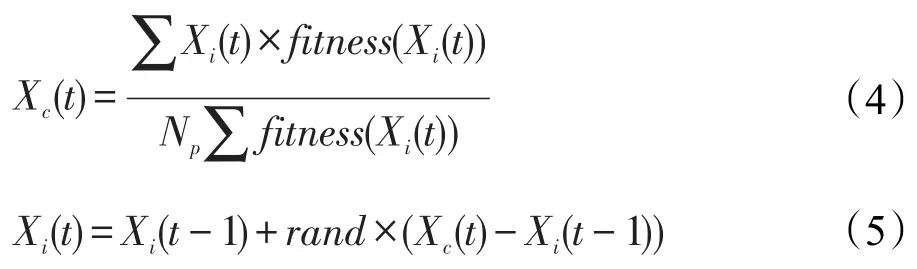
其中,fitness(Xi(t))是种群里每只鸽子的适应度。
2.2 PPI网络基本特征度量
蛋白质相互作用完成了特定的分子功能或者进程。PPI网络可以用图G(V,E)表示,其中V是顶点集,代表所有蛋白质结点的集合,E为边集,表示任意两蛋白质之间的相互作用集合。
结点的度degree表示在PPI网络中直接与此结点相连的边的条数,反映了当前结点与其周边结点的连接程度。
结点的聚集系数[21](node clustering coefficient,NCC)是指与该结点相邻的邻接结点之间实际存在的边的数目与可能存在的边的数目的比例,反映了网络中结点的聚集程度,结点v的聚集系数NCC(v)的计算公式为:

其中,kv表示结点v的度;nv表示v的kv个邻居结点之间存在的边数。
边的聚集系数[22](edge clustering coefficient,ECC)是指与该边构成的三角形个数和所有可能包括该边的三角形个数比例,计算过程如下:
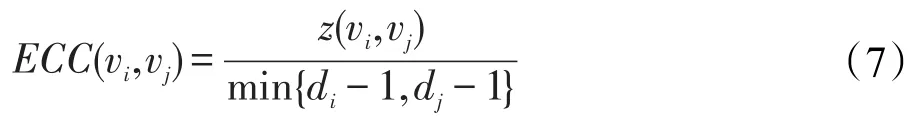
其中,Z(vi,vj)表示包含边(vi,vj)的三角形个数;min{di-1,dj-1}表示包含边(vi,vj)的可能存在的三角形的最大个数。
PPI网络子图G密度dens[21]的计算是用子图中存在的边数比上当子图为一个完全图时应该有的边数,其计算公式如下:

其中,m表示子图中存在的边的条数;n表示子图中的结点数。
2.3 蛋白质复合物的基本特征
对已有的酵母蛋白质复合物标准库中的蛋白质复合物的研究表明,蛋白质复合物存在以下3个特征:
(1)蛋白质复合物中的蛋白质之间的平均最短路径长度小于等于2的占90%以上[23]。
(2)大部分蛋白质复合物的密度在0.5以上[23]。
(3)蛋白质复合物都是以一个核心多个附件构成的[14]。
根据以上3个特征本文提出了簇的紧密邻接点的定义和附件与核心簇之间附着度的定义。
定义1一个簇C的紧密邻接点v为满足SP(u,v)≤2,u∈Vc的结点。其中Vc为簇C内包含的结点集合,SP(u,v)表示簇C的邻接点v到簇C内的任一结点u的最短路径。
定义2一个附件蛋白质结点v到一个核心簇C的附着度为DS(v,C),其计算公式如下:
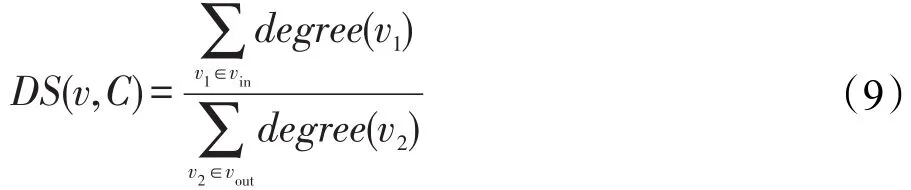
其中,vin表示核心簇中与v相连的结点集合;vout表示核心簇外与v相连的结点。DS(v,C)用来衡量一个结点v是核心C的附件的可能性,其值越大表示结点v属于核心C的附件的可能性越大。
3 基于鸽子优化算法的聚类算法PIOC
3.1 基于鸽子优化算法的聚类模型
PIOC算法主要分为两个阶段:地图罗盘操作阶段和地标操作阶段。第一阶段,先通过全局探索找到核心簇的种子结点,再通过地图罗盘操作中鸽子位置的更新形成核心簇。第二阶段,在地标算子的迭代过程中将附件蛋白质不断聚到核心簇中最终形成蛋白质复合物。该模型的设计中考虑的蛋白质复合特征有3个:核心附件结构,复合物内部蛋白质间最短距离不超过2,蛋白质复合物是个高密度的簇。
(1)地图罗盘操作
鸽子位置初始化:对整个PPI网络中的结点按照其NCC值降序排序,取前cn个结点作为核心簇的种子结点,每个种子结点看成一只鸽子。确定其下一步要飞向的候选位置:对于每一只鸽子,将其对应的核心簇的紧密邻接点(即到核心簇中每个结点的最短距离都不超过2的邻居结点)看作鸽子的候选位置。选最优位置:选出候选位置中优先权最高的位置作为最优位置,优先权是指该结点与簇内结点形成的边的聚集系数之和。位置更新:若最优位置优先权值大于0,则鸽子飞向最优位置,将优先权最高的紧密邻接点聚到鸽子所代表的核心簇中,否则,此鸽子的迭代结束。重复迭代上述过程,更新鸽子的位置,更新鸽子候选位置,更新最优位置,每更新一次位置就有一个最优邻接点聚到鸽子所代表的核心簇中,直到该核心簇的密度小于阈值dens_th时,结束第一阶段的更新。当每只鸽子完成地标罗盘操作里的迭代时就形成了一个核心簇。
(2)地标操作
对每只鸽子所代表的核心簇的紧密邻接点按附着度值DS进行排序。更新中心鸽子位置:将紧密邻接点中到核心簇的附着度最大的邻接点作为中心鸽子的位置。更新鸽子位置:若中心鸽子的位置处附着度DS值大于1,则更新鸽子位置,即将此紧密邻接点作为该鸽子的附件蛋白质聚到鸽子所代表的核心簇中,迭代更新中心鸽子位置和其他鸽子位置;若中心鸽子位置处的DS值小于1,则此只鸽子的地标操作结束,此鸽子对应的簇就是一个蛋白质复合物。
基于鸽子优化算法的聚类模型解决了当前大多数蛋白质复合物识别方法不能同时考虑网络的全局性和局部性的缺点,并考虑到了蛋白质复合物的内部核心附件结构。
鸽子算法聚类模型如图1所示。
3.2 PIOC算法描述
PIOC算法的输入是一个由蛋白质顶点和蛋白质相互作用边组成的网络图G=(V,E)。该算法由三部分组成:鸽子的初始化,地图罗盘算子和地标算子。整个算法的描述如下。
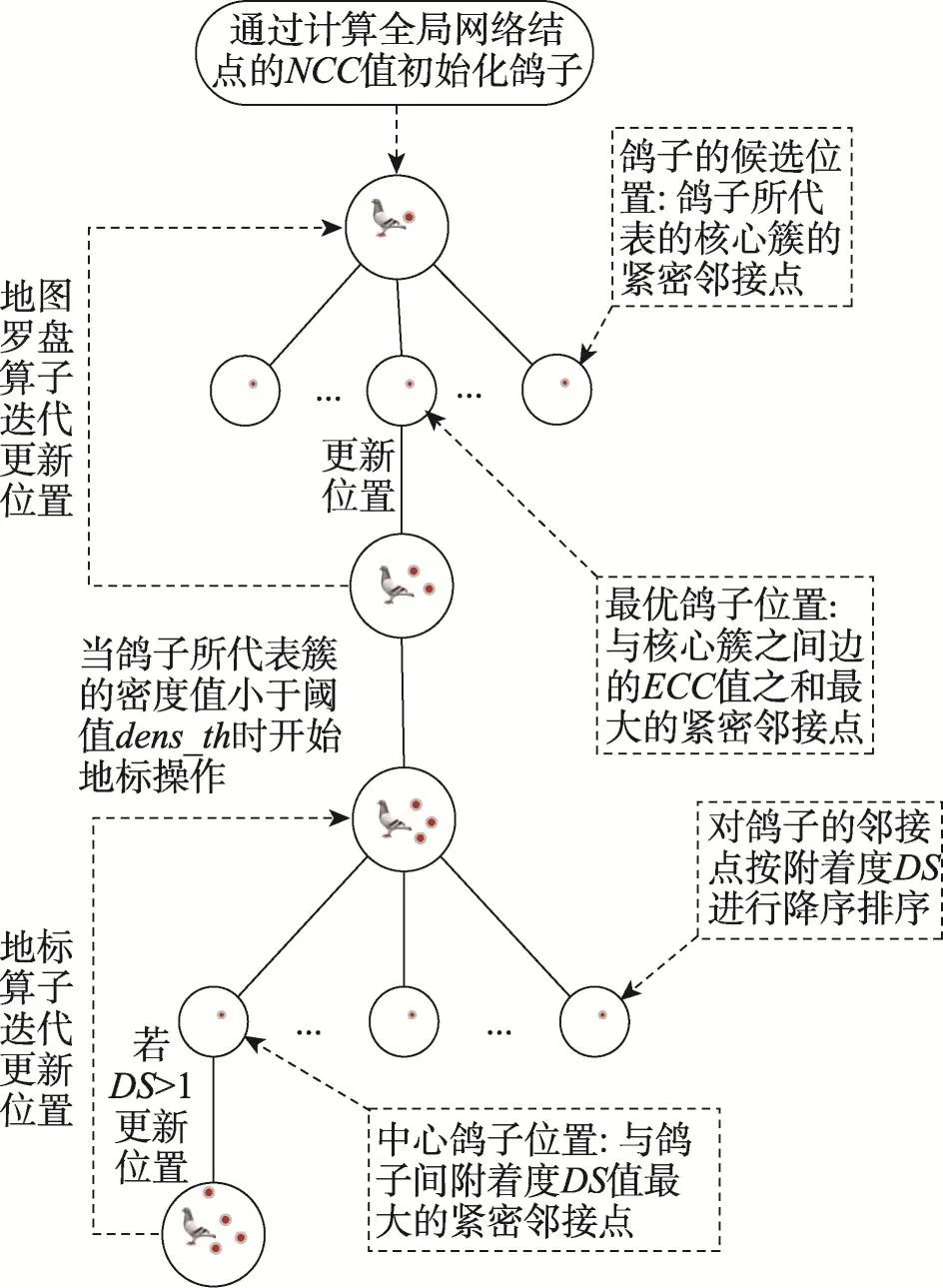
Fig.1 Clustering model of PIOC图1 PIOC聚类模型
算法PIOC聚类
输入:PPI网络对应的图G=(V,E)
输出:预测出来的蛋白质复合物C
初始化:计算出G图中每个顶点的聚集系数NCC,计算出图中每条边的聚集系数ECC,Xbest表示最优鸽子位置,Xc表示中心鸽子位置,cn表示鸽子的数量,dens_th表示密度阈值,C表示鸽子群,c表示每一只鸽子,标记 flag初始化为0。
(1)初始化鸽子
Sq←all nodes sorted in non-increasing order based on NCCvalue
C←the firstcnnode inSq(2)地图罗盘算子
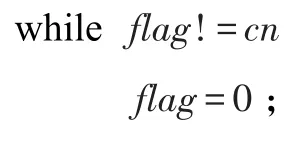

output C;//输出整个鸽子群,此时每只鸽子代表一个蛋白质复合物
鸽子优化算法PIO与聚类算法的对应关系如表1所示。
4 实验结果与分析
4.1 实验数据和评价标准
本文采用DIP数据库中20140427的酵母菌S.cerevisiae数据集测试PIOC算法的性能。静态的DIP蛋白质相互作用网络包含4 997个蛋白质结点和21 554对相互作用,网络的平均度是8.626 8。采用的标准数据集是CYC2008标准数据集,其中包含408个蛋白质复合物,1 628个蛋白质结点。

Table1 Corresponding relationships between PIO algorithm and clustering procedure of PPI network表1 PIO算法与PPI网络中聚类过程的对应关系
为了评估检测出的蛋白质复合物的性能,本文使用常见的3种统计评估方法:准确率Precision、查全率Recall和调和平均值F-measure[21]。Precision评估了预测的蛋白质复合物与标准库中已知蛋白质复合物相比的准确率;Recall评估了标准数据库中蛋白质复合物被识别出来的准确率;F-measure显示了准确率和查全率两者的调和平均值。这3个评价方法的计算公式如下所示:
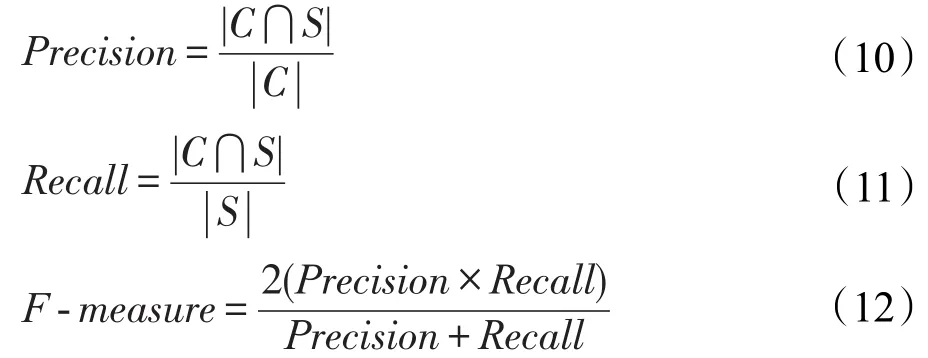
其中,C表示预测的蛋白质复合物;S表示标准库中的蛋白质复合物。
识别出来的复合物A与已知复合物B的匹配程度OS(A,B)[24]的计算公式如下:

其中,VA和VB分别表示蛋白质复合物A和蛋白质复合物B中的顶点集合。若两个复合物的匹配程度OS(A,B)超过给定的阈值,则称这两个复合物匹配。一般对于酵母蛋白质的数据集,取这个阈值为0.2[24],即被检测出来的蛋白质复合物与标准库中复合物的匹配程度值大于0.2时,这两个复合物被认为是相互匹配的。当OS的值为1时被认为是完美匹配。
4.2 参数分析
PIOC聚类算法中涉及两个参数:第一个是初始化鸽子时选取前cn个结点作为鸽子群体,则鸽子的群体数量cn是个针对不同PPI网络设置不同值的参数;第二个是核心簇的密度阈值dens_th,其在地图罗盘算子里控制迭代次数也控制着核心簇的大小。根据以前的研究工作,参数dens_th一般设为0.7[11]。当cn取由100到800的不同值时得到不同的聚类结果。图2显示了cn的取值与聚类结果性能之间的变化关系。
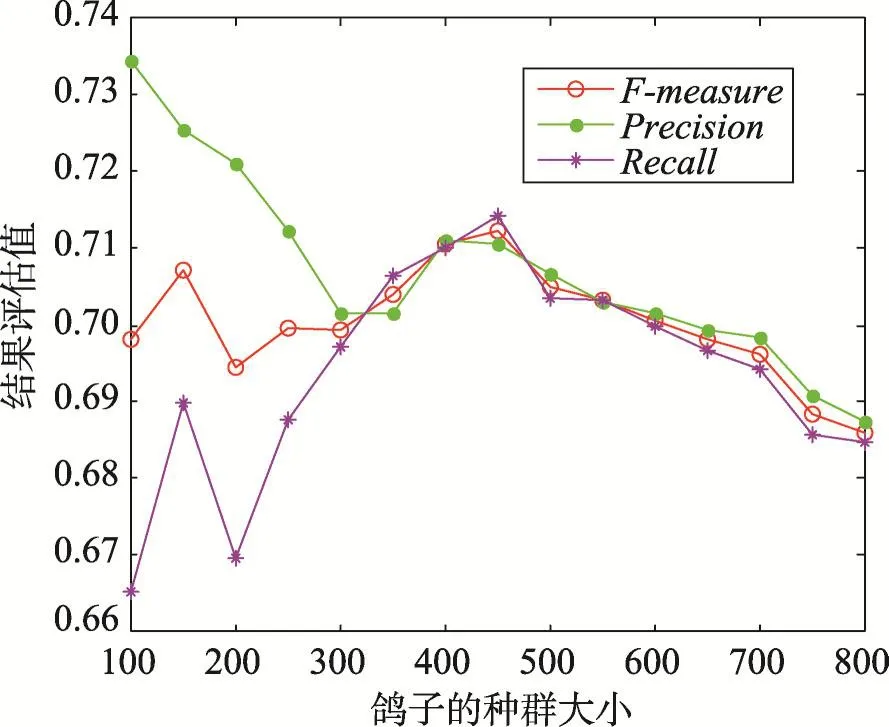
Fig.2 Influence of parametercnon clustering evaluation图2 参数cn对聚类结果的影响
由图2可以看出,随着参数cn的增大,准确度Precision呈现递减的趋势,而查全率Recall却呈现先增加再减小的趋势。因为当cn很小时,簇所取的核心种子结点都具有很高的点聚集系数,所以产生的蛋白质复合物都是很紧密的簇,具有很高的准确率。但与此同时,识别出来的蛋白质复合物对蛋白质的覆盖量少,因此查全率比较小。当cn越大,后面种子结点的聚集系数越小,故准确率下降。但随着蛋白质复合物数量的增加,其查全率也增加,当增加到一定值时,由于后面很多性能不好的种子结点的加入,产生很多错误蛋白质复合物,降低了查全率。由调和平均值F-measure可以看出,当cn取450左右时其总体性能最好。
4.3 与已知蛋白质复合物比较
将由PIOC得到的聚类结果与标准库中的蛋白质复合物进行比较,分析本文预测的蛋白质复合物中正确和错误的蛋白质。表2中随机选择PIOC识别的蛋白质复合物中的7个复合物进行分析。从表2中可以看出,PIOC聚类方法能正确识别蛋白质复合物中的绝大多数蛋白质,其中个别复合物会包含错误蛋白质或者漏掉个别蛋白质,但标准蛋白质复合物中的蛋白质基本上都能被识别出来。
图3可视化了表2中7号蛋白质复合物。图(a)表示标准库中的蛋白质复合物,不带背景颜色的结点为PIOC未识别出来的蛋白质。图(b)表示PIOC检测出来的蛋白质复合物,其中不带背景颜色的蛋白质为被错误识别的蛋白质。YIR025W和YGR-260C未被识别出来,是因为其与簇外的邻接点链接更紧密。YGR225W被错误识别出来,是因为其只与核心簇中蛋白质YKL022C相连。YLR451W被错误识别出来是因为其度为2,簇内的邻接点度值大于簇外的度值。

Table2 Analysis of 7 protein complexes in results表2 结果中7个蛋白质复合物的分析
4.4 性能比较
为了测试PIOC的性能,将PIOC的聚类结果与MCL[7]、CMC、MCODE、DPClus、HC-PIN、Clsuter-ONE、COACH[9-14]、F-MCL[16]这些典型算法的聚类结果进行比较。实验结果的获得都是基于DIP静态PPI网络和MIPS网络,并且被比较的算法都取各自的最优参数。PIOC的参数cn取450,核心密度dens_th取0.7。
表3展示了上面几种聚类算法得到的结果详情,表4对比了几种算法结果性能。
由表3可以看出,PIOC识别出来的蛋白质复合物数量和覆盖的蛋白质数量接近标准库中复合物的标准,但蛋白质复合物的平均大小偏大,这也说明PIOC识别出来的蛋白质复合物之间有很多重叠。与之对应,F-MCL和DPClus等方法识别出来的蛋白质复合物很多,覆盖的蛋白质数量也很大,但大多数蛋白质是错误的,导致性能低。
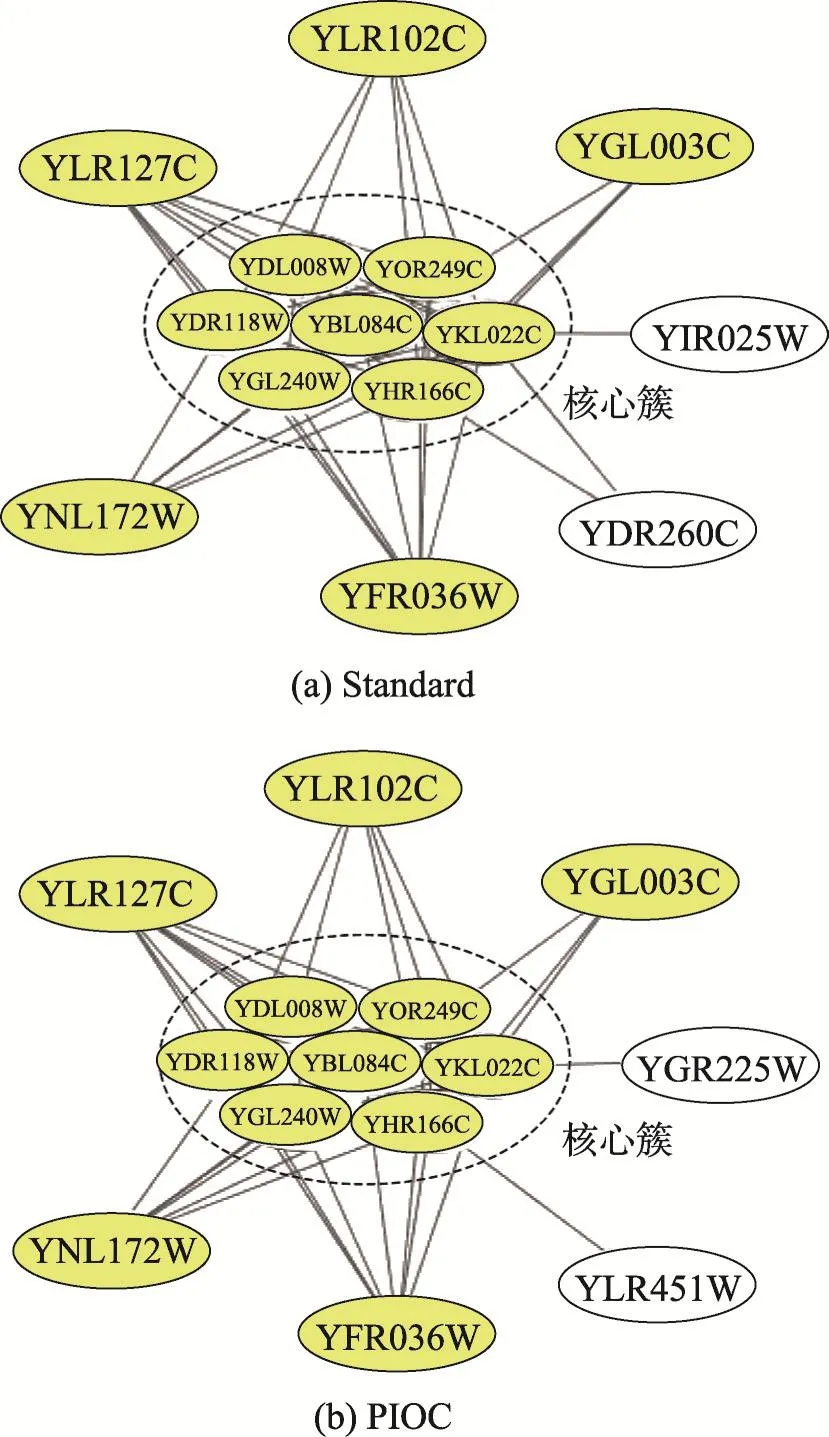
Fig.3 Visualization of comparison图3 可视化对比
表4显示了蛋白质复合物性能比较。可以看出PIOC在Precision、Recall和F-measure评价标准上都优于其他蛋白质复合物识别方法,也就是说PIOC比其他蛋白质复合物识别方法更能有效地识别蛋白质复合物。
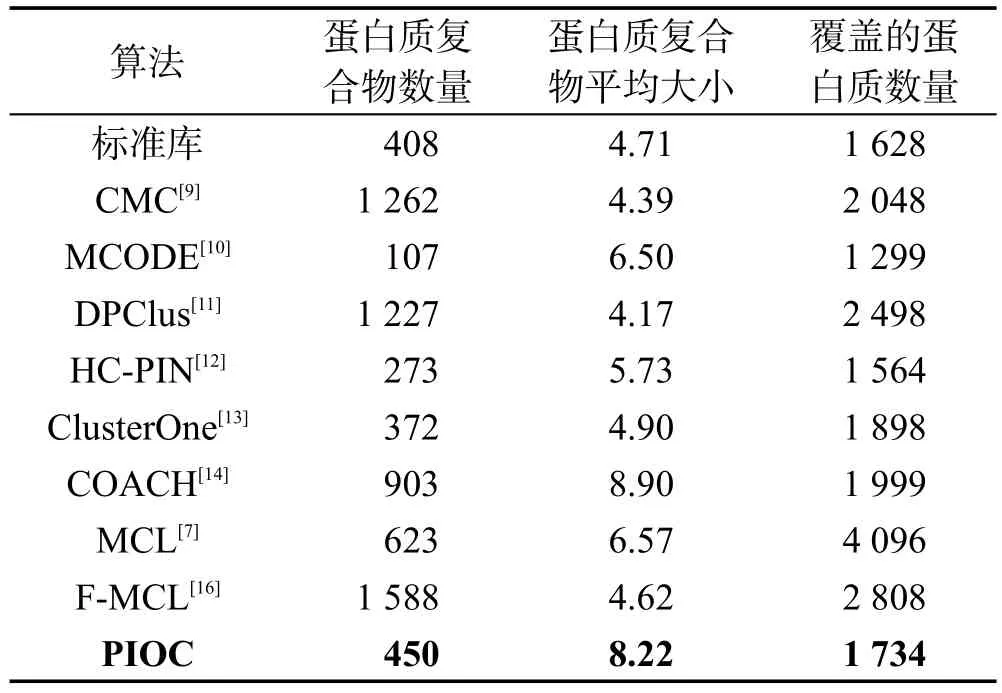
Table3 Clusters description of several clustering algorithms表3 几种聚类算法的聚类结果展示
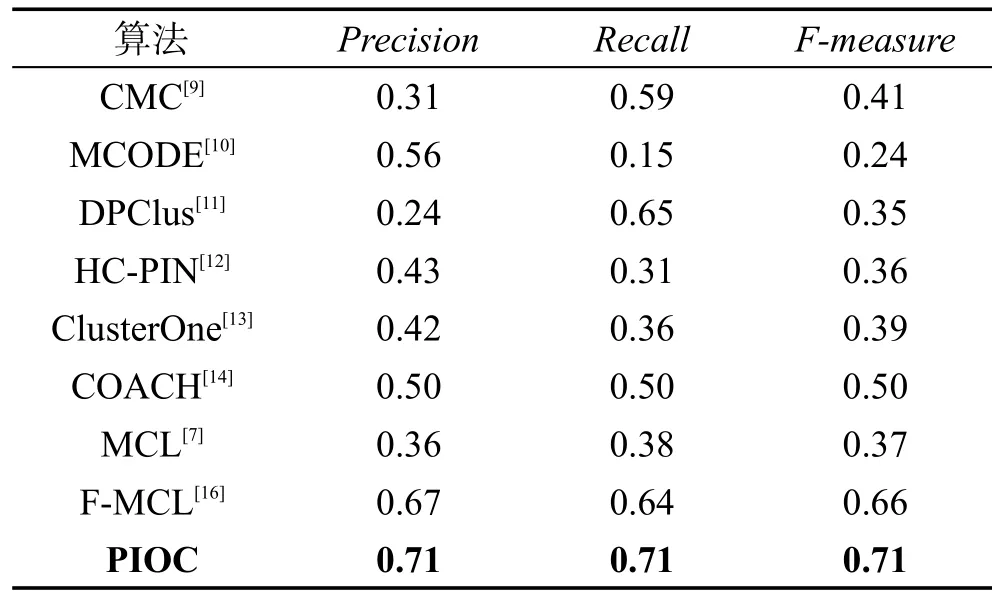
Table4 Performance comparison of different clustering algorithms with PIOC表4 几种聚类算法与PIOC的性能比较
5 结束语
本文将鸽子优化算法与蛋白质复合物的特性相结合,提出了一种蛋白质复合物识别算法PIOC。该算法将鸽子的全局寻优搜索和局部寻优搜索特性应用到PPI网络中寻找更优蛋白质复合物问题上,先通过全局搜索形成蛋白质复合物的核心,再通过局部搜索将附件蛋白质聚集到核心簇中形成蛋白质复合物。与传统的聚类算法相比,PIOC不仅考虑到PPI网络的全局性和局部性,还考虑到蛋白质复合物的核心-附件结构。与其他蛋白质复合物识别方法的性能比较显示,PIOC能更有效地识别蛋白质复合物。但是该算法的聚类数目需要人为控制以及蛋白质复合物间的重叠过多,这些问题还有待进一步研究。
[1]Spirin V,Mirny LA.Protein complexes and functional modules in molecular networks[J].Proceedings of the National Academy of Sciences of the United States of America,2003,100(21):12123-12128.
[2]DangerR,Pla F,MolinaA,et al.Towards a protein-protein interaction information extraction system:recognizing named entities[J].Knowledge-Based Systems 2014,57:104-118.
[3]Yu Liang,Gao Lin,Sun Penggang.Research on algorithms for complexes and functional modules prediction in proteinprotein interaction networks[J].Chinese Journal of Computers,2011,34(7):1239-1252.
[4]Guo Maozu,Dai Qiguo,Xu Liqiu,et al.One protein complexes identifying algorithm based on the novel modularity function[J].Journal of Computer Research and Development,2014,51(10):2178-2186.
[5]Tang Dongming,Zhu Qingxin,Yang Fan,et al.Efficient cluster analysis method for protein sequences[J].Journal of Software,2011,22(8):1827-1837.
[6]Girvan M,Newman M E J.Community structure in social and biological networks[J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(12):7821-7826.
[7]EnrightA J,Van Dongen S,Ouzounis CA.An efficient algorithm for large-scale detection of protein families[J].Nucleic Acids Research,2002,30(7):1575-1584.
[8]Li Xiaoli,Wu Min,Kwoh C K,et al.Computational approaches for detecting protein complexes from protein interaction networks:a survey[J].BMC Genomics,2010,11(1):S3.
[9]Liu Guimei,Wong L,Chua H N.Complex discovery from weighted PPI networks[J].Bioinformatics,2009,25(15):1891-1897.
[10]BaderGD,Hogue C W.An automated method for finding molecular complexes in large protein interaction networks[J].BMC Bioinformatics,2003,4:1-13.
[11]Altaf-Ul-Amin M,Shinbo Y,Mihara K,et al.Development and implementation of an algorithm for detection of protein complexes in large interaction networks[J].BMC Bioinformatics,2006,7:207-228.
[12]Wang Jianxin,Li Min,Chen Jian'er,et al.Afast hierarchical clustering algorithm for functional modules discovery in protein interaction networks[J].IEEE/ACM Transactionson Computational Biology and Bioinformatics,2011,8(3):607-620.
[13]Nepusz T,Yu Haiyuan,PaccanaroA.Detecting overlapping protein complexes in protein-protein interaction networks[J].Nature Methods,2012,9(5):471-472.
[14]Wu Min,Li Xiaoli,Kwoh C K,et al.Acore-attachment based method to detect protein complexes in PPI networks[J].BMC Bioinformatics,2009,10(1):169-185.
[15]Lei Xiujuan,Tian Jianfang,Ge Liang,et al.The clustering model and algorithm of PPI network based on propagating mechanism of artificial bee colony[J].Information Sciences,2013,247(15):21-39.
[16]Lei Xiujuan,Wang Fei,Wu Fangxiang,et al.Protein complex identification through Markov clustering with firefly algorithm on dynamic protein-protein interaction networks[J].Information Sciences,2016,329(6):303-316.
[17]Li Honghao,Duan Haibin.Bloch quantum-behaved pigeoninspired optimization for continuous optimization problems[C]//Proceeding of the Chinese Guidance,Navigation and Control Conference,Yantai,China,Aug 8-10,2014.Piscataway,USA:IEEE,2014:2634-2638.
[18]Duan Haibin,Qiao Peixin.Pigeon-inspired optimization:a new swarm intelligence optimizer for air robot path planning[J].International Journal of Intelligent Computing and Cybernetics,2014,7(1):24-37.
[19]Li Cong,Duan Haibin.Target detection approach for UAVs via improved pigeon-inspired optimization and edge potential function[J].Aerospace Science and Technology,2014,39:352-360.
[20]Sun hang,Duan Haibin.Member S:PID controller design based on prey-predator pigeon-inspired optimization algorithm[C]//Proceedings of the 2014 International Conference on Mechatronics and Automation,Tianjin,China,Aug 3-6,2014.Piscataway,USA:IEEE,2014:1416-1421.
[21]Zhang Aidong.Protein interaction networks[M].New York:Cambridge University Press,2009.
[22]Wang Jianxin,Li Min,Wang Huan,et al.Identification of essential proteins based on edge clustering coefficient[J].IEEE/ACM Transactions on Computational Biology&Bioinformatics,2012,9(4):1070-1080.
[23]Li Min,Wang Jianxin,Chen Jian'er.Distance measure-based algorithm for identification of protein complexes[J].Journal of Jilin University,2010,40(5):1318-1323.
[24]Wang Jianxin,Peng Xiaoqing,Li Min,et al.Construction and application of dynamic protein interaction network based on time course gene expression data[J].Proteomics,2013,13(2):301-312.
附中文参考文献:
[3]鱼亮,高琳,孙鹏岗.蛋白质网络中复合体和功能模块预测算法研究[J].计算机学报,2011,34(7):1239-1252.
[4]郭茂祖,代启国,徐立秋,等.一种蛋白质复合体模块度函数及其识别算法[J].计算机研究与发展,2014,51(10):2178-2186.
[5]唐东明,朱清新,杨凡,等.一种有效的蛋白质序列聚类分析方法[J].软件学报,2011,22(8):1827-1837.
[23]李敏,王建新,陈建二.基于距离测定的蛋白质复合物识别算法[J].吉林大学学报,2010,40(5):1318-1323.
Identification of Protein Complexes by Simulating Process of Pigeon-Inspired Optimization*
DING Yulian,LEI Xiujuan+,DAI Cai
College of Computer Science,Shaanxi Normal University,Xi’an 710062,China
+Corresponding author:E-mail:xjlei168@163.com
DING Yulian,LEI Xiujuan,DAI Cai.Identification of protein complexes by simulating process of pigeoninspired optimization.Journal of Frontiers of Computer Science and Technology,2017,11(8):1279-1287.
Detecting protein complexes is crucial to understand the principles of cellular organization and predict diseases.Yet up to now,the performance of existing protein complex detection algorithms is not very ideal for the deficiencies of low accuracy,sensitive to noisy data and so on.This paper proposes a novel algorithm named pigeoninspired optimization clustering(PIOC)algorithm.It puts forward the concepts of cluster's closely adjacent nodes and attachment's attachment degree on core,and identifies protein complexes by simulating the process of pigeons finding home based on these two concepts.The PIOC algorithm combines the characters of first global search then local search of pigeon-inspired optimization(PIO)algorithm and the core-attachment structure of protein complexes.Particularly,it first develops the cores of protein complexes by the global search of PIO's map and compass operator,and then forms the protein complexes by gathering in the attachment proteins to the cores based on the local search of PIO landmark operation.The experimental results on yeast protein DIP dataset demonstrate that PIOC is more effective in detecting protein complexes than the state-of-the-art complex detection algorithms.
an was born in 1975.She
the Ph.D.degree from Northwestern Polytechnical University in 2005.Now she is a professor and Ph.D.supervisor at Shaanxi Normal University.Her research interests include intelligent computing and bioinformatics. 雷秀娟(1975—),女,陕西长安人,2005年于西北工业大学获得博士学位,现为陕西师范大学教授、博士生导师,主要研究领域为智能计算,生物信息学。

DING Yulian was born in 1992.She is an M.S.candidate at Shaanxi Normal University.Her research interests include data mining and bioinformatics.丁玉连(1992—),女,河南信阳人,陕西师范大学计算机科学学院硕士研究生,主要研究领域为数据挖掘,生物信息。

DAI Cai was born in 1984.He received the Ph.D.degree from Xidian University in 2014.Now he is a lecturer at Shaanxi Normal University.His research interest is multi-objective optimization.代才(1984—),男,安徽阜南人,2014年于西安电子科技大学获得博士学位,现为陕西师范大学讲师,主要研究领域为多目标优化。
A
:TP391
*The National Natural Science Foundation of China under Grant Nos.61502290,61401263(国家自然科学基金);the Industrial Research Project of Science and Technology in Shaanxi Province under Grant No.2015GY016(陕西省工业科技攻关项目);the Postdoctoral Science Foundation of China under Grant No.2015M582606(中国博士后科学基金);the Graduated Student Innovation Foundation of Shaanxi Normal University under Grant No.2015CXS030(陕西师范大学研究生创新基金).
Received 2016-06,Accepted 2016-08.
CNKI网络优先出版:2016-08-15,http://www.cnki.net/kcms/detail/11.5602.TP.20160815.1659.010.html
ISSN 1673-9418 CODEN JKYTA8
Journal of Frontiers of Computer Science and Technology 1673-9418/2017/11(08)-1279-09
10.3778/j.issn.1673-9418.1607034
E-mail:fcst@vip.163.com
http://www.ceaj.org
Tel:+86-10-89056056
Key words:protein-protein interaction(PPI);pigeon-inspired optimization algorithm;protein complex;clustering
