福建漳浦凡纳滨对虾海水养殖中后期水体细菌群落多样性分析
2017-08-10胡东王丽萍赵苒邵宗泽
胡东,王丽萍,赵苒,邵宗泽*
(1.厦门大学 公共卫生学院,福建 厦门 361102;2.国家海洋局第三海洋研究所 国家海洋局海洋生物遗传资源重点实验室,福建 厦门 361005)
福建漳浦凡纳滨对虾海水养殖中后期水体细菌群落多样性分析
胡东1,2,王丽萍2,赵苒1,邵宗泽2*
(1.厦门大学 公共卫生学院,福建 厦门 361102;2.国家海洋局第三海洋研究所 国家海洋局海洋生物遗传资源重点实验室,福建 厦门 361005)

凡纳滨对虾;微生物群落多样性;16S rRNA基因高通量测序;水质因子
1 引言
凡纳滨对虾(Litopenaeusvannamei)因其具有生长快、抗病能力强、养殖经济效益显著,成为当今世界上水产养殖产量最高的三大优良对虾品种之一[1],其2011年产量占全球对虾产量的76%[2]。凡纳滨对虾养殖要经历幼虫到成体5个生长阶段[3],大约3~4个月的生长周期才可以上市。养殖动物肠道及养殖水体中微生物的种类和数量[4—5],水质因子[6](溶氧、温度、pH、化学需氧量、氮等),饲料投喂,藻类生长状况等是对虾能否健康成长的关键因素,了解养殖水体中微生物群落结构、多样性变化及微生物菌群与水体环境因子的相互作用对改善养殖水体、预防对虾疾病发生至关重要。
第二代测序技术为我们研究环境中微生物多样性提供了高效的方法,结合微生物独有的16S rRNA保守基因,这种方法被广泛应用到环境微生物多样性分析[5, 7—9]。16S rRNA高通量测序技术已用于淡水环境凡纳滨对虾养殖微生物多样性分析,Zhang等发现在凡纳滨对虾淡水养殖池中,变形菌门、蓝细菌、放线菌门和拟杆菌门是水体微生物的主要类群;变形菌门、蓝细菌、厚壁菌门、酸酐菌门、绿湾菌门、拟杆菌门是沉积物环境的主要类群[7]。但鲜有对虾海水养殖环境微生物菌群的相关研究报道,因此,我们的研究可以弥补这方面的空白,丰富人们对凡纳滨对虾养殖环境微生物菌群多样性的认识。
本研究利用Illumina高通量测序技术分析了凡纳滨对虾高位池养殖水体细菌多样性的动态变化过程,并分析了菌群与水质参数间的相互关系。研究结果将有助于了解对虾不同养殖时期水体菌群变化规律,认识影响对虾养殖水体菌群变化的环境因子,以期通过调控水质因子,达到改善养殖环境微生物的群落结构的目的,从而促进对虾健康养殖。
2 材料和方法
2.1 样品采集
采样点为福建漳浦县凡纳滨对虾海水养殖场高位池[5号池,0.9亩(1亩=666.67 m2),40万虾苗] (24°01′43″ N,117°50′39″E)。采样时间为2015年12月24日至2016年3月24日,即对虾虾苗度过“早期死亡综合症”到对虾收成,实验周期为3个月。在离岸2 m和虾池中心设置2个采样点(S1和S2,第131天只有S1点),使用经过高温湿热灭菌处理的有机玻璃采水器,采集上、中、下3层水样共3 L于无菌采样瓶中,共采集了7个时间点(养殖第40、59、67、77、95、115、131天 )的13个水体样品。采集的水样命名为P5D&S#,P5表示5号池,D&表示采样时间编号,S#表示采样点编号(下同)。水样立即置于放有冰袋的样品箱内保存,并运回实验室用0.22 μm滤膜过滤,滤膜于-80℃冰箱中保存。
2.2 水质参数检测

表1 主要水质指标及检测方法
2.3 基因组DNA提取
利用MOBIO水样基因组提取试剂盒(14900-100-NF,PowerWater® DNA Isolation Kit)进行基因组DNA提取。提取的基因组DNA质量和浓度分别用1%琼脂糖凝胶电泳和nanodrop2000超微量分光光度计检测,提取的DNA在-80℃冰箱中保存。
2.4 16S rRNA基因高通量测序
提取的基因组DNA送上海美吉生物医药科技有限公司,采用引物338F(5′-3′,ACTCCTACGGGAGGCAGCAG)和806R(5′-3′,GGACTACHVGGGTWTCTAAT)[10]对样品进行PCR扩增,引物两端带有特异的barcode序列,以识别不同样本的16S rRNA基因扩增产物;随后采用Miseq PE300测序平台对PCR产物测序。
2.5 微生物多样性分析
对原始数据进行拼接、过滤,得到优化序列。然后基于有效数据将相似性达97%的序列进行OTU(Operational Taxonomic Units)聚类。采用RDP classifier贝叶斯算法[11]对97%相似水平的OTU代表序列进行分类学分析,并在各水平统计每个样品的群落组成。基于OTU聚类分析结果,对OTU进行多种多样性指数分析,如Alpha多样性指数[12];基于分类学信息,进行群落结构的统计分析[13];基于系统发育,进行unifrac等分析[14];并结合水质因子进行RDA关联分析。在上述分析的基础上,进行一系列群落结构和系统发育等深入的统计学和可视化分析。
3 结果
3.1 养殖池水体的水质参数
为了探讨水质与水体环境微生物菌群多样性的关系,对采集水样进行水质分析(表2)。在养殖中后期,溶氧基本维持在饱和状态(6.0~8.0 mg/L);温度变化范围为 20~24.6℃,pH维持在弱碱性(7.2~7.5),变化较小;COD波动较大,变化范围为6.9~12.0 mg/L;硝酸盐氮值变化范围为0.96~1.49 mg/L,亚硝酸盐氮值变化范围为0.02~0.35 mg/L,氨态氮值变化较大,波动范围为0.11~4.96 mg/L,其中硝态氮的浓度变化与以往研究明显不同。本研究发现,硝酸盐浓度在养殖过程中总体上呈下降趋势,而以往的报道是养殖后期浓度升高[15—16]。

表2 养殖池水质参数的7次调查结果
3.2 Illumina 序列多样性评价
采用Illumina miseq测序平台对采集的7个时间点13个水样进行16S rRNA基因高通量测定,13个样品中共得到823 363条序列,序列长度在281~520 bp之间,每个样品的序列数目在55 449~73 485条之间,抽平后得到619 775条高质量序列,每个样品的序列数目为47 675条。稀释曲线分析(图1a)显示,随着测序数量的增加,稀释曲线逐渐趋于平缓,表明该测序量已经基本能够反映该实验中细菌群落多样性。按照97%相似度对OTU进行划分,其覆盖度均大于 0.99,每个样品得到的OTU数目在294~412之间。通过Shannon-Wiener多样性指数对养殖水体细菌群落多样性进行评估,如图1b所示,第40天样品微生物多样性最高,而第67天样品微生物多样性最低,其余时间点采集的样品微生物多样性大致相似。
3.3 主成分和聚类分析
主成分分析(Pcoa)结果显示(图2a),每个时间点采集的两个重复样本都能很好地聚在一起,说明相同时间采集的重复样本细菌群落多样性相似性较高。Unifrac聚类分析和科水平的细菌群落Heatmap分析(图2b,2c)也是如此。此外,其他水平的细菌群落Heatmap分析结果与科水平的细菌群落Heatmap分析结果基本一致,相同时间点采集的两个重复样本都能很好的聚在一起,不同时间差异较大。但是,不同时间段之间仍有一定的相似性,第40、77、95与131天之间的细菌组成相似,而第67天与115天细菌组成较为相似。
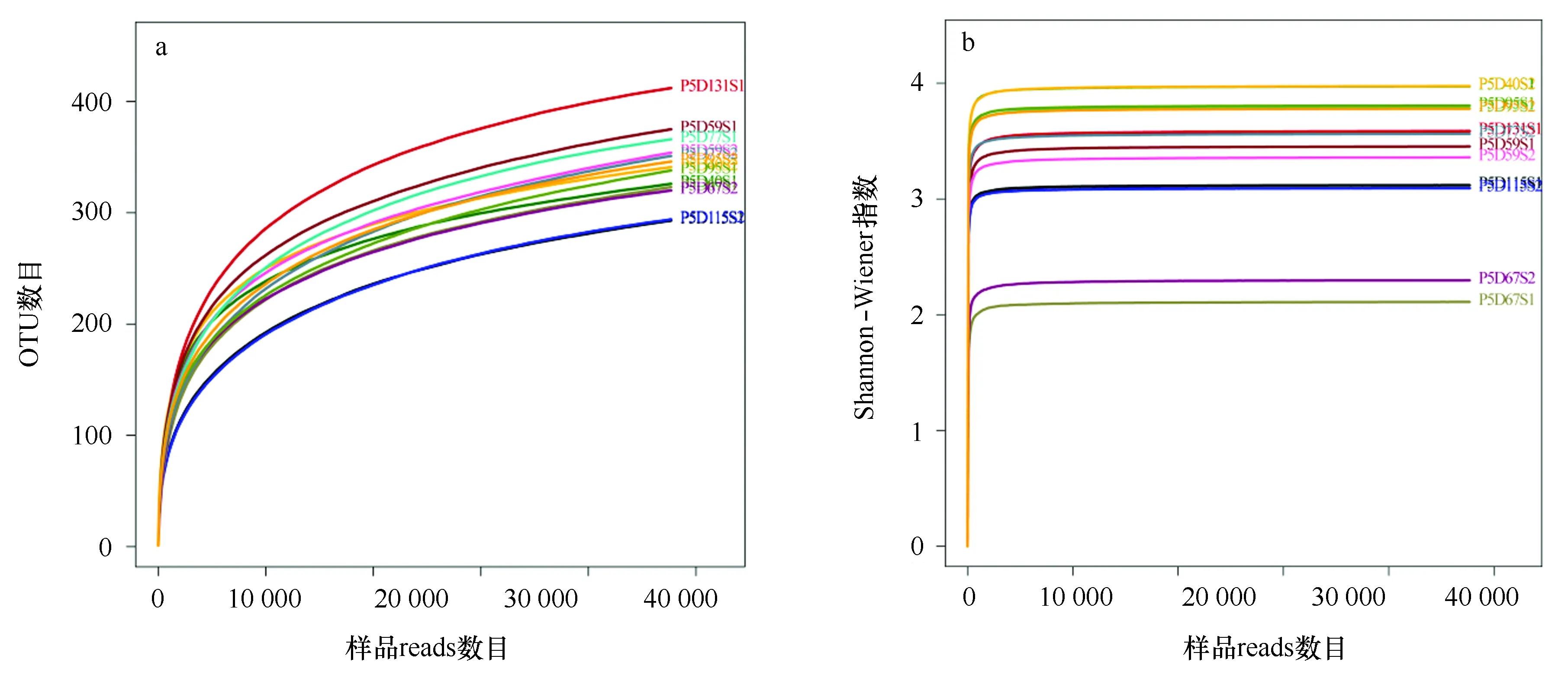
图1 各样品的细菌16S rRNA基因的α多样性分析Fig.1 Diversity analysis of 16S rRNA genes in different samplesa.不同样品的稀释曲线;b.不同样品的香农威纳指数图a.Rarefaction curve of different samples; b.Shannon-Wiener rarefaction curve plot of different samples
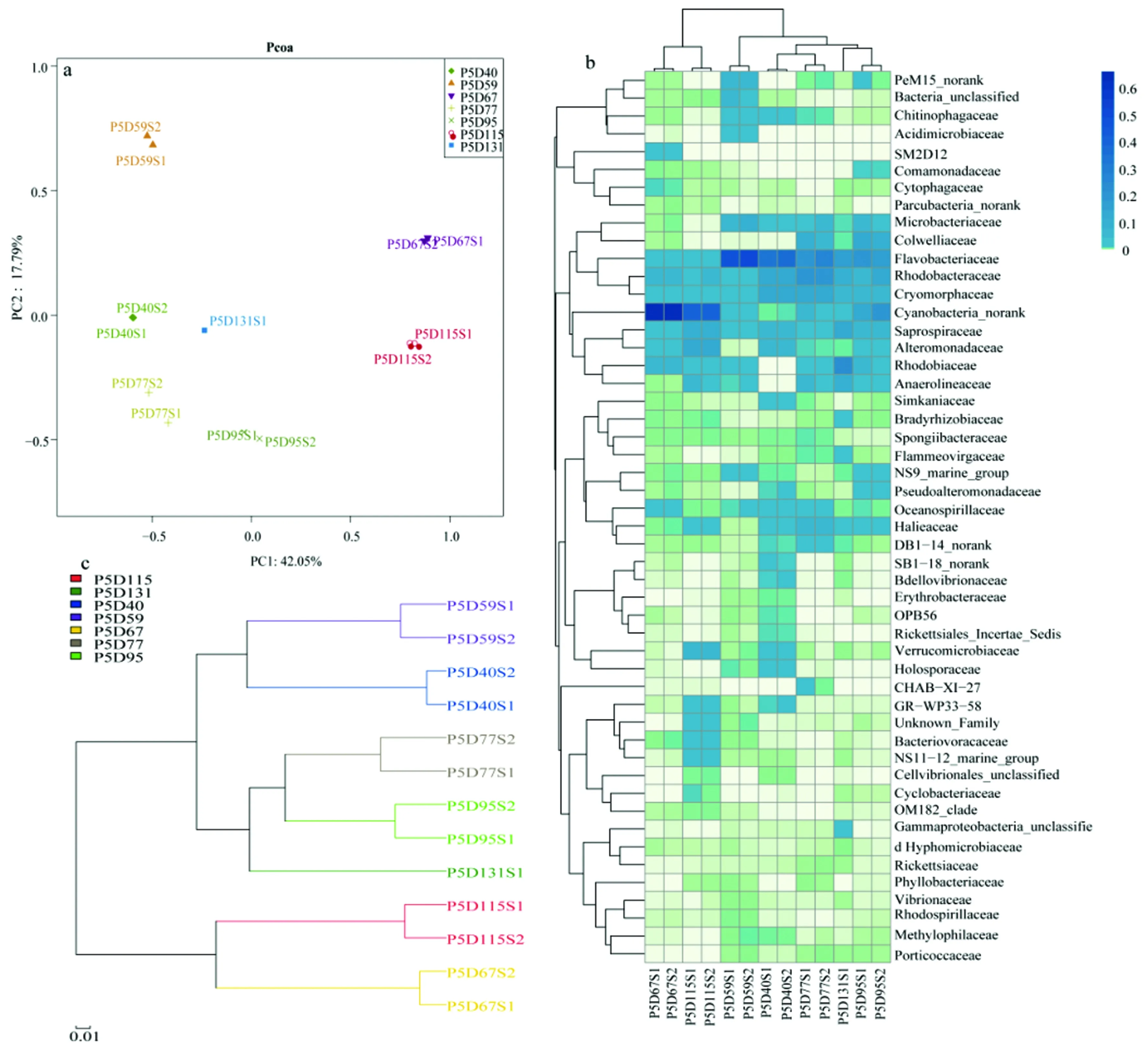
图2 不同样品细菌群落相似性分析Fig.2 Similarity analysis of microbial populations in different samplesa. 细菌群落的Pcoa分析; b. 科水平的细菌群落heatmap; c. 基于Unifrac的聚类分析a. Pcoa analysis; b. heatmap of bacterial populations at the family level; c. cluster analysis based Unifrac

图3 门水平细菌群落组成Fig.3 Bacterial community composition at the phylm level
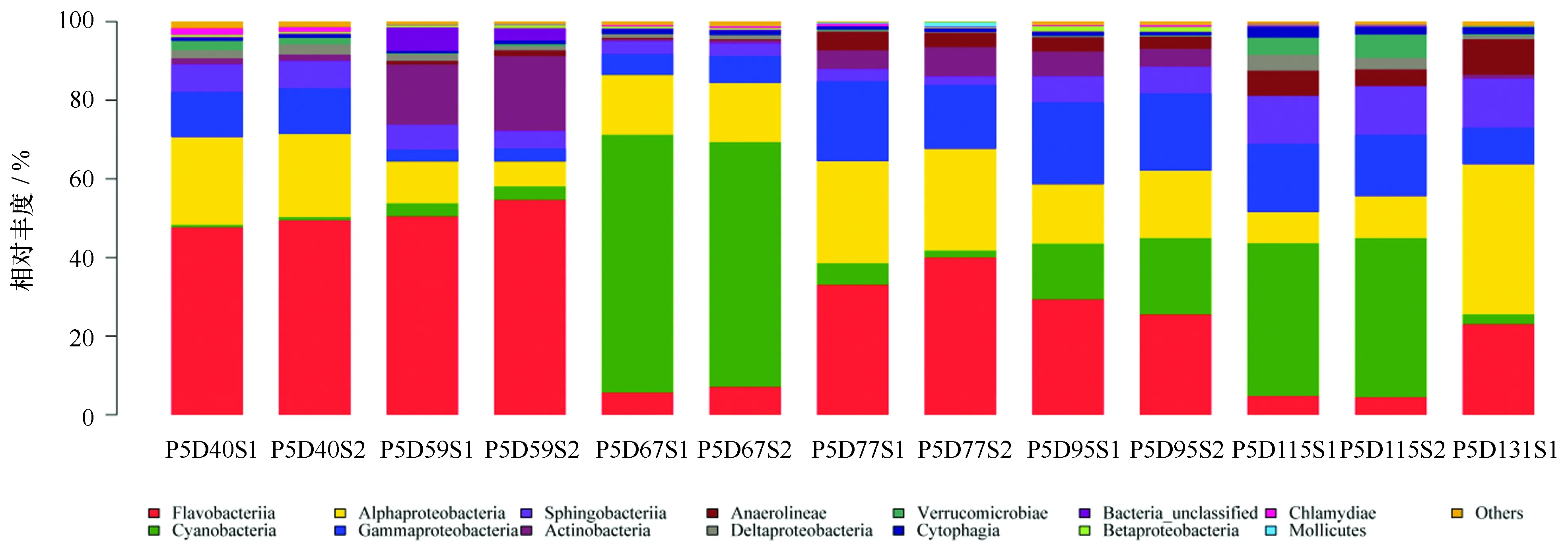
图4 纲水平上细菌群落变化Fig.4 Changes of bacterial community in all samples at the class level

图5 水质参数与细菌群落之间的关系Fig.5 The correlation between water quality parameters and bacterial community compositiona.细菌群落组成与水质的RDA分析; b.科水平上细菌群落与水质相关性热图a. Redundancy analysis (RDA) of the bacterial community composition with water quality parameters (red arrows); b. heatmap showing the correlation between bacterial community and the water quality parameters at the family level
3.4 细菌多样性组成
在本研究采集的7个时间点13个样品中,共检测到19个门、35个纲、80个目、135个科、254个属的细菌。另外,未能鉴定的序列数总共为5 337条,这表明样本中存在着大量未知的类群。根据样品中各微生物所测定的reads数,把每个样品中reads数大于总reads数10%的门定义为优势门。在不同养殖时期水样检测中,蓝细菌(Cyanobacteria)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、绿湾菌门(Chloroflexi)、疣微菌门(Verrucomicrobia)、放线菌门(Actinobacteria)、衣原体(Chlamydiae)等7个门是主要的优势类群(图3)。
根据细菌群落组成图可知,养殖水体中的绝大多数序列可以归类于变形菌门和拟杆菌门(图3),并且这2个门在采样期间一直是主要类群。其中,变形菌门在7个采样时间点所占的比例分别为36.2%、13.7%、22.7%、44.6%、38.0%、29.6%、48.8%,主要包括α-变形菌纲(Alphaproteobacteria)和γ-变形菌纲(Gmmaproteobacteria)(图4),α-变形菌纲在第59、115天样品丰度较低(分别为8.5%和9.3%),而在其他采样时间点丰度普遍较高(丰度变化为15.1%~38.1%),γ-变形菌纲则在第59、67天丰度较低(比例分别为3.2%和6.1%),其他采样时间点丰度普遍偏高(丰度变化为9.3%~20.1%);拟杆菌门在7个采样时间点所占的比例分别为56.3%、58.8%、10.7%、40.0%、35.4%、19.2%、37.9%,主要包括黄杆菌纲(Flavobacteriia)和Sphingobacteriia (图4),黄杆菌纲在第67、115天样品丰度较低(分别为6.3%和4.7%),而在其他采样时间点丰度普遍偏高(丰度变化为23.1%~52.6%),Sphingobacteriia在养殖后期丰度较高(各时间点所占比例为7.0%、5.4%、3.2%、2.6%、6.7%、12.2%、12.4%)。蓝细菌为养殖过程中处于次优势的一个类群,在7个采样时间点波动较大,所占的比例分别为0.7%、3.4%、63.9%、3.6%、16.8%、39.7%、2.5%(图3)。
整个采样期间,黄杆菌科(Flavobacteriaceae)、红杆菌科(Rhodobacteraceae)、Cryomorphaceae、Cyanobacteria_norank、腐螺旋菌科(Saprospiraceae)、红菌科(Rhodobiaceae)等优势类群(图2b),广泛存在于各个养殖时期,并且基本都是优势菌群。拟杆菌门的黄杆菌科在整个采样阶段占优势地位,最高比例可达48.0%,但第67、115天所占比例仅为2.5%、1.5%。α-变形菌纲的红杆菌科在整个采样期间也都被检测到,从第40天占整个种群的13.4%降低到第59、67天的3.1%和5.1%,在第77天又成为优势种群(19.2%),随后在第95、115、131天分别降低到9.4%、4.8%和10.7%;相似的现象在拟杆菌门的 Cryomorphaceae上被体现,其在整个种群中所占比例由第40天的13.8%减少到第59、67天的3.3%和3.2%,在第77天成为优势种群(13.6%),随后在第95、115、131天分别降低到5.8%、2.7%和5.3%。属于拟杆菌门的腐螺旋菌科在整个采样期间也是优势类群,其在整个菌群中所占比例由第40天到第95天的低比例(所占比例分别为5.0%、2.6%、2.7%、1.7%、6.4%)升高到第115、131天的10.4%和12.0%;相似的现象也在α-变形菌纲的红菌科上得到体现,其在整个种群中所占比例由采样前期低比例(第40天、59天所占比例分别为0%和2.5%)升高到第131天的23.1%。Cyanobacteria_norank表现出与门水平蓝细菌相同的变化,所占比例变化为0.7%~63.8%。
3.5 细菌群落与水质参数关系

4 讨论
本研究通过Illumina高通量测序技术初步揭示了凡纳滨对虾养殖中后期养殖水体中细菌群落多样性变化及其与水质之间的相互关系。Pcoa分析和聚类分析表明,养殖中期第40天和第59天细菌群落组成相似,第77、95天与养殖后期第131天细菌群落组成更相似,而第67天和第115天样品与其他时间点的样品差异较大。此外,同一个时间点采集的两个样品重复性较好,说明数据较为可靠。从7个时间点采集的13个水体样品中共获得619 775条高质量序列,检测到的细菌群落归属于19个门、35个纲、80个目、135个科、254个属,说明该养殖水体中细菌群落具有很高的多样性。注释结果表明,这些序列主要属于变形菌门、拟杆菌门和蓝细菌等3个门类。此外,未能鉴定的序列数总共为5 337条,占总序列数比例为0.86%,说明凡纳滨对虾养殖环境中还存在着大量未描述过的类群。
在本研究中,变形菌门、拟杆菌门广泛存在于凡纳滨对虾各个养殖时期的水体中,并且为优势类群。相似的结果在前人的研究中也发现,在凡纳滨对虾淡水养殖系统中,养殖水体温度高时,放线菌门、unclassified bacteria、变形菌门和拟杆菌门是主要的类群;养殖水体温度低时,主要的优势类群是放线菌门、变形菌门和拟杆菌门[18]。同样,Zhang等发现在凡纳滨对虾淡水养殖池中,变形菌门、蓝细菌、放线菌门和拟杆菌门是水体微生物的主要类群[7]。Huang等研究发现,不同生长时期凡纳滨对虾肠道的优势类群是变形菌门、拟杆菌门和放线菌门[5]。由此可见,变形菌门和拟杆菌门是凡纳滨对虾肠道和水体环境常见的优势类群。此外,在本研究中蓝细菌在第67、115天样品中占绝对优势,文献报道光照强并且营养盐丰富,有利于蓝细菌繁殖[19],但是实验记录同时期为阴雨天气,说明其他环境参数影响着蓝细菌丰度变化。
尽管不同养殖时期水体微生物在门水平上组成较相似,但在纲水平和科水平上,细菌类群构成变化较大。在纲水平上,黄杆菌纲、α-变形菌纲和γ-变形菌纲为第40天样品的优势类群,第59天样品的主要类群为黄杆菌纲和放线菌纲(Actinobacteria),而第67天样品的优势类群波动较大,转变为Cyanobacteria_norank和α-变形菌纲。第77、95、131天样品菌群以黄杆菌纲、α-变形菌纲、和γ-变形菌纲为主,而第115天样品的优势类群为蓝细菌、γ-变形菌纲和Sphingobacteriia。可以看出α-变形菌纲、γ-变形菌纲和黄杆菌纲是纲水平上的主要优势类群。变形细菌是所有细菌中最大和最具多样性的一个族群,具有较强的适应性,它们在水体中分布广泛[20],其中α-和γ-变形菌纲在海水浮游细菌中尤为重要。已有的关于黄杆菌的研究表明,黄杆菌可以改善底质环境状况,在维持菌落方面起重要作用,其多为有益菌[20],这可能是该虾池健康养殖的主要原因。在科水平上,黄杆菌科、红杆菌科、Cryomorphaceae、Cyanobacteria_norank、腐螺旋菌科、红菌科等类群,广泛存在于养殖水体中,并且为各个养殖时期的优势或者次优势菌。科水平上,黄杆菌科、红杆菌科和Cryomorphaceae为第40天样品的优势菌类群,黄杆菌科和微杆菌科(Microbacteriaceae)为第59天样品的优势类群,蓝细菌为第67天样品的优势菌,黄杆菌科、红杆菌科和Cryomorphaceae 为第77天样品的优势类群,黄杆菌科、蓝细菌和科韦尔氏科(Colwelliaceae)为第95天样品的优势类群,蓝细菌、腐螺旋菌科和交替单胞菌科(Alteromonadaceae)为第115天样品的优势菌类群,交替单胞菌科、红杆菌科、腐螺旋菌科和红菌科为第131天样品的优势类群。黄杆菌科在整个采样期间是占优势的微生物类群,研究报道表明黄杆菌(Flavobacteriumpsychrophilum)在鳟鱼(Trout)养殖中是一类严重的病原菌,可以导致虹鳟鱼鱼苗综合症(RTFS)和冷水病(CWT)[21];Tamlana作为一种潜在的益生菌可以产生琼脂酶[22];Tenacibaculummaritimum可以导致鱼类产生gliding bacterial disease和 tenacibaculosis/flexibacteriosis等疾病[23]。同样红细菌科在整个采样期间是占优势的微生物类群,研究表明玫瑰杆菌(Roseobacter)能在大菱鲆(Scophthatmusmaximus) 仔鱼的饲养环境中建立一个有益的微生物群落,可以抑制致病菌的生存[24],降低大菱鲆幼鱼的死亡率[25]。值得注意的是,上述这些主要的优势类群在养殖的第59、67、115天表现出相对较低的丰度,如纲水平上α-变形菌纲在第59、115天丰度较低,γ-变形菌纲在59、67天丰度较低,黄杆菌纲在67、115天丰度较低;科水平上黄杆菌科在第67、115天丰度较低,红杆菌科、Cryomorphaceae在第57、67、115天丰度较低。
在整个采样期间还检测到黄杆菌属(Flavobacterium),希瓦氏菌属(Shewanella),弧菌属(Vibrio),假单胞菌属(Pseudomonas)和Escherichia-Shigella等水生动物潜在的条件致病菌[7, 24, 26—27],除黄杆菌属和弧菌属外,其他菌属在所有样品中都维持低丰度(小于0.1%)。其中弧菌为对虾常见的致病菌,已知副溶血弧菌(Vibrioparahemolyticus)、哈维氏弧菌(V.harveyi)、坎贝氏弧菌(V.campbellii)都是凡纳滨对虾主要的致病菌[28],本研究中发现的弧菌主要为坎贝氏弧菌。潜在致病菌低reads数表明在进水温度适宜的情况下,增加换水频数不失为一种预防对虾病害发生的有效方法。此外,本研究在对各个时期养殖水体微生物的多样性分析中,同样检测到一定丰度的潜在益生菌,主要包括芽孢杆菌属(Bacillus)[29]、蛭弧菌属(Bdellovibrio)[24]、拟杆菌属(Bacteroides)[7]、乳球菌属(Lactococcus)[24],尽管它们的丰度低,但这些益生菌会降低潜在致病菌等危害因素导致对虾发病几率增加的可能性。
5 结论

致谢:感谢国家海洋局第三海洋研究所海化实验室林彩老师对水质参数COD测定提供的帮助;感谢国家海洋局第三海洋研究所海洋生物遗传资源重点实验室杨洋、杨硕、黄兆斌、刘阳对水样采集提供的帮助。感谢厦门大学公共卫生学院郭东北老师提供的水样采集仪器。
[1] Tu C, Huang H T, Chuang S H, et al. Taura syndrome in pacific white shrimp penaeus vannamei cultured in Taiwan[J]. Diseases of Aquatic Organisms, 1999, 38(2): 159-161.
[2] Yu Yang, Wei Jiankai, Zhang Xiaojun, et al. SNP discovery in the transcriptome of white Pacific shrimpLitopenaeusvannameiby next generation sequencing[J]. PLoS One, 2014, 9(1): e87218.
[3] Zheng Yanfen, Yu Min, Liu Yan, et al. Comparison of cultivable bacterial communities associated with Pacific white shrimp (Litopenaeusvannamei) larvae at different health statuses and growth stages[J]. Aquaculture, 2016, 451: 163-169.
[4] Boutin S, Bernatchez L, Audet C, et al. Network analysis highlights complex interactions between pathogen, host and commensal microbiota[J]. PLoS One, 2013, 8(12): e84772.
[5] Huang Zhaobin, Li Xiaoyi, Wang Liping, et al. Changes in the intestinal bacterial community during the growth of white shrimp,Litopenaeusvannamei[J]. Aquaculture Research, 2014, 47(6): 1737-1746.
[6] Berdjeb L, Ghiglione J F, Jacquet S. Bottom-up versus top-down control of hypo-and epilimnion free-living bacterial community structures in two neighboring freshwater lakes[J]. Applied and Environmental Microbiology, 2011, 77(11): 3591-3599.
[7] Zhang Hao, Sun Zhenli, Liu Bo, et al. Dynamic changes of microbial communities inLitopenaeusvannameicultures and the effects of environmental factors[J]. Aquaculture, 2016, 455: 97-108.
[8] Rosselli R, Romoli O, Vitulo N, et al. Direct 16S rRNA-seq from bacterial communities: a PCR-independent approach to simultaneously assess microbial diversity and functional activity potential of each taxon[J]. Scientific Reports, 2016, 6: 32165.
[9] Yan Qingyun, Bi Yonghong, Deng Ye, et al. Impacts of the three gorges dam on microbial structure and potential function[J]. Scientific Reports, 2015, 5: 8605.
[10] Xu Nan, Tan Guangcai, Wang Hongyuan, et al. Effect of biochar additions to soil on nitrogen leaching, microbial biomass and bacterial community structure[J]. European Journal of Soil Biology, 2016, 74: 1-8.
[11] Wang Qiong, Garrity G M, Tiedje J M, et al. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267.
[12] Schloss P D, Gevers D, Westcott S L. Reducing the effects of PCR amplification and sequencing artifacts on 16S rRNA-based studies[J]. PLoS One, 2011, 6(12): e27310.
[13] Oberauner L, Zachow C, Lackner S, et al. The ignored diversity: complex bacterial communities in intensive care units revealed by 16S pyrosequencing[J]. Scientific Reports, 2013, 3: 1413.
[14] Rivas N M, Burton O T, Wise P, et al. A microbiota signature associated with experimental food allergy promotes allergic sensitization and anaphylaxis[J]. Journal of Allergy and Clinical Immunology, 2013, 131(1): 201-212.
[15] 李卓佳, 李奕雯, 曹煜成, 等. 凡纳滨对虾海水高位池养殖水体理化因子变化与营养状况分析[J]. 农业环境科学学报, 2010, 29(10): 2025-2032.
Li Zhuojia, Li Yiwen, Cao Yucheng, et al. Analysis of physical-chemical factors variation and nutritional status of litopenaeus vannamei high level seawater ponds[J]. Journal of Agro-Environment Science, 2010, 29(10): 2025-2032.
[16] 张瑜斌, 章洁香, 詹晓燕, 等. 高位虾池养殖过程主要理化因子的变化及水质评价[J]. 水产科学, 2009, 28(11): 628-634.
Zhang Yubin, Zhang Jiexiang, Zhan Xiaoyan, et al. Changes in main physicochemical factors in water and assessment of water quality during culture in higher-level shrimp ponds[J]. Fisheries Science, 2009, 28(11): 628-634.
[17] Sheik C S, Mitchell T W, Rizvi F Z, et al. Exposure of soil microbial communities to chromium and arsenic alters their diversity and structure[J]. PLoS One, 2012, 7(6): e40059.
[18] Tang Yuyi, Tao Peiying, Tan Jianguo, et al. Identification of bacterial community composition in freshwater aquaculture system farming ofLitopenaeusvannameireveals distinct temperature-driven patterns[J]. International Journal of Molecular Sciences, 2014, 15(8): 13663-13680.
[19] 李爽, 姜华, 张德民, 等. 光合细菌及其在水产养殖上的应用[J]. 中国水产, 2007(1): 81-82.
Li Shuang, Jiang Hua, Zhang Demin, et al. Photosynthetic bacteria and its application in aquaculture[J]. China Fisheries, 2007(1): 81-82.
[20] 王姣姣, 李丹, 宋坚, 等. 不同时期刺参养殖池塘海水菌群结构分析[J]. 中国农业科技导报, 2015, 17(2): 134-140.
Wang Jiaojiao, Li Dan, Song Jian, et al. Structural analysis of seawater bacterial community from culture pond of apostichopus japonicus at different periods[J]. Journal of Agricultural Science and Technology, 2015, 17(2): 134-140.
[21] Stenholm A R, Dalsgaard I, Middelboe M. Isolation and characterization of bacteriophages infecting the fish pathogenflavobacteriumpsychrophilum[J]. Applied and Environmental Microbiology, 2008, 74(13): 4070-4078.
[22] 姜海峰, 刘小林, 常亚青, 等. 皱纹盘鲍(Haliotisdiscushannai)肠道潜在益生菌的筛选及对幼鲍生长的影响[J]. 海洋与湖沼, 2013, 44(2): 482-487.
Jiang Haifeng, Liu Xiaolin, Chang Yaqing, et al. Selection of potential probiotics from abalone intestional and their effects on growth of juvenile small abaloneHaliotisdiscushannal[J]. Oceanologia et Limnologia Sinica, 2013, 44(2): 482-487.
[23] Romero M, Avendano-Herrera R, Magarios B, et al. Acylhomoserine lactone production and degradation by the fish pathogen Tenacibaculum maritimum, a member of the Cytophaga-Flavobacterium-Bacteroides (CFB) group[J]. FEMS Microbiology Letters, 2010, 304(2): 131-139.
[24] Ninawe A S, Selvin J. Probiotics in shrimp aquaculture: avenues and challenges[J]. Critical Reviews in Microbiology, 2009, 35(1): 43-66.
[25] Bruhn J B, Nielsen K F, Hjelm M, et al. Ecology, inhibitory activity, and morphogenesis of a marine antagonistic bacterium belonging to theRoseobacterclade[J]. Applied and Environmental Microbiology, 2005, 71(11): 7263-7270.
[26] Duchaud E, Boussaha M, Loux V, et al. Complete genome sequence of the fish pathogenFlavobacteriumpsychrophilum[J]. Nature Biotechnology, 2007, 25(7): 763-769.
[27] Khashe S, Janda J M. Biochemical and pathogenic properties ofShewanellaalgaandShewanellaputrefaciens[J]. Journal of Clinical Microbiology, 1998, 36(3): 783-787.
[28] Wang Liping, Chen Yanwu, Huang Hui, et al. Isolation and identification ofVibriocampbelliias a bacterial pathogen for luminous vibriosis ofLitopenaeusvannamei[J]. Aquaculture Research, 2015, 46(2): 395-404.
[29] Nimrat S, Suksawat S, Boonthai T, et al. PotentialBacillusprobiotics enhance bacterial numbers, water quality and growth during early development of white shrimp (Litopenaeusvannamei)[J]. Veterinary Microbiology, 2012, 159(3/4): 443-450.
[30] Tas S, Yilmaz I N, Okus E. Phytoplankton as an indicator of improving water quality in the golden horn estuary[J]. Estuaries and Coasts, 2009, 32(6): 1205-1224.
[31] Tian Chuan, Tan Jing, Wu Xin, et al. Spatiotemporal transition of bacterioplankton diversity in a large shallow hypertrophic freshwater lake, as determined by denaturing gradient gel electrophoresis[J]. Journal of Plankton Research, 2009, 31(8): 885-897.
The diversity changes of bacterial community in mariculture water ofLitopenasusvannameiat Zhangpu, Fujian Province
Hu Dong1,2, Wang Liping2, Zhao Ran1, Shao Zongze2
(1.SchoolofPublicHealth,XiamenUniversity,Xiamen361005,China; 2.KeyLaboratoryofMarineBiogeneticResources,ThirdInstituteofOceanograph,StateOceanicAdministration,Xiamen361005,China)
Litopenasusvannamei; microbial community; 16S rRNA gene high throughput sequencing; water quality parameters
10.3969/j.issn.0253-4193.2017.08.009
2017-01-18;
2017-02-22。
厦门南方海洋研究中心项目(14CZP034HJ08);深远海(极地)微生物及其基因资源开发与产品应用示范 (201005032-1);国家自然科学基金项目(81673129)。
胡东(1990—),男,山东省新泰市人,主要研究方向为海洋环境微生物。E-mail:taianhudong@163.com
*通信作者:邵宗泽,男,研究员。E-mail:shaozz@163.com
S917.1
A
0253-4193(2017)08-0089-10
胡东,王丽萍,赵苒,等. 福建漳浦凡纳滨对虾海水养殖中后期水体细菌群落多样性分析[J].海洋学报,2017,39(8):89—98,
Hu Dong, Wang Liping, Zhao Ran,et al. The diversity changes of bacterial community in mariculture water ofLitopenasusvannameiat Zhangpu, Fujian Province[J]. Haiyang Xuebao,2017,39(8):89—98, doi:10.3969/j.issn.0253-4193.2017.08.009
