阿尔茨海默病淀粉样蛋白抗原表位`的生物信息学预测*
2017-08-07刘军莉汤峰刘川川崔森李占全
刘军莉,汤峰,刘川川,崔森,李占全
(1.青海大学高原医学研究中心;2.青海大学附属医院;3.高原医学应用基础重点实验室; 4.青海-犹他高原医学联合重点实验室,青海 西宁 810000)
阿尔茨海默病淀粉样蛋白抗原表位`的生物信息学预测*
刘军莉1,2,3,4,汤峰1,3,4,刘川川1,3,崔森2,3#,李占全2,3&
(1.青海大学高原医学研究中心;2.青海大学附属医院;3.高原医学应用基础重点实验室; 4.青海-犹他高原医学联合重点实验室,青海 西宁 810000)
目的 利用生物信息学预测阿尔茨海默病中淀粉样前体蛋白的二级结构及优势抗原表位,拟为今后基于Aβ蛋白表位疫苗的设计奠定基础。方法 用Genbank获取APP及氨基酸残基片段Aβ1~42蛋白的氨基酸序列,应用生物信息学在线软件SOPMA预测上述蛋白的二级结构,并通过IEDB、SYFPEITHI、Bcepred和ABCpred软件在线预测APP及Aβ1~42的T、B细胞抗原表位。同时对上述蛋白的亲水性、柔韧性、抗原倾向性及抗原暴露表面区域性进行预测。结果 APP及Aβ1~42蛋白二级结构中α-螺旋及延伸链比例分别为46.49%、52.38%,所占比例较高,有较强的抗原性,提示存在潜在的优势抗原性表位。进一步通过不同生物信息学方法分析出APP的T细胞表位位于37~49、16~28、1~13、11~24、30~43、42~0;Aβ1~42蛋白的T细胞表位位于3~11(675~683)、1~11(673~683)、22~31(696~705)、31~39(705~713)。APP蛋白潜在的B细胞表位位于49~62、72~85、350~365、637~652;Aβ1~42蛋白的B细胞表位位于4~19(676~691)、26~39(700~713)、11~26(683~698)、26~41(698~713)。结论 APP及Aβ1~42蛋白存在潜在的优势抗原性表位,且Aβ1~42的抗原优势表位与APP优势表位不同,针对Aβ1~42设计更具有特异性的表位疫苗对APP无影响从而发挥更好的预防、治疗作用。该结论为今后基于Aβ蛋白表位疫苗的设计奠定了理论基础。
阿尔茨海默病 APP蛋白 二级结构 表面抗原
β淀粉样蛋白(amyloid-β,Aβ)为淀粉样前体蛋白(amyloid precursor protein,APP)的酶解产物,由细胞分泌,Aβ在脑内的异常形成聚集产生的神经毒性作用已经被公认为阿尔茨海默病(Alzheimer’s disease,AD)形成和发展的关键因素[1-2]。APP经过β、γ分泌酶依次裂解为Aβ1~40 、Aβ25~35、Aβ1~42等多种片段。有研究表明AD患者脑内主要成分为Aβ1~42,而正常老年人和AD患者脑内均存在Aβ1~40[3],但Aβ1~42毒性更强,更容易聚集,形成Aβ沉积的核心,从而引发神经毒性作用[4]。因此以Aβ1~42为靶标研制疫苗阻断和清除Aβ沉积,是预防和治疗AD的一种有效策略。然而由于以Aβ1~42为抗原所产生的抗体会对APP产生相应的免疫识别,对正常的机体功能造成损伤。因此,如何激活机体免疫系统对Aβ1~42产生特异性高识别,并避免对APP蛋白正常功能的影响是AD预防与治疗的科学难题。
本研究拟利用在线生物信息学软件预测和分析APP及毒性片段Aβ1~42的二级结构特征,同时应用不同软件预测APP及Aβ1~42潜在的 T 细胞和B细胞的优势抗原表位,并寻找出Aβ1~42的优势表位避免引入APP蛋白的优势表位,为今后基于Aβ蛋白的表位疫苗的设计奠定理论基础。
1 材料与方法
1.1 获取APP蛋白的氨基酸序列
从GenBankhttps://www.ncbi.nlm.nih.gov/protein/178616?report=fasta)获取APP及Aβ1~42相应的氨基酸序列[5]。GenBank记录显示,APP蛋白由770个氨基酸组成,Aβ1~42由42个氨基酸组成。见表1。
表1 APP蛋白及A β1~42蛋白的氨基酸序列的组成

Table 1 Amino acid sequence of APP protein and A β1~42 protein
1.2 预测 APP蛋白及Aβ1~42蛋白的二级结构特征
应用在线生物信息学软件SOPMA(https://npsa-prabi.ibcp.fr/cgi- bin/secpred_sopma.pl)分析APP蛋白质序列的二级结构[6]。输入所获取APP蛋白及Aβ1~42蛋白的氨基酸序列,对这两个蛋白的二级结构的四个构象状态(α-螺旋结构、延长链结构、β-转角结构和无规则卷曲结构)分别进行分析。相似性阈值和窗口宽度的参数分别设置为8和17,其余参数为默认值[7]。
1.3 预测APP蛋白及Aβ1~42蛋白的T细胞抗原表位
明确APP及Aβ1~42蛋白的二级结构后,分别采用生物信息学软件[IEDB(http://tools.immuneepitope.org/main/index.html)[8]和Syfpeithi(http://www.syfpeithi.de)]进行人源 MHC1抗原 HLA-A*02:01 分析,以预测APP蛋白潜在的T细胞抗原表位。将APP及Aβ1~42蛋白质的氨基酸序列按要求分别输入上述网址,并且调整参数:“MHC allele(s)”设定为 HLA-A * 02:01,“length”设定为8、9、10,其余参数值不变。
1.4 预测APP蛋白及Aβ1~42蛋白的B细胞抗原表位
分别应用下列在线软件[Bcepred(http://www.imtech.resin/raghava/bcepred/bcepred_submission.html)和ABCpred(http://www.imtech.res.in/raghava/abcpred/)[9]分析APP及Aβ1~42蛋白质潜在的B细胞抗原表位。将上述蛋白的氨基酸序列按要求分别输入上述网址,并且调整参数:亲水性为 2;抗原性倾向为1.8;弹性为1.9;表面暴露面积为2.4;其余参数值不变。并设置 ABCpred 软件的抗原表位长度分别为10、12、14、16,其余参数不变。
2 结果
2.1 预测APP及Aβ1~42蛋白的二级结构特征
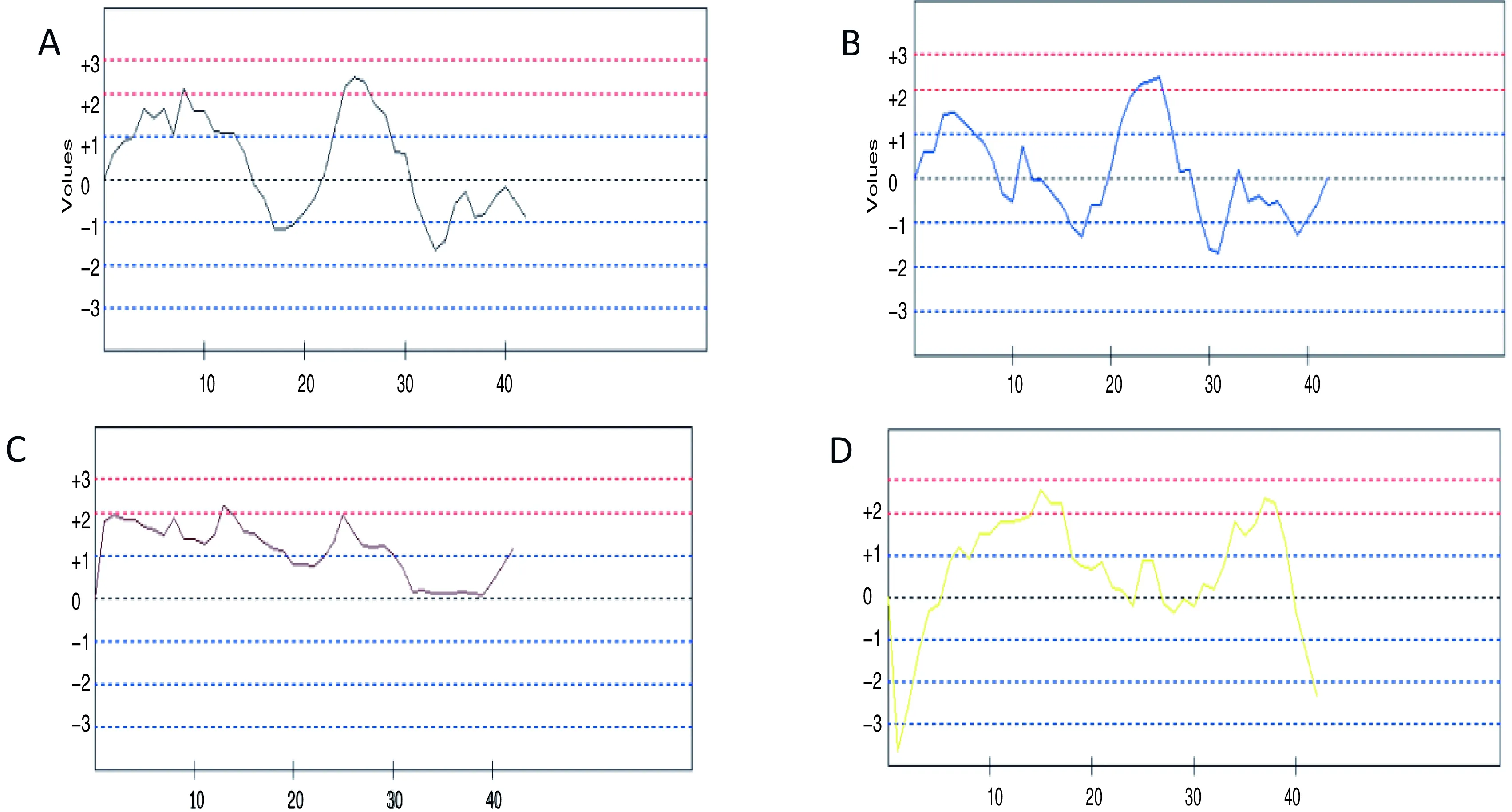
为评估APP及Aβ1~42蛋白的抗原特性,课题组应用生物信息学软件SOPMA预测上述蛋白的二级结构。APP蛋白质中的α螺旋结构和无规则卷曲结构极有可能是潜在的优势抗原表位。预测的二级结构特征见图1A、B。分析结果显示,APP的二级结构中α螺旋、无规则卷曲、延伸链、β转角结构的比例分别为46.69%、32.86%、13.38%、7.27%。见图1(A)。Aβ1~42蛋白中的延伸链结构和无规则卷曲结构极有可能是潜在的优势抗原表位。其二级结构中α螺旋、无规则卷曲、延伸链、β转角结构的比例分别为4.76%、23.81%、52.38%、19.05%。见图1(B)。
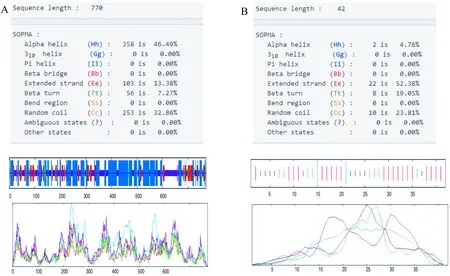
图中不同颜色的线条代表不同的二级结构:蓝色,α螺旋;绿色,β转角;红色,延伸链;紫色,无规卷曲.根据结构特征图可知APP蛋白在α螺旋最利于形成抗原表位,而Aβ1~42蛋白在延伸链有利于形成抗原表位.
Lines in different colors represent different secondary structures:Blue,α helix;green,β turn;red,extended strand;and purple, random coil.An increased number of α helix and extended strands in the protein corresponded with an increased likelihood of the protein forming an antigenic epitope.
图1 APP蛋白及Aβ1~42蛋白的二级结构预测
Figure 1 Secondary structure prediction results for the APP and Aβ1-42 protein
2.2 预测APP及Aβ1~42蛋白的T细胞抗原表位
若能明确APP及Aβ1~42蛋白抗原表位的精确位置,对于研发表位疫苗极其重要。在此次研究中,使用不同在线软件分别进行APP及Aβ1~42蛋白的T细胞表位预测。用IEDB软件预测高分值为99和100。然而,通过SYFPEITHI软件预测高分值为14和29。尽管这两种方法预测软件所采用的评分系统各不相同,但其共同点是分值较高的位点区域即为被预测的潜在抗原表位。见表2~3。
表2 使用IEDB和Syfpeithi在线预测APP的T细胞抗原表位
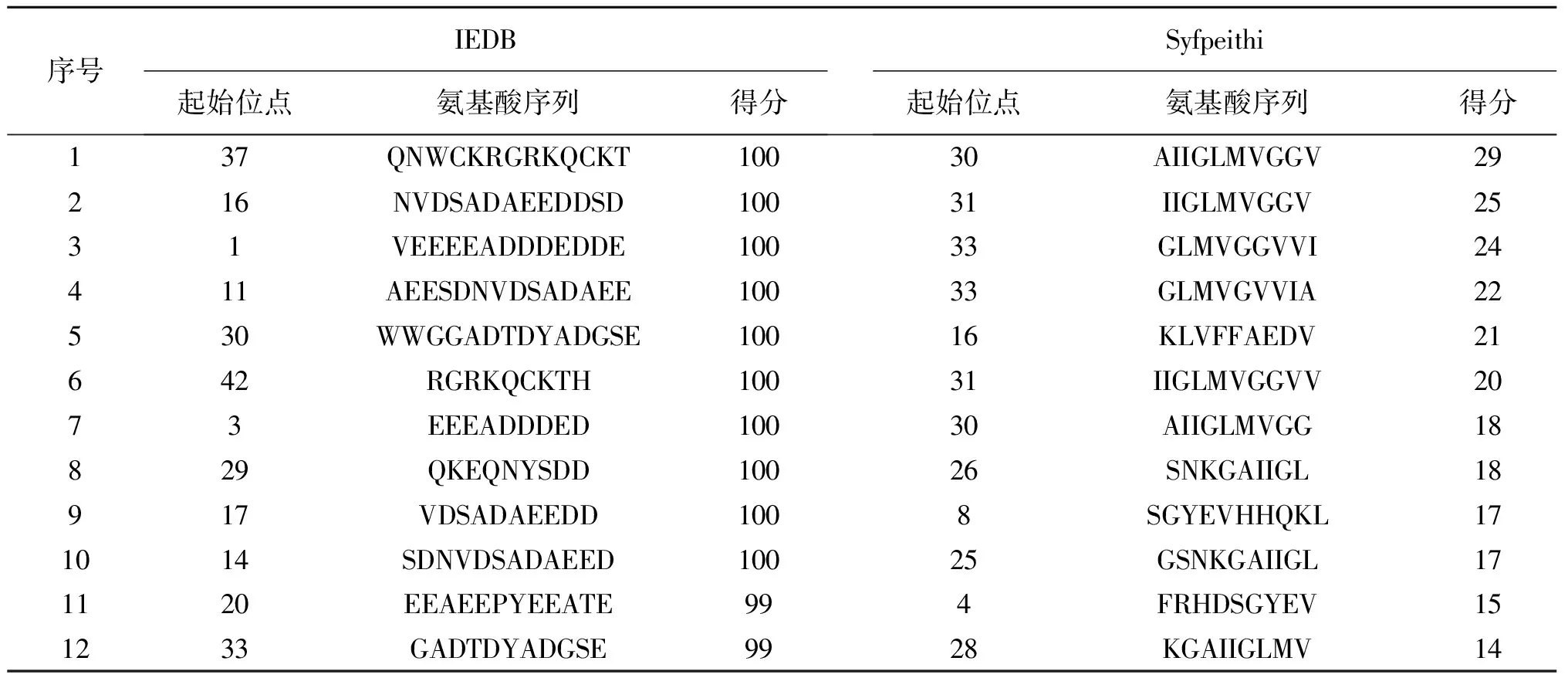
Table 2 Analysis of the T cell epitopes of APP protein using IEDB and Syfpeithi online prediction software
IEDE:http://tools.iedb.org/mhci/result_in_text/;Syfpeithi:http://www.syfpeithi.de.
表3 使用IEDB和Syfpeithi在线预测Aβ1~42的T细胞抗原表位
Table 3 Analysis of the T cell epitopes of Aβ1-42 protein using IEDB and Syfpeithi online prediction software
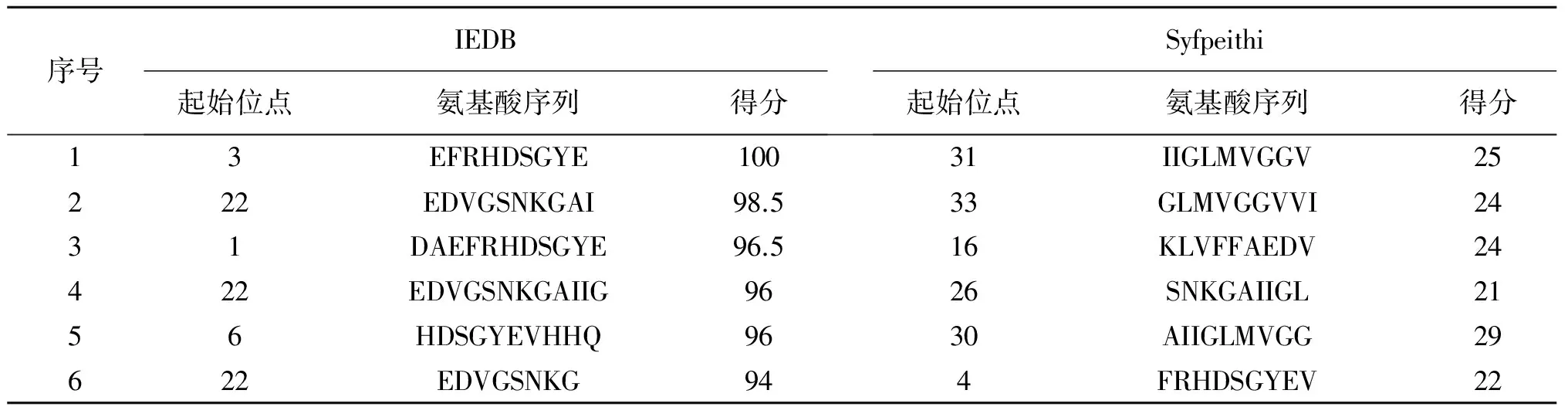
序号IEDB起始位点氨基酸序列得分Syfpeithi起始位点氨基酸序列得分13EFRHDSGYE10031IIGLMVGGV25222EDVGSNKGAI98.533GLMVGGVVI2431DAEFRHDSGYE96.516KLVFFAEDV24422EDVGSNKGAIIG9626SNKGAIIGL2156HDSGYEVHHQ9630AIIGLMVGG29622EDVGSNKG944FRHDSGYEV22
IEDE:http://tools.iedb.org/mhci/result_in_text/;Syfpeithi:http://www.syfpeithi.de.
2.3 预测APP及Aβ1~42蛋白的B细胞表位
使用Bcepred软件进行预测,对APP的氨基酸序列的亲水性、灵活性、抗原倾向性及抗原暴露表面区域性进行分析。预测分析结果见图2。预测分析出APP蛋白的有高亲水性的3个区域见图2 A:T192~205(氨基酸序列:ESDNVDSADAEEDD)、T214~222(氨基酸序列:DTDYADGSE)、T238~262(氨基酸序列:EEEEADDDEDDEDGDEVEEEAEEPV);预测出的2个弹性区域见图 2B:T53~57(氨基酸序列:DSDPS)、T98~102(氨基酸序列:CKRJR);2个可能的抗原倾向性区域见图 2C:T70~76(氨基酸序列:LQYCQEV)、110~121(氨基酸序列:HFVIPIRCLVGE)。2个可能的抗原表面暴露区域见图2D:T100~106(氨基酸序列:RGRKQCK)、T501~507(氨基酸序列:EQKDRQH)。对Aβ1~42蛋白的氨基酸序列的亲水性、灵活性、抗原倾向性及抗原暴露表面面积进行分析。分析出Aβ1~42蛋白的有高亲水性的1个区域见图3 A:T24~26(氨基酸序列:VGS);预测出的1个弹性区域见图3B:T23~25(氨基酸序列:DVG);1个抗原倾向性的区域见图3:T14~17(氨基酸序列:HKQL)。
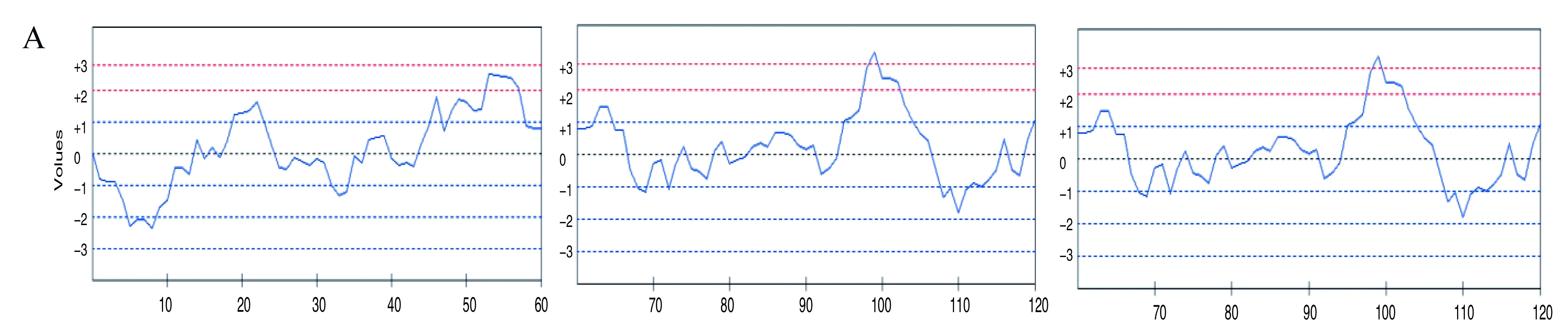
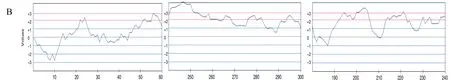
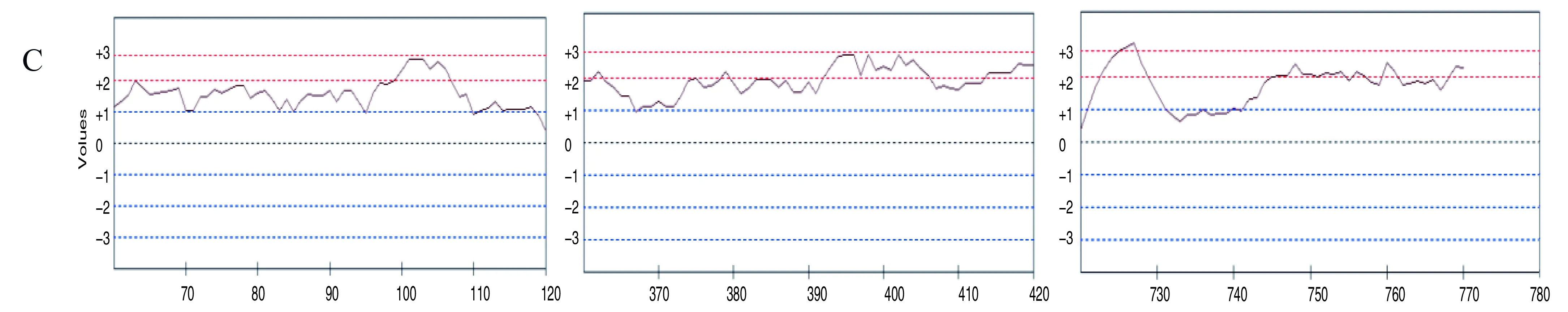
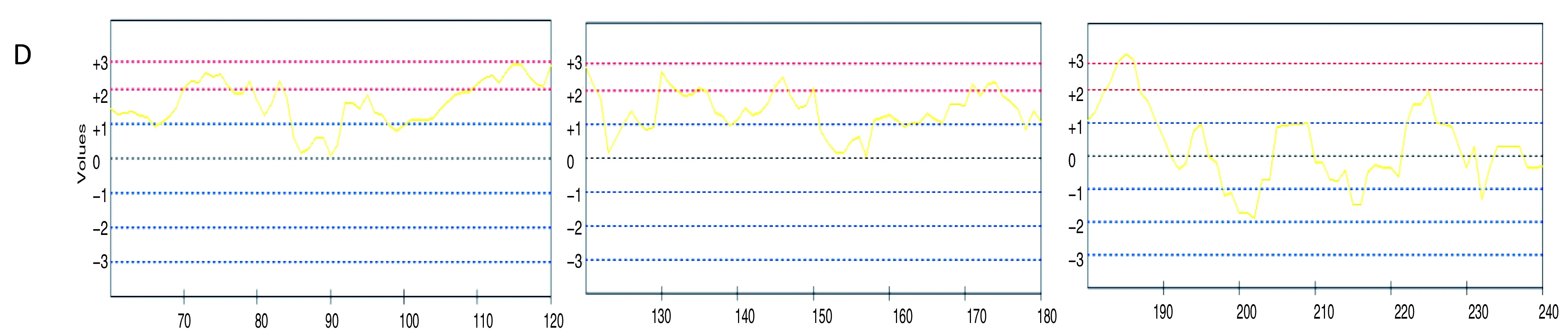
图2 生物信息学软件Bcepred预测APP蛋白的B细胞表位
A 亲水性 B 弹性 C 抗原暴露表面积 D 抗原的倾向性
表4 生物信息学软件ABCpred预测APP蛋白的B细胞抗原表位
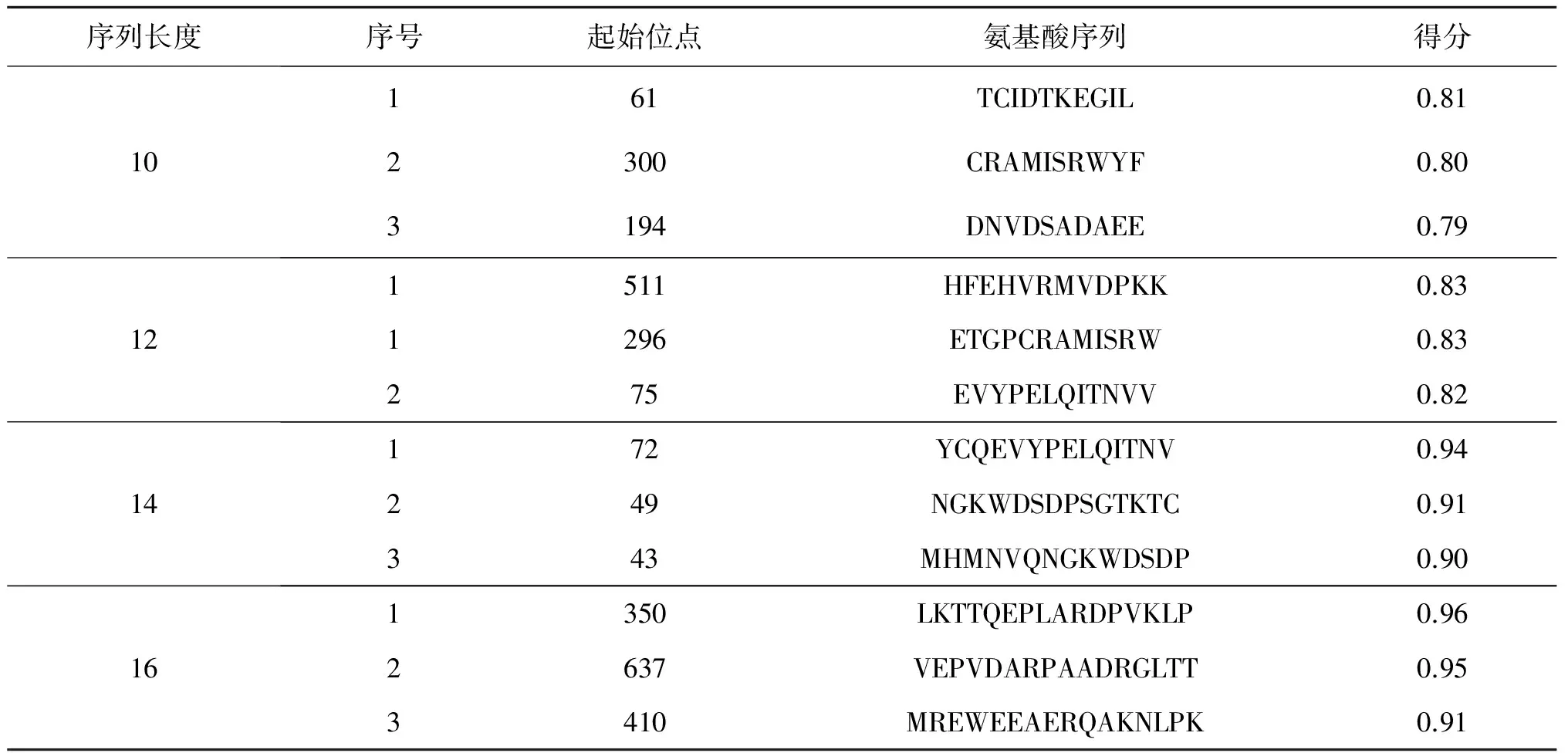
Table 4 Analysis of the B cell epitopes of APP protein using ABC pred online prediction software
http://www.imtech.res.in/raghava/abcpred/ABC_submission.html
表5 生物信息学软件ABCpred预测Aβ1-42蛋白的B细胞抗原表位
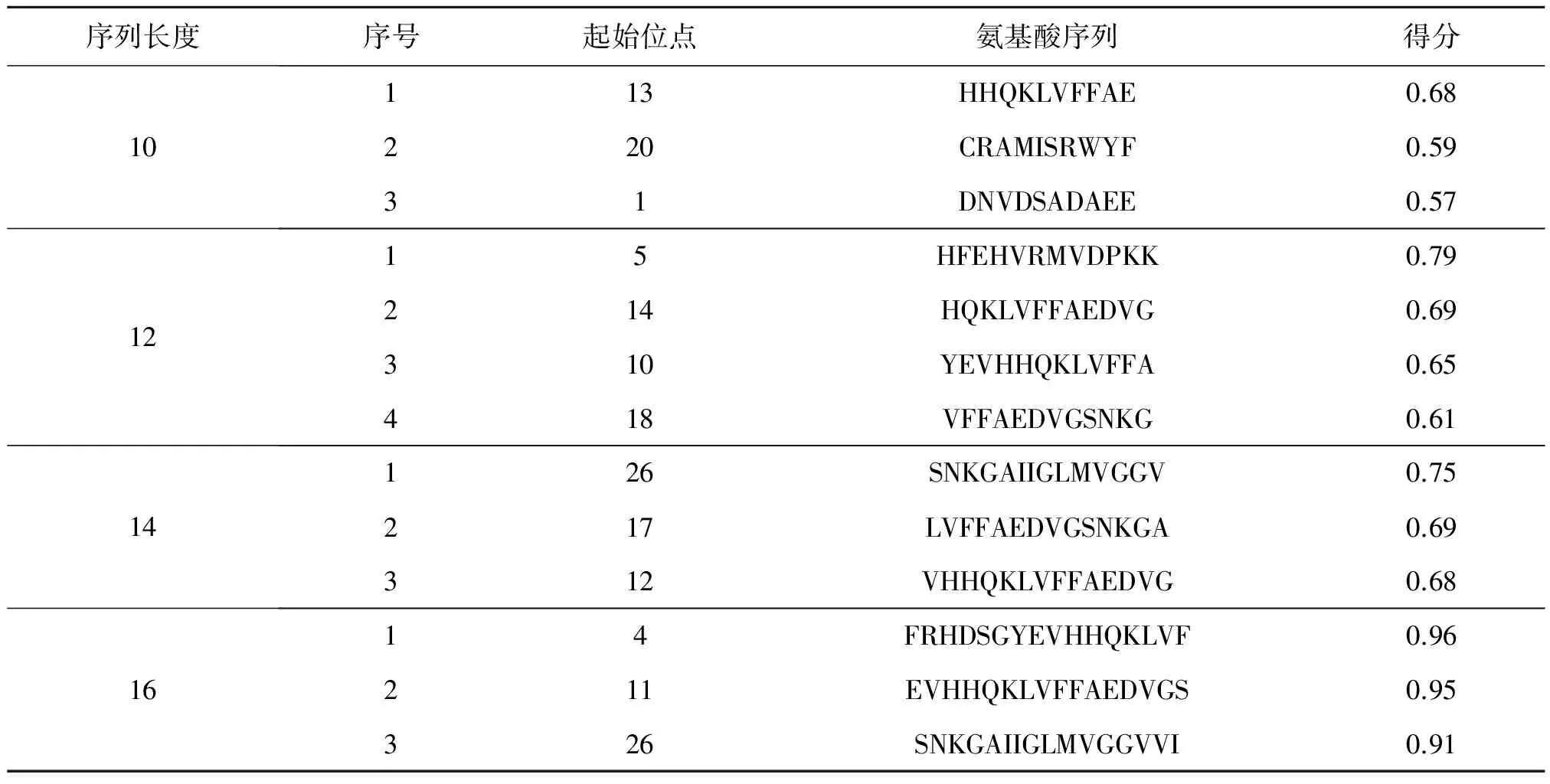
Table 5 Analysis of the B cell epitopes of Aβ1-42 protein using ABCpred online prediction software
http://www.imtech.res.in/raghava/abcpred/ABC_submission.html
3 讨论
本研究结果显示,APP及Aβ1~42蛋白存在潜在的优势抗原性表位,且Aβ1~42的抗原优势表位与APP优势表位不同。Aβ1~42的优势表位可避免引入APP蛋白的优势表位,从而激活机体免疫系统对Aβ1~42产生特异性高识别,避免对APP蛋白的正常功能产生不利影响。该结论为今后基于Aβ蛋白表位疫苗的设计奠定基础。
制备疫苗的最主要步骤是获得有关抗原表位的信息。近年来,随着生物信息学的快速发展,多参数、多方法分析预测提高了表位预测的准确性。申子刚等人[10]应用DNAStarProtein软件对FSHR胞外区的蛋白二级结构和表面特征进行分析,并在线进行B细胞表位预测,预测出FSHR胞外区蛋白可能的抗原表位。蛋白质的二级结构与其表位分布密切相关。研究证实,α螺旋结构在维持蛋白质二级结构的稳定中起重要作用,但α螺旋结构呈现于蛋白质的内部,因此不易与抗原分子结合[11]。与α螺旋结构不同,延伸链及无规卷曲等结构具有与配体特异性结合的结构区域,且多出现于蛋白质的表面,因而延伸链和无规卷曲结构所在的区域是抗原表位形成的[12]有利部位。
本研究使用不同方法、设置不同参数分析了APP及毒性片段Aβ1~42蛋白的二级结构。通过对前述蛋白的多种性质采用不同算法进行整合分析,从而提高所预测的抗原表位的准确性和特异性。
通过在线软件SOPMA分析,得出APP蛋白的二级结构中有利于形成表面抗原的α螺旋结构的比例为46.49%,Aβ1~42蛋白的延伸链结构比例较高,为52.38%,提示有较强的抗原性。研究显示,MHC-I 型的表位在预测的T细胞表位中的准确率高达90%[13]。在中国人群中HLA-A *02:01是最常见的HLA-I类分子,阳性率为55%。因此,在此项研究中,我们将前述两种蛋白抗原的HLA-A * 02:01 限制性表位使用 IEDB和SYFPEITHI在线软件进行预测分析。结果提示APP蛋白的T细胞表位位于37~49、16~28、1~13、11~24、30~43、42~50;Aβ1~42蛋白的T细胞表位位于3~11(675~683)、1~11(673~683)、22~31(696~705)、31~39(705~713)。B表位一般位于抗原分子的弹性区,具有可动性,这一特性有利于抗原表位和抗体结合部位表现出最佳的结构互补状态。如果互补性提高,结合的亲和力随之增强。我们通过在线生物性信息学软件和数据库,对APP及Aβ1~42毒性片段进行亲水性、柔韧性、抗原倾向性及抗原表面积暴露区域性进行B表位预测,分析得出APP蛋白潜在的B细胞表位位于49~62、 72~85、350~365、637~652;Aβ1~42蛋白的B细胞表位位于4~19(676~691)、26~39(700~713)、11~26(683~698)、26~41(698~713)。
本研究结果显示APP及Aβ1~42均存在抗原的优势表位,且Aβ1~42的抗原优势表位与APP优势表位不同。针对AD中毒性片段Aβ1~42设计出更具有特异性的表位疫苗,并寻找出Aβ1~42的优势表位避免引入APP蛋白的优势表位,从而发挥更好的预防、治疗作用。该结论为今后基于Aβ蛋白表位疫苗的设计奠定了理论基础。
[1]Santos AN,Ewers M,Minthon L,et.al.Amyloid-β oligomers in cerebrospinal fluid are associated with cognitive decline in patients with Alzheimer's disease[J].J Alzheimers Dis.2012;29(1):171-6.
[2]李大祥,鲜殊,杨卫,等.茶叶的神经保护作用研究进展[J].茶叶科学,2011,2:79-86.
[3]Gandy S.The role of cerebral amyloid beta accumulation in common forms of Alzheimer disease[J].J Clin Invest,2005,11 5(5):1121-9.
[4]王芳,周爱民.β淀粉样蛋白的研究进展[J].医学综述,2012,18(20):3377-9.
[5]庞明泉.多房棘球蚴抗原蛋白Emy162及TSP3的抗原表位的预测及鉴定[D].青海大学,2016.
[6]Geourjon C and Deléage G.SOPMA.significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J].Comput Appl Biosci,1995,11(6):681-4.
[7]庞明泉,汤峰,周虎,等.多房棘球蚴抗原蛋白TSP3抗原表位的生物信息学预测[J].青海医学院学报,2016,37(1):01-8.
[8]Vaughan K,Peters B,Larche M,et al.Strategies to query and display allergy-derived epitope data from the immune epitope database[J].Int Arch Allergy Immunol,2013,160(4):334-45.
[9]Sollner J,Grohmannr,Rapberger,et al.Analysis and prediction of protective continuous B-cell epitopes on pathogen proteins[J].Imm-unome Res,2008,4(1):1.
[10]Shen ZG,Yan P,He W,et al.Prediction of the secondary structure and the B cell epitope of the extracellular domain of FSHR[J].Journal of Chongqing Medical University,2010,35:1317-20.
[11]Li Y,Liu X,Zhu Y,et al.Bioin formatic prediction of epitopes in the Emy162 antigen of Echinococcus multilocularis[J].Exp Ther Med,2013,6(2):335-40.
[12]来鲁华,徐筱杰,唐有祺.蛋白质的结构预测与分子设计[J].大学化学,1993,8(5):49.
[13]Testa JS,Shetty V,Hafner J,et al.MHC class I presented T cell epitopes identified by immunoproteomics analysis are targets for a cross reactive influenza specific T cell response[J].PLoS One,2012,7(11):1-4.
Bioinformatic Prediction of Epitopes in The Amyloid β-Protein Antigen of Alzheimer′s Disease※
LIU jun-li1,2,3,4,TANG feng1,3,4,LIU chuan-chuan1,3,CUI sen2,3#,LI zhan-quan2,3&
(1.Research Center for Altitude Medicine;2.Qinghai University Affiliated Hospital; 3.The Key laboratory of high altitude medicine of Qinghai Province; 4.Qinghai-Utah Joint Research Lab for High Altitude Medicine,Xining 810000,China)
Objective The present study aims to predict the secondary structure and the T-and B-cell epitopes for the amyloid β-protein antigen,in order to reveal the dominant epitopes of the antigen.Methods The secondary structure of the protein was analyzed using SOPMA server.The T-cell and B-cell epitopes of APP and Aβ1~42 were predicted using IEDB,Syfpeithi,Bcepred and ABCpred online software.The characteristics of hydrophilicity,flexibility,antigenic propensity and exposed surface area were predicted.Result The α helix and extended strand accounted for 46.49% and 52.38% of the secondary structure of the APP and Aβ1~42 protein,respectively.This was indicative of the presence of potential dominant antigenic epitopes in these two proteins.The T-cell epitopes of APP and Aβ1~42 were analyzed by different bioinformatics methods.The high scoring T-cell epitopes of APP were located at positions 37~49,16~28,1~13,11~24,30~43 and 42~50;Aβ1~42 were 3~11(675~683,1~11(673~683),22~31(696~705),31~39(705~713);B-cell epitopes of APP and Aβ1~42 were located at positions 49~62,72~85,350~365,637~652 and 4~19(676~691),26~39(700~713),11~26(683~698),26~41(698~713),respectively.Conclusions APP and Aβ1~42 have the potential advantage of antigenic epitopes,and their antigenic epitopes were different.If the epitope vaccine designed for A 1~42 has no effect on APP,it will provide a theoretical basis for the study of epitope vaccines in the future.
Alzheimer′s disease Amyloid Precursor protein Secondary structure Epitopes
※:青海省科技厅自然科学基金项目(2017-ZJ-771);#:通信作者,教授,博士研究生导师,E-mail 13897284366@139.com;&:通信作者,教授,博士研究生导师 ,E-mail Li-zhanquan@163.com 刘军莉(1977~),女,汉族,甘肃籍,副主任医师,在读博士
R741
A
10.13452/j.cnki.jqmc.2017.02.004
2017-4-26
