水稻白条纹突变体st13的表型分析及基因定位
2017-07-31孙立亭林添资王云龙牛梅胡婷婷刘世家王益华万建民
孙立亭 林添资 王云龙 牛梅 胡婷婷 刘世家 王益华 万建民, 2,*
水稻白条纹突变体的表型分析及基因定位
孙立亭1, 3林添资1, 3王云龙1牛梅1胡婷婷1刘世家1王益华1万建民1, 2,*
(1南京农业大学作物遗传与种质创新国家重点实验室/农业部长江中下游粳稻生物学与遗传育种重点实验室/长江流域杂交水稻协同创新中心/江苏省现代作物生产中心,南京 210095;2中国农业科学院作物科学研究所,北京100081;3江苏丘陵地区镇江农业科学研究所,江苏句容 212400;*通讯联系人,E-mail: wanjm@njau.edu.cn, wanjianmin@caas.cn)
【目的】叶色突变相关基因鉴定和克隆有助于研究光合作用,补充并完善叶绿体发育机理和色素合成代谢途径,为开展水稻的高光效育种提供理论依据。【方法】从粳稻品种Dongjin的组培后代中分离出一个白条纹突变体,成熟期测定野生型和的主要农艺性状,苗期测定色素含量并观察叶绿体的超微结构;将和Dongjin进行正反交,观察F1植株表型,并对F2表型分离进行卡方检验,对进行遗传分析;利用×南京11(籼稻品种)的F2和F2:3群体,对突变基因定位;采用qPCR分析叶绿体发育和叶绿素合成相关基因在与野生型相对表达量。【结果】与野生型Dongjin相比,该突变体的株高、单株有效穗数、穗长、结实率和千粒重等主要农艺性状显著下降。苗期的色素含量降低,分蘖期无差异。突变体的叶绿体中既有含丰富的类囊体膜结构的正常叶绿体,也存在无类囊体结构的叶绿体。遗传分析和基因定位结果表明,的突变表型受1对隐性核基因控制,突变基因位于第3条染色体长臂InDel(Insertion-Deletion)标记I3-21和I3-22之间。进一步在这两个标记之间设计了6对InDel标记,最终将基因定位在94 kb区间内,此区间共有8个候选基因。【结论】这8个候选基因中,有5个假定的蛋白,其他三个都是有功能注释的蛋白,而这三个蛋白在水稻中均未见报道,因此,突变是由一个新的叶色基因突变引起的;同时中叶绿体发育、叶绿素合成和光合系统相关基因的表达也发生了显著改变,推测可能是调控叶绿体发育的关键基因。
水稻;白条纹突变体;叶绿体;精细定位
水稻叶色突变是指叶片颜色发生变异,可通过直接观察表型进行区分。通常叶色变异发生在苗期或分蘖期之前,易于鉴别、相对容易获得。由于大多数叶色突变表现出光合速率下降、作物减产,过去常被认为是无意义的突变。随着功能基因组学的发展,众多研究结果都显示叶色突变与植物光合作用[1]、激素生理[2]、光形态建成[3]等密切相关,因此,克隆和鉴定叶色突变相关基因有助于研究光合作用,补充和完善叶绿体发育机理和色素合成代谢途径,为开展水稻的高光效育种提供理论基础。
叶色突变体的发生机制主要有以下两类: 一是叶绿素的生物合成及分解代谢途径受阻。高等植物中叶绿素的生物合成途径已基本明确[4-6]。叶绿素的合成从谷氨酰-tRNA开始到最终合成叶绿素a和叶绿素b共有15步,分别由15种酶催化,其中任何一步发生突变,都会导致叶色变异。这15种酶在水稻中克隆报道的有CHLD(镁离子螯合酶D亚基)、CHLI(镁离子螯合酶I亚基)[7]、CHLH(镁离子螯合酶H亚基)[8]、YGL8(镁离子原卟啉Ⅸ单酯环化酶)[9]、DVR(脱酯基叶绿素酸酯a乙烯基还原酶)[10]、POR(NADPH-原叶绿素酸酯氧化还原酶)[11]、CHLG (叶绿素合成酶)[12]和CAO(叶绿素a氧化酶)[5,13]。叶绿素的降解过程对叶色的变异也有着重要的影响,NOL和NYC1都编码短链脱氢酶/还原酶,可形成叶绿素b还原酶复合体,行使叶绿素b还原酶的功能,在叶片衰老过程中调节叶绿素b和捕光复合体Ⅱ的降解,功能缺失后表现出滞绿表型[14]。二是叶绿体发育途径受阻。编码质体RNA聚合酶(PEP)的一个必需的非核心亚基,PEP是成熟叶绿体中一种主要的RNA聚合酶,通过与PEP复合体亚基互作调控叶绿体发育;编码Val-tRNA合成酶,调控叶绿体中核糖体的合成。和在早期叶绿体的发育中发挥重要的作用[15-16]。叶绿体的发育受核基因、叶绿体基因、光照和器官特异性的共同调节[17-18],例如编码光系统Ⅱ和Ⅰ蛋白复合物的和基因家族、、,影响叶绿体内光系统的形成[19-20];编码转运RNA 聚合酶的族的、和。此类基因缺失后,植株色素合成量降低[21];编码NADH脱氢酶的基因家族中的和,参与光合作用的等,这些基因调控叶绿体内的能量代谢[22-23]。目前已分离和鉴定的叶色突变体超过180个,其中成功克隆的达80多个,但水稻叶色变化过程和调控机制并未阐明,因此需要挖掘、鉴定新的叶色突变相关基因。本研究对白条纹突变体进行了主要农艺性状、色素含量和叶绿体超微结构等表型分析,同时利用×南京11(籼稻品种)的F2和F2:3群体进行定位,将基因定位在第3条染色体长臂87.4 cM和91.1 cM之间,两者相距3.7 cM。进一步设计InDel标记,最终将基因定位在94 kb区间内,共有8个候选基因,为基因的克隆和功能研究及育种应用奠定基础。
1 材料与方法
1.1 试验材料与田间试验
是从粳稻品种Dongjin的组培后代中分离出的一个白条纹突变体。野生型和突变体正季种植于南京农业大学牌楼实验基地,在苗期和分蘖期进行表型观察和色素含量测定。成熟期调查突变体和野生型的株高、单株有效穗数、剑叶长、穗长、结实率和千粒重等主要农艺性状。每个样品重复5次。
1.2 色素含量测定
取苗期(3叶期)完全展开叶,参照Wu等[12]测定叶片单位鲜质量的叶绿素a(Chl a)、叶绿素b(Chl b)和类胡萝卜素(Car)的含量。
1.3 叶绿体超微结构观察
取3叶期的野生型和突变体相同部位叶片,去掉中间叶脉,固定于2.5%的戊二醛溶液中(2.5%的戊二醛用0.1 mol/L磷酸缓冲液配制,pH 7.2),4 h后冲洗,再在1%锇酸中固定4 h,之后用乙醇(30%、50%、70%、80%、90%、100%)梯度脱水,最后用环氧树脂SPURR包埋、聚合、修块,经醋酸铀染色后在日立JEM-1230透射电子显微镜下观察、拍照。
1.4 水稻总RNA的提取及实时荧光定量PCR
选取3叶期野生型和突变体相同部位叶片提取总RNA。RNA的提取采用天根公司的植物RNA小量提取试剂盒,提取步骤详见说明书。反转录步骤参照TaKaRa公司的说明书,基因为内参。实时PCR 20 μL反应体系如下: 反转录产物2 μL,10 µmol/L的正、反向引物各0.4 μL,SYBRGreen混合物(TaKaRa)10 μL,最后补水至20 μL。反应在7500实时PCR仪(Applied Biosystems)上进行,反应程序参照操作手册,按2–ΔΔCt方法分析基因的相对表达量[24],每个样本重复3次。

A-3叶期幼苗;B-A图中的第2片真叶;C-分蘖期植株。
Fig. 1. Phenotype of the wild-type and mutantat the seedling and tillering stages.
1.5 遗传分析及定位群体构建
用突变体和野生型Dongjin进行正反交,同年冬季在海南陵水加代获得F2,第二年正季在南京农业大学牌楼实验基地种植,苗期统计F2群体中正常表型和白条纹表型单株数,计算分离比,并进行χ2测验。
用与南京11杂交,获得Fl种子,自交获得F2种子,将F2种子种植于南京农业大学牌楼基地,苗期鉴定植株白条纹分离比例,并选取白条纹表型的单株进行基因初定位。同时部分F2种子在海南陵水基地繁种。正季播种 F2:3群体种子,苗期分株系选取白条纹植株,进行基因的精细定位。
1.6 基因定位
基因组DNA提取采用SDS法[25],用实验室均匀分布于水稻12条染色体上的InDel标记(Insertion-Deletion)(由南京金斯瑞生物科技有限公司合成)对和南京11进行多态性分析,采用筛得的有多态的引物对/南京11的F2中235份白条纹单株和F2:3中2000个白条纹单株进行连锁分析和基因定位。PCR扩增体系参照Shi等[26],扩增产物用8%聚丙烯酰胺凝胶电泳进行检测,银染显色。将具有的带型记作“1”,具有南京11的带型记作“2”,杂合带型记作“3”,计算遗传距离,构建分子标记连锁图谱。
2 结果与分析
2.1 突变体的表型分析
是从粳稻品种Dongjin的组培后代中分离出的一个白条纹突变体(图1)。与野生型相比,突变体在3叶期表现出白条纹症状(图1-A、B),分蘖期转绿,但株高比野生型降低(图1-C)。主要农艺性状调查结果表明,突变体的株高、穗长、有效穗、结实率和千粒重与Dongjin相比均显著下降(表1),这表明该基因对植株生长发育十分重要。
2.2 野生型Dongjin和突变体的色素含量测定
为了确定突变体的白条纹表型是否由色素含量变化引起,我们分别测定了Dongjin和在苗期和分蘖期的色素含量。结果表明,苗期突变体的叶绿素a、叶绿素b和类胡萝卜素的含量均比野生型Dongjin显著降低(图2-A);当幼苗生长至分蘖期时,的叶片转绿(图1-C),此时其叶绿素a、叶绿素b和类胡萝卜素含量与野生型无显著差异(图2-B)。这表明的突变症状是由色素含量变化引起的。

表1 野生型Dongjin与突变体st13的主要农艺性状比较
**表示经测验后,野生型与突变体在0.01水平下差异显著。
**Difference between the wild type and themutant was significant at 0.01 level by Student’s-test.

平均值±标准差, n=3。**P<0.01,*P<0.05 (t测验)。
Fig. 2. Pigment contents of Dongjin andat the seedling and tillering stages.

A, D-野生型的叶绿体超微结构; B, C, E, F-突变体st13中同时含有具正常结构的叶绿体(B, E)和具不正常结构的叶绿体(C, F)。 A, B, C图标尺为10 μm,D, E图标尺为1 μm,F图标尺为1 μm。Cp-叶绿体; Thy-类囊体; OB-嗜锇体。
Fig. 3. Transmission electron microscopy (TEM) analysis of Dongjin andleaves at the seedling stage.
2.3 叶绿体的超微结构观察
为进一步探究叶片白条纹形成的原因,我们利用透射电镜观察了Dongjin和苗期叶绿体的超微结构,突变体中,少数细胞中的叶绿体与野生型无显著差异(图3-A、B),但大部分叶肉细胞观察不到叶绿体(图3-C),表现出空泡状结构,而突变体的叶绿体结构也观察到两种类型(图3-E、F),一种含有丰富的类囊体片层结构,与野生型相比无明显差异(图3-D、E),另一种叶绿体细胞中无类囊体片层结构(图3-F); 这一结果与白条纹突变性状(白绿相间)一致。
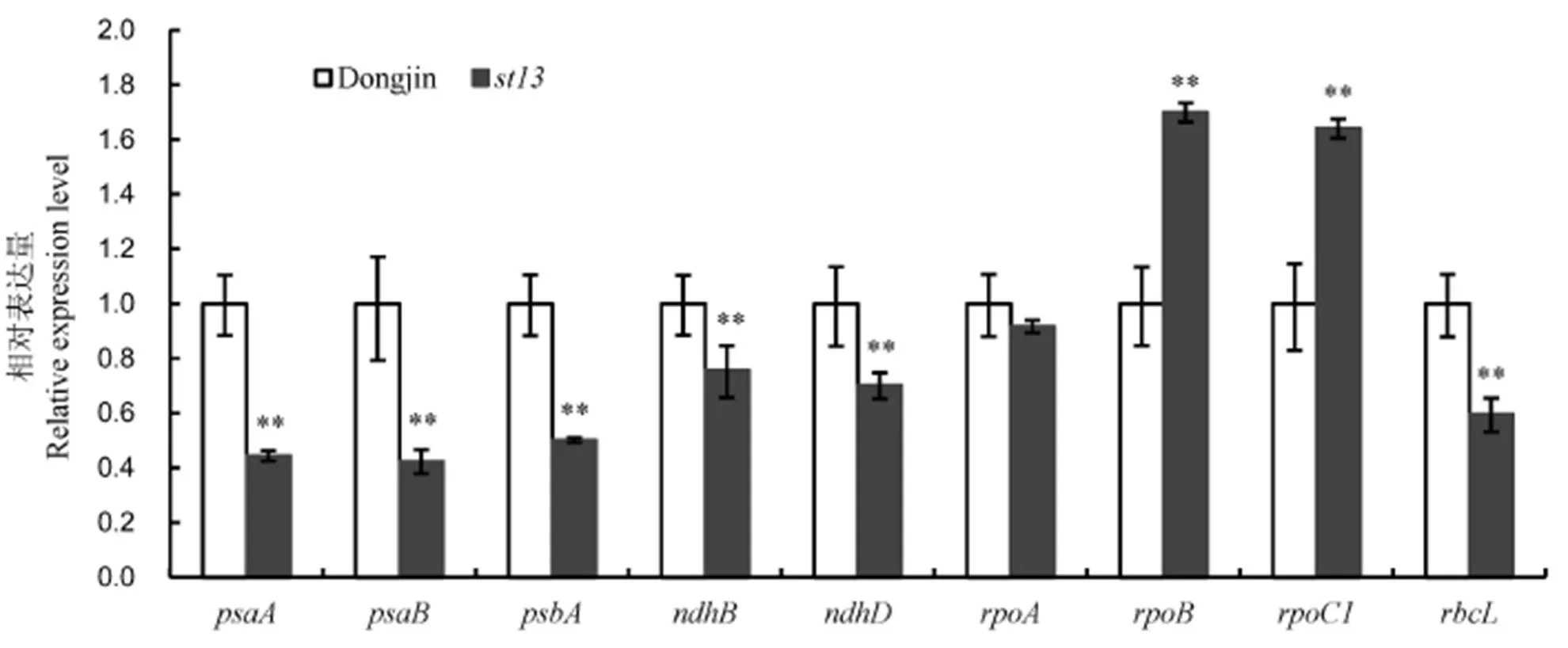
平均值±标准差, n=3。**P<0.01(t测验)。
Fig. 4. Quantitative RT-PCR analyses of genes associated with chloroplast development and photosynthetic system in wild-type Dongjin andmutant at the seedling stage.
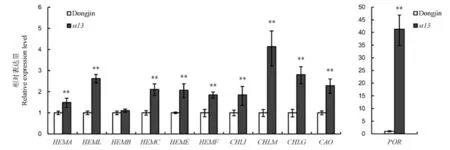
平均值±标准差, n=3。**P<0.01(t测验)。
Fig. 5. Quantitative RT-PCR analyses of genes associated with chlorophyll biogenesis in wild-type Dongjin andmutant at the seedling stage.
2.4 突变体中叶绿体发育及光合系统相关基因的表达分析
叶绿体是植物进行光合作用的场所,为明确的白条纹症状是否影响叶绿体发育及光合系统相关基因的表达,我们进行了qRT-PCR分析(图4)。结果发现突变体中,编码转运RNA聚合酶的族基因和的表达显著上调;编码1,5-二磷酸核酮糖羧化酶/加氧酶的基因表达下调;同时编码NADH脱氢酶的基因家族和均显著下调,而编码光系统Ⅱ和Ⅰ蛋白复合物的、和表达量下调。这表明白条纹这一突变性状影响了叶绿体发育,同时抑制了光合系统相关基因的表达,这与透射电镜观察结果一致。

表2 野生型Dongjin与st13突变体正反交组合的F2分离比
2.5 突变体中叶绿素合成相关基因的表达分析
为探究叶绿体发育缺陷是否影响叶绿素合成,我们分析了叶绿素合成相关基因的表达(图5)。与野生型相比,叶绿素合成途径中的(编码谷氨酸-tRNA还原酶)、(编码谷氨酸-1-半缩醛氨基转移酶)、(编码原脱氨酶)、(编码尿卟啉原Ⅲ脱羧酶)、(编码粪卟啉Ⅲ氧化酶)、(编码螯合酶I亚基)、(编码镁离子原卟啉Ⅸ甲基转移酶)、(NADPH-原叶绿素酸酯氧化还原酶)、(编码叶绿素合成酶)和(编码叶绿素a氧化酶)表达量均显著上调。这表明突变体的叶绿素合成受到影响。
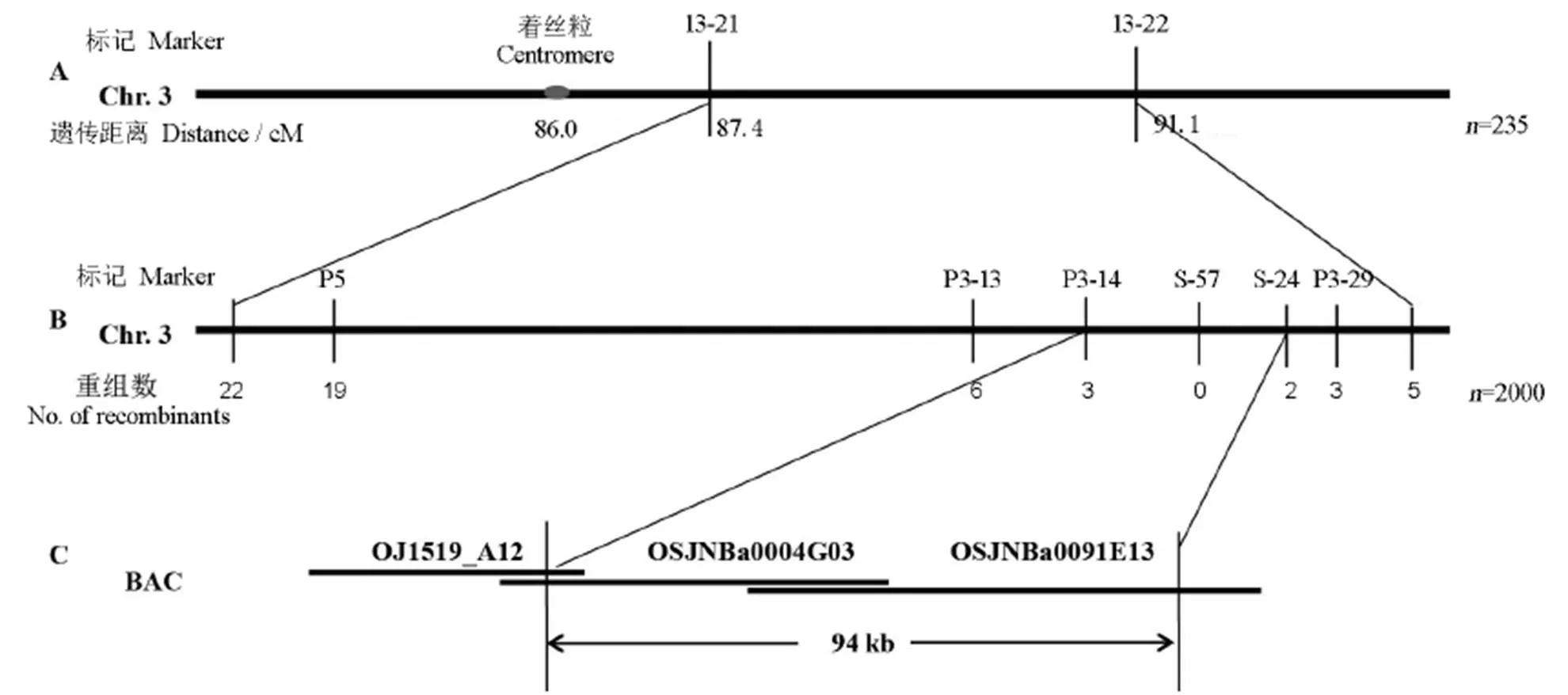
A-基因位点初定位到第3条染色体长臂的InDel标记I3-21和I3-22之间;B-基因位点精细定位到标记P3-14和S-24之间;C-定位区间位于BAC克隆OJ1519_A12、OSJNBa0004G03和OSJNBa0091E13之间94 kb的物理区间。
Fig. 6. Fine mapping of.
2.6 遗传分析
为了确定突变性状的遗传特性,我们配制了野生型和突变体的正反交杂交组合,无论突变体用作母本还是父本,相应的F1植株叶片颜色均表现为正常绿色,其自交后代F2群体表型出现明显分离,且正常绿色与突变体植株数比值符合3∶1(表2)。这说明该性状是由一对隐性核基因控制的。

表3 目标区域内有多态的InDel标记
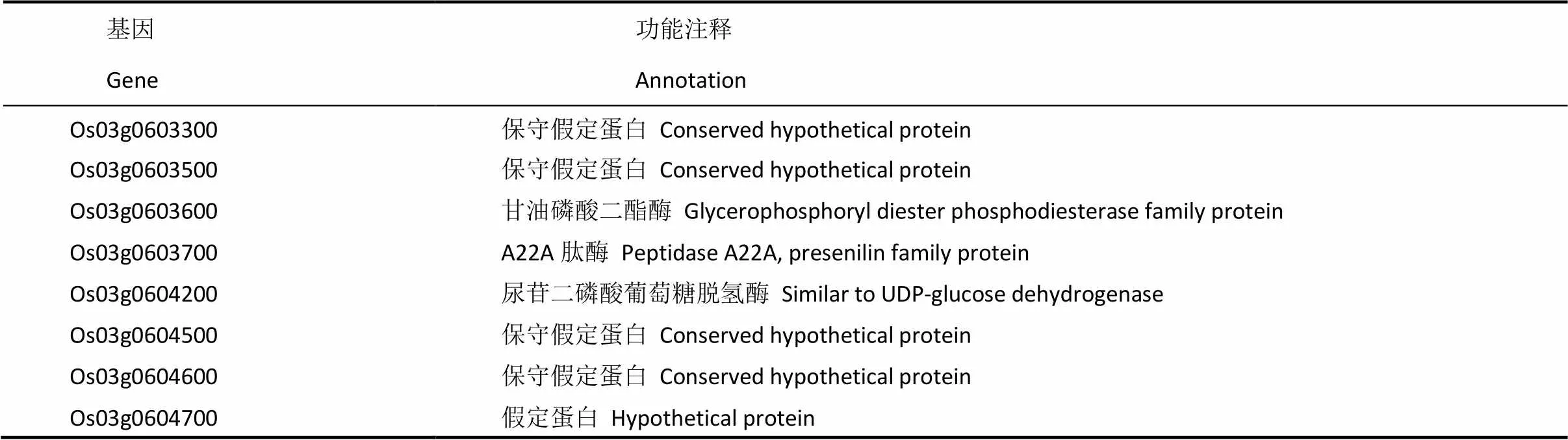
表4 94 kb区间内的预测基因及其可能的功能
2.7 突变性状的基因定位及候选基因
将突变体与籼稻品种南京11进行杂交,挑取其F2群体分离出的白条纹单株235株及F2:3群体分离的白条纹单株2000株作为定位群体,利用本实验室均匀分布在12条染色体上的InDel标记将基因初定位在第3染色体长臂的I3-21标记(87.4 cM)与I3-22标记(91.1 cM)之间(图6-A)。为进行精细定位,利用Primer premier 5.0软件,开发出两亲本之间有多态的引物6对(表3),最终将基因定位在标记P3-14与标记S-24之间,位于BAC OSJNBa0004G03和OSJNBa0091E13上,两者相距94 kb(图6-B、C)。利用水稻表达谱数据库(http:// ricexpro.dna.affrc.go.jp/index.html)预测此区间内含有8个开放阅读框(ORF),其功能预测见表4。
3 讨论
叶绿体的发育是在细胞核和细胞质基因的协同调控下完成的,这个过程分为3个步骤: 第一步是叶原基分化为分生组织,激活质体复制和质体DNA的合成;第二步是叶绿体遗传系统建立,分生细胞增大,叶绿体翻译和转录活性大大增加;第三步是叶绿体分化形成,开始转录光合作用元件[27]。光反应相关蛋白对叶绿体的结构和功能的完整起着重要的作用,和编码PSⅠ合成相关亚基,编码PSⅡ核心复合物[28-29],突变体中,这三个基因的表达下调。光系统复合物形成过程中,类囊体也在形成,突变体的叶绿体细胞中存在大量无类囊体的空泡状结构,类囊体膜无法正常形成,叶绿体的分化阶段受到影响,叶绿体发育不良。编码转运RNA聚合酶的族基因功能缺失后,植株色素含量减少,质体内膜系统发育不完整[21],而突变体中,表达量与野生型比无显著差异,和表达量上升,可能tRNA编码产物大量积累,导致tRNA聚合酶消耗量减少,其编码基因表达上调。突变体中,,和均显著下调,从而导致叶绿体内能量代谢失衡。突变体的叶绿体发育受损,导致色素含量降低。突变体中编码Mg-原卟啉Ⅸ螯合酶的Ⅰ亚基的基因发生突变,导致出现浅黄绿叶的表型[7],在突变体中,此基因表达上调,同时突变体中编码叶绿素a加氧酶的基因[13]在突变体中表达上调,其他叶绿素生物合成途径中的基因、、、、、、和表达上调,推测可能是调控叶绿体发育的关键基因。
水稻中已克隆14个白条纹或白化基因,有的表现出白化致死,如[30]、[31]、[32]、[16]、[33],有的属于温度依赖型,如[6]、、[34]、[35]、[15]等,有的表现出白化转绿,如[36]等。本研究中,突变体在苗期表现为叶片白条纹,分蘖期转绿,与以往报道的水稻白化转绿突变体相比具有相似的表型,但该突变体主要农艺性状如株高、单株有效穗数、穗长、千粒重和结实率都显著降低,说明基因具有一因多效。通过对×南京11(籼稻品种)的F2和F2:3群体进行定位,最终将基因定位在第3条染色体长臂94 kb区间内,此区间内预测到8个候选基因,突变体可能是由一个新的叶色基因突变引起的。在该区间内,有5个假定的蛋白,其他三个都是有功能注释的蛋白。Os03g0604200编码尿苷二磷酸葡萄糖脱氢酶,在拟南芥中,此酶催化细胞壁中UDP-葡萄糖不可逆的氧化为UDP-葡萄醛酸[37],在水稻中此基因未见报道。Os03g0603700基因编码A22A肽酶,在植物中报道较少。Os03g0603600编码甘油磷酸二酯酶,在哺乳动物中涉及信号转导、神经分化及发育和细胞骨架调控等[38],植物中未见报道。因此,需要进一步研究开展测序、表达分析及转基因互补等研究以确定的候选基因,为阐明基因的功能奠定基础。
[1] Fambrini M, Castagna A, Dalla V F, Degl’Innocenti E, Ranieri A, Vernieri P, Pardossi A, Guidi L, Rascio N, Pugliesi C. Characterization of a pigment-deficient mutant of sunflower (L.) with abnormal chloroplast biogenesis, reduced PSII activity and low endogenous level of abscisic acid., 2004, 6: 645-650.
[2] Agrawal G K, Yamazaki M, Kobayashi M, Hirochika R, Miyao A, Hirochika H. Screening of the rice viviparous mutants generated by endogenous retrotransposoninsertion: Tagging of a zeaxanthin epoxidase gene and a novel ostatc gene., 2001, 125(3): 1248-1257.
[3] Parks B M, Quail P H. Phytochrome-deficientandlong hypocotyl mutants ofare defective in phytochrome chromophore biosynthesis., 1991, 3(11): 1177-1186.
[4] Kusumi K, Sakata C, Nakamura T,Kawasaki S, Yoshimura A, Iba K. A plastid protein NUS1 is essential for build-up of the genetic system for early chloroplast development under cold stress conditions., 2011, 68(6): 1039-1050.
[5] Lee S, Kim J H, Yoo E S,Lee C H, Hirochika H, An G. Differential regulation of chlorophyll a oxygenase genes in rice., 2005 57: 805-818.
[6] Sugimoto H, Kusumi K, Noguchi K, Yano M, Yoshimura A, Iba K. The rice nuclear gene,, is essential for chloroplast development and encodes a novel type of guanylate kinase targeted to plastids and mitochondria., 2007, 52: 512-527.
[7] Zhang H, Li J, Yoo J, Yoo S, Cho S, Koh H, Seo H S, Paek N. Riceandencode ChlD and ChlI subunits of Mg-chelatase, a key enzyme for chlorophyll synthesis and chloroplast development., 2006, 62: 325-337.
[8] Jung K H, Hur J, Ryu C H, Choi Y, Chung Y Y, Miyao A, Hirochika H, An G. Characterization of a rice chlorophyll- deficient mutant using the T-DNA gene-trap system., 2003, 44: 463-472.
[9] Kong W, Yu X, Chen H, Liu L, Xiao Y, Wang Y, Wang C, Lin Y, Yu Y, Wang C, Jiang L, Zhai H, Zhao Z, Wan J. The catalytic subunit of magnesium-protoporphyrin IX monomethyl ester cyclase forms a chloroplast complex to regulate chlorophyll biosynthesis in rice., 2016, 92(1-2): 177-191.
[10] Wang P, Gao J, Wan C, Zhang F, Xu Z, Huang X, Sun X, Deng X. Divinyl chlorophyll (ide) a can be converted to monovinyl chlorophyll (ide) a by a divinyl reductase in rice.2010, 153: 994-1003.
[11] Sakuraba Y, Rahman M L, Cho S H, Kim Y S, Koh H J, Yoo S C, Paek N C. The rice faded green leaf locus encodes protochlorophyllide oxidoreductase B and is essential for chlorophyll synthesis under high light conditions., 2013, 74: 122-133.
[12] Wu Z M, Zhang X, He B, Diao L, Sheng S, Wang J, Guo X, Su N, Wang L, Jiang L, Wang C, Zhai H, Wan J. A chlorophyll-deficient rice mutant with impaired chlorophyllide esterification in chlorophyll biosynthesis., 2007, 145: 29-40.
[13] Yang Y, Xu J, Huang L, Leng Y, Dai L, Rao Y, Chen L, Wang Y, Tu Z, Hu J, Ren D, Zhang G, Zhu L, Guo L, Qian Q, Zeng D., encoding chlorophyllide a oxygenase 1, impacts leaf senescence and indirectly affects grain yield and quality in rice., 2016, 67(5): 1297-1310.
[14] Sato Y, Morita R, Katsuma S, Nishimura M, Tanaka A, Kusaba M. Two short-chain dehydrogenase/reductases, NON-YELLOW COLORING 1 and NYC1-LIKE, are required for chlorophyll b and light-harvesting complex II degradation during senescence in rice., 2009, 57: 120-131.
[15] Wang L, Wang C, Wang Y, Niu M, Ren Y, Zhou K, Zhang H, Lin Q, Wu F, Cheng Z, Wang J, Zhang X, Guo X, Jiang L, Lei C, Wang J, Zhu S, Zhao Z, Wan J. WSL3, a component of the plastid-encoded plastid RNA polymerase, is essential for early chloroplast development in rice., 2016, 92(4-5): 581-595.
[16] Wang Y, Wang C , Zheng M, Lyu J, Xu Y, Li X, Niu M, Long W, Wang D, Wang H Y, William T, Wang Y, Wan J. WHITE PANICLE1, a Val-tRNA synthetase regulating chloroplast ribosome biogenesis in rice, is essential for early chloroplast development., 2016, 170(4): 2110-2123.
[17] Lopez-Juez E. Plastid biogenesis between light and shadows., 2007, 58: 11-26.
[18] Sakamoto W, Miyagishima S Y, Jarvis P. Chloroplast biogenesis: Control of plastid development, protein import, division and inheritance., 2008, 6: e110.
[19] Webber A N, Malkin R. Photosystem I reaction-centre proteins contain leucine zipper motifs. A proposed role in dimer formation., 1990, 264: 1-4.
[20] Rutherford A W, Faller P. Photosystem II: evolutionary perspectives.. 2003, 358:245-253.
[21] Santis-Maciossek G D, Kofer W, Bock A, Schoch S, Maier R M, Wanner G, Rüdiger W, Koop Hans-Ulrich, Herrmann R G. Targeted disruption of the plastid RNA polymerase genes,and: molecular biology biochemistry and ultrastructure., 1999,18: 477-489.
[22] Peng L W, Yamamoto H, Shikanai T. Structure and biogenesis of the chloroplast NAD(P)H dehydrogenase complex., 2011, 1807: 945-953.
[23] Andersson I, Backlund A. Structure and function of Rubisco., 2008, 46: 275-291.
[24] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2 [Delta Delta C(T)] method., 2001, 25: 402-408.
[25] McCouch S R, Kochert G, Yu Z H, Wang Z Y, Khush G S, Coffman W R, Tanksley S D. Molecular mapping of rice chromosome., 1998, 76: 815-829.
[26] Shi Y F, Chen J, Liu W Q, Huang Q N, Shen B, Leung H, Wu J L. Genetic analysis and gene mapping of a new rolled-leaf mutant in rice (L.).:, 2009, 52: 885-890.
[27] Kusumi K, Chono Y, Shimada H, Shimada H, Gotoh E, Tsuyama M, Iba K. Chloroplast biogenesis during the early stage of leaf development in rice., 2010, 27: 85-90.
[28] Allen J F, Wilson B M, Puthiyaveetil S, Nield J. A structural phylogenetic map for chloroplast photosynthesis., 2011, 16: 645-655.
[29] Pakrasi H B. Genetic analysis of the form, function of photosystem I and photosystem II., 1995, 29: 755-776.
[30] 王兴春, 王敏, 季芝娟, 陈钊, 刘文真, 韩渊怀, 杨长登. 水稻糖苷水解酶基因在叶绿体发育中的功能. 作物学报, 2014, 40(12): 2090-2097.
Wang X C, Wang M, Ji Z J, Chen Z, Liu W Z, Han Y H, Yang C D. Functional characterization of the glycoside hydrolase encoding geneduring chloroplast development in., 2014, 40(12): 2090-2097.(in Chinese with English abstract)
[31] Gothandam K M, Kim E S, Cho H, Chung Y Y. OsPPR1, a pentatricopeptide repeat protein of rice is essential for the chloroplast biogenesis., 2005, 58(3): 421-433.
[32] Lin D, Jiang Q, Zheng K, Chen S, Zhou H, Gong X, Xu J, Teng S, Dong Y. Mutation of the ricegene encoding plastid ribosomal protein L21 causes chloroplast developmental defects and seedling death., 2015, 17(3): 599-607.
[33] Zhang Z, Tan J, Shi Z, Xie Q, Xing Y, Liu C, Chen Q, Zhu H, Wang J, Zhang J, Zhang G. Albino Leaf1 that encodes the sole octotricopeptide repeat protein is responsible for chloroplast development., 2016, 171(2): 1182-1191.
[34] Yoo S C, Cho S H, Sugimoto H, Li J, Kusumi K, Koh H J, Iba K, Paek N C. Rice virescent3 and stripe1 encoding the large and small subunits of ribonucleotide reductase are required for chloroplast biogenesis during early leaf development., 2009, 150(1): 388-401.
[35] Wang Y, Zhang J, Shi X, Peng Y, Li P, Lin D, Dong Y, Teng S. Temperature-sensitive albino gene, encoding a monooxygenase, affects chloroplast development at low temperatures., 2016, 67(17): 5187-5202.
[36] Su N, Hu M L, Wu D X, Wu F Q, Fei G L, Lan Y, Chen X L, Shu X L, Zhang X, Guo X P, Cheng Z J, Lei C L, Qi C K, Jiang L, Wang H, Wan J M. Disruption of a rice pentatricopeptide repeat protein causes a seedling- specific albino phenotype and its utilization to enhance seed purity in hybrid rice production., 2012, 159(1): 227-238.
[37] Klinghammer M, Tenhaken R. Genome-wide analysis of the UDP-glucose dehydrogenase gene family in, a key enzyme for matrix polysaccharides in cell walls., 2007, 58(13): 3609-3621.
[38] 孙兰茜, 常平安, 曾鑫, 韩丽萍, 冉凤, 王超颖, 王玲, 黄飞飞. 甘油磷酸二酯酶家族蛋白的分子进化. 基因组学与应用生物学, 2015, 34(27): 172-178.
Sun L X, Chang P A, Zeng X, Han L P, Ran F, Wang C Y, Wang L, Huang F F. Molecular evolution of the glycerophospho- diesterase family proteins., 2015, 34(27): 172-178. (in Chinese with English abstract)
Phenotypic Analysis and Gene Mapping of a White Stripe Mutantin Rice
SUN Liting1,3, LIN Tianzi1,3, WANG Yunlong1, NIU Mei1, HU Tingting1, LIU Shijia1, WANG Yihua1, WAN Jianmin1,2,*
(1State Key Laboratory of Crop Genetics and Germplasm Enhancement, Nanjing Agricultural University/Key Laboratory of Biology, Genetics and Breeding of japonica Rice in Mid-lower Yangtze River, Ministry of Agriculture/The Yangtze River Valley Hybrid Rice Collaboration Innovation Center/Jiangsu Collaboration Innovation Center for Modern Crop Production, Nanjing 210095, China;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3Zhenjiang Agricultural Research Institute, Jurong 212400, China;*Corresponding author, E-mail:wanjm@njau.edu.cn, wanjianmin@caas.cn)
【Objective】Rice leaf color mutation identification and cloning of related genes is helpful to research photosynthesis and further understand the mechanism of chloroplast development and pigment synthesis pathway, providing theoretical basis for the breeding of rice with high photosynthetic efficiency.【Method】A somaclonal mutant (designated as,) was isolated from tissue-cultured progenies of arice variety Dongjin. The main agronomic traits of wild type andwere determined at maturity, and the pigment contents and ultrastructure of chloroplast were observed at the seedling stage. Dongjin andwere reciprocally crossed to observe the phenotype of F1and genetic analysis was carried out. An F2population and F2:3population derived from the cross× Nanjing11 (anrice variety) were used for gene mapping of. Quantitative RT-PCR was carried out to analyze the relative expression of genes associated with chloroplast development and chlorophyll biogenesis in the wild-type Dongjin andmutant. 【Result】Compared to the wild type, the plant height, number of effective panicles, panicle length, seed-setting rate, and 1000-grain weight were all significantly reduced in the mutant. In agreement with leaf color, the pigment content ofmutant was reduced at the seedling stage but rose to almost the same level as the wild type at the tillering stage. With a transmission electron microscopy, defective chloroplasts with no thylakoid were observed in themutant. Genetic analysis indicated the mutant phenotype was controlled by a single recessive nuclear gene.was restricted to an interval between the InDel (insertion/deletion) markers I3-21 and I3-22 on chromosome 3. Six InDel markers were further developed and thegene was narrowed down to a 94-kb region, containing eight putative open reading frames. 【Conclusion】These eight candidate genes encode three putative proteins and five functional proteins respectively, while none of them have been reported in rice. Thus,was an unreported gene in rice. Relative to wild type, the expression levels of genes associated with chloroplast development, chlorophyll biosynthesis, and photosynthetic system were significantly altered in the mutant. So we hypothesized thatmight be the key gene for regulating chloroplast development.
rice; white stripe mutant; chloroplast; fine mapping
10.16819/j.1001-7216.2017.6165
Q343.5; S511.01
A
1001-7216(2017)04-0355-09
2016-12-20;
国家重点研发项目七大农作物育种专项(2016YFD0100101-08); 国家863计划资助项目(2014AA10A603-15); 江苏省科技支撑计划资助项目(BE2014394, BE2015363); 江苏省农业科技自主创新资金资助课题[CX(16)1029]。
修改稿收到日期:2017-02-07。
