转基因棉花对土壤细菌群落的影响
2017-07-24范巧兰李永山王慧席凯鹏席吉龙史俊东张建诚
范巧兰,李永山,王慧,席凯鹏,席吉龙,史俊东,张建诚
(1.山西省农业科学院棉花研究所,山西运城044000;2.运城农业科学院,山西运城044000)
转基因棉花对土壤细菌群落的影响
范巧兰1,2,李永山1,2,王慧1,2,席凯鹏1,2,席吉龙1,2,史俊东1,2,张建诚1,2
(1.山西省农业科学院棉花研究所,山西运城044000;2.运城农业科学院,山西运城044000)
为了评价转基因棉花对土壤细菌群落结构的影响,试验采用转基因棉花品种晋棉26号及其非转基因亲本晋棉7号以及传统棉花品种中棉所12号进行了长期定位试验,利用高通量基因测序法研究了转基因棉花对土壤细菌的群落结构的影响。结果表明,3个棉花土壤细菌共检测到1 585个OTUs、除1个未分类门和4个待定候选门外,其余分别属于已知的21个门;主要优势种群有变形菌门、酸杆菌门、放线菌门、浮霉菌门、绿弯菌门、拟杆菌门;每个品种有独特的种群,转基因棉花晋棉26号独有的种群为栖热菌门,晋棉7号独有种群为WCHB1-60;转基因棉花土壤细菌的多样性降低,但没有改变土壤细菌种群结构。
转基因棉花;土壤;细菌;群落结构;高通量测序
2015年的生物技术作物商业化20周年,全球种植转基因作物面积是1996年的100多倍,达1.797亿hm2。2015年全球种植棉花3 200万hm2,75%的种植面积是转基因棉花,其中,我国种植370万hm2转基因棉花,占全国棉花总面积的96%[1]。随着转基因作物种植面积的不断扩大,转基因植物的生态安全风险评价日益受到人们的关注,转基因对土壤生态安全评价成为研究热点[2],国内外学者研究了转Bt基因抗虫棉对土壤微生物的影响[3-12]。由于根系分泌物和残体中的Bt毒素在土壤中的积累,连续种植棉花会导致土壤微生物种群结构变化。由于大田和实验室采用的试验方法(传统微生物培养法、PFLA和DGGE等)、试材等条件的不同,导致试验结果不一致,多数结果认为,连续种植转基因作物对土壤微生物没有影响或影响很小[13-16],但也有相反的结果[17-22]。近年来,高通量测序技术是一种新的技术,在微生物分子生态学等方面得到了广泛应用[23]。
本研究应用Illumina平台的Miseq高通量测序土壤细菌16S rDNA,以转Bt基因棉花晋棉26号及其非转基因棉花亲本晋棉7号以及传统品种中棉所12号为试材,进行田间定位试验,旨在探索连续多年种植转基因棉花对棉花土壤细菌组成、结构及多样性的影响,为转基因棉花安全评价提供依据。
1 材料和方法
1.1 试验地概况
试验设在山西省农业科学院棉花研究所牛家凹试验农场。试验地为黄壤土,肥力中等偏上,有机质10.91 g/kg,全氮2.13 g/kg,速效氮186.04 mg/kg,速效磷6.52 mg/kg,速效钾145.14 mg/kg,土壤pH值为7.2。
1.2 试验材料
供试棉花品种为转基因棉花晋棉26号(JM26)及其非转基因棉花亲本品种晋棉7号(JM7)和非转基因品种中棉所12号(CRI12)。
1.3 试验设计
试验采用随机区组设计,3次重复,小区面积30 m2。从2008年开始进行定位试验,于2014年棉花收获期采集土样,采集的土样放在无菌封口塑料袋中,拣出根系,立即将其放置在-80℃冰箱中保存待测。
1.4 土壤微生物总DNA的提取及细菌的高通量测序及分析
称取土壤样品0.5 g,用E.Z.N.A Soil DNA(OMEGA,USA)试剂盒进行土壤总DNA提取后,用核酸定量仪(NanoDrop ND-1000)和1%琼脂糖凝胶电泳检测土壤总DNA的浓度和纯度。
高通量测序主要步骤:按指定测序区域,合成带有bar code的特异引物。PCR采用TransGenAP221-02:TransStart Fastpfu DNA Polymerase;PCR仪:ABI GeneAmpR9700型。
土壤细菌的PCR的扩增引物:采用16S rRNA的扩增引物515F(5′-GTGCCAGCMGCCGCGG-3′)和907R(5′-CCGTCAATTCMTTTRAGTTT-3′)来扩增土壤细菌的16S rRNA基因。PCR反应体系共20 μL:4 μL 5×FastPfu Buffer,2 μL 2.5 mmol/L dNTPs,0.8 μL Forward Primer(5 μmol/L),0.8 μL Reverse Primer(5 μmol/L),2 μL Template(10 ng DNA),0.4 μL FastPfu Polymerase和10 μL ddH2O。PCR扩增条件:95℃预变性5 min;95℃变性30 s,55℃退火30 s,72℃下延伸45 s,共有27个循环;最后在72℃下终延伸10 min结束。每个样本3次重复,将同一样本的PCR产物混合后再用2%的琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物,Tris-HCl洗脱;2%琼脂糖电泳检测PCR产物的纯度和浓度。
将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行定量检测,之后按照每个样品的测序量要求,进行相应比例的混合,进行Miseq测序。
通过利用QIIME软件技术,依照序列的相似度方法把序列分为数个OTUs(operational taxonomic unit)。统计出每个测试样品中OTU情况以及各个OTU中序列的个数。在Silva(Release115 http://www. arb-silva.de)数据库中将序列进行比对和物种分类。采用RDP classifier贝叶斯算法在97%相似水平的OTU代表序列进行生物信息分类学分析,分别在phylum(门)和genus(属)水平上统计各样本的群落组成,并对OTU进行生态学分析,计算土壤Shannon指数、Simpson指数、物种丰富度Chaol指数等。
土样样品由上海美吉生物医药科技有限公司应用Illumina平台的MiSeq进行测序。
2 结果与分析
2.1 土壤样品的测序结果
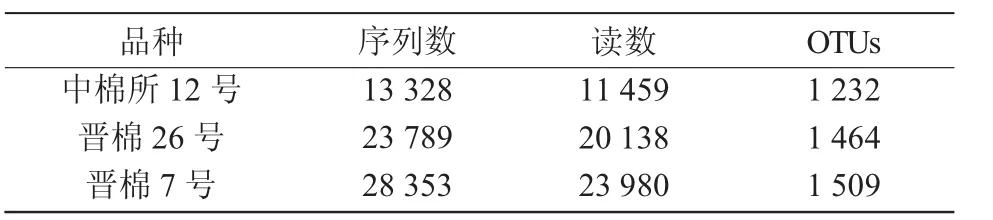
表1 不同棉花品种土壤细菌DNA测序结果
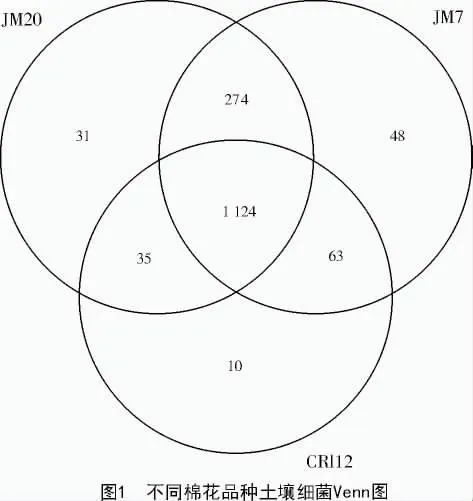
通过对3种棉花土壤细菌16S rDNA进行测序,过滤掉样品中低质量的原始序列后,得到总数为65 470个的有效序列,共获得55 577条读数(reads),并在97%相似度下将其聚类为用于物种分类的OUT。由表1可知,晋棉7号的土壤中含有最多数量的OTUs,其次是转基因棉花晋棉26号,中棉所12号OTUs数量最少;与晋棉7号相比,晋棉26号的土壤OTUs数量减少2.98%,中棉所12号土壤减少18.36%;3个棉花土样共产生1 585个OTUs,其中,3个棉花品种共享1 124个OTUs,共享率达70.91%(图1)。
2.2 土壤细菌的种类分析
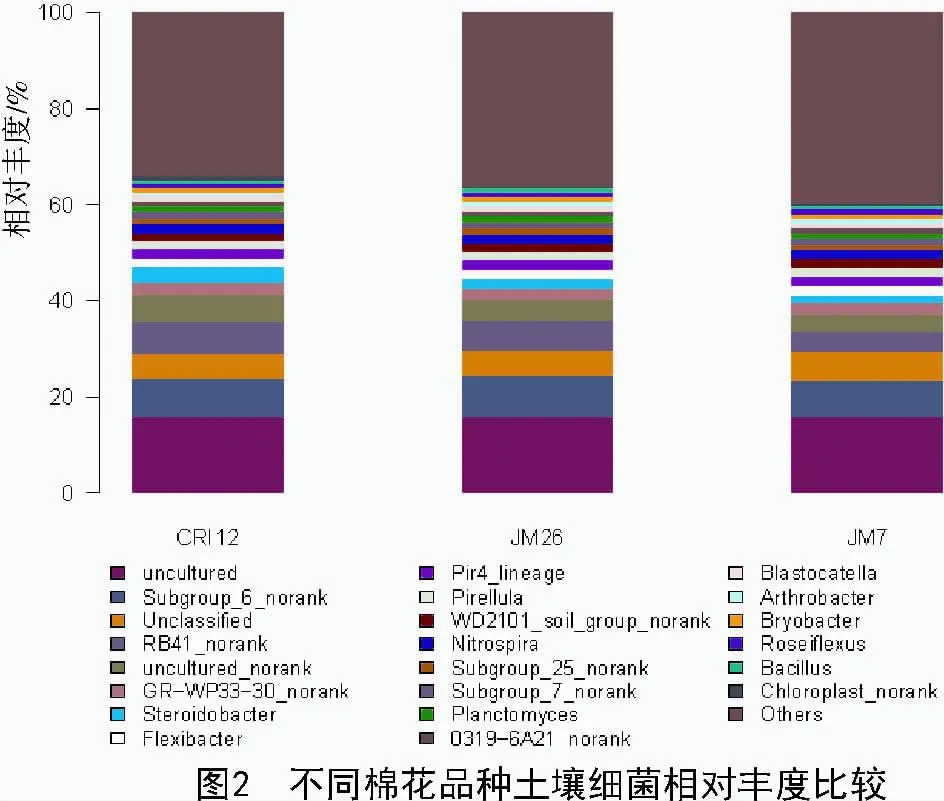
在门水平上,3个棉花土壤共检测到21个细菌门,除1个未被分类(unclassified)群体外,还有4个候选细菌门。其中,相对丰度较大的有变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)、芽单胞菌门(Gemmatimonadetes)、硝化螺菌门(Nitrospirae)、厚壁菌门(Firmicutes)、蓝藻门(Cyanobacteria)等10个细菌门,占到3个处理各自土壤细菌总量的94.9%以上。不同相对丰度大于5%的主要优势种群有变形菌门、酸杆菌门、放线菌门、浮霉菌门、绿弯菌门、拟杆菌门,分别占总量的26.6%~29.2%,19.1%~22.6%,8.8%~11.9%,9.2%~10.8%,7.4%~9.2%,6.6%~8.7%。其中,晋棉26号独有的种群为栖热菌门(Deinococcus-Thermus,相对丰度0.059 6%),晋棉7号独有的种群为WCHB1-60(相对丰度0.033%)。
在属水平上,3种棉花土壤细菌共有相对丰度>1%的优势属数量19个,3个棉花品种中均有分布的优势种属有12个。晋棉26号、晋棉7号土壤中优势属的种类最多,均为15个;中棉所12号土壤中优势属的种类最少,为14个(图2)。每个品种皆有自己特有的种群,但相对丰度很小,其中,转基因棉花晋棉26号土壤细菌有Truepera,D05-2_ norank,Methylohalomonas,34P16_norank,Tepidibacter种群,相对丰度0.004%~0.060%,而晋棉7号土壤细菌有Aliihoeflea,PAUC26f_norank,Mucilaginibacter,Martelella,Nubsella,WCHB1-60_norank,Propionibacterium种群,相对丰度为0.012 5%~0.060 0%,中棉所12号有Parasegetibacter种群,相对丰度为0.02%等。
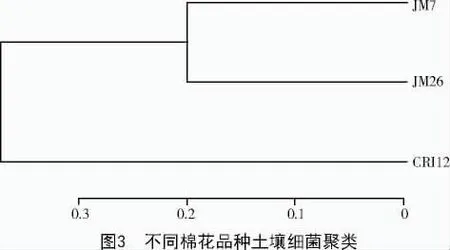
聚类分析结果表明,3个棉花品种可分为2大类,其中一类为传统棉花品种中棉所12号,另一类为转基因棉花品种晋棉26号及其非转基因亲本晋棉7号。表明转基因晋棉26号土壤细菌种群结构与亲本晋棉7号没有明显差异(图3)。
2.3 转基因棉花土壤细菌的多样性分析
试验结果表明(表2),转基因棉花晋棉26号的Shannon指数低于其非转基因亲本晋棉7号,但高于传统品种中棉所12号,说明转基因棉花土壤细菌多样性低于非转基因;Chaol指数大小依次为晋棉7号>晋棉26号>中棉所12号,说明转基因棉花丰富度低于非转基因亲本;Simpson指数依次为中棉所12>晋棉26>晋棉7,说明转基因棉花晋棉26多样性低于晋棉7号。
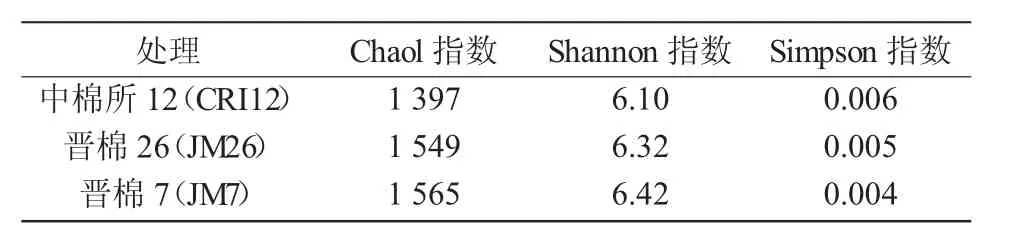
表2 不同棉花品种对土壤细菌多样性的影响
3 结论与讨论
本研究利用高通量的测序技术对土壤细菌16S rDNA进行扩增测序,结果表明,在相似度为97%的条件下,OTUs囊括了土壤细菌量的94%以上;而且测序还发现了很多未被分类细菌,表明高通量测序可以得到更为全面的微生物信息,很多未知细菌或未分类菌也能通过测序发现。因此,利用高通量基因测序研究微生物是一种有效的方法。
转基因作物主要通过根系分泌物或残体进入土壤生态环境,从而影响特定的微生物种群。HU等[14]采用传统微生物培养法进行研究,结果表明,连续5 a种植转基因棉花,其根际土壤功能细菌没有受到明显影响。LI等[15]研究发现,连续3 a种植转基因棉花对土壤微生物没有影响(传统微生物培养法)。ZHANG等[16]采用PCR-DGGE进行3 a田间研究,结果表明,NC33B棉花对土壤细菌、真菌、放线菌没有影响。本研究表明,棉花土壤细菌的优势种群有变形菌门、酸杆菌门、放线菌门、浮霉菌门、绿弯菌门、拟杆菌门,转基因晋棉26号有独有的种群栖热菌门,晋棉7号独有的种群为WCHB1-60;在属水平,转基因棉花晋棉26号土壤细菌有Truepera,D05-2_norank,Methylohalomonas,34P16_ norank,Tepidibacter,而晋棉7号土壤细菌有Aliihoeflea,PAUC26f_norank,Mucilaginibacter,Martelella,Nubsella,WCHB1-60_norank,Propionibacterium,这些独特的种群相对丰度都很低,没有影响细菌的种群结构。SINGH等[23]用RFLP-PCR检测出转Bt基因茄子土壤独有蓝藻细菌(Cyanobacteria)和拟杆菌(Bacteroidetes),而非转基因茄子土壤中独有β变形菌、绿屈挠菌属、梭杆菌门和浮霉菌门,并没有改变土壤细菌菌落结构。但也有研究表明,连续种植转基因棉花会对土壤细菌菌落产生影响[17-21]。因此,有关转基因棉花对土壤微生物的影响需要建立多学科、多种方法、长期定位进行综合评价,才能获得更加有效准确的评价。
[1]JAMES CLIVE.2015年全球生物技术/转基因作物商业化发展态势[J].中国生物工程杂志,2016,36(4):1-11.
[2]SINGH AMIT KISHORE,SURESH,KUMAR DUBEY.Current trends in Bt crops and their fate on associated microbial community dynamics:review[J].Protoplasma,2016,253:663-681.
[3]DONEGAN K K,PALM C J,FIELAND V J,et al.Changes in levels,species,and DNA fingerprints of soil microorganisms associated with cotton expressing the Bacillus thuringiensis var.kurstaki endotoxin[J].Applied Soil Ecology,1995,2:111-124.
[4]李永山,范巧兰,陈耕,等.转Bt基因棉花对土壤微生物的影响[J].农业环境科学学报,2007,26(Z):533-536.
[5]李永山,范巧兰,陈耕,等.利用PLFA研究转Bt基因棉花对土壤微生物群落结构变化的影响[J].棉花学报,2009,21(6):503-507.
[6]范巧兰,陈耕,李永山,等.转Bt基因棉花不同生育期对土壤微生物的影响[J].山西农业科学,2010,38(12):34-36.
[7]范巧兰,陈耕,李永山,等.转基因棉花不同生物量对土壤微生物群落结构的影响[J].棉花学报,2012,24(1):85-90.
[8]娜布其,红雨,杨殿林,等.利用根箱法解析转双价(Bt+CpTI)基因棉花对土壤微生物数量及细菌多样性的影响[J].棉花学报,2011,23(2):160-166.
[9]沈法富,韩秀兰,范术丽.转Bt基因抗虫棉根际微生物区系和细菌生理群多样性的变化[J].生态学报,2004,24(3):432-437.
[10]雒珺瑜,崔金杰,李树红,等.不同棉花品种对土壤微生物的影响[J].中国棉花,2005,32(9):10.
[11]SHEN REN FANG,CAI HONG,GONG WAN H E.Transgenic Bt cotton has no apparent effect on enzymatic activities or functional diversity of microbial communities in rhizosphere soil[J].Plant and Soil,2006,285:149-159.
[12]SUN C X,CHEN L J,WU Z J,et al.Soil persistence of Bacillus thuringiensis(Bt)toxin from transgenic Bt cotton tissues and itseffect on soil enzyme activities[J].Biology and Fertility of Soils,2007,43,617-620.
[13]Head G,Surber J B,Watson J A,et al.No detection of Cry1Ac protein in soil after multiple years of transgentic Bt cotton(Bollgard)use[J].Environmental Entomology,2002,31:30-36.
[14]HU H Y,LIU X X,ZHAO Z W.Effects of repeated cultivation of transgenic Bt cotton on functional bacterial populations in rhizosphere soil[J].World J Microbiol Biotechnol,2009,25:357-366.
[15]LI X G,LIU B,CUI J J,et al.No evidence of persistent effects of continuously planted transgenic insect-resistant cotton on soil microorganisms[J].Plant and Soil,2011,339:247-257.
[16]ZHANG YANJUN,XIE MING,WU GANG,et al.A 3-year field investigation of impacts of Mansanto's transgenic Bt-cotton NC33B on rhizosphere microbial communities in northern China[J].Applied Soil Ecology,2015,89:18-24.
[17]崔金杰,雒珺瑜,李树红,等.转基因抗虫棉对土壤微生物影响的初步研究[J].河北农业大学学报,2005,28(6):73-75.
[18]张金国,刘翔,崔金杰,等.转基因(Cry1Ac)抗虫棉对土壤微生物的影响[J].中国生物工程杂志,2006,26(5):78-80.
[19]CHEN Z H,CHEN L J,ZHANG Y L,et al.Microbial properties,enzyme activities and the persistence of exogenous proteis in soil under consecutive cultivation of transgenic cottons(Gossypium Hirsutum L.)[J].Plant and Soil Environment,2011,57(2):67-74.
[20]CHEN Z H,CHEN L J,WU Z J.Relationships among persistence of Bacillus thuringiensis and Cowpea trypsin inhibitor proteins,microbial properties and enzymatic activities in rhizosphere soil after repeated cultivation with transgenic cotton[J].Applied Soil E-cology,2012,53:20-23.
[21]李永山,范巧兰,刘凯,等.种植转Bt基因棉花不同年限土壤PLFA指纹特征与差异[J].中国农业大学学报,2015,20(3):30-37.
[22]BARTRAM A K,LYNCH M D J.Generation of multimillion-sequence 16S rRNA gene libraries from complex microbial communities by assembling paired-end Illuminareads[J].Applied and Environmental Microbiology,2011,77(11):3846-3852.
[23]SINGH AMIT KISHORE,GOVIND KUMAR RAI.Bacterial community structure in the rhizosphere of a Cry1Ac Bt-brinjal crop and comparison to its non-transgenic counterpart in the tropical soil[J].Microb Ecol,2013,66:927-933.
Effects of Transgenic Cotton on Soil Bacteria Community
FANQiaolan1,2,LI Yongshan1,2,WANGHui1,2,XI Kaipeng1,2,XI Jilong1,2,SHI Jundong1,2,ZHANGJiancheng1,2
(1.Institute ofCotton,Shanxi Academy ofAgricultural Sciences,Yuncheng 044000,China;2.Yuncheng Academy ofAgricultural Sciences,Yuncheng 044000,China)
The experiement was conducted to evaluate the effects of transgenic cotton on soil bacterial community structure and diversity.Transgenic Bt cotton Jinmian 26 and its non-Bt parental line Jinmian 7,and traditional cultivar CRI 12 were used in long-term experiment site to evaluate the effects of transgenic cotton on soil bacterial community structure by high throughput sequencing method. High throughput sequencing analysis showed 1 585 OTUs were detected in soil bacterial communities.Twenty one phylum,one unclassified and 4 candidate bacteria were detected in soils.Dominant phylum were Proteobacteria,Acidobacteria,Actinobacteria, Planctomycetes,Chloroflexi,Bacteroidetes,Gemmatimonadetes,Nitrospirae/Firmicutes,Cyanobacteria,in all soils.Deinococcus-Thermus in transgenic Jinmian 26 soil and WCHB1-60 in non-Bt Jinmian 7 were found uniquely in phylum.Shannon index in Bt-soil bacteria was lower than non-Bt soil,and soil bacteria community structure was not affected by transgenic cotton.
transgenic cotton;soil;bacteria;community structure;high throughput sequencing
S154.38+1
:A
:1002-2481(2017)07-1124-04
10.3969/j.issn.1002-2481.2017.07.21
2016-12-29
山西省自然科学基金项目(2014011029-2);国家自然科学基金项目(31372143,30871601)
范巧兰(1965-),女,山西夏县人,助理研究员,主要从事植物保护和转基因植物安全评价研究工作。李永山为通信作者。
