新德里金属-β-内酰胺酶-1的结构、催化机理及其抑制剂研究进展
2017-07-06胡建平
胡建平, 左 柯, 万 华
(1.成都大学 药学与生物工程学院, 四川 成都 610106;2.成都大学 四川抗菌素工业研究所, 四川 成都 610052;3.华南农业大学 数学与信息学院, 广东 广州 510642)
新德里金属-β-内酰胺酶-1的结构、催化机理及其抑制剂研究进展
胡建平1,2, 左 柯1,2, 万 华3
(1.成都大学 药学与生物工程学院, 四川 成都 610106;2.成都大学 四川抗菌素工业研究所, 四川 成都 610052;3.华南农业大学 数学与信息学院, 广东 广州 510642)
β-内酰胺类抗生素,作为一线抗生素被广泛用于各类细菌感染的临床治疗.由位于质粒的基因blaNDM-1编码的新德里金属-β-内酰胺酶-1可高效水解几乎所有β-内酰胺类抗生素,对β-内酰胺类抗生素的应用造成了极大的威胁,开发有效的抑制剂新德里金属-β-内酰胺酶-1抑制剂迫在眉睫.而抑制剂分子的设计改造工作的开展有赖于对新德里金属-β-内酰胺酶-1结构和催化功能的研究,本综述旨在总结目前有关新德里金属-β-内酰胺酶-1结构、催化机理及其抑制剂的相关研究进展,为基于新德里金属-β-内酰胺酶-1结构的药物分子设计、改造等研究提供帮助.
细菌耐药;NDM-1;催化机理;抑制剂;药物设计
0 引 言
自1929年,人类发现第一种抗生素——青霉素以来,抗生素已挽救了无数的生命.目前,以青霉素和头孢菌素为代表的β-内酰胺类抗生素,是治疗细菌感染的常用药物,该类抗生素的化学结构中均含有一个发挥药效必需的四元β-内酰胺环,其化学结构如图1所示.当其作用于细菌时,β-内酰胺类抗生素的β-内酰胺环开环,与细菌细胞壁合成途径中的羧肽酶和转肽酶发生酰化作用,抑制该酶的生物活性而使合成的细胞壁出现缺陷,最终导致菌体裂解死亡[1-2].
随着抗生素的广泛应用,细菌耐药问题也日益凸显[3],同时携带多种耐药基因的“超级细菌",如泛耐药鲍曼不动杆菌、耐碳青霉烯肺炎克雷伯菌、铜绿假单胞菌等也频频被报道[4-6].在细菌耐药机制中,产生一种或多种水解酶或钝化酶灭活抗菌药物最为常见,而β-内酰胺酶是一种可催化β-内酰胺环水解开环的水解酶.基于氨基酸序列的同源性[7],β-内酰胺酶可分为A、B、C和D 4类.其中,A、C和D类酶的活性位点为丝氨酸残基,故又统称为丝氨酸-β-内酰胺酶(serine-β-lactamase,SBL);而B类酶则依靠1个或2个金属离子行使催化功能,因此又称为金属-β-内酰胺酶(metallo-β-lactamase,MBL),具体可细分为B1、B2和B3 3个亚类.

图1 β-内酰胺类抗生素的化学结构
新德里金属-β-内酰胺酶-1(New Delhi metallo-β-lactamase-1,NDM-1)是一种广谱高效的B1 MBL,于2009年首次被报道[8-9].目前,研究人员已发现同类的另外14个变种[10],依次命名为NDM-2~NDM-15.NDM-1由位于质粒的blaNDM-1基因编码,可水解除氨曲南和美西林外几乎所有的β-内酰胺类抗生素[8,10-11].研究发现,blaNDM-1基因并不仅限于在医院获得性菌种间传递,同时也可在社区高度流行的菌种间传播[12-13],现已发现近10种细菌表达NDM-1[14].这些表达NDM-1的细菌可引起包括尿道和肺部感染、腹膜炎、软组织感染及败血症等疾病,为相关病症的临床治疗造成了严重威胁.
随着检测技术的发展,比如,改良Hodge试验[15-16]和基于显色培养基筛选法[17-19]等表型方法,基于PCR的体外蛋白表达技术[20]、基质辅助激光解吸电离飞行时间质谱法[21-22]等基因型方法,以及分子方法均可有效检测出产NDM-1的菌株.同时,在治疗方面,虽然有报道联用多种抗菌药物并口服磷霉素成功治愈产NDM-1肺炎克雷伯菌引发的尿道感染的个案[23],但目前仍缺乏对产NDM-1菌株特效的治疗药物.
考虑到β-内酰胺类抗生素良好的药效和较低的毒副作用,开发有效的NDM-1抑制剂并与β-内酰胺类抗生素配伍使用是目前主要的研发思路.而掌握NDM-1的化学结构、催化机理等生物化学信息,可为其抑制剂的设计、改造等提供有力的技术支撑.
1 NDM-1的分子结构
独特的分子结构是NDM-1发挥水解功能的基础,同时也是筛选、设计抑制剂的重要依据.截至2017年3月,RCSB蛋白质数据库(http://www.rcsb.org/)共收录了30个NDM-1晶体结构,除3S0Z(PDB ID)为大肠杆菌NDM-1外,其余均来自肺炎克雷伯菌.NDM-1为双金属MBL[24-27],在30个NDM-1晶体结构中,活性口袋中含有金属离子的结构有23个(3SFD为单锌离子结构),同时结合β-内酰胺类抗生素的结构缩为14个,其中结合抑制剂的结构有3个(5A5Z、4U4L、4EXS).表1列出了NDM-1-底物/抑制剂复合物晶体结构的具体信息.

表1 NDM-1-底物/抑制剂复合物的晶体结构信息

1.1 NDM-1的整体结构
NDM-1由单多肽链构成,共含有270个氨基酸残基,呈典型的αβ/βα夹层结构[28-30],具体如图2所示.

图2 NDM-1的整体结构
从图2中可以看出,分子暴露在溶剂中的两侧分别为2段α螺旋,从N端往C端依次为α1、α2、α5和α6;2个β折叠通过一段α-β-α的结构相连接位于分子的内层.其中,靠近N端的β折叠由7股平行或反平行的β折叠片构成,而靠近C端的β折叠则由4股组成[26].3段loop区L3(L65-V73)、L7(T119~M126)和L10(C208~L221)组成了活性口袋的主体部分,具有催化活性的2个Zn2+(Zn1与Zn2)则位于口袋底部[31].

此外,研究还发现,在细菌体内,NDM-1以单体形式发挥催化活性[31],而目前解析得到的NDM-1晶体结构则多为二聚体,其在膜结合与纯化状态下能通过疏水与范德华相互作用进行二聚被推测与该酶独特的耐药机制有关[28].
1.2 NDM-1的活性位点
图3给出了NDM-1活性位点的结构:位于活性中心的Zn1与H120、H122、H189及水分子/OH-(与晶体生长时的pH有关[27,35])结合构成四面体构型,即组氨酸位点;Zn2与D124、C208和H250结合构成三角锥形结构形成半胱氨酸位点;Zn1与Zn2通过D124的侧链相联系[26,30-31].
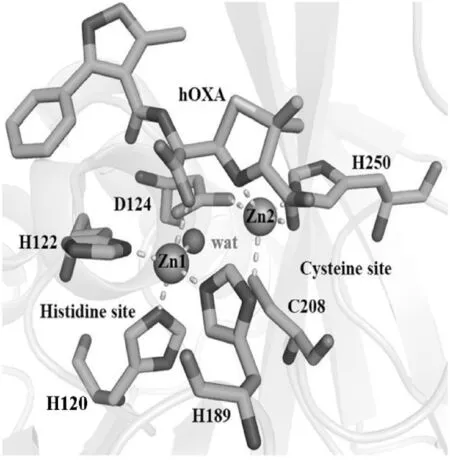
图3 NDM-1活性位点的局部放大图
从图3可以看出,Zn1与水解后的苯唑西林(oxacillin,OXA)的β-羧基间存在相互作用,Zn2还与噻唑环上的N和羧基配位结合,提示Zn2+在NDM-1与底物的结合和催化水解反应的过程中均具有重要作用.Zn2+与底物间的这种相互作用,使β-内酰胺环维持在易被进攻的空间取向状态,从而使得水解反应得以高效进行[36].晶体学实验也发现,Zn2+可以被其他金属离子,如Cd2+、Mn2+、Co2+等,替换[30].
Yang等[37]测定了野生型NDM-1和Zn/Co、Co/Co和Co/Cd 3种金属离子替换后的NDM-1水解chromacef的动力学常数,以及紫外—可见光光谱(UV-vis)、核磁共振氢谱(1H NMR)、电子顺磁共振谱(EPR)、X射线吸收精细结构谱(EXAFS)等数据,结果显示:金属替换后,NDM-1的酶促动力学常数出现明显变化,具有顺磁性的金属取代NDM-1,比如Co/Cd和Zn/Co,与底物反应时有显著的结构变化;与其他B1 MBLs相比,NDM-1的活性位点拥有更大的表面积,这显然与构成活性位点的L3和L10均距离锌离子中心较远有关[28].Yuan等[38]通过分子对接预测了160种β-内酰胺类抗生素在NDM-1活性位点处的构象,发现NDM-1在结合底物后,其活性位点构象并未出现较为显著的变化.这为NDM-1水解底物的广谱性提供了重要的结构基础.
此外,科研人员对活性位点的结构分析还发现一些关键识别残基,例如:K125通过与周围残基形成的氢键网络稳定L7的构象,同时对与Zn1结合的残基有一定的空间位阻排斥效应[27];Y229对稳定活性位点的构象同样具有重要意义[27,31];定点突变L65A导致氨苄西林和一些头孢菌素的最低抑菌浓度下降,而定点突变N220A则导致NDM-1对氨苄西林、头孢吡肟、美罗培南和亚胺培南的水解能力大大降低[39].Zhu等[40]对NDM-1与氨苄西林、头孢硝噻吩和美罗培南形成的3个复合物体系进行了30 ns的分子动力学模拟发现,位于L3上的L65、M67和F70均与氨苄西林和头孢硝噻吩的侧链形成了较强的疏水作用,此对于维持复合物的稳定性具有重要意义,并给出了L3在整个模拟过程中具有3簇主要构象,并呈现出较大的片段柔性.相关研究表明:Q123与D124侧链的氧原子可与临近的β内酰胺类抗生素侧链形成氢键[27];K211、G219、N220与H189在IMP-1、CcrA等同类MBLs中高度保守,K211除了参与底物的识别与水解反应外,还可通过与底物形成盐键维持其有利于水解反应的正确取向;N220可与底物的羰基氧原子形成氢键,还可与Zn1形成相互作用,后者可能与维持水解反应中的四面体中间产物的稳定有关[26-28,39,41].
2 NDM-1的催化机理
作为B1类MBL成员之一,NDM-1催化底物水解的机理与同类MBLs类似[27],主要包括,β-内酰胺羰基与Zn1结合,OH-的亲核进攻,质子向N原子的转移和C-N键的断裂,具体如图4所示.但其水解机理中的具体细节尚未被全部阐明,还有待进一步的探究.
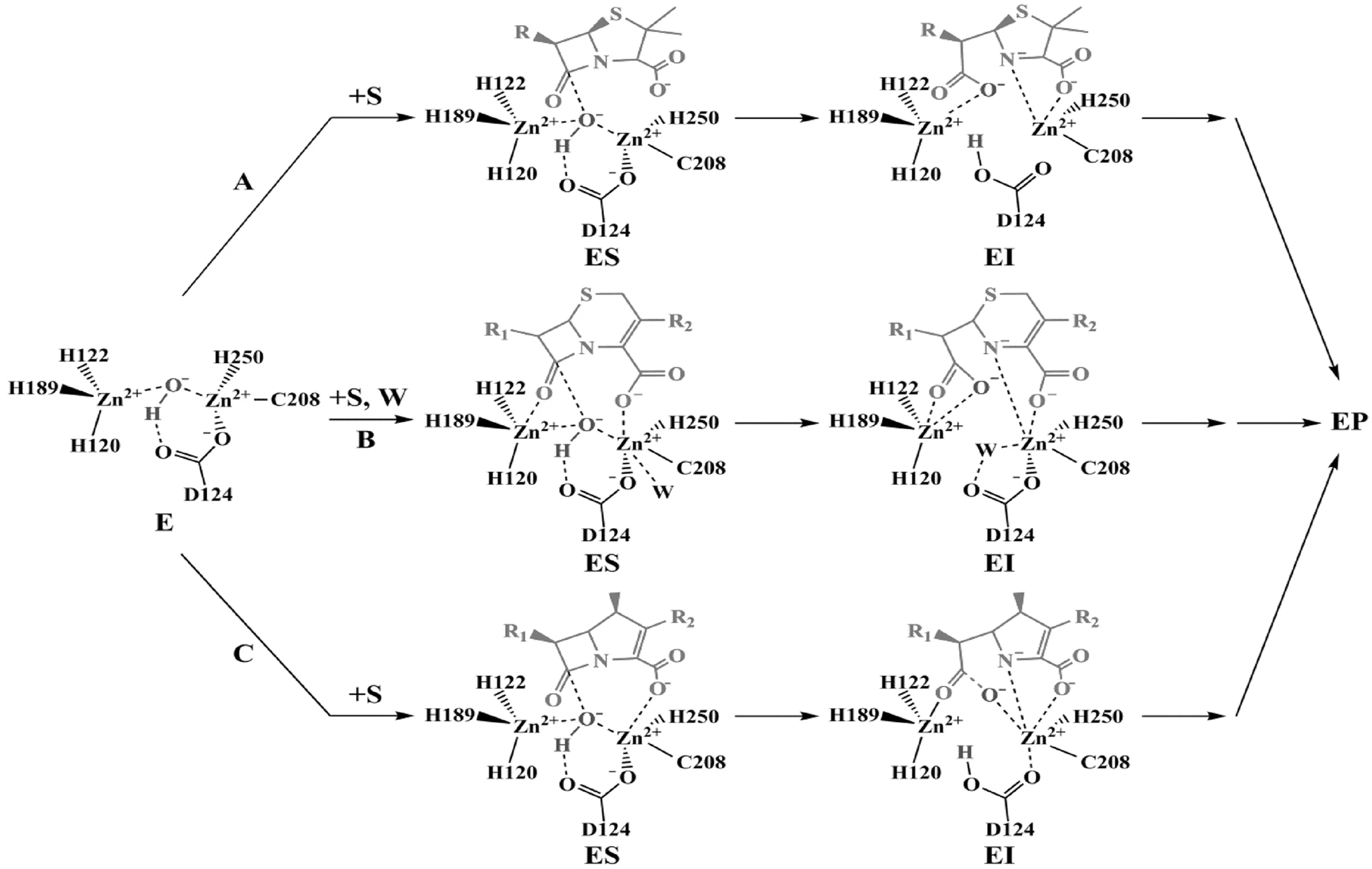
A表示青霉素类β-内酰胺类抗生素时可能的水解途径;B表示头孢菌素类β-内酰胺类抗生素时可能的水解途径;C表示碳青霉烯类β-内酰胺类抗生素时可能的水解途径.E表示NDM-1,S表示底物,I表示中间体,P表示产物,W表示水.机理的绘制参考文献[37,42,45].
图4 NDM-1可能的催化机理
关于反应中的亲核试剂和广义碱,有学者认为,溶剂环境中的水分子在水解反应中作为亲核试剂,而位于活性位点中的OH-作为广义碱,溶剂环境中的水分子向OH-给出质子后生成新的OH-,然后进攻β-内酰胺环的羰基碳原子,该反应的发生需要越过约80 kJ/mol的能垒[27,35];也有学者认为,亲核试剂可能是位于活性位点的水分子,而D124或环境中的水分子作为广义碱接受质子,该情况下的反应能垒大于或等于200 kJ/mol[42].
有学者认为,按照其他MBLs的反应机理,位于Zn1和Zn2间的OH-作为亲核试剂进攻β-内酰胺环后,C-N键可立即断裂形成氮负离子中间体[43].但Zhang等[42]提出,β-内酰胺环受到OH-进攻后,C-N键的断裂可能与负离子的质子化反应协同进行,并不产生氮负离子中间体.但无论是上述哪种情况,氮原子上的质子均来自亲核进攻的OH-.而科研人员在NDM-1Δ6、NDM-1Δ21和NDM-1Δ36(分别表示移除分子的前6、21和36个氨基酸)的酶促动力学研究过程中发现,NDM-1的催化机理中可能存在不只一个中间体[44].在探究NDM-1对头孢菌素类β-内酰胺类抗生素的水解机制中,Feng等[25]通过1H NMR监测到了二氢噻嗪环中双键转移的情况,最终质子并不是转移至N原子,而是根据底物的结构迁移至不同的部位,并得到了反应中间体的晶体结构.
此外,研究人员在对金属取代NDM-1催化机理的研究中还发现:金属离子的种类并不能显著影响反应的能垒;产物的释放速率与半胱氨酸位点的金属路易斯酸性大小有关(Zn>Co>Cd),但同时也受到组氨酸位点处金属种类的影响;当用Co2+离子取代Zn1时,生成产物的速率比未取代时快2~3倍,这可能与Co2+离子改变了与底物反应时的电子结构,降低了电子结构对称性有关;当用Cd2+离子取代Zn2时,中间体的生成速率有明显影响,提示Zn2在亲核进攻和稳定中间体的过程中具有重要意义[37].
目前,分子动力学模拟和混合量子力学/分子力学模拟等理论研究方法在酶催化反应的机理研究中应用广泛.Zheng等[42]使用SCC-DFTB/CHARMM混合力场模拟了NDM-1与底物氨苄西林和抑制剂L-卡托普利的结合模式,并根据密度泛函理论对氨苄西林的开环过程进行了理论计算,模拟结果显示:OH-位于两个Zn2+的中间位置,并通过氢键与D124相连;氨苄西林的羧基并未与Zn2相连,而是通过氢键与K211和N220连接;后续OH-亲核进攻、C-N键断裂、形成氮负离子中间体以及开环后羧基与Zn2配位键的形成几乎同时进行(见图4).Zhu等[45]使用ONIOM算法对NDM-1催化美罗培南的水解机理研究发现,离子化的D124是C-N键断裂的有利因素;在没有水分子与Zn2结合时,反应过程中更倾向于形成氮负离子中间体,且K211在底物结合和催化反应的过程中均具有重要意义.由于NDM-1高效的水解速率,目前很难通过实验对理论计算得出的过渡态进行验证.相关研究也表明,针对不同类型的底物NDM-1的催化机理各不相同.为了指导设计开发具有临床应用价值的抑制剂,NDM-1催化下的水解机理还有待在分子层面做进一步探究.
3 NDM-1抑制剂的研究现状
目前,科研人员为应对NDM-1带来的耐药问题采取的主要策略为:在了解NDM-1结构及可能水解机制的情况下,不断开发或寻找不易被NDM-1水解的新抗生素;筛选设计NDM-1抑制剂,并与传统抗生素制成合剂.
3.1 含硫类抑制剂
硫元素是生命活动中的重要元素之一,也常出现在各类药物分子中.含硫类抑制剂的结构式及IC50值如图5所示.
血管紧张素转化酶抑制剂卡托普利中含有巯基结构,是经典的抗高血压药物.研究发现,D-卡托普利1对NDM-1表现出较好的抑制活性(IC50=7.9 μM),而L-卡托普利的抑制活性相对较弱,其IC50为202.0 μM[31].Li等[46]为进一步探究NDM-1抑制剂的药效基团,设计合成并测试了系列D-卡托普利类似物2-6的抑制活性,发现R构型的苄胺类似物6a表现出了较高的抑制活性,其IC50值为1.5 μM,远高于S型旋光异构体.Shen等[47]发现噻吩羧酸类衍生物7-9对NDM-1具有一定的抑制作用,与美罗培南联合给药可提高美罗培南的抗菌活性.此外,Wang等[48]通过多步虚拟筛选从含有280万个类药分子的ZINC化合物库中筛出3个磺胺类NDM-1抑制剂10-12,其分子动力学模拟显示,分子中的磺酰胺基直接与Zn1结合,从而阻碍水解反应的发生,提示磺酰胺基可能是开发NDM-1抑制剂式可选的优势骨架结构.
3.2 苯并吡喃类抑制剂
苯并吡喃类NDM-1抑制剂的结构如图6所示.
利用分子对接方法,Proschak等[49]将SPECS数据库中的分子对接至NDM-1等4种MBLs晶体结构中.分子对接同时使用MOE、GOLD和PLANTS 3款软件进行,以3款软件预测结合模式的均方根偏差小于或等于0.2 nm为判据进行分子片段筛选;在筛得的分子片段中,仅保留MBLs中结合模式均方根偏差值小于或等于0.2 nm的片段进行Fluorocillin试验评价活性,由此得到抑制活性最好的苯并吡喃类化合物13(IC50=50±9 μM)[50].此外,Gan等[51]还从青霉菌株(Penicillium sp. 109F 484)中分离得到2种具有微弱NDM-1抑制活性的聚酮化合物14和15,二者的IC50分别为94.9 μM和87.9 μM.抗菌实验显示,化合物14和15对产NDM-1肺炎克雷伯菌均无抑制活性,与美罗培南联合使用时,也无法提高美罗培南的抗菌活性.
图5 含硫类NDM-1抑制剂的结构
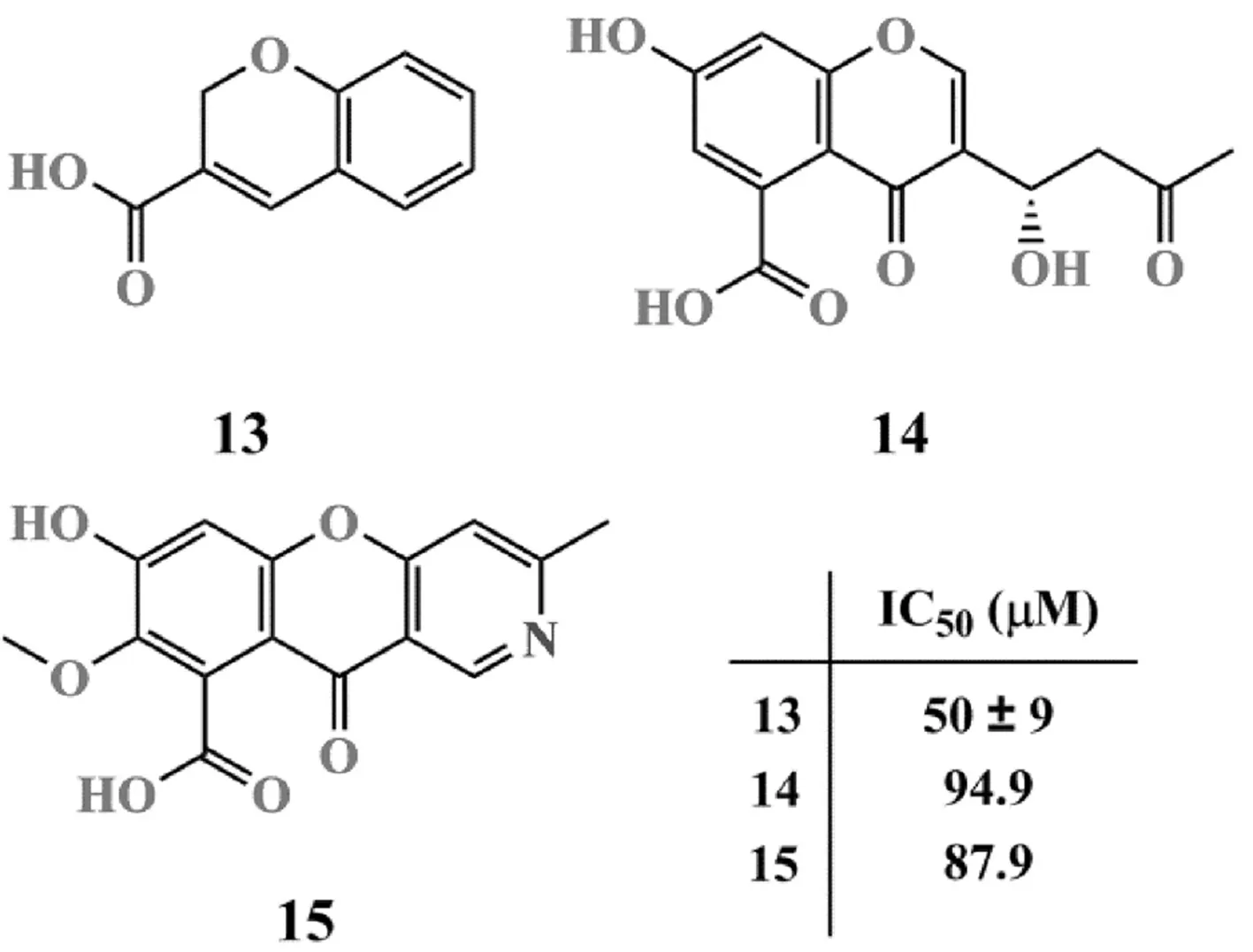
图6 苯并吡喃类NDM-1抑制剂的结构
3.3 共价结合型抑制剂
共价结合型NDM-1抑制剂的结构如图7所示.
研究发现,通过氢键、范德华等非键相互作用与靶点可逆性结合是大多数药物在发挥药效时与靶点的结合特点,但也不乏通过形成共价键与靶点不可逆结合的例子,如细胞毒类抗肿瘤药物.在NDM-1共价结合型抑制剂的开发过程中,Christopeit等[52]报道了2种可逆共价结合型非β-内酰胺类抑制剂16(Kd=181 μM)和17(Ki=580 nM),其电喷雾离子化质谱和氨基酸定点突变实验显示,化合物16和17通过单键与NDM-1活性位点的K224结合;通过共价分子对接还发现,化合物17的酮羰基与Zn2之间存在极性相互作用,并与W87、I35和V67间形成输水相互作用.Thomas等[53]通过高通量筛选也得到了2种共价结合型NDM-1抑制剂,对氯汞苯甲酸18和硝普钠19,两者的抑制活性(IC50)分别为2.3 μM和9.0 μM,通过质谱联用技术检测和C208D突变实验发现,化合物18可以选择性共价修饰NDM-1活性位点结合Zn2的C208,从而发挥抑制作用.另外,Thomas等[54]还报道了头孢克洛20通过K211介导的多步反应,不可逆地共价抑制NDM-1的活性(Ki=2.3±0.1 mM),提示K211可能是开发共价结合型抑制剂的关键氨基酸残基.依布硒啉21是一种新型非甾体抗炎药,临床上主要用于缺血和中风的治疗,研究发现:依布硒啉可与键合Zn2的C208形成S-Se键,从而影响NDM-1的催化功能;以(1.3~1.4)∶1的比例联合使用依布硒啉和氨苄西林或美罗培南,可使得β-内酰胺类抗生素的MICs分别下降4和35倍[55].

图7 共价结合型NDM-1抑制剂的结构
3.4 金属螯合剂
金属离子作为NDM-1发挥催化功能的关键辅助因子,若使用金属螯合剂封闭NDM-1活性位点的Zn2+,理论上可有效抑制其催化活性.典型NDM-1金属螯合剂的结构如图8所示.

图8 金属螯合剂的结构
研究发现,乙二胺四乙酸22是常见的金属螯合剂,可有效抑制NDM-1(IC50=1.6 μM),但由于其具有较强的毒性,并不适合直接用于临床治疗[56].Azumah等[57]评价了3种金属螯合剂23~25在与美罗培南联合使用时,NDM-1对美罗培南的耐药情况:当化合物23用量在4和8 mg/L时活性最佳,可使美罗培南的MICs分别降低至0.5和0.06 mg/L;化合物24在8和16 mg/L的剂量时,同样也可将美罗培南的MICs分别降至1和0.125 mg/L;但化合物25对NDM-1的抑制效果较差,需要将用量提至32 mg/L才可将美罗培南的MICs降低至0.06 mg/L.此外,Falconer等[58]还报道了一类二氢吲哚螺噻二唑类金属螯合剂26,并通过产NDM-1肺炎克雷伯菌小鼠感染模型证明了该类化合物对NDM-1的抑制活性,其单晶结构显示,原化合物26的S-Cspiro键断裂,噻二唑环开环形成亚胺结构,暴露的S与亚胺N构成二齿配体与Zn2+进行螯合,从而发挥抑制作用.
3.5 其 他
其他NDM-1抑制剂分子的结构如图9所示.
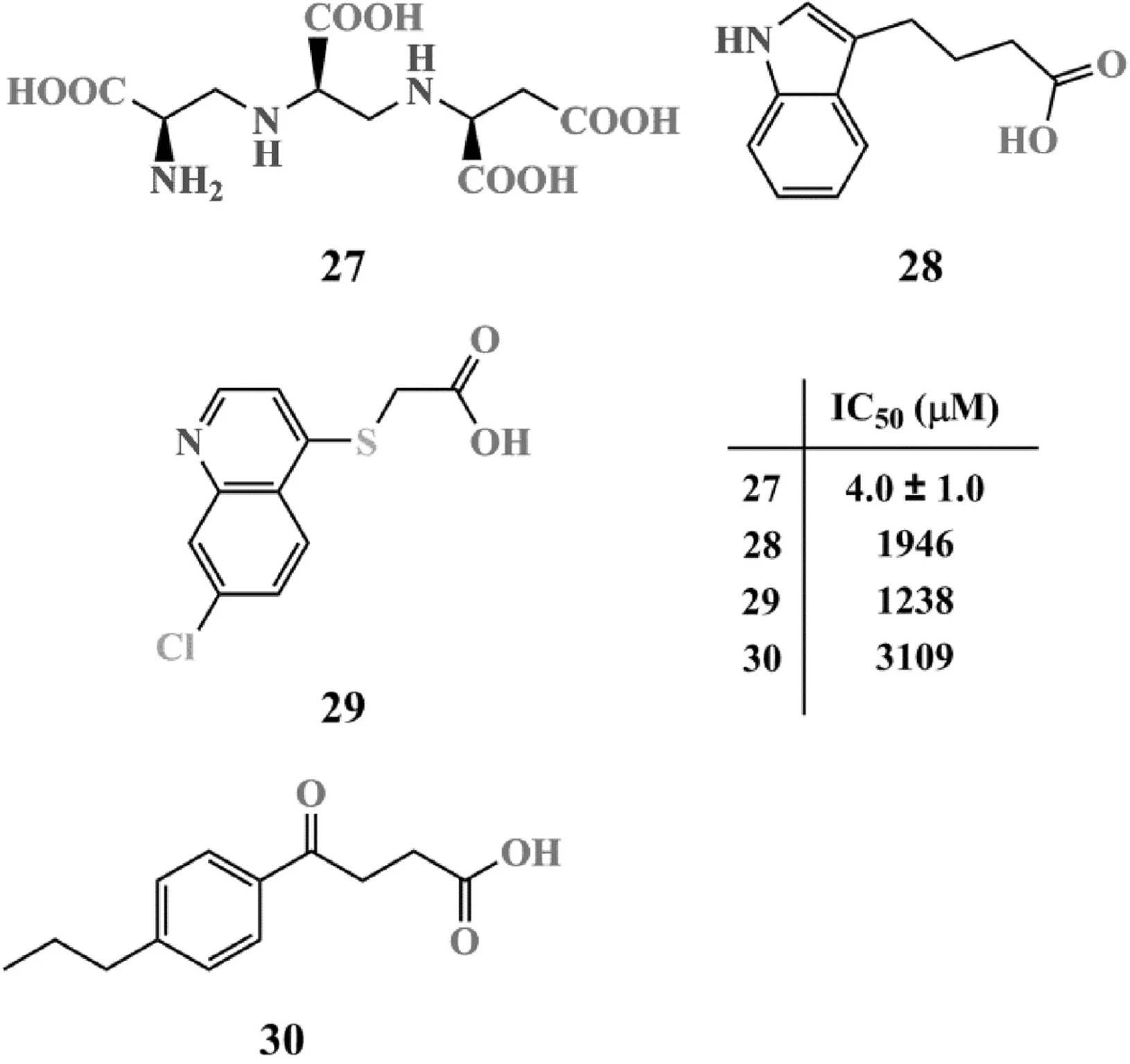
图9 其他NDM-1抑制剂分子的结构
曲霉明A 27是一种从杂色曲霉菌株中分离得到的一种天然产物.研究发现,曲霉明对血管紧张素转化酶和内皮素转化酶均有良好的抑制作用,其IC50值分别为1.2 μM和3.4 μM[59-61].进一步研究发现,曲霉明对NDM-1的抑制作用同样出色(IC50=4.0±1.0 μM)[62].研究人员利用电感耦合质谱证实,曲霉明对NDM-1的抑制主要通过耗尽NDM-1活性位点中的Zn2+实现,属于一种不可逆抑制作用,但这种抑制作用造成的失活状态可通过加入过量的ZnSO4实现完全复活[62].虽然曲霉明在抑制活性和毒性等方面的表现令人满意,但由于其较高的亲水性,其药动学性质还有待进一步改善[62].Chrestopeit等[63]使用正交筛选联合表面等离子共振技术得到了另外3种对NDM-1有一定抑制作用的片段分子28~30,其QM-polarized分子对接结果显示:28~30的羧基可与Zn2结合,并与K224和N233形成氢键;28和30与W87、L59和V67间还存在一定的疏水作用.这些结合力可能是维持这些片段分子发挥抑制活性的关键因素.
4 展 望
产NDM-1的耐药菌感染对使用β-内酰胺类抗生素的临床治疗造成了严重威胁.自2009年首次报道NDM-1至今,各国学者已在NDM-1的结构、催化机理及其抑制剂等领域开展了一系列研究,并基于不同底物提出了NDM-1多种可能的催化机理,但对于催化机理中的碱、亲核试剂及中间体质子化过程等具体细节仍在探讨之中.就目前开发的抑制剂而言,抑制活性普遍维持在微摩尔级,虽在体外或体内均取得了较为满意的结果,但又由于分子固有的毒性或较差的药代动力学性质等,尚未能成功开发为药物上市销售.
随着各学科的交叉融合及大数据时代的来临,对NDM-1研究而言,根据量子或分子力学等理论,通过计算机模拟NDM-1的水解机理或其与抑制剂分子间的结合模式,并通过数学建模定量探究分子结构与活性之间的关系,为NDM-1抑制剂分子的设计与改造提供理论指导等已取得了一定的成效.随着计算科学的飞速发展,模拟精度的逐步提高,药学科研工作中计算机辅助的应用将会越来越多,相信在不久的将来,基于理论的计算可望成为应对诸如细菌耐药问题等各类医、药学难题时不可缺少的主要工具之一.
[1]Wilke M S,Lovering A L,Strynadka N C.Beta-lactamantibioticresistance:acurrentstructuralperspective[J].Curr Opin Microbiol,2005,8(5):525-533.
[2]Rawat D,Nair D.Extended-spectrumβ-lactamasesingramnegativebacteria[J].J Glob Infect Dis,2010,2(3):263-274.
[3]World Health Organization.Antimicrobialresistance:globalreportonsurveillance2014[R].Geneva,Switzerland:World Health Organization,2014.
[4]Protic D,Pejovic A,Andjelkovic D,et al.Nosocomialinfectionscausedbyacinetobacterbaumannii:arewelosingthebattle?[J].Surg Infect,2016,17(2):1-7.
[5]Stoesser N,Mathers A J,Moore C E,et al.Colistinresistancegenemcr-1andpHNSHP45plasmidinhumanisolatesofescherichiacoliandklebsiellapneumoniae[J].Lancet Infect Dis,2016,16(3):285-286.
[6]Labarca J A,Salles M J C,Seas C,et al.Carbapenemresistanceinpseudomonasaeruginosaandacinetobacterbaumanniiinthenosocomialsettinginlatinamerica[J].Crit Rev Microbiol,2016,42(2):276-292.
[7]Bush K,Jacoby G A.Updatedfunctionalclassificationofbeta-lactamase[J].Antimicrob Agents Chemother,2010,54(3):969-976.
[8]Yong D,Toleman M A,Giske C G,et al.Characterizationofanewmetallo-beta-lactamasegene,bla(NDM-1),andanovelerythromycinesterasegenecarriedonauniquegeneticstructureinKlebsiellapneumoniaesequencetype14fromIndia[J].Antimicrob Agents Chemother,2009,53(12):5046-5054.
[9]Moellering R C J.NDM-1—acauseforworldwideconcern[J].New Engl J Med,2010,363(25):2377-2379.
[10]Groundwater P W,Xu S,Lai F,et al.Newdelhimetallo-β-lactamase-1:structure,inhibitorsanddetectionofproducers[J].Future Med Chem,2016,8(9):993-1012.
[11]Marrs E C,Day K M,Perry J D.InvitroactivityofmecillinamagainstenterobacteriaceaewithNDM-1carbapenemase[J].J Antimicrob Chemother,2014,69(10):2873-2875.
[12]Denis C,Poirel L,Carricajo A,et al.NosocomialtransmissionofNDM-1-producingescherichiacoliwithinanon-endemicareainFrance[J].Clin Microbiol Infec,2012,18(5):E128-E130.
[13]Walsh T R,Weeks J,Livermore D M,et al.DisseminationofNDM-1positivebacteriainthenewdelhienvironmentanditsimplicationsforhumanhealth:anenvironmentalpointprevalencestudy[J].Lancet Infect Dis,2011,11(5):355-362.
[14]Berrazeg M,Diene S,Medjahed L,et al.Newdelhimetallo-beta-lactamasearoundtheworld:aneReviewusinggooglemaps[J].Euro Surveil,2014,19(20):2-15.
[15]Girlich D,Poirel L,Nordmann P.Valueofthemodifiedhodgetestfordetectionofemergingcarbapenemasesinenterobacteriaceae[J].J Clin Microbiol,2012,50(2):477-479.
[16]Abidin N Z Z,Sulong A,Alfizah H,et al.Screeningfornewdelhimetallo-β-lactamase-1inenterobacteriaceae:istherearoleforthemodifiedhodgetest?[J].Pak J Med Sci,2015,31(6):1340-1343.
[17]Perry J D,Naqvi S H,Mirza I A,et al.PrevalenceoffaecalcarriageofenterobacteriaceaewithNDM-1carbapenemaseatmilitaryhospitalsinpakistan,andevaluationoftwochromogenicmedia[J].J Antimicrob Chemother,2011,66(10):2288-2294.
[18]Nordmann P,Poirel L,Walsh T R,et al.TheemergingNDMcarbapenemases[J].Trends Microbiol,2011,19(12):588-595.
[19]Nordmann P,Poirel L,Carrr A,et al.Howtodetectndm-1producers[J].J Clin Microbiol,2011,49(2):718-721.
[20]Huang L,Hu X,Zhou M,et al.RapiddetectionofNewdelhimetallo-β-lactamasegeneandvariantscodingforcarbapenemaseswithdifferentactivitiesbyuseofaPCR-basedinvitroproteinexpressionmethod[J].J Clin Microbiol,2014,52(6):1947-1953.
[21]Hrabák J,Studentová V,Walková R,et al.DetectionofNDM-1,VIM-1,KPC,OXA-48,andOXA-162carbapenemasesbymatrix-assistedlaserdesorptionionization-timeofflightmassspectrometry[J].J Clin Microbiol,2012,50(7):2441-2443.
[22]Hrabák J,Chudácková E,Walková R.Matrix-assistedlaserdesorptionionization-timeofflight(maldi-tof)massspectrometryfordetectionofantibioticresistancemechanisms:fromresearchtoroutinediagnosis[J].Clin Microbiol Rev,2013,26(1):103-114.
[23]Wilkowski P,Ciszek M,Dobrzaniecka K,et al.SuccessfultreatmentofurinarytractinfectioninkidneytransplantrecipientscausedbymultiresistantklebsiellapneumoniaeproducingnewDelhimetallo-beta-lactamase(NDM-1)withstrainsgenotyping[J].Transplant Proc,2016,48(5):1576-1579.
[24]Klingler F,Wichelhaus T A,Frank D,et al.Approveddrugscontainingthiolsasinhibitorsofmetallo-β-lactamases:strategytocombatmultidrug-resistantbacteria[J].J Med Chem,2015,58(8):3626-3630.
[25]Feng H,Ding J,Zhu D,et al.StructuralandmechanisticinsightsintoNDM-1catalyzedhydrolysisofcephalosporins[J].J Am Chem Soc,2014,136(42):14694-14697.
[26]King D T,Worrall L J,Gruninger R,et al.NewDelhimetallo-β-lactamase:structuralinsightsintoβ-lactamrecognitionandinhibition[J].J Am Chem Soc,2012,134(28):11362-11365.
[27]Zhang H,Hao Q.CrystalstructureofNDM-1revealsacommonβ-lactamhydrolysismechanism[J].FASEB J,2011,25(8):2574-2582.
[28]King D,Strynadka N.Crystalstructureofnewdelhimetallo-β-lactamaserevealsmolecularbasisforantibioticresistance[J].Protein Sci,2011,20(9):1484-1491.
[29]Kim Y,Tesar C,Mire J,et al.Structureofapo-andmonometalatedformsofNDM-1—ahighlypotentcarbapenem-hydrolyzingmetallo-β-lactamase[J].Plos One,2011,6(9):e24621.
[30]Green V L,Verma A,Owens R J,et al.Structureofnewdelhimetallo-β-lactamase1(NDM-1)[J].Acta Crystallogr Sect F Struct Biol Cryst Commun,2011,67(10):1160-1164.
[31]Yu G,Jing W,Niu G,et al.AstructuralviewoftheantibioticdegradationenzymeNDM-1fromasuperbug[J].Protein Cell,2011,2(5):384-394.
[32]Aitha M,Moller A J,Sahu I D,et al.InvestigatingthepositionofthehairpinloopinNewDelhimetallo-β-lactamase,NDM-1,duringcatalysisandinhibitorbinding[J].J Inorg Biochem,2015,156(7):35-39.
[33]Rehman M T,Khan A U.Roleofnon-activesiteresidueTrp-93inthefunctionandstabilityofNewDelhiMetallo-β-Lactamase-1(NDM-1)[J].Antimicrob Agents Chemother,2015,60(1):356-360.
[34]Marcoccia F,Bottoni C,Sabatini A,et al.KineticstudyoflaboratorymutantsofNDM-1metallo-β-lactamase:theimportanceofanisoleucineatposition35[J].Antimicrob Agents Chemother,2016,60(4):2366-2372.
[35]Kim Y,Cunningham M A,Mire J,et al.NDM-1,theultimatepromiscuousenzyme:substraterecognitionandcatalyticmechanism[J].FASEB J,2013,27(5):1917-1927.
[36]Wang Z.Metallo-beta-lactamase:structureandmechanism[J].Curr Opin Chem Biol,1999,3(5):614-622.
[37]Yang H,Aitha M,Marts A R,et al.Spectroscopicandmechanisticstudiesofheterodimetallicformsofmetallo-β-lactamaseNDM-1[J].J Am Chem Soc,2014,136(20):7273-7285.
[38]Yuan Q,He L,Ke H.Apotentialsubstratebindingconformationofβ-lactamsandinsightintothebroadspectrumofNDM-1activity[J].Antimicrob Agents Chemother,2012,56(10):5157-5163.
[39]Chiou J,Leung T Y,Chen S.MolecularmechanismsofsubstraterecognitionandspecificityofNewDelhimetallo-β-lactamase[J].Antimicrob Agents Chemother,2014,58(9):5372-5378.
[40]Zhu K,Lu J,Ye F,et al.Structure-basedcomputationalstudyofthehydrolysisofNewDelhimetallo-β-lactmase-1[J].Biochem Biophys Res Commun,2013,431(1):2-7.
[41]Liang Z,Li L,Wang Y,et al.MolecularBasisofNDM-1,anewantibioticresistancedeterminant[J].PloS One,2010,6(8):e23606.
[42]Zheng M,Xu D.NewDelhimetallo-β-lactamaseI:substratebindingandcatalyticmechanism[J].J Phys Chem B,2013,117(39):11596-11607.
[43]Wang Z,Walter Fast A,Benkovic S J.Onthemechanismofthemetallo-β-lactamasefromBacteroidesfragilisn[J].Biochemistry,1999,38(31):10013-10023.
[44]Yang H,Aitha M,Hetrick A M,et al.Mechanisticandspectroscopicstudiesofmetallo-β-lactamaseNDM-1[J].Biochemistry,2012,51(18):3839-3847.
[45]Zhu K,Lu J,Liang Z,et al.Aquantummechanics/molecularmechanicsstudyonthehydrolysismechanismofNewDelhimetallo-β-lactamase-1[J].J Comput Aid Mol Des,2013,27(3):247-256.
[46]Li N,Xu Y,Xia Q,et al.SimplifiedcaptoprilanaloguesasNDM-1inhibitors[J].Bioorg Med Chem Lett,2014,24(1):386-389.
[47]Shen B,Yu Y,Chen H,et al.Inhibitordiscoveryoffull-lengthnewdelhimetallo-β-lactamase-1(NDM-1)[J].PloS One,2013,8(5):e62955.
[48]Wang X,Lu M,Shi Y,et al.DiscoveryofnovelNewDelhimetallo-β-lactamases-1inhibitorsbymultistepvirtualscreening[J].Plos One,2015,10(3):e0118290.
[49]Rydzik A M,Brem J,van Berkel S S,et al.MonitoringconformationalchangesintheNDM-1metallo-beta-lactamaseby19FNMRspectroscopy[J].Angew Chem Int Edit,2014,53(12):3129-3133.
[50]Klingler F M,Moser D,Büttner D,et al.Probingmetallo-β-lactamaseswithmolecularfragmentsidentifiedbyconsensusdocking[J].Bioorg Med Chem Lett,2015,25(22):5243-5246.
[51]Gan M,Liu Y,Bai Y,et al.Polyketideswithnewdelhimetallo-β-lactamase1inhibitoryactivityfromPenicilliumsp[J].J Nat Prod,2013,76(9):1535-1540.
[52]Christopeit T,Albert A,Leiros H K.Discoveryofanovelcovalentnon-β-lactaminhibitorofthemetallo-β-lactamaseNDM-1[J].Bioorg Med Chem,2016,24(13):2947-2953.
[53]Thomas P W,Spicer T,Cammarata M,et al.Analteredzinc-bindingsiteconfersresistancetoacovalentinactivatorofNewDelhimetallo-beta-lactamase-1(NDM-1)discoveredbyhigh-throughputscreening[J].Bioorg Med Chem,2013,21(11):3138-3146.
[54]Thomas P W,Cammarata M,Brodbelt J S,et al.Covalentinhibitionofnewdelhimetallo-β-lactamase-1(NDM-1)bycefaclor[J].ChemBioChem,2014,15(17):2541-2548.
[55]Chiou J,Wan S,Chan K F,et al.Ebselenasapotentcovalentinhibitorofnewdelhimetallo-β-lactamase(NDM-1)[J].Chem Commun,2015,51(46):9543-9546.
[56]Ma J,McLeod S,MacCormack K,et al.Real-timemonitoringofnewdelhimetallo-β-lactamaseactivityinlivingbacterialcellsby1HNMRspectroscopy[J].Angew Chem Int Edit,2011,126(8):2162-2165.
[57]Azumah R,Dutta J,Somboro A M,et al.Invitroevaluationofmetalchelatorsaspotentialmetallo-β-lactamaseinhibitors[J].J Appl Microbiol,2016,120(4):860-867.
[58]Falconer S B,Reid-Yu S A,King A M,et al.Zincchelationbyasmall-moleculeadjuvantpotentiatesmeropenemactivityinvivoagainstNDM-1-producingklebsiellapneumoniae[J].Acs Infect Dis,2015,1(11):533-543.
[59]Mikami Y,Suzuki T.Novelmicrobialinhibitorsofangiotensin-convertingenzyme,aspergillomarasminesAandB[J].Agric Biol Chem,1983,47(11):2693-2695.
[60]Arai K,Ashikawa N,Nakakita Y,et al.AspergillomarasmineAandB,potentmicrobialinhibitorsofendothelin-convertingenzyme[J].Biosci Biotechnol Biochem,2014,57(11):1944-1945.
[61]Matsuura A,Okumura H,Asakura R,et al.Pharmacologicalprofilesofaspergillomarasminesasendothelinconvertingenzymeinhibitors[J].Jpn J Pharmacol,1993,63(2):187-193.
[62]King A M,Reidyu S A,Wang W,et al.AspergillomarasmineAovercomesmetallo-β-lactamaseantibioticresistance[J].Nature,2014,510(7506):503-506.
[63]Christopeit T,Leiros H S.Fragment-baseddiscoveryofinhibitorscaffoldstargetingthemetallo-β-lactamasesNDM-1andVIM-2[J].Bioorg Med Chem Lett,2016,26(8):1973-1977.
Advance Research on NDM-1:Structure,Catalytic Mechanism and Inhibitors
HUJianping1,2,ZUOKe1,2,WANHua3
(1.School of Pharmacy and Bioengineering,Chengdu University, Chengdu 610106, China;2.Sichuan Industrial Institute of Antibiotics,Chengdu University, Chengdu 610052, China;3.College of Mathematics and Informatics, South China Agricultural University, Guangzhou 510642, China)
As the first-line antibiotics,β-lactam antibiotics are widely used in the clinical treatment of various infections.New Delhi metallo-β-lactamase-1(NDM-1) encoded by gene blaNDM-1in plasmid DNA can hydrolyze nearly all kinds of β-lactam antibiotics,posing a critical threat to the application of β-lactamase.It is of great urgency to design novel NDM-1 inhibitors.The design and modification of inhibitor molecules are closely related to the research of both structural information and catalytic mechanism of NDM-1.In this review,structure,catalytic mechanism and inhibitors of NDM-1 all are summarized,which will be helpful for the design and modification of drug molecules targeting NDM-1.
antimicrobial resistance;NDM-1;catalytic mechanism;inhibitor;drug design
1004-5422(2017)02-0115-10
2017-03-02.
国家自然科学基金(31600591、 11247018)资助项目.
胡建平(1978 — ), 男, 博士, 教授, 从事计算机辅助药物设计研究.
Q55;R915
A
