基于NRPS基因筛选鉴定产生环脂肽葡萄附生细菌的研究
2017-07-05,,,,
, , ,,
(南京农业大学食品科技学院,江苏南京 210095)
基于NRPS基因筛选鉴定产生环脂肽葡萄附生细菌的研究
王傲,杨柯,吕曼,史雅凝,辛志宏*
(南京农业大学食品科技学院,江苏南京 210095)
本研究采用平板稀释法和斜面分离纯化技术,从夏黑葡萄中筛选分离到5株附生细菌,提取菌株的基因组DNA,PCR扩增16S rDNA序列,产物纯化后进行克隆测序,利用MEGA 6.0软件对序列进行同源性比对分析,构建系统进化树后,将5株附生细菌分别鉴定为克雷伯肺炎杆菌(Klebsiellapneumoniae)、枯草芽孢杆菌(Bacillussubtilis)、克考氏菌(Kocuriamarina)、嗜气芽孢杆菌(Bacillusaerophilus)、类芽孢杆菌(Paenibacillusagaridevorans)。在此基础上,以非核糖体多肽合成酶基因(non-ribosomal peptide compounds synthetase,NRPS)为靶点,筛选到含有目的条带的菌株PTWA2,经系统发育树预测该菌株能够产生环脂肽类化合物。该菌株发酵产物经有机溶剂萃取、常压硅胶柱层析、Sephedex LH-20等分离手段纯化得到一个单体化合物,采用核磁共振和质谱方法鉴定该化合物为环脂肽Surfactin A。该研究结果为以功能基因定向筛选产生环脂肽化合物的菌株提供了新的研究方法与理论依据。
附生菌,16S rDNA,序列分析,非核糖体多肽合成酶,系统发育树,结构鉴定
植物附生菌(epiphyte)是一类附着在植物表面、以植物分泌物为营养的微生物类群[1]。大量研究表明,多数植物附生菌能够产生结构新颖、活性独特的次级代谢产物,从中分离到的多种物质也被证实具有各种各样的生物活性[2-3]。如Shigemori[4]从海绵附生菌中分离到的大环内酰胺类化合物Alteramide A被证明具有显著的抗肿瘤细胞活性。2002年,Hornschuh[5]等从德国卡塞尔郡的森林Funaria树叶表面分离出一株附生菌(Methylobacteriummesophilicum),能产生特定的激素从而促进植物生长发育。张学君[6]研究发现,苹果附生菌中存在着多种对苹果常见病症——轮纹病和炭疽病起拮抗作用的微生物。植物附生菌已成为寻找和发现各种生物活性物质的重要资源,特别是植物附生细菌,因其易于培养、生长速率快、能产生结构多样的次级代谢产物,已经成为附生微生物的研究热点。
夏黑葡萄[7]原产日本,属欧美杂种三倍体品种,由日本山梨县果树实验场用巨峰与无核白杂交选育所得,1997年获得品种登记。夏黑葡萄果粒着生紧密,果穗大小整齐,果皮紫黑色,果实酸甜适口,被誉为“东方黑珍珠”。研究表明,在表皮完整且温度适宜的情况下,夏黑葡萄的贮藏期可达到60 d[8]。除了葡萄皮自身的保护屏障,也取决于表面的附生微生物。这些附生微生物利用葡萄表面的营养物质进行繁殖,可产生小分子量的抗菌物质、抗菌蛋白或多肽等活性物质抑制了病原微生物的生长。因此,开展葡萄附生菌的筛选及其代谢产物的研究,对于阐明葡萄的生物防控机制具有重要意义。
在附生微生物的研究过程中,通常采用的方法以活性筛选、化学筛选或者二者结合为主。但是,这些方法的主要缺点是无法预测微生物次级代谢产物的类型,难以实现定向筛选[9]。近年来,随着分子生物信息学的发展,许多微生物次级代谢产物的全基因组序列与生物合成机制得到解析,通过功能基因保守功能域设计引物,目标直接指向合成多肽类化合物的目标基因,为研究和开发现代天然产物提供了新的战略依据。非核糖体多肽合成酶(nonribosomal peptide synthetases,NRPSs),是催化合成多肽类物质(nonribosomal peptide,NRP)的关键酶,由多模块组成,不同的模块按照起始、延伸和终止的空间顺序排列而成,每个模块均有腺苷酰化结构域(A结构域),肽酰载体蛋白结构域(T结构域)和缩合结构域(C结构域)3个核心结构域[10]。A结构域负责氨基酸腺苷化,它在每一个反应循环的第一步从底物池中选择特异性的氨基酸,在ATP的作用下,使氨基酸活化成氨酰-AMP,被誉为特异性的“守门者”,是NRPS的核心保守模件[11]。因此可以通过A结构域设计引物进行PCR筛选,产物测序后利用系统发育分析预测化合物的结构类型。
本研究以夏黑葡萄为研究材料,采用稀释梯度法和平板划线分离技术筛选葡萄附生细菌,通过分子生物学方法对其进行分类鉴定。在此基础上,以NRPS功能基因为靶点,筛选含有NRPS基因的菌株,利用常压硅胶柱层析、Sephedex LH-20等技术分离纯化得到单体化合物,通过核磁共振和质谱方法鉴定化合物结构,以期为定向筛选有价值的环脂肽化合物并阐明葡萄的生物防控机制提供参考。
1 材料与方法
1.1材料与仪器
夏黑葡萄 采自江苏省南京市玄武区童卫路,装入塑料保鲜袋,置于4 ℃冰箱保存;Taq酶、DNA Marker及PCR相关试剂 南京诺唯赞生物科技有限公司;Omega细菌基因组试剂盒(Bacteria DNA Kit 50)及引物(16SF/16SR) 上海捷瑞生物工程有限公司;Ver.3.0 D823A琼脂糖凝胶DNA提取试剂盒、D102A pMD19-T载体 日本TaKaRa公司;其他生理生化试剂 南京寿德试剂器材有限公司;LB液体培养基(g/L) 胰蛋白胨10、酵母粉5、NaCl 10,琼脂20,自来水配制,pH7.2。
SW-CJ-1FD无菌工作台 苏州净化集团设备有限公司;Microfuge 22R台式微量冷冻离心机 美国Beckman公司;TP600型梯度PCR仪 日本TaKaRa公司;DYCP-31DN电泳仪 北京市六一仪器厂;JS-380C全自动数码凝胶成像分析仪 上海培清科技有限公司;JNM-ECP600型核磁共振仪 日本JEOL公司;Mariner API-TOF型质谱仪 美国应用生物系统公司;EYELAN-N型立式旋转蒸发仪 日本Tokyo Rikakikai有限公司;ULVAC DTC-22B型隔膜真空泵 上海万库真空设备有限公司。
1.2实验方法
1.2.1 细菌的分离培养 取数颗葡萄置于无菌研钵中研磨出汁液,得到葡萄原液。取1 mL原液置于9 mL无菌水中,制成10-1的葡萄稀释液,同理,依次制备10-2、10-3、10-4、10-5、10-6葡萄稀释液。用无菌吸管吸取0.2 mL,分别涂布于LB培养基上,用无菌玻璃棒涂布均匀,将培养皿在37 ℃恒温培养箱中倒置培养1~2 d,挑取形态有差异的单菌落,转移到斜面试管中,4 ℃保存。
1.2.2 基因组DNA的提取 从保藏菌种的斜面培养基中挑取单菌落转接到LB平板上,37 ℃培养24 h。基因组DNA的提取采用细菌基因组试剂盒(Bacteria DNA Kit 50),按照说明书操作。提取的基因组DNA在1%琼脂糖凝胶中电泳检测(120 V,30 min),溴化乙锭(EB)染色。4 ℃保存备用,或于-20 ℃中长期保存。
1.2.3 16S rDNA以及NRPS基因的PCR扩增 16S区域的扩增选择原核生物16S rDNA的通用扩增引物16S-F(5′-AGAGTTTGATCCTGGCTCAG-3′)/16S-R(5′-ACGGTTACCTTGTTACGACTT-3′),PCR反应条件为:94 ℃预变性3 min,94 ℃变性30 s,50 ℃退火45 min,72 ℃延伸100 s,共35个循环,最后72 ℃延伸7 min。NRPS功能基因筛选引物A3F(5′-GCSTACSYSATSTACACSTCSGG-3′)/A7R(5-SASGTCVCCSGTSCGGTAS-3′),PCR反应条件为:95 ℃预变性5 min,95 ℃变性1 min,59 ℃退火2 min,72 ℃延伸4 min,共35个循环,最后72 ℃延伸10 min[12-13]。PCR扩增反应均采用25 μL的反应体系,包括ddH2O 9.5 μL、10 μmol/L 16S-F 1 μL、10 μmol/L 16S-R 1 μL、DNA 1 μL、5 U/μL Taq聚合酶Mix 12.5 μL。PCR扩增产物经1%琼脂糖凝胶电泳检测,4 ℃保存备用。
1.2.4 PCR产物的回收和克隆 采用琼脂糖凝胶DNA提取试剂盒回收PCR产物,纯化的PCR产物与pMD19-T载体连接,然后转化至E.coliDH5α感受态细胞中。在-70 ℃保存的100 μL感受态细胞E.coliDH5α中加入10 μL连接产物,冰中放置30 min,取出后42 ℃热激90 s,加入890 μL LB液体培养基37 ℃振荡培养1 h。菌液涂布于含100 μg/mL氨苄青霉素AMP的LB培养基平板,倒置过夜培养,挑取白色单菌落培养。取1 μL菌液直接做PCR,引物为M13RV和M13-47,电泳检测是否含有目的片段。PCR采用25 μL的反应体系,包括ddH2O 9.5 μL、10 μmol/L Primer-F 1μL、10 μmol/L Primer-R 1 μL、DNA 1 μL、5 U/μL Taq聚合酶Mix 12.5 μL。
1.2.5 DNA序列测序和系统发育学分析 将含有目的DNA序列的菌液交由上海美吉生物有限公司进行测序。获得序列后,利用DNAMAN进行序列拼接,在 EzBioCloud(http://eztaxon-e.ezbiocloud.net/)中进行EzTaxon比对,下载与供试菌株序列同源性相近的模式菌株序列,利用MEGA 6.0软件Neighbor-Joining(N-J)方法构建系统发育树,自展数为1000。

表1 5株葡萄附生细菌鉴定结果Table 1 The results of five bacteria from grapes
1.2.6 菌种发酵 在无菌条件下,用接种环从保存的固体斜面培养基中刮取菌体,接种于内装15 mL无菌水的小三角瓶内,振摇均匀形成孢子液,按1%接种量(1 mL)取孢子液接种于100 mL液体培养基的三角瓶中,在37 ℃、120 r/min的条件下摇床培养48 h,作为种子培养液。将种子培养液接种至15瓶含300 mL液体发酵培养基的1000 mL三角瓶中,接种量2.5%,发酵14 d。
1.2.7 化合物的分离与纯化 发酵液用纱布过滤后得到发酵液和菌丝体,发酵液减压浓缩至1 L,加入等体积丙酮,萃取3次,用乙酸乙酯萃取丙酮中的脂溶性物质,减压浓缩至干,得到粗提物。将粗提物(9 g)上正向减压柱硅胶柱纯化,经石油醚/丙酮/甲醇(100∶0/0∶100/100∶0,v/v/v)梯度洗脱,得到组分(Fraction,简写Fr):Fr-1~Fr-5,经抑菌实验发现仅有组分Fr-2对金色葡萄球菌具有抑制作用,该组分进一步经凝胶层析Sephadex LH-20纯化,用氯仿∶甲醇(1∶1,v/v)洗脱,得到一单体化合物A(10 mg)。
1.2.8 化合物A的波普学分析 分离纯化的样品过滤后进样分析,质谱参数:电喷雾离子源(ESI),500 ℃,气帘气25.0 psi,喷雾电压5500.0 V,离子源气1:55.0 psi,离子源气2:50.0 psi;结合核磁(1H、13C、1H-1H COSY、HMBC等)分析手段,最终确定化合物A的结构。
2 结果与分析
2.1 16S rDNA扩增及序列测定
采用平板分离与斜面纯化相结合的方法共从夏黑葡萄中分离得到5株附生细菌,菌株经DNA提取,通过PCR扩增16S rDNA基因片段,构建系统发育树进行鉴定。16S rDNA扩增电泳结果如图1所示,5株菌的条带清晰单一,大小约1500 bp,与理论值一致[12]。

图1 5株菌16S rDNA序列PCR扩增结果Fig.1 PCR results of 16S rDNA sequences from the five strains注:M:DNA Marker DL2000;1~5:代表5株葡萄附生细菌PTWA1~PTWA5。
以菌株PTWA2 16S rDNA为例,说明附生菌的鉴定过程。PTWA2 16S rDNA经Blast比对,发现与枯草芽孢杆菌(Bacillussubtilissubsp. inaquosorum KCTC 13429)16S rDNA序列的同源性最高,相似性达98.63%。进一步利用MEGA6.0软件通过邻位连接法构建16S序列系统发育树(如图2所示),结果显示,与所选取的外组菌株短芽孢杆菌(Brevibacillusbrevis)相比,所有菌株聚为一个大组,不同菌种分别聚在不同的分枝上,菌株PTWA2与枯草芽孢杆菌杆菌(B.subtilis)处于同一分枝,自展值为98,亲缘关系最近,鉴定PTWA2为枯草芽孢杆菌(B.subtilis)。据此方法,将5株葡萄附生细菌PTWA1~PTWA5分别被鉴定为K.pneumoniae、B.subtilis、K.marina、B.aerophilus、P.agaridevorans。结果如表1所示。
2.2 NRPS基因筛选结果及系统发育分析
对分离到的5株葡萄附生细菌进行NRPS基因扩增只有菌株PTWA2扩增结果为阳性,得到大小约750 bp的目的条带(图3),与理论值一致[14]。对NRPS基因克隆测序后,经orf finder在线转化为氨基酸序列后进行BLASTp搜索,下载同源蛋白序列,采用MEGA6.0软件,用邻接法(Neighbor-Joining)构建NRPS进化树,结果如图4所示。从图4中可见,能够产生相同化学类型化合物的细菌分别聚为一枝,说明同种细菌的不同菌株或不同细菌能够产生相同类型的化合物,如不同的大肠杆菌能够产生Enterobactin,而贝莱斯芽孢和枯草芽孢杆菌都能产生Plipastatin。PTWA2蛋白序列与能合成Surfactin的菌株聚为一类,提示该菌株可能产生环脂肽Surfactin。

图2 基于菌株PTWA2的16S rDNA区序列构建的系统发育树Fig.2 Phylogenetic tree of strain PTWA2 based on 16S rDNA gene sequence

图3 5株葡萄附生细菌NRPS序列扩增结果Fig.3 Amplification result of five strains based on NRPS genes注:M.DNA Marker DL2000;1~5:代表5株葡萄附生细菌PTWA1~PTWA5。

图5 化合物A的质谱图Fig.5 ESI-MS spectrum of compound A
2.3化合物A的结构解析

图4 菌株PTWA2基于NRPS构建的系统发育树Fig.4 Phylogenetic tree of strain PTWA2 based on NRPS functional gene
化合物A:白色针状晶体,阴离子质谱HR-ESI-MS在m/z 1034.1[M-H]-处出现准离子峰(图5A),阳离子质谱HR-ESI-MS在m/z 1058.7[M+Na]+处出现准离子峰(图5B),表明该化合物的分子质量为1035,结合1H-NMR、13C-NMR波普数据(表2),分析该化合物分子式为C53H93N7O13。1H-NMR(600 MHz C3D6O)谱显示7个-NH信号分别为δ 7.96(s,1H,H-1)、7.67(s,1H,H-7)、7.44(s,1H,H-14)、7.68(s,1H,H-21)、7.73(s,1H,H-27)、7.96(s,1H,H-32)、7.70(s,1H,H-39),进一步分析发现在δ 4.02(d,J=5.82 Hz,1H,H-2)、4.38(t,J=7.32 Hz,1H,H-8)、4.47(t,J=8.94 Hz,1H,H-15)、4.24(d,J=5.70 Hz,1H,H-22)、4.77(t,J=4.86 Hz,1H,H-28)、4.53(t,J=8.1 Hz,1H,H-33)、4.40(t,J=6.18 Hz,1H,H-40)出现7个特征的氨基酸α-H信号,提示该化合物可能为环七肽。高场区δ 1.84~1.36出现一组密集信号,推测该化合物中可能含有脂肪酸链。13C-NMR(150 MHz,C3D6O)谱显示7个羰基碳信号分别为δ 175.0(C-31)、174.3(C-20)、174.3(C-26)、174.0(C-45)、172.9(C-38)、171.7(C-13)、171.6(C-6)、172.8(C-5)、172.2(C-30)、171.1(C-1′),同时,δ C 61.6(C-2)、58.0(C-22)、53.0(C-40)、52.9(C-8)、52.6(C-33)、52.5(C-15)、50.8(C-28)出现7个特征的氨基酸α-C信号;进一步证实该化合物为环脂肽。经文献数据比对[15-17],表明该化合物是由谷氨酸(Glu)-亮氨酸(Leu)-亮氨酸(Leu)-缬氨酸(Val)-天冬氨酸(Asp)-亮氨酸(Leu)-亮氨酸(Leu)组成的环脂肽,确定该化合物的结构为Surfactin。结构式如图6所示。

表2 化合物A的NMR数据Table 2 NMR data of compound A
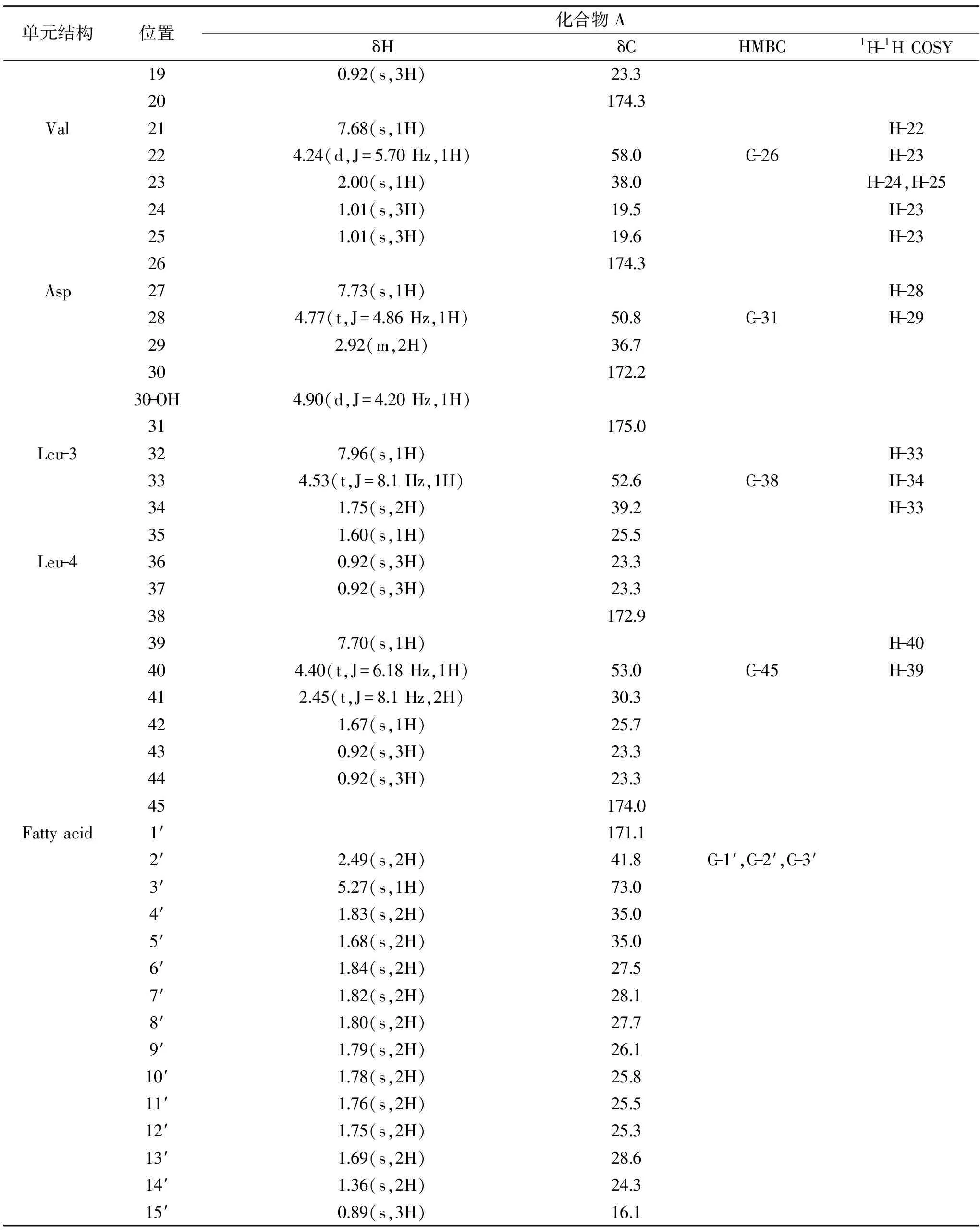
续表

图6 化合物A的结构Fig.6 The structure of compound A
Surfactin是一类由非核糖体途径(NPRS)产生的小分子量脂肽类化合物,具有低临界胶束浓度(CMC)、高生物降解性、抗病毒和抗细菌性等作用[18]。因为选择性广、对环境影响小,Surfactin在食品工业、医药以及农业生物防治方面有着广阔的应用潜力,是一种新型的对环境友好的绿色表面活性剂。近年来,人们一直采用血平板法和扩油圈法筛选代谢Surfactin的菌株[19],但这样不仅工作效率低下,且不能有效将代谢脂肽的微生物从中区分开。本研究以NRPS基因为靶点,从葡萄表皮筛选到一株产环脂肽化合物的菌株,说明该方法能够实现脂肽类化合物从基因型到化学型的定向筛选,为实现产环脂肽化合物菌株的快速筛选提供了新的理论依据和研究方法。
3 结论
本实验运用16S rDNA序列分析技术对夏黑葡萄附生菌进行分类鉴定,5株附生细菌分别鉴定为Klebsiellapneumoniae、Bacillussubtilis、Kocuriamarina、Bacillusaerophilus、Paenibacillusagaridevorans。在此基础上,以NRPS基因为靶点,从5株细菌中筛选到一株含有NPRS基因的菌株,编号为PTWA2。通过NRPS序列系统发育分析,提示该菌株可产生环脂肽化合物Surfactin。进一步发酵该菌株,从发酵产物中分离纯化得到一个单体化合物,经ESI-MS、1H-NMR、13C-NMR等综合分析,确定其化学结构为环脂肽C15-Surfactin A。该研究结果说明以NRPS基因为靶点,能够实现脂肽类化合物从基因型到化学型的定向筛选,为以功能基因为靶点,定向筛选产生环脂肽化合物的菌株提供了新的研究方法与理论依据。
[1]林文翰. 红树附生微生物的化学成分研究[C]. 国家“863”计划资源环境技术领域海洋生物高技术论坛,2005.
[2]Andrews JH,Harris RF. The ecology and biogeography of microorganisms on plant surfaces[J]. Annual Review of Phytopathology,2000,38(1):145-180.
[3]Cueto M,Jensen PR,Kauffman C,et al. Pestalone,a new antibiotic produced by a marine fungus in response to bacterial challenge[J]. Journal of Natural Products,2001,64(11):1444
-1446.
[4]Shigemori H,Bae M A,Yazawa K,et al. Alteramide A,a new tetracyclic alkaloid from a bacteriumAlteromonassp. associated with the marine spongeHalichondriaokadai[J]. J Org Chem,1992,57(15):4317-4320.
[5]Hornschuh M,Grotha R,Kutschera U. Epiphytic Bacteria Associated with the BryophyteFunariahygrometrica:Effects of Methylobacterium,Strains on Protonema Development[J]. Plant Biology,2002,4(4):682-687.
[6]张学君,徐盈. 苹果表面微生物数量及其与两种主要病菌的关系[J]. 果树学报,1995(4):232-236.
[7]单洪友. 夏黑葡萄特征特性及栽培技术[J]. 农技服务,2007,24(9):95.
[8]施南芳,楼宇涛,吾建祥. 不同品种葡萄贮藏特性研究[J]. 农技服务,2014(2):119.
[9]张俊楠,彭洁,刘天行,等. 基于PKSⅠ基因海芦笋内生真菌及其次级代谢产物筛选鉴定[J]. 食品科学,2016,37(1):114-119.
[10]Bruner S D,Weber T,Kohli R M,et al. Structural basis for the cyclization of the lipopeptide antibiotic surfactin by the thioesterase domain SrfTE[J]. Structure,2002,10(3):301-310.
[11]王世媛. 非核糖体肽合成酶(NRPSs)作用机理与应用的研究进展[J]. 微生物学报,2007,47(4):734-737.
[12]W G Weisburg,S M Barns,D A Pelletier,et al. 16S ribosomal DNA amplification for phylogenetic study[J]. Journal of Bacteriology,1991,173(2):697-703.
[13]Gontang E A,Gaudêncio S P,Fenical W,et al. Sequence-based analysis of secondary-metabolite biosynthesis in marine actinobacteria[J]. Applied & Environmental Microbiology,2010,76(8):2487-2499.
[14]唐晓敏. 红树林土壤微生物卤代酶基因和宏基因组非核糖体肽合酶基因的克隆和表达[D].厦门:厦门大学,2008.
[15]Liu X-Y,Yang S-Z,Mu B-Z. Production and characterization of a C15-surfactin-O-methyl ester by a lipopeptide producing strainBacillussubtilisHSO121[J]. Process Biochemistry,2009,44(10):1144-1151.
[16]Hideji Itokawa,Toshiko Miyashita,Hiroshi Moita,et al. Structure and Conformational Studies of[Ile7]and[Leu7]Surfactins fromBacillussubtilisnatto[J]. Chem PharmBull,1994,42(3):604-607.
[17]刘丽霞. Surfactin合成酶相关基因研究[D]. 南京:南京农业大学,2012.
[18]Thimon L PF,Maget Dana R,Roux B G M. Interactions of bioactive lipopeptides,iturin A and surfaction fromBacillussubtilis[J]. Biotechnol Appl Biochem,1992,16(2):144-151.
[19]王大威,刘永建,林忠平,等. 一株产生脂肽的枯草芽孢杆菌的分离鉴定及脂肽对原油的作用[J]. 微生物学报,2008,48(3):304-311.
ScreeningandidentificationofanepiphyticbacteriumproducinglipopeptidesbasedonNRPSgene
WANGAo,YANGKe,LVMan,SHIYa-ning,XINZhi-hong*
(College of Food Science and Technology,Nanjing Agricultural University,Nanjing 210095,China)
In this study,five epiphytic bacteria were isolated from "summer black" grapes using plate streaking and slant culture methods. After extraction of DNA,16S rDNA from the five strains were amplified for sequence homology analysis,and the phylogenetic trees were constructed with MEGA 6.0.The results showed that the five strains were characterized asKlebsiellapneumoniae,Bacillussubtilis,Kocuriamarina,Bacillusaerophilus,Paenibacillusagaridevorans,respectively. On this basis,the strain PTWA2 was screened with the non-ribosomal peptide compounds synthetase(NRPS)gene. After construting the NRPS phylogenetic tree,the results showed that the PTWA2 was capable of producing lipopeptide. As a result,a pure compound was obtained from the fermentation product of the PTWA2 by solvent extraction,silica gel column chromatography and Sephedex LH-20 chromatography and its chemical sturture was identified as surfactin A by physiochemical analysis and spectroscopic techniques. The study highlights new research methods and theoretical basis for target-screening strains which produced cyclic lipopeptide.
epiphytic bacteria;16S rDNA;gene sequence analysis;NRPS;phylegenetic tree;structure identificaiton
2016-12-12
王傲(1991-),女,硕士研究生,研究方向:食品营养与化学,E-mail:2014108056@njau.edu.cn。
*通讯作者:辛志宏(1974-),男,博士,教授,研究方向:食品营养与安全,E-mail:xzhfood@njau.edu.cn。
2016年农产品质量安全风险评估项目(GJFP201601205)。
TS201.2
:A
:1002-0306(2017)12-0164-07
10.13386/j.issn1002-0306.2017.12.030
