基于SSR分子标记的大蒜种质资源遗传多样性研究
2017-06-24孙亚丽史庆华刘世琦孙秀东陈亚霏钱胜艳刘星辰
孙亚丽,史庆华,刘世琦,孙秀东,陈亚霏,钱胜艳,刘星辰
山东农业大学园艺科学与工程学院,作物生物学国家重点实验室,农业部黄淮地区园艺作物生物学与种质创制重点实验室,山东 泰安 271018
基于SSR分子标记的大蒜种质资源遗传多样性研究
孙亚丽,史庆华,刘世琦*,孙秀东,陈亚霏,钱胜艳,刘星辰
山东农业大学园艺科学与工程学院,作物生物学国家重点实验室,农业部黄淮地区园艺作物生物学与种质创制重点实验室,山东 泰安 271018
大蒜为无性繁殖作物,其生物学特征在经历气候变迁、自然选择和人工选择后发生了多方面变异,从而形成了众多地方品种。因此,研究大蒜种质资源的遗传多样性为大蒜的科学分类及品种鉴定、保存、遗传改良提供科学依据。采用SSR(简单重复序列,simple sequence repeats)分子标记技术对55份大蒜种质的基因组DNA进行多态性扩增。用POPGENE version1.32软件计算有效等位基因数Ne、Nei’s遗传相似系数(GS)、遗传距离(GD)、Nei’s遗传多样性指数、Shannon信息指数等指标,并采用NTSYS pc 21-2软件进行种质间的UPGMA(非加权组平均法,un-weighted pair-group method)聚类分析和基于Nei’s遗传距离的组群间聚类分析。结果表明,7对引物共扩增出75条带,多态性条带为73条(多态性比率为97.33%),每对引物平均扩增出10.71个位点和10.43个多态性位点,说明SSR分子标记揭示的多态性强。55份大蒜种质的平均Nei’s遗传多样性指数和平均Shannon信息指数分别为0.1335和0.2278。大蒜具有较高的遗传多样性,聚为一类的大蒜多来源于相同或相近区域。
大蒜;SSR;遗传多样性
大蒜(Allium sativum L.)为百合科葱属两年生草本植物,原产于亚洲西部的高原地带。我国大蒜种质资源丰富,有世界上最大的栽培面积,约占全球大蒜种植面积的85%以上,总生产量以及出口量均居世界第一[1,2]。大蒜味道鲜美,营养丰富,据有特殊的香辛气味,能促进消化,增进食欲,具有明显的保健作用和药用价值。
近年来,分子标记技术被广泛应用于种质鉴定与遗传多样性分析[3-6]广泛应用在葱属类植物、大麦、花生、甘蓝、西瓜、苹果、大豆[7-15]等作物的连锁图构建或亲缘关系分析上。简单序列重复(Simple sequence repeats,SSR)又称微卫星(Microsatellite),是指以1~6个核苷酸为单位重复DNA序列。SSR标记数量丰富,具有较多的等位变异,且技术重复性好,易于操作,结果可靠[16],使其成为遗传多样性分析中最常用的分子标记技术之一[17,18]。
大蒜虽为无性繁殖作物,但由于地理环境和气候条件差异大,在不同的生态环境下,通过人为定向选择培育和自然淘汰,使大蒜呈现多方面的变异。因此研究大蒜种质资源遗传多样性,掌握其种源及分布规律,发掘优良基因,合理利用现有的大蒜种质资源培育新品种具有重要意义。本研究选用分布范围较大的大蒜资源,采用SSR技术对其进行遗传多样性分析和亲缘关系分析,旨在为大蒜种质资源的收集、保护及利用提供依据。
1 材料与方法
1.1 供试材料
试验于2013~2014年山东农业大学园艺作物生物学实验室进行。供试的55份大蒜品种及来源见表1,收自国内37份种质材料,其中山东26份,河南3份,四川2份,新疆5份,台湾1份,引进国外种质材料18份,包括美国8份,日本2份,泰国2份,加拿大2份,乌克兰2份,俄罗斯1份,欧洲1份。

表1 供试的55个大蒜品种及来源地Table 1 Names and sources of 55 garlic cultivars used in this study
1.2 基因组DNA提取
取植物新鲜组织100 mg或者干粉样品30 mg,加入液氮充分研磨成粉末状。样品量不应超过100 mg(干粉样品30 mg),过多的样品使得裂解不充分,最终会导致基因组DNA提取量减少。实验前在1.5 mL离心管中加入800 μL的Lysis Buffer,于65℃下预热,然后加入β-巯基乙醇至终浓度为0.1%。将研磨好的粉末加到以上预备好的Lysis Buffer中,65℃水浴20~30 min,其间颠倒混匀样品数次。加入500 μL氯仿,充分混匀,12000 rpm离心5 min。小心吸取上层水相转入一个新的离心管中,加入700 μL Binding buffer,充分混匀。将混匀的液体转入离心柱中(可分次转入),12000 rpm离心1 min。加入700 μL Wash BufferA,12000 rpm离心1 min,弃废液。加入700 μL Wash buffer B,12000 rpm离心1 min,弃废液。加入500 μLl Wash buffer B,12000 rpm离心1 min,弃废液。再次12000 rpm离心2 min,将离心柱置于新的离心管中,并打开离心柱盖,于室温或37℃放置5~10 min,至无明显乙醇味。加入50~200 μL TE buffer(事先预热55~65℃),置于室温2~5 min,12000 rpm离心2 min,离心管中即为所提取的基因组DNA溶液。
1.3 SSR-PCR扩增
1.3.1 SSR引物信息 本文采用Kyung-Ho[19]和Lee[20]开发的SSR引物,筛选出的7对引物(表2)。

表2 引物信息Table 2 Primer Information
1.3.2 反应体系 PCR反应设3次重复,体系为模板DNA 2 μL,上下游引物各0.5 μL,Dntp 0.5 μL,Taq酶0.5 μL,10×PCR buffer 2.5 μL,dd H2O 18.5 μL,总体积25 μL。
1.3.3 反应条件 PCR扩增程序为94℃5 min;94℃30 s,50℃30 s,72℃30 s,35个循环;72℃10 min,4℃保存。PCR产物采用6%变性聚丙烯酰胺凝胶电泳分离,恒功率50 W电泳2.5 h,采用银染色法显色。
1.4 数据分析
根据电泳结果,凝胶相同迁移率位置上,有条带的记为“1”,无条带的记为“0”,构成“0,1”矩阵。利用POPGENE version1.32软件计算有效等位基因数Ne、Nei’s遗传相似系数(GS)、遗传距离(GD)、Nei’s遗传多样性指数、Shannon信息指数等指标,并采用NTSYS pc 21-2软件进行种质间的UPGMA(非加权组平均法,unweighted pair-group method)聚类分析和基于Nei’s遗传距离的组群间聚类分析。并利用NTSYS pc 21-2软件进行种质间的UPGMA(非加权组平均法)聚类分析和基于Nei’s遗传距离的组群间的聚类分析。
2 结果与分析
2.1 SSR引物扩增的多态性
利用7对SSR引物对55份大蒜种质材料进行扩增,共扩增出75条带,其中73条为多态性条带,多态性比率为97.33%,扩增多态性片段的长度在47~287 bp之间,每对引物的扩增效率存在一定的差异,平均每对引物扩增出10.71个位点和10.43个多态性位点,说明SSR分子标记揭示的多态性强且大蒜品种间的遗传多样性较为丰富。
2.2 大蒜种质资源的遗传多样性
利用POPGENE version1.32软件对试验数据进行各项参数的统计分析,结果表明,样品间的平均Nei’s遗传多样性指数为0.1335,平均Shannon信息指数为0.2278,大蒜虽为无性繁殖植物,但具有一定的遗传多样性。
基于Nei’s相似性系数运用UPGMA法进行聚类分析(图1),结果表明,在遗传相似系数为0.773处,可将供试材料分为三类:i类分为30个品种,其中国内品种25个,国外品种5个;ⅱ类分为24个品种,其中国内品种11个,国外品种13个;ⅲ类仅新疆野蒜一个品种,与其他品种的相似系数为0.77。
在相似系数为0.861时,i类又可以分为4个亚类,A亚类23个品种来源广泛,来自山东、四川、河南、台湾以及日本;B亚类4个品种,均来自新疆;C亚类只有一个品种,为瑞士的欧洲小蒜;D亚类的2个品种均来自泰国。在相似系数为0.857处,ⅱ类又可以分为4个亚类,A亚类的13个品种分为来自山东、俄罗斯和美国;B亚类的4个品种来自加拿大和乌克兰;C亚类的5个品种和D亚类的2个品种,均来自美国。经分析发现,除i-A和ⅱ-A外,每一类的大蒜多来源于相同或相近区域,表明大蒜遗传多样性受地域来源影响较大。
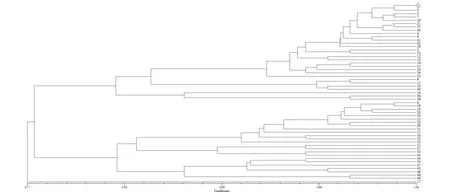
图1 55份大蒜种质基于7对引物的SSR分析和UPGMA聚类结果Fig.1 Dendrogram of 55 garlic accessions based on SSR analysis with 7 pair primers combination
2.3 不同地域来源大蒜种质资源的遗传一致度和聚类结果
利用POPGENE version1.32软件计算不同地域来源大蒜种质资源群体间的遗传距离和遗传相似系数(Nei's,1978),从表3中可以看出,8个资源群的遗传相似系数分布为0.8546~0.9980,其中中南群体和西南群体的遗传相似系数最高,为0.9980,遗传距离最近;而西北群体和北美群体的遗传相似系数最低,为0.8546,遗传距离最远。
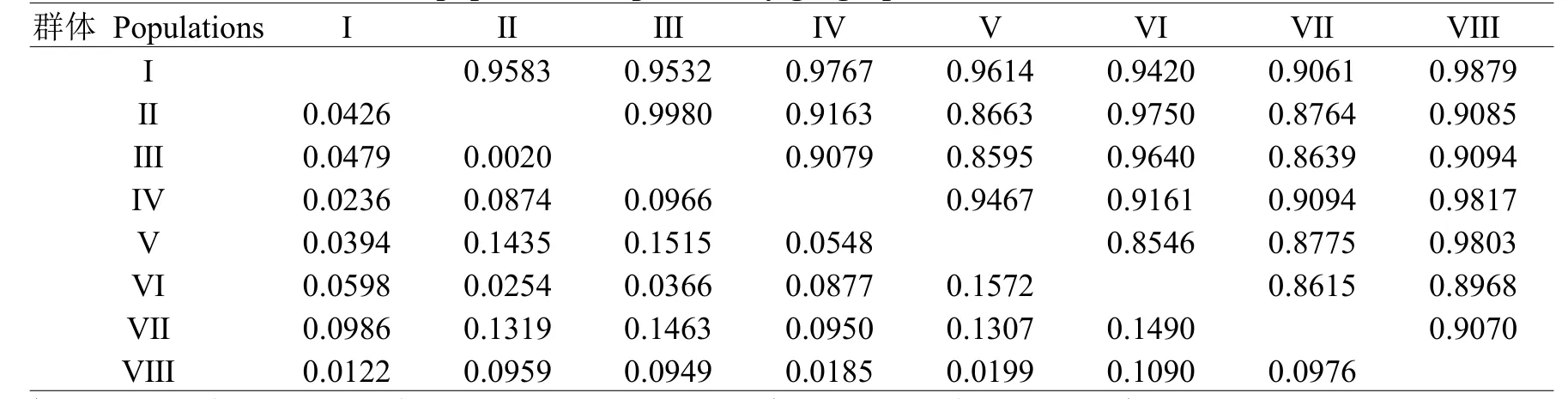
表3 10个大蒜种质资源群间的Nei's遗传相似系数(对角线上方)、遗传距离(对角线下方)Table 3 Nei’s genetic identity(above diagonal)and genetic distance(below diagonal)among the ten groups of garlic populations separated by geographical distributions
2.4 不同地域来源大蒜种质资源的遗传多样性
为进一步分析大蒜种质资源遗传多样性与其地域来源的关系以及不同区域大蒜种质资源的亲缘关系,将55份材料按照地理区域划分为8个(来自台湾的只有一个品种,除外)群体。经POPGENE version1.32软件计算Shannon信息指数(表4)。8个群体总体Shannon信息指数变化范围是0.0242-0.1792,各群体多样性指数由高到低依次为华东群体、欧洲群体、北美群体、西北群体、中南群体、西南群体、日本群体、泰国群体,总体来看,国内群体的Shannon信息指数略大于国外群体,其中,华东群体Shannon信息指数最高,遗传多样性最为丰富。

表4 10个大蒜种质资源群间的SSR分析Table 4 Ten groups of garlic populations separated by SSR analysis
根据群体间遗传相似系数,运用NTSYS软件进行聚类(图2),可以看出,8个种质资源群体在遗传相似系数0.91处聚成3类,其中第一类包括华东群体、中南群体、西南群体和日本群体;第二类包括西北群体、北美群体和欧洲群体;第三类是泰国群体。
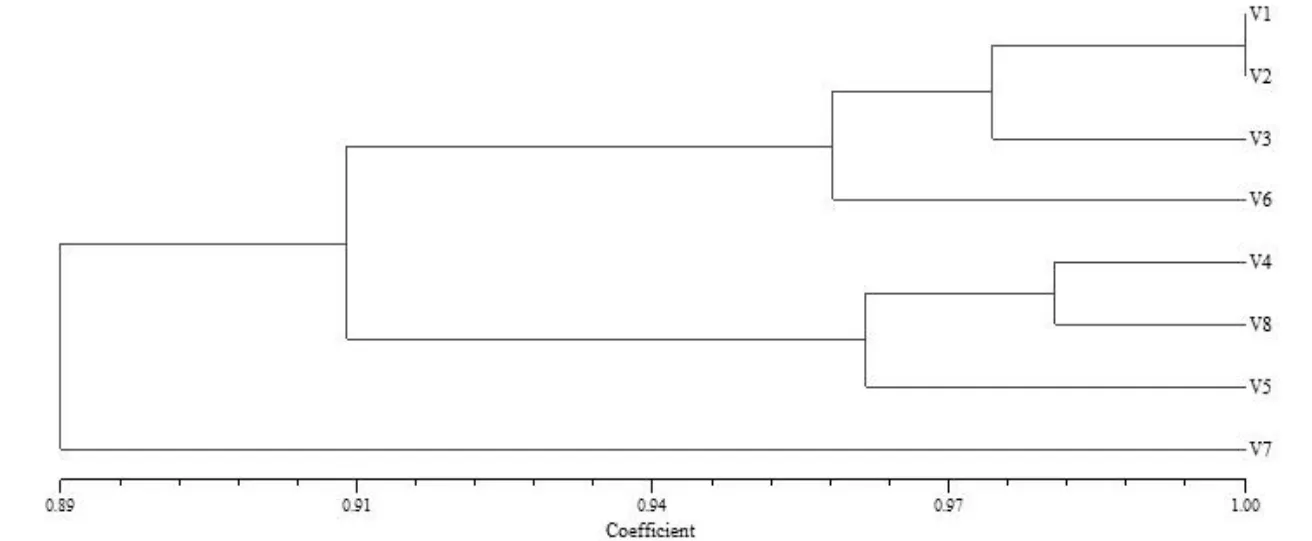
图2 基于Nei’s遗传相似系数对8个大蒜群体的UPGMA聚类结果Fig.2 Dendrogram of 8 garlic populations based on Nei’s genetic identity
3 讨论
试验采用的7对SSR引物共扩增出73条多态性条带,平均每对引物10.43条,揭示的多态性强。
采用NTSYS pc 21-2软件对55份大蒜种质资源进行种质间的UPGMA聚类分析,通过对聚类图的分析发现,国内的某些大蒜种质之间的亲缘关系与其地域分布有一定的关系,但不存在必然联系。聚类图中的第一大类中,除欧洲小蒜外,第一大类中的所有品种均来自亚洲;第二大类中除山东金乡系列大蒜外,其他种质材料均属于国外品种。
对55份大蒜种质聚类图分析发现,同一地域来源的种质大都被聚在一起,如新疆区域的四份种质材料,与王海平等[21]和陈书霞[16]试验结果相同;不同地域来源的种质也有可能被聚在一类,如加拿大的2份种质材料和乌克兰的2份种质材料,加拿大和乌克兰虽然不在同一区域,但是纬度相近,温度和光照等环境条件相似,说明相似的地理环境对大蒜遗传多样性的影响相同;同一地域来源的种质也可能不被聚到一类,如山东的苍山系列大蒜和金乡系列大蒜,虽然同属于山东区域且两地地理位置较近,但是在相似系数0.811处,分别聚类到两个不同的大类中,说明大蒜种质材料本身具有复杂的遗传多样性,金乡系列大蒜和美国大蒜、加拿大大蒜、乌克兰大蒜、俄罗斯大蒜被聚到第二大类中,说明金乡系列大蒜可能是大蒜区域相互引种交流的结果,1991年之前李文家从苏联引进苏联大蒜,而金乡系列大蒜与俄罗斯大蒜又被聚到同一亚类中,说明金乡系列大蒜和俄罗斯大蒜有一定的亲缘关系,金乡系列大蒜应该是苏联大蒜引进的选育品种。大蒜区域间的品种交流使得大蒜种质来源复杂。
本研究利用SSR分子标记分析大蒜种质资源的遗传多样性,从大蒜本身遗传信息分析了其亲缘关系,为大蒜种质的收集、保护和新品种选育提供理论依据和技术支持。
4 结论
大蒜品种间的遗传多样性受地域影响较大,大蒜区域间的品种交流频繁使种质来源复杂,SSR分子标记技术对大蒜基因组检测多态性水平高,可有效揭示其遗传多样性。
[1]刘世琦.蔬菜栽培学简明教程[M].北京:化学工业出版社,2007:173
[2]王 震.中国大蒜出口存在的问题及对策探讨[J].网友世界,2013(6):46-47
[3]Lombard V,Baril CP,Dubreuil P,et al.Genetic relationships and fingerprinting of rapeseed cultivars by AFLP: Consequences for varietal registration[J].Crop Science,2000,40(5):1417-1425
[4]Métais I,Aubry C,Hamon B,et al.Description and analysis of genetic diversity between commercial bean lines (Phaseolus vulgaris L.)[J].Theoretical andApplied Genetics,2000,101(8):1207-1214
[5]Sun GL,William M,Liu J,et al.Microsatellite and RAPD polymorphism in Ontario corn hybrids are related to the commercial sources and maturity ratings[J].Molecular Breeding,2001,7(1):13-24
[6]Chen S,Chen WF,Shen XQ,et al.Analysis of the genetic diversity of garlic(Allium sativum L.)by simple sequence repeat and inter simple sequence repeat analysis and agro-morphological traits[J].Biochemical Systematics and Ecology,2014,55(8):260-267
[7]翟亚辉,马蓉丽,成 妍,等.37份洋葱遗传多样性的RAPD和SSR分析[J].华北农学报,2013,28(6):115-120
[8]吴凤芝,金 雪.葱属作物遗传多样性研究进展[J].东北农业大学学报,2014,45(1):118-122
[9]赖 勇,王鹏喜,范贵强,等.大麦SSR标记遗传多样性及其与农艺性状关联分析[J].中国农业科学,2013,46(2):233-242
[10]成良强,唐 梅,任小平,等.栽培种花生遗传图谱的建及主茎高和总分枝数QTL分析[J].作物学报,2015,41(6):979-987
[11]王庆彪,张扬勇,庄 木,等.中国50个甘蓝代表品种EST-SSR指纹图谱的构建[J].中国农业科学,2014,47(1):111-121
[12]张法惺,栾非时,盛云燕.不同生态类型西瓜种质资源遗传多样性的SSR分析[J].中国蔬菜,2010(14):36-43
[13]左力辉,韩志校,梁海永,等.不同产地中国李资源遗传多样性SSR分析[J].园艺学报,2015,42(1):111-118
[14]左力辉,张文林,邱 彤,等.新疆野苹果叶形性状变异及其与SSR标记关联分析[J].园艺学报,2015,42(4):759-768.
[15]许占友,邱丽娟,常汝镇,等.利用SSR标记鉴定大豆种质[J].中国农业科学,1999,32(增刊):40-48
[16]陈书霞,常燕霞,周 静,等.大蒜种质遗传多样性的SSR分析[J].农业生物技术学报,2012,20(4):372-381
[17]Sajib AM,Hossain MM,Mosnaz ATMJ,et al.SSR marker-based molecular characterization and genetic diversity analysis of aromatic landraces of rice(Oryza sativa L.)[J].Journal of Bioscience and Biotechnology,2012,1(2):107-116
[18]罗 兵,徐港明,孙海燕,等.利用简单重复序列(SSR)标记分析太湖稻区现代粳稻品种的遗传多样性[J].农业生物技术学报,2014,22(12):1502-1513
[19]Lee GA,Kwon SJ,Park YJ,et al.Cross-amplification of SSR markers developed from Allium sativumto other Allium species[J].Scientia Horticulturae,2011,128(4):401-407
[20]Kyung HM,Jae-Gyun K,Zhao WG.Isolation and characteristics of eight novel polymorphic microsatellite loci from the genome of garlic(Allium sativum L.)[J].Scientia Horticulturae,2009,122(3):355-361
[21]王海平,PhilippWS,李锡香,等.中国大蒜种质资源遗传多样性和群体遗传结构分析[J].中国农业科学,2012,45(16):3318-3329
The Study on the Genetic Diversity of Garlic Germplasm Based on SSR Molecular Marker
SUN Ya-li,SHI Qing-hua,LIU Shi-qi*,SUN Xiu-dong,CHEN Ya-fei,QIAN Sheng-yan,LIU Xing-chen
College of Horticulture Science and Engineering,State Key Laboratory of Crop Biology,Agriculture Ministry Key Laboratory of Biology and Genetic Improvement of Horticultural Crops(Huanghuai Region)/Shandong Agricultural University,Tai’an 271018,China
Garlic is an asexual reproductive crop,a lot of local varieties were formed due to the mutation induced by climate change,natural selection and artificial selection.The research on genetic diversity of Garlic Germplasm Resources provided scientific basis for garlic scientific classification,identification,preservation and genetic improvement.Simple sequence repeats(SSR)molecular marker technique was used for polymorphic amplification for genomic DNA of 55 garlic germplasm. POPGENE version1.32 software calculated the effective bit gene number,Nei’s genetic similar coefficient,genetic distance, Nei’s genetic diversity index and Shannon's information index and other indicators.NTSYS PC software was used for UPGMA(non weighted average method)cluster analysis and inter cluster analysis of genetic distance based on Nei's.The results show that 75 bands were obtained from 7 pairs of primers,among which 73 bands showed polymorphic bands (97.33%).Each pair of primers amplified 10.71 loci and 10.43 polymorphic loci,SSR molecular markers have strong polymorphism.The genetic diversity index and the average information index of 55 garlic accessions were 0.1335 and 0.2278. Garlic has a high genetic diversity,the most of garlic known as one category was from the same or similar area.
Garlic;SSR;genetic diversity
S633.4
:A
:1000-2324(2017)03-0384-06
2015-10-22
:2015-11-20
国家自然科学基金(31372084);国家公益性行业科研专项(200903018);“十二五”农村领域国家科技计划(2013BAD01B04)
孙亚丽(1991-),女,硕士研究生.研究方向为蔬菜学.E-mail:yalisun2013@163.com
*通讯作者:Author for correspondence.E-mail:liusq99@sdau.edu.cn
