乳腺癌分子病理诊断的机遇与挑战
2017-06-24郑唯强
郑唯强
·专家论坛·
乳腺癌分子病理诊断的机遇与挑战
郑唯强
乳腺肿瘤;分子诊断;预后;靶向治疗
乳腺癌的临床病理特征是由肿瘤组织学类型、癌细胞增殖活性和淋巴血管浸润等形态学确定的[1]。传统的乳腺癌组织学分类对小管癌、浸润性筛状癌、单纯性黏液癌、腺样囊性癌等,无论是其临床预后还是治疗方式均无太大差异。现阶段临床医师已不满足于单纯组织形态学诊断的信息,乳腺癌的治疗已从循证医学模式向个体化医学模式转变。
鉴于乳腺癌对治疗的反应及肿瘤的异质性,目前ER、PR和HER-2已经作为全身性治疗的主要依据。临床病理实践中除主要运用形态学诊断以外,已经开始结合分子生物学技术如免疫组化、原位杂交、RT-PCR等进行评估,提供临床需要的生物学标记状态如HER-2,并试图通过这些技术对乳腺癌作出比较正确的诊断、分类和预后评估。随着分子生物学技术的迅猛发展,基因表达谱、蛋白组学、DNA拷贝数改变或甲基化、染色体改变、基因突变和microRNAs分析等为我们更准确地对乳腺癌生物学特性的了解和个体化治疗的评估提供良好的机遇。二代测序技术的运用也为乳腺癌分子生物学的解码、分子学分类的重新定义提供新途径[2],但准确的运用和临床效果还有待于临床实践的检验。
1 乳腺病理诊断和预后评估的分子生物学主要标记
乳腺病理诊断和预后评估标记通常主要用以良性和恶性病变、原位癌及浸润性癌、某些癌的亚型定义(如基底细胞样癌)和肿瘤起源(如肌上皮癌)的鉴定,实践工作中主要依赖免疫组化的生物学标记进行[3-4]:如梭形细胞病变、肌上皮细胞鉴别、乳头状病变分类、导管癌与小叶癌的鉴别(如E-cadherin和p120)以及增生性和肿瘤性上皮(如CK5和ER)的鉴别等。其它免疫组化标记如CK可用于淋巴结微转移的鉴别,Pax-8、WT1可用于卵巢癌的鉴别,TTF-1可用于甲状腺癌、肺癌的鉴别,Melan-A、HMB-45 和S-100可用于黑色素瘤的鉴别等。有时还需借助特殊的遗传学易位加以鉴别,如乳腺肿块实施粗针穿刺仅获得恶性叶状肿瘤的间质成分时,则需与其它软组织肉瘤鉴别[5]。
精准医学主要目的是预测患者对治疗的反应,它最早起始于20世纪70年代基于ER表达的内分泌治疗反应。实践证明ER和PR的表达对乳腺癌治疗有重要意义;HER-2蛋白过表达或基因的扩增可作为靶向治疗的依据。尽管诸如21-基因复发评分等多基因的检测还未作为常规检测内容,但作为指导临床治疗的决策和预后判断有较好的参考意义。
1.1 乳腺癌治疗的常规检测
1.1.1 ER、PR检测 目前,日常病理工作常采用免疫组化法检测ER、PR的表达;但不同的实验室有不同的阳性评估标准,常用有Allred免疫组化得分法(即H-score法),其既评估了阳性细胞的比例,又结合了细胞核阳性着色的强度。美国临床肿瘤学会/美国病理学会指南推荐的阳性标准为1%[6],理由是只要大于该阈值的受体阳性患者对内分泌治疗有良好疗效。但是对转移性乳腺癌的鉴别可能需要结合每个病理医师的实际临床经验和综合分析确定其阳性标准,不能仅依赖于1%的阳性程度进行鉴别。
1.1.2 HER-2检测 15%~20%的乳腺癌有HER-2的过表达(3+)或扩增,由于其对靶向治疗的指导意义已非常明确,国内外均有相应的检测指南和诸多文献报道[7],在此不作赘述。
1.1.3 Ki-67增殖指数 免疫组化是检测Ki-67增殖指数的常规方法,其可以鉴定各类型乳腺癌的增殖状态[8],Ki-67增殖指数可作为预后判断的指标之一[9-10]。然而,无论是室内还是室间质控的标准化问题,对Ki-67评估的正确性始终存在争议。2011年St. Gallen共识中将Ki-67阳性率设定为14%,作为乳腺癌分子亚型(Luminal A型、Luminal B型)的阈值曾引起较大争议。2016年美国临床肿瘤学会在临床实践指南中并不建议Ki-67用以新辅助化疗的标记[11]。因此,免疫组化检测Ki-67 增殖指数的指标运用尚有待于进一步探讨。
1.2 肿瘤的遗传学检测和诊断 乳腺癌在基因水平(基因组杂交和显微芯片的基因组杂交)存在明显的异质性,在导管原位癌和浸润性导管癌有基因拷贝数的差异,在组织学I级浸润性导管癌通常为二倍体或近二倍体,频发16q的缺失,但III级的癌则具有复杂且众多的拷贝数异常,常为非整倍体,而16q的缺失仅限于ER阴性病例,且只有30%的发生频率。
乳腺癌的原发灶和转移灶的配伍采用全基因组测序显示,肿瘤是由多种癌细胞亚克隆嵌合构成,不仅含有原发性的遗传学改变,也可出现继发遗传学改变,如乳腺癌的转移灶可起源于遗传学有别于原发瘤的亚克隆。有些显微芯片的诊断基因表达检测可用来明确肿瘤组织的来源,包括使用2000基因分类模型Path work和应用RT-PCR的Theros CancerType ID 92基因检查可确定低分化恶性肿瘤的原发部位[12]。某些特殊类型的乳腺癌显示特殊易位:如分泌性癌显示特征性染色体的t(12;15)的遗传学易位,导致 ETV6的5′段与NTRK3 的3′段的融合,形成ETV6-NTRK3融合基因[13];黏液表皮样癌中发现有特征性的t(11;19)(q21;p13),形成MECT1-MAML2 融合基因[14];腺样囊性癌出现特征性的t(6;9)(q22-23; p23-24),形成 MYB-NFIB融合基因[15]。在腺样囊性癌混合高级别的三阴型乳腺癌中也发现有MYB-NFIB融合基因。因此,可以假设从腺样囊性癌进展到三阴型乳腺癌的过程中可能涉及肿瘤性克隆的选择和(或)影响某些特殊基因,如FGFR1突变的遗传学改变[16]。
2 乳腺癌的分子学分类
除了对乳腺癌的杂合性缺失、比较基因谱杂交等检测外,按ER、PR和HER-2表达可以对乳腺癌进行分子分型(Luminal A型、Luminal B型、HER-2过表达型和基底细胞样型等)的方法已被临床普遍接受[17],使用非监督分层聚类分析全基因表达谱技术可以使分类更为准确。通过“固有”基因的差异表达分析显示:在转录组水平上乳腺癌不是单一性的疾病,就是富于ER表达相关的独特性而被命名为“腔样”表型。尽管每种乳腺癌分类具有独特的全基因表达谱与它的生物学特征和异常有关,其它的分子类型(HER-2阳性、基底样型等)均是根据该基因表达谱判断的。腔样型乳腺癌又可依据它的异质性分为Luminal A型、Luminal B型,有文献报道[18-19]其也曾进一步分为Luminal C型、Luminal N型,由于未能被证实而弃用。Burstein等[20]报道基底样型乳腺癌可以根据基因表达谱细分为基底样型1、基底样型2、免疫调节型、间叶型、间叶干细胞型和腔样雄激素受体型。这些不同的类型显示不同的临床意义,如Luminal A型具有最多和最散的突变基因,其PIK3CA的基因突变率达45%;Luminal B型显示既有PIK3CA又有TP53的较高突变频率,而在基底样型中GATA3和PIK3CA等突变相对少见,TP53突变较多[21]。癌症基因谱协作机构(TCGA)[22]曾报道使用5种不同的平台(基因表达谱DNA拷贝数分析、DNA甲基化、外显子测序、mRNA阵列、microRNA测序和逆相蛋白阵列)分析466例乳腺癌患者,识别2个新的涉及间质/微环境的蛋白表达以及特殊的信号途径。
为了克服使用新鲜组织、阵列分析技术和费用等问题,我们可以把RT-PCR和免疫组化等检测技术运用到常规病理工作中。目前临床实践主要有2种方式:(1)使用显微芯片检测由基因表达谱定义的分类,如PAM50(即检测50个分类基因和5个参照基因)[23];(2)采用组织芯片和免疫组化对ER和HER-2的表达进行检测。WHO(2012)乳腺癌分类提出预测临床预后的多基因表达谱标识,显微芯片分析是检测乳腺癌治疗反应的有效方法。其中70-基因预后标识可用于Ⅰ~Ⅱ期淋巴结阴性且肿瘤直径小于5 cm的乳腺癌预后,该二元分类法把预后分为“好”和“差”,可作为远处转移的独立预后指标,但它不能用于ER阴性肿瘤的预后判断,而且必须要用术后新鲜标本。21-基因复发评分是根据21个基因qRT-PCR结果的评分系统,包括16个癌相关基因和5个参照基因,该系统为数学函数,用于评估ER阳性但淋巴结转移阴性乳腺癌的10年远处复发风险。它是一个连续变量,由此将患者分为3组:低危组、中危组和高危组[24]。该分组可以使低危组患者避免不必要的化疗,其优点是它可使用10%中性福尔马林固定后的石蜡包埋标本进行实验,缺点是该评分系统对于ER阴性或HER-2阳性等乳腺癌患者的临床运用有限。
3 特殊类型乳腺癌的分子病理学特征
浸润性小叶癌是最常见的特殊类型,除E-cadherin的缺失,还有特殊的PTEN、TBX3和FOXA1突变。PTEN缺失与增加的AKT磷酸化有关,基因表达谱分析将其分为反应样、免疫相关和增殖型3个亚型,这些亚型显示它们在mRNA和蛋白/磷酸蛋白水平上的基因谱特征,其中增殖型的预后最差,反应样型预后较好[25],在DNA水平上可检测到浸润性小叶癌富于CDH1突变,其次是GATA3突变。乳腺的纯黏液癌实验显示有相对较低的遗传学不稳定性并且倾向于同源性而非异质性。遗传学主要表现为1q和16p的获得、16q和22q的缺失,而混合型黏液癌不同成分基因表达谱与单纯黏液癌相似。浸润性微乳头状癌除了它的特殊乳头形态,还有特殊的拷贝数异常、Cyclin D1高表达和MYC(8q24)扩增,其特征性的一系列基因拷贝数异常有别于分级和ER状态相匹配的浸润性导管癌。伴大汗腺分化的癌通过基因表达微阵列分析识别的“大汗腺分子标识”,以雄激素信号增加以及与HER-2过表达组显著重叠为特征。提示伴大汗腺分化乳腺癌并非一个独特的组别,它由“大汗腺”和“腔样”两种分子亚型构成。值得注意的是,在混合型微乳头状癌中含微乳头的区域和非微乳头区域显示相同的基因异常,这种形态上不同但基因异常却相同的现象值得思考。基底样型的包括髓样特征的癌(可出现体细胞突变和BRCA1启动子甲基化)、化生性癌(10%~25%可出现EGFR的扩增和多态性,可发生PIK3CA及Wnt通路基因频发突变)和唾液腺样肿瘤如腺样囊性癌[(频发易位t(6;9)(q22-23;p23-24)](图1、2)[26]。采用大规模平行测序检测嗜酸细胞癌有高频率的TP53突变,常显示11q13.1-q13.2和19p13获得;其它的体细胞突变包括PIK3CA、mTOR、CTNNB1、BRCA1、ERBB4、ERBB3、INPP4B和FGFR2等。
4 引入二代测序技术
二代测序技术对乳腺癌的个体化治疗,尤其是对于识别源于“乘客”基因和“驱动”基因的不同突变有很大帮助。二代测序技术证实乳腺癌的突变频率低于肺鳞状细胞癌或膀胱尿路上皮癌,但类似于卵巢癌和肾透明细胞癌。约10%不同类型、不同组织学分级、ER、PR、HER-2阴阳性的乳腺癌染色体异常频率以上的乳腺癌40基因中有7个(TP53、PIK3CA、MYC、ERBB2、 FGFR1、CCND1和GATA3)发生突变,与58%的驱动突变有明显关联;但三阴型和基底样乳腺癌有较高的肿瘤内异质性,常见的驱动基因如TP53、PIK3CA和PTEN突变有较高的克隆发生率,而体细胞突变仅出现在少量癌细胞中。二代测序技术也显示若出现乳腺癌转移后的一系列体细胞突变,提示该克隆遗传异质性对乳腺癌治疗的耐受性。
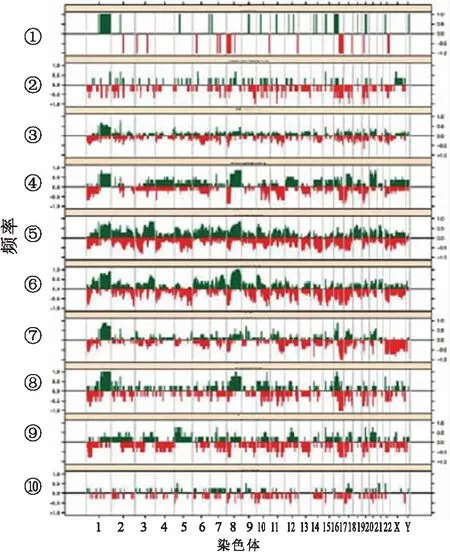
图1 部分特殊类型的乳腺癌染色体基因拷贝数异常分布图
1.小管癌;2.神经内分泌癌;3.黏液癌;4.微乳头状癌;5.化生性癌;6.伴髓样特征癌;7.小叶癌;8.伴破骨细胞样间质巨细胞的癌;9.大汗腺癌;10.腺样囊性癌;其中绿色示染色体片段的扩增;红色示缺失
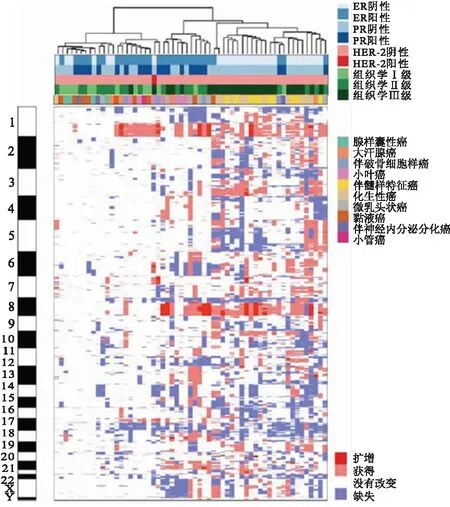
图2 特殊类型乳腺癌的基因表达谱聚类分析
采用二代测序技术检测微腺性腺病/非典型性微腺性腺病,结果显示至少有1个与TP53突变一致的体细胞非同义突变[27];在放射状瘢痕/复杂性硬化性病变中也有类似现象。相反,纯微腺性腺病则缺乏克隆性的类似突变,其拷贝数改变也有限。提示前者这些病变可能是肿瘤性的而非增生性改变。有证据表明与浸润性癌并存的导管原位癌和前者有相似的基因谱、转录谱和表型[28],但遗憾的是预测复发或进展到浸润性癌的癌前病变分子学分析还有所欠缺。
5 结语
由于日益增加的高通量测序或二代测序技术的运用,新的具有靶向治疗意义的驱动基因不断被发现,为曾经治疗失败的病例寻找发生的机制,为寻找更具潜力的有效靶点提供参考。乳腺癌分子病理学检测在其诊断尤其是在分子分型和治疗中的地位逐渐显现。然而,组织病理学的诊断是我们病理医师一切工作的根本,病理医师在评估乳腺肿瘤大小、淋巴结转移、组织学类型等仍需对肿瘤TNM分期、分子分型、预后与疗效进行判断。随着分子生物学新技术运用的不断推进,大量的可供分析的临床检测数据会持续涌现[29],由此必然会导致新的治疗靶点出现,并促进对乳腺癌形态学与分子病理学相结合的更精细分类以指导临床实践。
[1] Rakha E A, Reis-Filho J S, Baehner F,etal. Breast cancer prognostic classification in the molecular era: the role of histological grade[J]. Breast Cancer Res, 2010,12(4):207.
[2] Ng C K, Schultheis A M, Bidard F C,etal. Breast cancer genomics from microarrays to massively parallel sequencing: paradigms and new insights[J]. J Natl Cancer Inst, 2015,107(5):djv015.
[3] Lee A H. Use of immunohistochemistry in the diagnosis of problematic breast lesions[J]. J Clin Pathol, 2013,66(6):471-477.
[4] Rakha E A, Aleskandarany M A, Lee A H,etal. An approach to the diagnosis of spindle cell lesions of the breast[J]. Histopathology, 2016,68(1):33-44.
[5] Fisher C. The diversity of soft tissue tumours with EWSR1 gene rearrangements: a review[J]. Histopathology, 2014,64(1):13450.
[6] Hammond M E, Hayes D F, Dowsett M,etal. American society of clinical oncology/college of American pathologists guideline recommendations for immunohistochemical testing of estrogen and progesterone receptors in breast cancer[J]. J Clin Oncol, 2010,28(16):2784-2795.
[7] 杨 莹,魏 兵,步 宏. 乳腺癌HER-2检测的现状及存在的问题[J]. 临床与实验病理学杂志, 2010,26(5):602-605.
[8] Aleskandarany M A, Rakha E A, Macmillan R D,etal. MIB1/Ki-67 labelling index can classify grade 2 breast cancer into two clinically distinct subgroups[J]. Breast Cancer Res Treat, 2011,127(3):591-599.
[9] Wishart G C, Rakha E, Green A,etal. Inclusion of Ki-67 significantly improves performance of the PREDICT prognostication and prediction model for early breast cancer[J]. BMC Cancer, 2014,14:908.
[10] Yerushalmi R, Woods R, Ravdin P M,etal. Ki-67 in breast cancer: prognostic and predictive potential[J]. Lancet Oncol, 2010,11(2):174-183.
[11] Harris L N, Ismaila N, McShane L M,etal. Use of biomarkers to guide decisions on adjuvant systemic therapy for women with early-stage invasive breast cancer: American Society of Clinical Oncology Clinical Practice Guideline[J]. J Clin Oncol, 2016,34(10):1134-1150.
[12] Ma X J, Patel R, Wang X,etal. Molecular classification of human cancers using a 92-gene real-time quantitative polymerase chain reaction assay[J]. Arch Pathol Lab Med, 2006,130(4):465-473.
[13] Tognon C, Knezevich S R, Huntsman D,etal. Expression of the ETV6-NTRK3 gene fusion as a primary event in human secretory breast carcinoma[J]. Cancer Cell, 2002,2(5):367-376.
[14] Tonon G, Modi S, Wu L,etal. t(11;19)(q21;p13) translocation in mucoepidermoid carcinoma creates a novel fusion product that disrupts a Notch signaling pathway[J]. Nat Genet, 2003,33(2):208-213.
[15] Mitani Y, Li J, Rao P H,etal. Comprehensive analysis of the MYBNIB gene fusion in salivary adenoid cystic carcinoma: incidence, variability, and clinicopathologic significance[J]. Clin Cancer Res, 2010,16(19):4722-4731.
[16] Fusco N, Geyer F C, De Filippo M R,etal. Genetic events in the progression of adenoid cystic carcinoma of the breast to high-grade triplenegative breast cancer[J]. Mod Pathol, 2016,29(11):1292-1305.
[17] Rakha E A, Reis-Filho J S, Ellis I O. Combinatorial biomarker expression in breast cancer[J]. Breast Cancer Res Treat, 2010,120(2):293-308.
[18] Sotiriou C, Neo S Y, McShane L M,etal. Breast cancer classification and prognosis based on gene expression profiles from a population-based study[J]. Proc Natl Acad Sci USA, 2003,100(18):10393-10398.
[19] Rakha E A, Soria D, Green A R,etal. Nottingham prognostic index plus (NPI+): a modern clinical decision making tool in breast cancer[J]. Br J Cancer, 2014,110(7):1688-1697.
[20] Burstein M D, Tsimelzon A, Poage G M,etal. Comprehensive genomic analysis identifies novel subtypes and targets of triple-negative breast cancer[J]. Clin Cancer Res, 2015,21(7):1688-1698.
[21] Santarpia L, Bottai G, Kelly C M,etal. Deciphering and targeting oncogenic mutations and pathways in breast cancer[J]. Oncologist, 2016,21(7):1063-1078.
[22] Cancer Genome Atlas Network. Comprehensive molecular portraits of human breast tumours[J]. Nature, 2012,490(7418):61-70.
[23] Parker J S, Mullins M, Cheang M C,etal. Supervised risk predictor of breast cancer based on intrinsic subtypes[J]. J Clin Oncol, 2009,27(8):1160-1167.
[24] van de Vijver M J, He Y D, van’t Veer L J,etal. A gene-expression signature as a predictor of survival in breast cancer[J]. N Engl J Med, 2002,347(25):1999-2009.
[25] Ciriello G, Gatza M L, Beck A H,etal. Comprehensive molecular portraits of invasive lobular breast cancer[J]. Cell, 2015,163(2):506-519.
[26] Horlings H M, Weigelt B, Anderson E M,etal. Genomic profiling of histological special types of breast cancer[J]. Breast Cancer Res Treat, 2013,142(2):257-269.
[27] Guerini-Rocco E, Piscuoglio S, Ng C K,etal. Microglandular adenosis associated with triple-negative breast cancer is a neoplastic lesion of triple-negative phenotype harbouring TP53 somatic mutations[J]. J Pathol, 2016,238(5):677-688.
[28] Vincent-Salomon A, Lucchesi C, Gruel N,etal. Integrated genomic and transcriptomic analysis of ductal carcinoma in situ of the breast[J]. Clin Cancer Res, 2008,14(7):1956-1965.
[29] 杨举伦,王 丽. 乳腺癌的分子病理学研究[J]. 临床与实验病理学杂志, 2011, 27(1):10-14.
国家自然科学基金(81272905)
第二军医大学附属长海医院病理科,上海 200433
郑唯强,男,主任医师,教授。 E-mail: zheng6947@126.com
时间:2017-5-17 23:52 网络出版地址:http://kns.cnki.net/kcms/detail/34.1073.R.20170517.2352.001.html
R 737.9
A
1001-7399(2017)05-0473-04
10.13315/j.cnki.cjcep.2017.05.001
接受日期:2017-04-12
