杉木磷转运蛋白基因ClPht1;1的克隆及表达分析*
2017-06-23苏烁烁吴鹏飞马祥庆
苏烁烁 李 明,2 吴鹏飞,2 张 颖 马祥庆,2
(1.福建农林大学林学院 福州 350002; 2.国家林业局杉木工程技术研究中心 福州 350002)
杉木磷转运蛋白基因ClPht1;1的克隆及表达分析*
苏烁烁1李 明1,2吴鹏飞1,2张 颖1马祥庆1,2
(1.福建农林大学林学院 福州 350002; 2.国家林业局杉木工程技术研究中心 福州 350002)
【目的】PHT1基因家族是影响植物吸收磷营养的重要磷转运子之一。从杉木32号磷高效家系cDNA中克隆得到PHT1基因家族的1个杉木磷转运蛋白基因ClPht1;1,并对不同程度磷胁迫下ClPht1;1的时空表达进行研究,为杉木PHT1基因序列特征和功能结构的研究以及磷高效利用杉木基因型的选育奠定基础。【方法】 根据PHT1基因家族序列保守性设计简并引物,以32号磷高效杉木基因型根系cDNA为模板进行扩增获得目的基因ClPht1;1的cDNA序列,使用RACE 技术对目的基因进行全长克隆,并对其序列特征、同源性和编码磷转运蛋白结构进行分析。实时荧光定量PCR检测ClPht1;1在32号磷高效杉木家系根、茎、叶中的表达,检测中度缺磷胁迫下ClPht1;1在不同磷利用效率杉木4号、15号、25号、27号、28号、32号家系根系中的表达差异,以及在中度、重度缺磷胁迫下ClPht1;1在32号磷高效杉木家系根系中随时间序列的表达量变化。【结果】 克隆得到1个杉木磷转运蛋白PHT1基因,命名为ClPht1;1(GenBank登录号: KX302006),基因序列编码区长1 638 bp,编码545 aa的蛋白质。ClPht1;1所编码蛋白质由12个疏水的跨膜区域组成,1个疑似跨膜域。每个跨膜结构域基本由17~25个氨基酸残基组成螺旋,同时跨膜蛋白的N 端和C端均位于细胞质内,保守序列位于第4个跨膜域。构成蛋白质的主要骨架是α-螺旋,无信号肽序列。ClPht1;1基因编码蛋白与日本柳杉PHT基因编码蛋白的氨基酸序列相似性达到87.0%,与胡杨、油茶、马尾松等PHT家族基因编码蛋白的氨基酸序列相似性均在75%以上。ClPht1;1基因在杉木的根、茎、叶组织中均有表达,其中在根中的表达量最高,在叶中的表达量最低。在中度缺磷胁迫下,ClPht1;1基因在杉木不同家系根部的表达量为25号>27号>4号>15号>32号>28号。在中度和重度缺磷胁迫下,ClPht1;1基因在32号杉木家系根部的表达量随胁迫时间的延长而逐渐上升; 恢复供磷后,ClPht1;1基因表达量逐渐下降至正常水平; 重度缺磷胁迫下,ClPht1;1基因表达量要高于其在中度缺磷胁迫下的表达量。【结论】ClPht1;1基因具有PHT1基因家族的典型结构,其编码蛋白的氨基酸序列与日本柳杉等磷转运蛋白氨基酸序列具有高度相似性,为杉木高亲和磷转运蛋白PHT1基因家族成员。ClPht1;1基因主要在杉木的根部表达,在叶片中的表达量较低; 杉木磷利用效率越强,ClPht1;1基因在其根部的表达量越高。在不同磷利用效率的杉木家系中ClPht1;1基因表达量存在较大差异。ClPht1;1 基因的表达受低磷胁迫的诱导,缺磷胁迫下ClPht1;1基因表达量明显升高,恢复供磷后ClPht1;1基因表达量明显降低。
杉木; 磷转运蛋白基因;Pht1;1; RACE; RT-qPCR; 磷利用效率
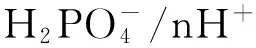
植物PHT1家族成员基因表达水平的升高是植物适应低磷胁迫的普遍机制。对拟南芥(Arabidopsisthaliana)、小麦(Triticumaestivum)、水稻(Oryzasativa)、大豆(Glycinemax)等植物PHT1基因的表达研究表明: 在介质磷浓度满足植物生长情况下,PHT1基因表达受到抑制,少量表达或不表达; 在介质磷浓度较低时,植物高亲和力转运系统启动,从而诱导PHT1基因强烈表达; 对PHT1 基因表达进行干扰将会导致根系吸收磷能力的降低(Yang etal., 2010)。因此,PHT1基因的高表达是植物磷高效吸收利用的重要指标之一,挖掘植物磷高效吸收利用的基因资源进行遗传改良,是缓解土壤有效磷缺乏和提高磷肥利用效率的有效途径之一。研究表明,植物PHT1家族的成员组成和表达受植物种及品种的影响,水稻、玉米(Zeamays)等植物不同品种在低磷胁迫下的PHT1 基因表达具有明显差异(Aietal., 2009; Calderon-Vazquezetal., 2008)。在水稻、大豆、欧洲油菜(Brassicanapus)、烟草(Nicotianatabacum)、油茶(Camelliaoleifera)、蒺藜苜蓿(Medicagotruncatula)等农作物中都克隆得到了部分PHT1 家族的磷转运蛋白基因成员,并根据PHT1基因的表达量变化来筛选和培育了一些磷高效农作物品种或无性系,为揭示植物的磷转运机制和提高磷素利用效率奠定了基础。目前,对植物PHT1基因的研究主要集中在1年生农作物上,在森林树种上的研究报道较少,仅见毛果杨(Populustrichocarpa)、橡胶树(Heveabrasiliensis)等乔木中。
杉木(Cunninghamialanceolata)是我国南方最主要的用材林造林树种之一,其面积和蓄积量均居我国人工林树种的首位(俞新妥, 2006)。由于南方酸性土壤对磷具有强烈化学固定作用,土壤磷素多以Fe-P、Al-P和O-P等形态存在(贾兴永等, 2011),这使得南方林区土壤固定态磷丰富,但是有效磷极度缺乏,杉木等南方林木表现出“遗传学缺磷”现象(梁霞等, 2006)。长期以来,南方森林土壤有效磷不足一直是限制杉木人工林产量最重要的因素之一。因此筛选和培育磷高效杉木基因型,提高杉木对土壤磷素的吸收和利用效率,是培育杉木速生高效人工林和缓解多代连栽下杉木人工林地力衰退的重要方向。然而目前在杉木磷转运蛋白基因方面研究报道较少,据此课题组在长期的杉木耐低磷胁迫试验研究中,选育了一些磷高效吸收利用杉木基因型,这些杉木基因型或主要通过根系皮层细胞溶解、加快体内磷素的转运来抵抗磷胁迫逆境(4号、32号家系等); 或主要通过在贫磷斑块中的根系向四周拓展增生和分泌化学物质,增强对介质中磷的吸收效率来适应环境的磷胁迫(25号、32号家系等)(汪攀, 2015; 吴鹏飞等, 2012)。以这些磷高效利用杉木基因型为研究材料,对于发掘杉木高亲和磷转运子PHT1基因起到重要作用并具有实践意义。
本试验以磷高效利用杉木4号、15号、25号、27号、28号、32号家系为试验材料,根据植物PHT1基因的保守性氨基酸序列设计简并引物,利用RT-PCR和RACE技术对杉木PHT1基因全长进行克隆,对其序列特征、同源性和编码磷转运蛋白结构进行分析,并使用荧光定量PCR检测杉木PHT1基因在磷高效杉木基因型中的时空表达情况,以期为揭示杉木吸收磷营养的分子机制和选育磷高效利用杉木基因型奠定基础。
1 材料与方法
1.1 试验材料
选取种植在福建农林大学妙峰山试验苗圃的1年生不同利用效率的磷高效杉木4号(1.14±0.08)kg·g-1、15号(1.10±0.06)kg·g-1、25号(1.77±0.22)kg·g-1、27号(1.38±0.03)kg·g-1、28号(0.73±0.06)kg·g-1、32号(1.09±0.02)kg·g-1(汪攀, 2015)家系幼苗,根系经纯水清洗后固定于塑料小桶进行水培供磷试验。幼苗经过7天正常供磷营养液培养后,进行不同程度缺磷胁迫处理。分别在缺磷胁迫0,12,24,48 h,以及恢复正常供磷12,24,48 h时采集幼苗的根、茎和叶,液氮速冻后-80 ℃保存待用。
利用KH2PO4设置3个供磷水平: 正常供磷(1.0 mmol·L-1KH2PO4)、中度缺磷(0.5 mmol·L-1KH2PO4,0.5 mmol·L-1KCl)和重度缺磷(0 mmol·L-1KH2PO4,1.0 mmol·L-1KCl),胁迫中不足的K+用等量KCl代替。其他营养元素含量根据吴鹏飞(2009)改良的1/3Hoagland营养液配方进行补充,即5.0 mmol·L-1KNO3、 2.0 mmol·L-1MgSO4·7H2O、5.0 mmol·L-1Ca(NO3)2·4H2O、l mL·L-1Fe-EDTA, Arnon微量元素(46.3 μmol·L-1H3BO3、0.3 μmol·L-1CuSO4·5H2O、0.8 μmol·L-1ZnSO4·7H2O、9.1 μmol·L-1MnC12·4H2O、0.4 μmol·L-1H2MoO4·4H2O),调节营养液pH为5.8。
1.2ClPht1;1基因全长的克隆
1.2.1ClPht1;1基因保守区域扩增 采用RNA plant plus植物总RNA提取试剂盒(天根公司)提取重度缺磷处理24 h的32号磷高效杉木根系总RNA,并使用Fermentas公司RT-PCR试剂盒合成cDNA第1条链。
根据油茶(GenBank: EU496869.1)、拟南芥(GenBank: AB005746)、玉米(GenBank: NM001111799.1)等植物PHT1基因氨基酸序列中的保守区,利用Primer Premier 5.0软件设计简并引物ClPht1-R1∶5′-GTGCTGAATGCACTCGATGT-3′和ClPht1-F1∶5′-CCTAGCTGGGAAAATCTCAGC-3′对杉木根系cDNA进行PCR扩增,反应程序为: 95 ℃预变性3 min; 94 ℃ 变性40 s,54 ℃退火40 s,72 ℃ 延伸100 s,35个循环; 72 ℃延伸 7 min。扩增产物经天根公司DNA凝胶回收试剂盒回收、纯化后,连接到pGEM-T Easy(TaKaRa公司)载体上进行测序,获得目的基因的核心片段。
1.2.2ClPht1;1基因全长cDNA的获得 采用Invitrogen公司5′RACE(Version2.0)试剂盒,设计特异引物(5′-1:5′-TCGGAGGCAGAGAACC-3′; 5′-2:5′-TCCCAGGAGCCTCGTCAC-3′; 5′-3:5′-GCAGAAG AGGTCATACGCATC-3′)克隆目的基因的5′端序列。使用Superscript Ⅱ RT 酶和引物5′-1对总RNA进行目的基因第1链cDNA的合成,使用引物5′-2和桥连铆钉引物AAP(5′-GGCCACGCGTCGACTAGT ACGGGIIGGGIIGGGIIG-3′)对已经加dC尾的cDNA进行PCR第1轮扩增,使用引物5′-3和桥连通用扩增引物AUAP(5′-GGCCACGCGTC GACTAGTAC-3′)进行巢式PCR第2轮扩增,扩增产物连接载体及测序过程同上。
采用Clontech 公司的SMARTerTMRACE试剂盒,设计特异引物(3′-1:5′-ACCTGGTTTCTTCT GGACATCGCCTACT-3′; 3′-2: 5′-AACCTGTTTCAA AAGGACATCTTCACGG-3′)克隆目的基因的3′端序列。使用逆转录酶SmartScribeTMReverse Transcriptase 和引物3′CDS primer A(5′-AAGCAGTGGTATC AACGCAGACTAC(T)30 V N-3′)对总RNA进行逆转录合成cDNA,并以此cDNA为模板,使用引物3′-1和UPM(5′-CTAATACGACTCACTATAGGGCA AGCAGTGGTATCAACGCAGAGT-3′)进行第1轮PCR扩增。扩增产物稀释50倍后,用引物3′-2和 UPM进行第2轮PCR扩增,扩增产物连接载体及测序过程同上。
根据获得的目的基因核心片段、3′末端和5′末端3 个序列信息,利用Vector NTI10.3.0软件进行序列拼接,获得目的全长基因的cDNA 序列。设计上游引物QC288F:5′-TTGAACCATGGCGGAGAA GC-3′和下游引物QC288R:5′-CTAAGCTGGT GCGGTTCTGG-3′,采用Takara Taq polymerase对目的基因全长进行PCR扩增,反应程序为94 ℃预变性5 min; 94 ℃变性60 s,50 ℃退火60 s,72 ℃ 延伸180 s,30个循环; 72 ℃延伸 10 min。扩增产物连接载体及测序过程同上。
1.2.3ClPht1;1基因序列分析 通过DNAMAN软件预测测序所得目的基因编码的氨基酸序列,并使用ORF Finder预测杉木PHT1基因的最长开放阅读框。使用NCBI在线Blast检索其他植物PHT1基因编码氨基酸序列,并采用Clustal X2.1软件进行多序列比对。使用ProtParam软件和ProtScale软件预测目的基因编码蛋白质的分子量、等电点、不稳定系数、疏水性等理化性质; 使用TMpredServer软件和MobylePortal软件预测目的基因编码蛋白质的跨膜结构和拓扑结构; 使用SOPMA软件和Swiss-model软件预测目的基因编码蛋白质的二级和三级结构。
1.3ClPht1;1基因表达分析
采用实时荧光定量PCR检测目的基因在32号磷高效杉木家系根、茎、叶中的表达情况; 检测中度缺磷胁迫下目的基因在不同磷利用效率杉木4号(1.14±0.08)kg·g-1、15号(1.10±0.06)kg·g-1、25号(1.77±0.22)kg·g-1、27号(1.38±0.03)kg·g-1、28号(0.73±0.06)kg·g-1、32号(1.09±0.02)kg·g-1家系根系中的表达差异; 检测中度、重度供磷胁迫下0,12,24,48 h,以及恢复正常供磷12,24,48 h目的基因在32号磷高效杉木家系根系中的表达量变化,以上试验均设置3次生物学重复。实时荧光定量PCR在Roch480荧光定量PCR仪上进行,荧光染料采用TakaRa公司SYBR Green PCR Master Mix,上游引物5′-GGCCATTCCGTACAACCACTG-3′,下游引物5′-AATTCTTGACCCCAATACCCT-3′。 使用β-Actin基因作为内参基因,其上游引物为5′-TACATTGCCGGTGTATTGAACGTC-3′,下游引物为5′-AGTTGCTCCAGAAGAACATCC-3′。PCR反应程序为95 ℃预变性3 min; 95 ℃变性20 s,60 ℃退火30 s,72 ℃延伸30 s,45个循环; 95 ℃ 30 s,60 ℃ 30 s,逐渐升温到95 ℃,速度0.11 ℃·s-1。扩增结果采用2-△△Ct法计算目的基因的相对表达水平。
2 结果与分析
2.1ClPht1;1基因全长克隆
以重度缺磷处理24 h的32号磷高效杉木根系cDNA为模板,采用设计的简并引物ClPht1-R1和ClPht1-F1进行PCR扩增测序,获得1个长度为1 004 bp的杉木PHT1基因保守片段序列(图1A)。根据获得的杉木PHT1基因保守序列分别设计目的基因的5′和3′ RACE特异引物,5′RACE-PCR扩增得到长度为250 bp的片段(图1B),3′RACE-PCR扩增得到长度为1 000 bp片段(图1C)。将目的基因保守序列、5′RACE片段和3′RACE片段拼接后获得全长为1 967 bp的基因序列。依据拼接获得的基因序列设计全长引物H1和H2,扩增目的基因全长并测序验证,在预测位置获得单一清晰的目的条带(图1D),测序结果与拼接的序列一致,命名为杉木高亲和磷转运蛋白基因ClPht1;1(GenBank登录号: KX302006)。
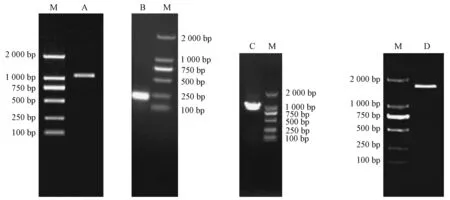
图1 杉木PHT1基因保守片段(A)、cDNA 5′RACE(B)、cDNA 3′RACE(C)和全长基因(D)Fig.1 Cunninghamia lanceolata PHT1 gene conservative fragment (A), cDNA 5′RACE (B), cDNA 3′RACE (C) and full length gene (D)
2.2ClPht1;1基因生物信息学分析
2.2.1ClPht1;1基因序列分析ClPht1;1基因(GenBank登录号: KX302006)全长序列分析表明,其长度为1 967 bp,开放阅读框为1 638 bp,编码1个含545个氨基酸的蛋白质,编码蛋白分子量为59.95 kDa,等电点为9.04。ClPht1;1基因编码多肽的原子组成为C2750H4198N698O751S28,不稳定系数是33.44,表明其为稳定蛋白质分子。ClPht1;1基因的脂肪系数为82.57,总平均亲水性为0.226,ProtScale 软件分析表明ClPht1;1基因编码蛋白含有较多的疏水区域,为疏水性蛋白(图2)。
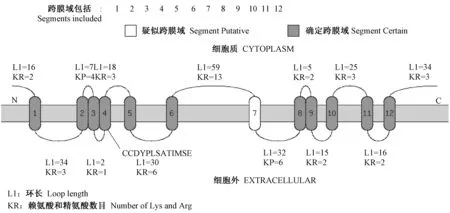
图3 ClPht1;1蛋白跨膜拓扑结构预测Fig.3 Prediction of ClPht1;1 protein transmembrane topology

图2 ClPht1;1疏水性曲线Fig.2 ClPht;1 hydrophobic curve
2.2.2ClPht1;1基因编码蛋白的结构预测 利用在线TMpred Server 和Mobyle portal软件对ClPht1;1基因编码蛋白进行跨膜结构和跨膜区拓扑结构预测,结果表明ClPht1;1基因编码蛋白具有12个跨膜域(TM),其中含1个疑似跨膜域,包括6 个N端的跨膜区以及6个C端的跨膜区,中部(TM6和TM7之间)由1个中央亲水环(hydrophilic loop)分隔(图3)。每个跨膜结构域由17~25个氨基酸残基组成的螺旋构成,跨膜蛋白的N 端、C端和中间的大环都分布在细胞质中,保守序列位于第4个跨膜域,ClPht1;1基因编码蛋白的跨膜区结构与其他物种PHT1基因编码蛋白的跨膜区结构基本一致(Nussaumeetal., 2011)。使用SOPMA软件和Swiss-model软件对ClPht1;1基因编码蛋白质结构进行分析,结果表明其蛋白质二级结构中41.65%为α-螺旋,26.42%为与蛋白质功能相关的无规则卷曲,21.47%为延伸链,10.46%为β-转角; 三级结构中构成蛋白质的主要骨架是α-螺旋(图4); 保守域分析表明ClPht1;1基因编码蛋白有典型的MFS结构域。
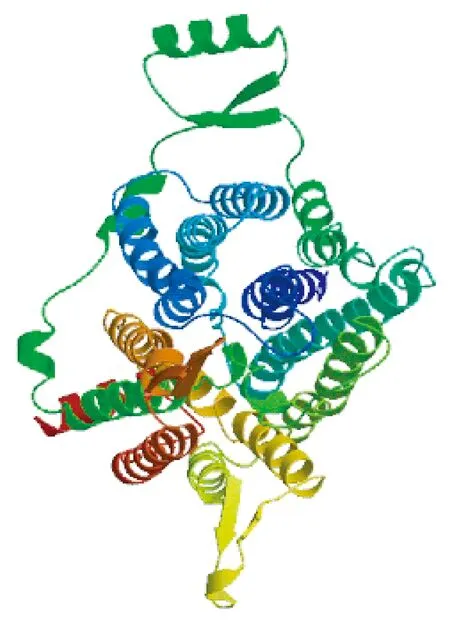
图4 ClPht1;1蛋白三级结构预测Fig.4 Prediction of ClPht1;1 protein tertiary structure
2.2.3 ClPht1;1蛋白序列同源性分析 使用ClustalX2.1软件对ClPht1;1基因和Blast获得的其他植物PHT基因编码氨基酸序列进行多重比对(图5)。结果表明,ClPht1;1基因编码产物与日本柳杉(Cryptomeriajaponica)PHT基因编码氨基酸序列的相似性为87%,与其他植物PHT基因编码氨基酸序列的相似性均在70%以上。其中,与马尾松(Pinusmassoniana)和可可树(Theobromacacao)等南方乔木PHT基因编码氨基酸序列相似性达到77%,与油茶、葡萄(Vitisvinifera)、胡杨(Populuseuphratica)等相似性达到76%。
2.3ClPht1;1基因的表达分析
采用实时荧光定量PCR检测正常供磷情况下ClPht1;1基因在32号磷高效杉木家系根、茎、叶中的表达情况,结果表明ClPht1;1基因在杉木各组织部位均有表达,但在根中的表达量要远高于茎和叶,在叶片中的表达量较低(图6)。中度缺磷胁迫(0.5 mmol·L-1KH2PO4)下,ClPht1;1基因在不同磷利用效率杉木4号(1.14±0.08)kg·g-1、15号(1.10±0.06)kg·g-1、25号(1.77±0.22)kg·g-1、27号(1.38±0.03)kg·g-1、28号(0.73±0.06)kg·g-1、32号(1.09±0.02)kg·g-1家系根系中的表达量为25号>27号>4号>15号>32号>28号(图7),表明ClPht1;1基因在杉木根部的表达量随着不同家系杉木磷利用效率的增强而升高。中度、重度(0 mmol·L-1KH2PO4)缺磷胁迫下ClPht1;1基因在32号杉木家系根系中的表达量随诱导时间(0,12,24,48 h)的延长均呈现明显的上调趋势; 恢复供磷12 h后ClPht1;1基因表达量明显下降,并在恢复供磷24 h和48 h后呈现与正常供磷培养相近的表达量水平。重度缺磷胁迫下,ClPht1;1基因表达量要高于其在中度缺磷胁迫下的表达量,表明ClPht1;1基因的表达明显受介质低磷胁迫诱导(图8)。


图5 ClPht1;1及部分植物PHT基因编码蛋白的氨基酸序列多重比对Fig.5 Multiple alignments of the amino acid sequences of ClPht1;1 and some plant PHT genes encoding proteins罗马数字Ⅰ-Ⅻ表示12 个跨膜区,虚框内表示疑似跨膜区; ■表示N-糖基化位点; ▲表示蛋白激酶C磷酸化位点; ★表示酪蛋白激酶Ⅱ磷酸化位点。Roman numerals Ⅰ-Ⅻ represent 12 transmembrane regions, virtual boxes represent the suspected transmembrane areas; ■ represents N-glycosylation site; ▲ represents protein kinase C phosphorylation site; ★ represents casein kinase Ⅱ phosphorylation site.

图6 ClPht1;1在32号杉木不同组织中的表达分析Fig.6 Expression analysis of ClPht1;1 in different tissues of Cunninghamia lanceolata No. 32
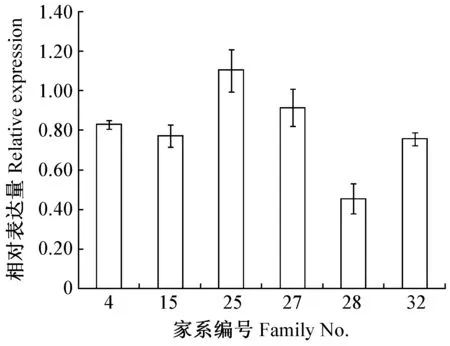
图7 ClPht1;1在不同家系杉木根部的表达分析Fig.7 Expression analysis of ClPht1;1 in roots in different families of Cunninghamia lanceolata

图8 不同磷胁迫和处理时间下ClPht1;1在32号杉木根系的表达分析Fig.8 Expression analysis of ClPht1;1 in roots of Cunninghamia lanceolata No. 32 under different P stresses at different treatment time
3 讨论
PHT1 基因家族成员大多主要在植物根部表达,一些PHT1 基因不仅在植物根部表达,还在植物的茎、叶、花、块茎和子叶中表达,这些PHT1基因可能涉及到植株体内的磷转运功能(Liuetal., 2005)。在拟南芥中,PHT1;1和PHT1;9均受缺磷胁迫的诱导表达,除了PHT1;6在花粉中表达外,其他成员均主要在根中表达(Bayleetal., 2011)。马铃薯(Solanumtuberosum)StPT1和番茄(Lycopersiconesculentum)LePT1(Nagyetal., 2005)在缺磷或不缺磷的根部和地上部均能大量表达,但缺磷能诱导增强其表达量;StPT2和LePT2则仅在植株的根部受缺磷环境的诱导表达(Leggewieetal., 1997; Liuetal., 1998; Gordon-Weeksetal., 2003)。油茶BhPHT1;4基因在嫩根、老根、茎、新叶、老叶中都有表达,但表达量依次递减(赵彩芝, 2014)。本试验结果表明ClPht1;1基因与大部分植物PHT1基因类似,主要在杉木根部表达,叶片中仅能检测到少量表达信号。ClPht1;1基因在杉木根、茎和叶片中均有表达,说明其不仅参与杉木对土壤磷的吸收,而且可能参与杉木体内磷素的转运。植物对磷的转运效率受细胞膜上磷转运蛋白的数量及其对磷的亲和性决定,在中度缺磷胁迫下,ClPht1;1基因在不同磷利用效率4号、15号、25号、27号、28号、32号家系杉木根系中的表达量存在差异,表达量随着不同家系杉木磷利用效率的增强而升高,这可能与不同磷利用效率杉木根部细胞膜表面ClPht1;1磷转运蛋白数量多少有关。ClPht1;1基因的表达量随根系缺磷胁迫时间的延长和胁迫程度的加重而升高,在恢复供磷培养后表达量下降逐渐至正常水平,表明ClPht1;1基因的表达受低磷胁迫的诱导。综上研究,ClPht1;1基因的时空表达情况符合PHT1家族基因的表达特性,对杉木PHT1基因的表达量分析为磷高效转运基因型的筛选提供了参考。
4 结论
磷是我国杉木人工林产量和质量的主要限制因子,由于南方红壤区土壤有效磷长期不足,杉木长期处于低磷的生长环境中。本试验从32号磷高效利用杉木家系的根部克隆获得一个高亲和磷转运蛋白基因ClPht1;1,其具有植物PHT1基因家族的典型结构特点和表达模式。对于长期处于低磷环境中的杉木,ClPht1;1在杉木高亲和转运土壤低浓度有效磷中起到重要的作用。目前,杉木PHT1基因家族的成员组成和各自的磷转运功能仍有待研究,深入解析杉木高亲和磷转运蛋白基因的结构和功能,对于了解杉木磷吸收的代谢机制,培育磷高效利用杉木品种和增强林分磷素管理都具有重要意义。
贾兴永, 李菊梅. 2011. 土壤磷有效性及其与土壤性质关系的研究. 中国土壤与肥料, (6): 76-82.
(Jia X Y, Li J M. 2011. Study on the relationship between soil phosphorus availability and soil properties. Soil and Fertilizer in China, (6): 76-82. [in Chinese ])
李 慧,丛 郁,常有宏,等. 2013. 豆梨磷转运蛋白质基因(PcPht1)的克隆、表达及启动子分析. 江苏农业学报,29 (4): 842-850.
(Li H, Cong Y, Chang Y H,etal. 2013. Cloning, expression and promoter analysis of phosphorus transport protein gene (PcPht1) of soybean and pear. Jiangsu Journal of Agricultural Sciences,29 (4): 842-850. [in Chinese ])
李立芹. 2011. 农作物Pht1 家族磷转运体蛋白的生物信息学分析. 作物杂志,(3): 320-324.
(Li L Q. 2011. Bioinformatics analysisi ofPht1 phosphate transporter protein family in crops. Crops, (3): 320-324. [in Chinese ])
梁 霞,刘爱琴,马祥庆,等. 2006. 不同杉木无性系磷素特性的比较. 植物生态学报,30(6): 1005-1011.
(Liang X, Liu A Q, Ma X Q,etal. 2006. Comparison of phosphorus characteristics of different clones of Chinese fir. Journal of Plant Ecology, 30(6): 1005-1011. [in Chinese ])
汤银辉,何 鹏. 2010. 巴西橡胶树磷转运蛋白基因的克隆及生物信息学分析. 热带作物学报,31(5): 758-766.
(Tang Y H,He P. 2010. Molecular cloning and bioinformatic analysis of a gene encoding a phosphate transporter fromHeveabrasiliensis. Chinese Journal of Tropic Crops,31(5): 758-766. [in Chinese ])
王 策,秦静静,甘红豪,等. 2012. 毛果杨全基因组磷酸根转运蛋白家族成员序列分析. 浙江农林大学学报,29 (4): 516-526.
(Wang C,Qin J J,Gan H H,etal. 2012. Genome wide analysis of phosphate transporter gene family inPopolustrichocarpa. Journal of Zhejiang Ariculure and Forestry University,29 (4): 516-526. [in Chinese ])
王 萍, 陈爱群, 余 玲, 等. 2006. 植物磷转运蛋白基因及其表达调控的研究进展. 植物营养与肥料学报, 12 (4): 584-591.
(Wang P,Chen A Q,Yu L,etal. 2006. Advance of plant phosphate transporter genes and their regulated expression. Plant Nutrition and Fertilizer Science,12 (4): 584-591. [in Chinese ])
汪 攀. 2015. 低磷胁迫下杉木根系形成通气组织与磷利用效率的关系研究. 福州: 福建农林大学硕士学位论文.
(Wang P. 2015. Study on the relationship between the formation of the root system of Chinese fir and the utilization efficiency of phosphorus under low phosphorus stress. Fuzhou: MS thesis of Fujian Agriculture and Forestry University. [in Chinese ])
吴鹏飞,马祥庆,陈友力,等. 2012. 杉木无性系测定林磷素利用效率的比较. 福建农林大学学报: 自然科学版, 41 (1): 40-45.
(Wu P F, Ma X Q, Chen Y L,etal. 2012. Comparison of phosphorus use efficiency among clonal test plantations of Chinese fir. Journal of Fujian Agriculture and Forestry University: Natural Science Edition, 41 (1): 40-45. [in Chinese ])
吴鹏飞. 2009. P高效利用杉木无性系适应环境磷胁迫的机制研究. 福州: 福建农林大学博士学位论文.
(Wu P F. 2009. Adaptation mechanism of Chinese fir clones with high phosphorus-use-efficiency to environmental phosphorus stress. Fuzhou: PhD of Fujian Agriculture and Forestry University. [in Chinese ])
杨存义, 刘 灵, 沈 宏, 等. 2006. 植物Pht1 家族磷转运子的分子生物学研究进展. 分子植物育种, 4(2): 153-159.
(Yang C Y, Liu L, Shen H,etal. 2006. Research progress on molecular biology of phosphorus transport in plantPht1 family. Molecular Plant Breeding, 4(2): 153-159.[in Chinese ])
俞新妥. 2006. 中国杉木研究进展 (2000—2005) I. 杉木生理生态研究综述. 福建林学院学报, 26 (2): 177-185.
(Yu X T. Progress in research on Chinese fir (2000—2005) I. Chinese fir physiological and ecological research review. Journal of Fujian Forestry College, 26 (2): 177-185. [in Chinese ])
赵彩芝. 2014. 油菜高亲和磷转运蛋白基因BhPHT1;4的功能研究. 武汉: 华中师范大学硕士学位论文.
(Zhao C Z. 2014. High affinity phosphate transporter geneBhPHT1;4 function of oilseed rape. Wuhan: MS thesis of Huazhong Normal University. [in Chinese ])
周俊琴,谭晓风,袁 军,等. 2013. 油茶Pht1;2的克隆及生物信息学分析. 生物技术, (1): 37-42.
(Zhou J Q, Tan X F, Yuan J,etal. 2013. Isolation and bioinformatics analysis ofCoPht1;2 from oiltea. Biotechnology, (1): 37-42. [in Chinese ])
Ai P,Sun S,Zhao J,etal. 2009. Two rice phosphate transporters,OsPht1;2 and OsPht1;6,have different functions and kinetic properties in uptake and translocation. Plant Journal for Cell & Molecular Biology, 57 (5): 798-809.
Bayle V, Arrighi J F, Creff A,etal. 2011.Arabidopsisthalianahigh affinity phosphate transporters exhibit multiple levels of posttranslational regulation. Plant Cell, 23(4): 1523-1535.
Calderon-Vazquez C, Ibarra-Laclette E, Caballero-Perez J,etal. 2008. Transcript profiling ofZeamaysroots reveals gene responses to phosphate deficiency at the plant- and species-specific levels. Journal of Experimental Botany, 59(9): 2479-2497.
Gordon-Weeks R, Tong Y, Davies T G,etal. 2003. Restricted spatial expression of a high-affinity phosphate transporter in potato roots. Journal of Cell Science, 116(15): 3135-3144.
Leggewie G, Willmitzer L, Riesmeier J W. 1997. Two cDNAs from potato are able to complement a phosphate uptake-deficient yeast mutant: Identification of phosphate transporters from higher plants. Plant Cell, 9(3): 381- 392.
Liu C M, Muchhal U S, Uthappa M,etal. 1998. Tomato phosphate transporter genes are differentially regulated in plant tissues by phosphorus. Plant Physiology, 116(1): 91-99.
Liu J Q,Samac D A,Bucciarelli B,etal. 2005. Signaling of phosphorus deficiency induced gene expression in white lupin requires sugar and phloem transport. Plant Journal, 41 (2): 257 -268.
Mitsukawa N,Okumura S,Shirano Y,etal. 1997. High-affinity phosphate transporter genes ofArabidopsisthaliana. Proceedings of the National Academy of Sciences, 94(13): 7098-7102.
Nagy R, Karandashov V, Chague V,etal. 2005. The characterization of novel mycorrhiza-specific phosphate transporters fromLycopersiconesculentumandSolanumtuberosumuncovers functional redundancy in symbiotic phosphate transport in solanaceous species. Plant Journal, 42 (2): 236-250.
Nussaume L, Kanno S, Javot H,etal. 2011. Phosphate import in plants: focus on thePHT1 transporters. Frontiers in Plant Science, 2(12): 1085-1091.
Poirier Y, Jung J. 2015. Phosphate transporters// Plaxton W C, Lambers H. Annual plant reviews Volume 48: Phosphorus metabolism in plants. Wiley,125-158.
Preuss C P,Huang C Y,Gilliham M,etal.2010. Channel-like characteristics of the low-affinity barley phosphate transporterPHT1;6 when expressed inXenopusoocytes.Plant Physiology,152(3): 1431-1441.
Scheible W R, Rojas-Triana M. 2015. Sensing, signaling, and control of phosphate starvation in plants: molecular players and applications// Plaxton W C, Lambers H. Annual plant reviews Volume 48: Phosphorus metabolism in plants. Wiley, 25-64.Wu Z Y,Zhao J M,Gao R F,etal. 2011. Molecular cloning, characterization and expression analysis of two members of thePht1 family of phosphate transporters inGlycinemax. PLoS One, 6 (6): e19752.Yang X J,Finnegan P M. 2010. Regulation of phosphate starvation responses in higher plants. Annals of Botany, 105(4): 513-526.
(责任编辑 徐 红)
Cloning and Expression Analysis of Phosphate Transporter GeneClPht1;1 inCunninghamialanceolata
Su Shuoshuo1Li Ming1,2Wu Pengfei1,2Zhang Ying1Ma Xiangqing1,2
(1.College of Forestry, Fujian Agriculture and Forestry University Fuzhou 350002; 2.State Forestry Administration Engineering Research Center of Chinese Fir Fuzhou 350002)
【Objective】PHT1 gene family is one of the important phosphorus transporters which affect the uptake of phosphorus in plants. ACunninghamialanceolataphosphate transporter gene ofPHT1 gene family was cloned from No.32 phosphorus efficientC.lanceolatacDNA,and the temporal and spatial expression ofClPht1;1 under different levels of phosphorus stress was studied to lay the foundation for the research ofPHT1 gene sequence characteristics, functional structure and the selection of theC.lanceolatagenotype which is efficient in phosphorus utilization.【Method】According to the designed degenerate primer based on the conserved sequence ofPHT1 gene family, the target geneClPht1;1 cDNA sequence was obtained by amplification of the root cDNA of the P-efficient family No.32 ofC.lanceolataas the template, the full length of the target gene was cloned by RACE technique, and analyzed its sequence characteristics, homology and protein structure were then analyzed. The real-time fluorescence quantitative PCR was used to detect the expression ofClPht1;1 in roots, stems and leaves of No.32C.lanceolatawith high P-efficiency, differential expressions ofClPht1;1 in the roots ofC.lanceolatafamilies of No.4,No.15,No.25,No.27,No.28 and No.32 in different P-utilization efficiency under moderate P deficiency stress, and theClPht1;1 expression change with time in the root system of phosphorus efficientC.lanceolataNo.32 under moderate and severe phosphorus deficiency stress.【Result】AC.lanceolataPHT1 gene was cloned and namedClPht1;1 (GenBank accession number KJ493165). The coding region of the gene sequence was 1 638 bp, code 545 aa protein.ClPht1;1 encoded proteins were composed of 12 hydrophobic transmembrane domains, including 1 suspected transmembrane domains. Each transmembrane domain was composed of 17-25 amino acid residues, and the N and C ends of transmembrane proteins were located in the cytoplasm, and the conserved sequences were located in the fourth transmembrane domains. The main skeleton of the protein was α-helix, no signal peptide sequence. The similarity of amino acid sequences of encoding proteins ofClPht1;1 was 87% withCryptomeriajaponicaPHTgene, and above 75% withPopulus,Camellia,Pinusmassonianaetc.PHTfamily gene.ClPht1;1 genes were expressed in root, stem and leaf ofC.lanceolata, and the highest expression was in root,the lowest was in leaf; Under moderate P deficiency stress, the expression level ofClPht1;1 in the root ofC.lanceolatavaried among families in a order of No.25>No.27>No.4>No.15>No.32>No.28. Under moderate and severe P deficiency stresses, the expression ofClPht1;1 in the root of No.32 increased with the extension of stress time; The expression level ofClPht1;1 was gradually decreased to normal level after the recovery of phosphorus supply;ClPht1;1 gene expression in severe P deficiency stress was higher than that under moderate P deficiency stress.【Conclusion】ClPht1;1 gene has the typical structure ofPHT1 gene family, and its protein amino acid sequence is highly similar to the amino acid sequences ofCryptomeriajaponicaphosphate transporter protein, it is a member of the high affinity phosphate transporterPHT1 gene family ofC.lanceolata.ClPht1;1 gene was mainly expressed in root ofC.lanceolata, and its expression was lower in leaf. The stronger the phosphorus use efficiency, the higher theClPht1;1 gene expression in its roots.There were large differences in the expression level ofClPht1;1 gene among families with different phosphorus use efficiencies.ClPht1;1 gene expression was induced by low phosphorus stress; Under P deficiency stress, the expression level ofClPht1;1 was significantly increased, and the expression ofClPht1;1 gene was significantly decreased after the recovery of phosphorus supply.
Cunninghamialanceolata; phosphate transporter;PHT1;1; RACE; RT-qPCR; phosphorus use efficiency
10.11707/j.1001-7488.20170505
2016-06-15;
2016-08-12。
国家自然科学基金海峡联合基金项目(U1405211); 国家林业局杉木工程技术研究中心科技成果孵化基金(6213C0111)。
S718.46
A
1001-7488(2017)05-0033-10
*李明为通讯作者。
