基于神经网络的微生物生长环境关系抽取方法*
2017-06-21王健李虹磊林鸿飞杨志豪张绍武
王健 李虹磊 林鸿飞 杨志豪 张绍武
(大连理工大学 计算机科学与技术学院, 辽宁 大连 116024)
基于神经网络的微生物生长环境关系抽取方法*
王健 李虹磊 林鸿飞 杨志豪 张绍武
(大连理工大学 计算机科学与技术学院, 辽宁 大连 116024)
提出一种基于神经网络的方法实现细菌和栖息地的关系抽取,充分利用神经网络的特性实现对隐含的深层特征的自动学习,以避免传统人工特征设计的复杂性和冗余性.该方法利用单词以及实体属性的分布式向量丰富句法和语义信息,使用两个不同神经网络模型从不同角度进行关系抽取,并融合文档级别的分类结果,在生物医学自然语言处理BioNLP-ST 2016共享任务的BB-event语料上进行实验,取得了不错的F1值,表明该方法在微生物生长环境关系抽取上具有良好的性能.
微生物生长环境关系抽取;卷积神经网络;长短时记忆神经网络;分布式向量
生物领域内的文献数量急剧增长,自动准确地从这些文献中抽取出有价值的信息,构建完善的信息检索系统,对于辅助生物学家利用其中潜在的信息进行相关研究来说至关重要.因此,致力于识别生物实体之间的关系或事件的生物信息抽取技术吸引了大量研究学者的注意[1].然而,随着信息抽取技术的日益成熟,通用的信息抽取,如蛋白质关系抽取,已不能满足生物学家的需求,所以研究者的焦点也慢慢转移到了涉及生物多样性知识的横向问题上.例如,基因调控、疾病、代谢和环境等多种生物学问题在文献中出现得非常广泛[2],但是几乎不能以结构化的形式展现给生物学家,造成定位所需的有用信息非常困难,在一定程度上影响了从生物文献中挖掘潜在的知识,因此,针对这种具体的生物学问题进行特定的信息抽取研究,对生物学领域的研究有着重要的作用.
现如今,针对非结构化文本的信息抽取技术已经应用到了生物学的多个领域中,许多生物信息抽取系统应运而生.近年来出现了许多用于蛋白质关系抽取的公开评测任务,如LLL[3]、BioCreative[4]等,在此背景下就产生了许多蛋白质交互作用关系抽取系统[5],对于蛋白质复杂网络的构建、预测隐含的蛋白质关系有重要应用.进一步地,2009年首次提出了关于生物医学事件抽取的BioNLP共享任务,与简单的蛋白质关系抽取等二元关系抽取任务不同的是,该任务旨在抽取细粒度的生物实体之间的复杂关系,如催化、基因表达以及磷酸化等,最具有代表性的事件抽取系统是UturKu,该系统将事件抽取任务视为多分类问题,使用基于机器学习的方法人工设计大量特征,采用SVM模型进行事件抽取,F1值达到51.95%,在所有参赛队伍中位居第一[6],并且该系统的设计思路在BioNLP 2011和BioNLP 2013任务中被许多参赛队伍所沿用并改进,都取得了不错的性能.然而,标注数据的规模限制了传统机器学习方法的进一步提升,所以为能够利用包含领域知识的大量未标注语料,文献[7]提出了基于对偶分解和词向量的方法进行事件抽取.词向量能够从大量未标注语料中学习到丰富的语义特征,将其集成到大量的传统特征中可以更好地检测事件,在BioNLP2013语料中F1值有2.22%的提高.
为更好地适应生物学家的需求,生物信息抽取领域提出了许多新的热点问题,如癌症遗传学、通路管理、基因调控网络构建、微生物生长环境关系抽取等生物领域的热点.其中,针对微生物生长环境的识别就是在此背景下产生的,它旨在从生物文献中自动地抽取微生物和栖息地之间的复杂关系,这不仅对构建全面的、可理解的细菌及其栖息地的关系数据库有重要指导作用,而且能够促进微生物、健康科学和食物加工等领域的发展与实际应用.2011年,Alvis系统[8]利用大量语言学和词法学知识,通过人工设计模式进行关系匹配,实现细菌和栖息地的关系抽取;Uturku系统[9]将微生物生长环境抽取看作二分类问题,抽取大量特征,利用机器学习方法进行关系抽取.
总体来看,针对微生物生长环境关系抽取任务的方法主要分为基于规则的方法和基于机器学习的方法[10].这两种方法都需要人工设计大量的规则和特征,精心选择分类器,而且没有利用未标注语料,存在一定的局限性,因此文中提出了一种基于神经网络的微生物生长环境关系抽取方法,实现特征的自动学习,避免了过多的人工干预,同时能够利用大量未标注语料中的领域知识.首先,运用word2vec工具[11]训练收集到的PubMed文献摘要,得到单词的分布式向量表示,同时对实验语料集进行分句、确定实体在句中位置等预处理.其次确定细菌-栖息地实体对,并通过实体对之间的单词以及实体属性特征,共同构建候选实例.之后使用分布式向量初始化候选实例,作为卷积神经网络(CNN)和长短时记忆神经网络(LSTM)的输入,进行特征自动学习并通过softmax分类器[12]进行分类,并将分类结果进行融合,得到最终答案文档.最后在BioNLP-ST 2016评测任务的BB-event数据集上进行实验.
1 抽取方法概述
文中提出的基于神经网络的微生物生长环境关系抽取系统流程如图1所示,主要分为3个模块:输入实例表示(分布式向量学习和候选实例构建)、神经网络模型(CNN和LSTM)训练、分类结果融合.

图1 系统流程图
1.1 输入实例表示
在机器学习以及深度学习方法中,输入对于模型学习来说至关重要.为了从不同角度丰富输入表示,文中分别从句子级别和文档级别两个角度对输入实例建模,减少了跨句子关系的缺失;同时舍弃了大量的人工设计特征,仅使用实体对上下文信息、实体属性特征以及分布式向量初始化来对输入实例建模.
(1)学习分布式向量.从PubMed数据库中下载大量的文献摘要,构成训练词向量的输入语料.对语料进行过滤等预处理之后,利用word2vec工具对收集到的语料进行词向量的训练,最后得到蕴含丰富句法和语义信息的单词的分布式向量表示[13].
(2)对实验语料进行预处理.由于跨句子的关系存在的比例较少,而且鉴于跨句子关系处理的复杂性,初步实验设计只考虑单个句子中的关系抽取,称为Sen级别的关系抽取,即先对实验语料进行分句,然后以句子为单位确定细菌-栖息地实体对.
此外,为了在一定程度上弥补跨句子的关系缺失,实验增加了文档级别的关系抽取,称为Doc级别的关系抽取,即不对语料进行分句,以全文档为单位确定细菌-栖息地实体对.为了简化实验的复杂性和避免负例的大幅度增加,实验不考虑指代消解,并且如果细菌和栖息地之间的单词距离过大,实体对将会被过滤.
(3)提取实体属性特征.实体本身的信息对于明确实体对之间的关系有很重要的影响.实体的类别信息、实体对之间的单词距离信息,可以指明实体对之间的亲疏关系,将作为补充信息加入候选输入实例的构建中,称作实体属性特征,记为T=(a1,a2,…,am),m为实体属性特征的数量.
(4)模型输入实例表示.仅选取实体对及其之间的单词以及实体对的属性特征作为候选实例表示,简化了人工特征设计的复杂性,并通过明确候选实例实体本身的属性来提高模型对关系的辨别能力.同时,使用分布式向量初始化候选实例中的单词,使其在输入模型之前就保留有单词之间的原始语义信息,促进模型更好地进行特征自动学习.
最后,得到的模型输入实例的向量化表示为(e1,x1,x2,…,xj,…,xn,e2,T),其中e1和e2是两个实体向量,(x1,x2,…,xj,…,xn)是实体对之间的单词向量表示,T=(a1,a2,…,am),是该实例对应的实体属性特征的特征向量表示.在实验中,实体属性特征向量随机初始化,并在训练过程中进行动态调整.
1.2 神经网络模型
近几年,随着深度学习模型的不断成熟与发展,它已成功应用到了不同的研究领域,如图像分类、音视频识别[14]、自然语言处理[15]等,并且都取得了比较好的效果,在生物领域的信息抽取中同样引起了广泛的关注,文中选取卷积神经网络和循环神经网络两个深度学习模型框架进行生物领域的微生物生长环境关系抽取.下面简要介绍一下这两种深度学习模型的相关知识.
1.2.1 卷积神经网络
数学上定义的卷积,是指其中一个函数翻转并平移后与另一个函数的乘积的积分,是一个对平移量的函数.对于函数y和函数g的卷积,可以表示为
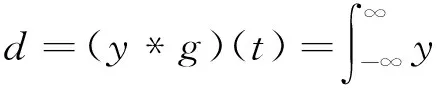
(1)
卷积操作被看作是滑动平均的推广,在实际应用中,可以减少局部数据或信号源的噪音干扰,使得获取的信息最有效.卷积神经网络[16]中加入了卷积操作,与数学上的卷积有异曲同工之妙.利用卷积核去获得局部数据的最显著特征,然后通过组合局部低层特征形成更高层的特征.如式(2)所示:
dj=σ(WTxj∶j+w-1+b)
(2)
式中:W代表一种卷积核,xj∶j+w-1代表窗口大小为w的文本的分布式向量[xj,xj+1,…,xj+w-1],b为偏倚项,σ为激活函数;通过卷积操作,得到该窗口下文本的特征值dj.滑动窗口在输入实例上依次滑动就可以得到不同窗口下的特征值,最后卷积神经网络通过池化操作来筛选这组特征值,从而获得最有价值的特征.通常采用的池化操作有两种,一是最大化操作,即选取同一组特征中的最大值;二是平均操作,即对同一组特征值取平均操作.同时,卷积神经网络为了提取不同类型的特征,会通过增加不同的卷积核来学习输入的隐含特征.
1.2.2 循环神经网络
循环神经网络引入了时序和记忆机制,即神经网络学习了历史信息并进行选择性的记忆,对当前时刻的输出产生影响.具体的表现形式为隐藏层之间的节点不再是无连接的,而是有连接的,这样神经网络就会对前面的信息进行记忆并应用于当前输出的计算中.循环神经网络在不断演化和改进中产生了许多不同的模型[17- 18],其中,使用较为广泛且应用成功的模型是LSTM,即长短时记忆模型,它的成功之处在于弥补了传统循环神经网络中不能很好地处理长距离依赖的缺点[19].
LSTM引入了区块的概念,这样的区块可以看成是一种智能网络单元,有记忆不定时间长度内的信息的功能.区块中包含的主要元素有输入门i、忘记门f、输出门o和记忆细胞单元c,3个门用来保护和控制记忆细胞单元,决定输入是否重要、是否被记忆以及是否允许输出[20].对于某一时刻t,给定输入xt,隐藏层节点状态ht的计算和更新操作如式(3)所示:
(3)
其中,σ(·)和tanh(·)是激活函数,W(·)、U(·)、V(·)是权重矩阵,b(·)是偏倚项.
通过LSTM神经网络对原始输入的学习,最终可以学习到输入的特征向量表示,更好地表达原始输入隐含的有价值信息和特征.
1.2.3 模型学习
微生物生长环境关系抽取任务实质上是确定微生物领域中细菌及栖息地之间是否存在“居住”关系的二元分类问题,最终目的是确定出有关系的细菌-栖息地实体对.对于分类问题,传统的方法是使用基于机器学习的方法,这需要人工设计大量的特征,过程复杂且易造成特征冗余.为了避免复杂的人工特征设计,文中利用单词以及实体属性的分布式向量表示,基于神经网络的方法实现特征的自动学习.对于候选输入实例的构建,文中使用实体对之间的上下文信息以及实体属性特征,设计简单且有效,同时用单词的分布式向量初始化输入,使输入包含丰富的语义信息,最终将其输入到神经网络模型中进行特征的自动学习.
文中利用卷积神经网络能够学习局部特征的特性,分别构造句子级别和文档级别的候选输入实例,然后分别输入到包含多个不同窗口的并行卷积层的卷积神经网络中,通过神经网络对隐含特征的自动学习,动态调整原始输入的分布式向量表示和卷积核参数,最终学习到原始输入的特征向量表示,以此作为预测细菌-栖息地实体对是否存在关系的特征表示输入到softmax分类器进行分类.
此外,文中还利用LSTM神经网络模型对候选输入实例进行特征自动学习,利用该模型区块的记忆和时序特性挖掘输入中的潜在句法和语义信息,最终可以将输入实例表示成一个特征向量,使用softmax分类器实现细菌-栖息地关系的分类.
1.3 结果融合
不同的神经网络模型对于特征的学习角度有所差异,卷积神经网络偏向于学习输入中所有可能短语的局部特征,LSTM神经网络更偏向于记忆历史信息对当前单词预测的影响.通过不同神经网络学习的特征向量能够从不同方面突出输入的特点,作为分类依据各有优缺点,产生的结果互相补充.文中采取对分类结果进行简单的并集融合的方法,提高细菌和栖息地关系抽取的整体性能.系统通过增加文档级别的关系抽取方法CNN-Doc来弥补句子级别关系抽取方法CNN-Sen对跨句子关系抽取能力的缺失,通过使用两个不同的深度学习模型CNN和LSTM来弥补单一模型分类性能的局限性,从而减少分类错误的正例.两个不同的模型从不同角度考虑实体之间的关系,各有优点,使得分类出来的结果可以互相补充,从而提高召回率.
2 实验与分析
2.1 数据集与评测指标
文中采用BioNLP-ST2016评测任务的BB-event数据集,该数据集主要描述的是细菌及其栖息地的关系,数据的来源是相关科学文献的英文摘要.数据集包括训练集、调试集和测试集,全部数据均已经标注出细菌和栖息地实体名称及其在文档中的位置,训练集和调试集给出了实体之间的关系类别信息.表1给出了数据集的详细统计信息.
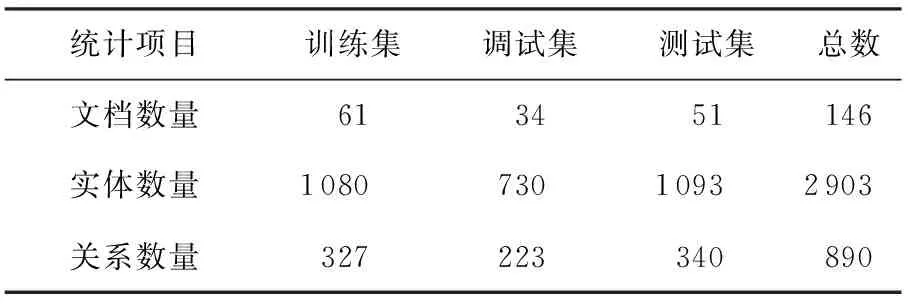
表1 BB-event数据集统计
文中通过BioNLP-ST2016共享任务在线评测平台对分类结果进行度量,评测指标有召回率(R)、准确率(P)和F1值.式(4)给出了各个评测指标的具体计算方法,其中S代表模型预测正确的正例数,N代表语料中的正例数,M代表模型预测的所有正例数.
(4)
2.2 实验结果与分析
为了验证神经网络模型对微生物生长环境关系抽取的有效性,文中同时增加支持向量机(SVM)模型在BB-event语料上进行实验作为比照.此外,文中根据CNN、LSTM和SVM的特点,给定不同的特征输入到模型中.对于CNN和SVM,每个输入实例的原始信息包含实体本身、两个实体之间的单词、实体类别和两个实体间距;对于LSTM,由于其保留历史信息的特点,实例单词之间应该存在关联,所以输入实例的原始信息只保留实体本身及其之间的单词.对于CNN和LSTM两个神经网络模型,都使用单词的分布式向量丰富输入实例的语义信息;而对于SVM模型,使用传统的one-hot特征向量表示原始输入实例.不同模型及其组合在BB-event语料上的具体结果见表2.
表2BB-event测试集上的实验结果
Table2ExperimentalresultsonthetestdatasetsforBB-eventtask
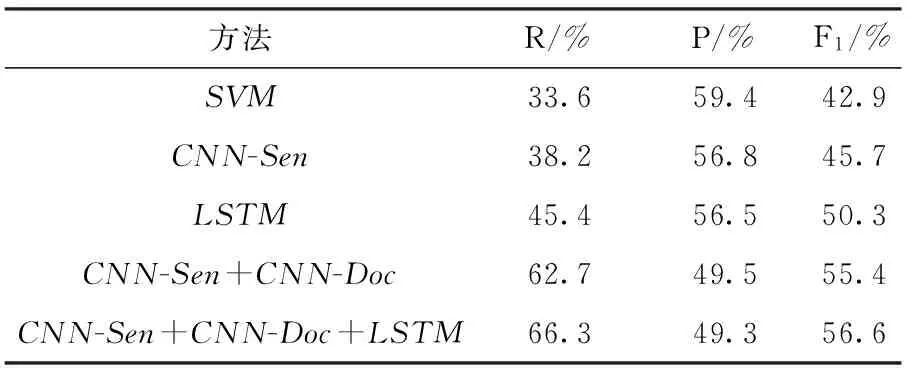
方法R/%P/%F1/%SVM33.659.442.9CNN-Sen38.256.845.7LSTM45.456.550.3CNN-Sen+CNN-Doc62.749.555.4CNN-Sen+CNN-Doc+LSTM66.349.356.6
从表2的实验结果可以看出,神经网络模型CNN-Sen和LSTM的分类效果优于SVM模型,F1值提高幅度分别为2.8个百分点和7.4个百分点,尤其是召回率提升明显.这表明,神经网络模型对于特征的自动学习是有效的,能够挖掘出句子中潜在的深层句法和语义信息,更好地辨别细菌-栖息地之间的关系.对于文中提出的融合文档级别的关系抽取方法CNN-Doc的分类结果的思路,从实验结果来看此方法是有效的,F1值达到了55.4%,相比较CNN-Sen方法,F1值提高了近10个百分点.这表明语料中存在不少的跨句子关系,通过融合文档级别的抽取结果,能够弥补句子级别关系抽取方法CNN-Sen跨句子关系抽取能力的缺失.进一步地,将LSTM模型的分类结果融合在其中,F1值也有所提升,这表明通过使用两个不同的深度学习模型(CNN和LSTM)可以弥补单一模型分类性能的局限性,从而减少分类错误的正例.综上所述,文中提出的结果融合方法在微生物生长环境关系抽取的性能上优于单个模型,也表明了神经网络模型在细菌及其栖息地关系抽取任务上的有效性.
表3给出了BioNLP-ST2016评测中BB-event任务参赛队伍的性能,这里只列出了前三名参赛队伍的结果.从表中可以看出,文中方法在细菌群落栖息地关系抽取上取得了很好的效果,优于其他3个系统,而且在4种系统中,文中方法的召回率最高,再次说明多模型融合方法对于召回率的正向作用.再者,文中没有沿用传统的人工特征设计的方法,而是使用简单的输入、强大的特征自动学习模型,减少了人工干预,具有很好的泛化性和可移植性.从表3的F1值还可以发现,这些系统的F1值都没有超过60%,对于微生物领域的实际应用需求来说,还是远远不够的;这可能是由于细菌及其栖息地的高度多样性、以及语料数量相对小所导致的.所以,对于微生物生长环境关系抽取技术的研究还有很大的改进空间,既是挑战也是机遇,是未来微生物信息抽取领域需要不断努力的方向.
表3 不同方法在BB-event测试集上的实验结果
Table3ExperimentalresultsofdifferentmethodsonthetestdatasetsforBB-eventtask

方法R/%P/%F1/%文中方法66.349.356.6VERSE61.551.055.8TurkuNLP44.862.352.1LIMST64.638.848.5
3 结语
文中提出了一种基于神经网络的微生物生长环境抽取方法,利用细菌和栖息地实体之间的上下文信息、实体属性特征对候选关系实例进行表示,并通过大量未标注语料训练单词的分布式向量表示,用其初始化候选实例的每个单词,使其含有丰富句法和语义信息.其次,文中提出了3种模型实现关系识别,系统通过增加文档级别的关系抽取方法来弥补句子级别关系抽取方法对跨句子关系抽取能力的缺失,通过使用两个不同的深度学习模型来弥补单一模型分类性能的局限性,最后对分类结果进行简单的并集融合的方法,提高了关系抽取的整体性能.对于如何进一步提高系统的准确率,如何解决语料稀疏问题,如何更充分利用神经网络的自主学习能力,进一步地提高微生物生长环境关系抽取的整体性能,是下一步需要探索和深入研究的工作重点.
[1] 肖春,周建龙. 生物医学领域中的文本信息抽取技术与系统综述 [J]. 计算机应用研究,2007,24(9): 1- 7. XIAO Chun,ZHOU Jian-long. Overview of information extraction techniques and systems in biomedical domain [J]. Application Research of Computers,2007,24(9): 1- 7.
[2] NÉDELLEC C,BOSSY R,KIM J D,et al. Overview of BioNLP shared task 2013 [C]∥Proceedings of the Bio-NLP Shared Task 2013 Workshop.Sofia∶Association for Computational Linguistics,2013: 1- 7.
[3] NÉDELLEC C. Learning language in logic-genic interaction extraction challenge [C]∥Proceedings of the 4th Learning Language in Logic Workshop (LLL05).Bonn: ICML,2005: 1- 7.
[4] KRALLINGER M,LEITNER F,RODRIGUEZ-PENAGOS C,et al. Overview of the protein-protein interaction annotation extraction task of BioCreativeII [J]. Genome bio-logy,2008,9(Suppl 2): S4.
[5] MADKOUR A,DARWISH K,HASSAN H,et al. Bio-Noculars: extracting protein-protein interactions from bio-medical text [C]∥Proceedings of the Workshop on BioNLP 2007: Biological,Translational,and Clinical Language Processing.Morristown:Association for Computational Linguistics,2007: 89- 96.
[6] BJÖRNE J,HEIMONEN J,GINTER F,et al. Extracting complex biological events with rich graph-based feature sets [C]∥Proceedings of the Workshop on Current Trends in Biomedical Natural Language Processing:Shared Task.Boulder:Association for Computational Linguistics,2009:10- 18.
[7] LI L,LIU S,QIN M,et al.Extracting biomedical event with dual decomposition integrating word embeddings [J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics (TCBB),2016,13(4): 669- 677.
[8] RATKOVIC Z,GOLIK W,WARNIER P,et al. BioNLP 2011 task bacteria biotope: the Alvis system [C]∥Proceedings of the BioNLP Shared Task 2011 Workshop.Portland:Association for Computational Linguistics,2011:102- 111.
[9] KARADENIZ I,OZGÜR A. Bacteria biotope detection,ontology-based normalization,and relation extraction using syntactic rules [C]∥Proceedings of the BioNLP Shared Task 2013 Workshop. Sofia:Association for Computational Linguistics,2013:170- 177.
[10] BOSSY R,GOLIK W,RATKOVIC Z,et al. BioNLP Shared Task 2013-an overview of the bacteria biotope task [C]∥Proceedings of the BioNLP Shared Task 2013 Workshop.Sofia:Association for Computational Linguistics,2013:161- 169.
[11] MIKOLOV T,YIH W,ZWEIG G.Linguistic regularities in continuous space word representations [C]∥Proceedings of NAACL-HLT 2013.Atlanta:Association for Computational Linguistics,2013:746- 751.
[12] DUAN K,KEERTHI S S,CHU W,et al. Multi- category classification by soft-max combination of binary classifiers[C]∥Proceedings of International Workshop on Multiple Classifier Systems.Guildford:Springer,2003:125- 134.
[13] HINTON G E. Learning distributed representations of concepts [C]∥Proceedings of the Eighth Annual Conference of the Cognitive Science Society.Amherst:Lawrence Erlbaum Associates,1986 :12.
[14] LECUN Y,BENGIO Y,HINTON G. Deep learning [J]. Nature,2015,521(7553):436- 444.
[15] COLLOBERT R,WESTON J,BOTTOU L,et al. Natural language processing (almost) from scratch [J]. Journal of Machine Learning Research,2011,12:2493- 2537.
[16] KIM Y. Convolutional neural networks for sentence classification [C]∥Proceedings of Emprical Methods in Natural Language Processing.Doha:Association for Computational Linguistic,2014:1746- 1751.
[17] SUTSKEVER I,MARTENS J,HINTON G E. Generating text with recurrent neural networks [C]∥Proceedings of the 28th International Conference on Machine Learning (ICML-11).Bellevue:International Machine Learning Society,2011:1017- 1024.
[18] LIU S,YANG N,LI M,et al. A recursive recurrent neural network for statistical machine translation [C]∥ Proceedings of the 52nd Annual Meeting of the Association for Computational Linguistics. Baltimore:Association for Computational Linguistics,2014:1491- 1500.
[19] HOCHREITER S,SCHMIDHUBER J. Long short- term memory [J]. Neural Computation,1997,9(8):1735- 1780.
[20] RAVURI S V,STOLCKE A. Recurrent neural network and LSTM models for lexical utterance classification [C]∥Proceedings of Sixteenth Annual Conference of the International Speech Communication Association. Dresden:International Speech Communication Association(ISCA),2015:135- 139.
Bacteria Biotope Extraction on the Basis of Neural Network
WANGJianLIHong-leiLINHong-feiYANGZhi-haoZHANGShao-wu
(School of Computer Science and Technology, Dalian University of Technology, Dalian 116024, Liaoning, China)
Proposed in this paper is a neural network-based method for extracting the relationship between bacteria and their habitats. In this method, implicit senior features are learnt automatically to avoid the complexity and redundancy of the traditional artificial design of features, and, distributed vector representation with rich syntactic and semantic knowledge for words and entities, two different neural network models, as well as integrated document- level extraction results, are comprehensively employed to make an evaluation on the BB- event corpus from BioNLP- ST 2016. Experimental results show that the proposed method achieves preferableF1score, which means that it is effective in bacteria biotope extraction.
bacteria biotope extraction; convolutional neural network; long short- term memory neural network; distributed vector representation
2016- 11- 18
国家自然科学基金资助项目(61572098,61572102,61562080);国家重点研发计划项目(2016YFB1001103) Foundation items: Supported by the National Natural Science Foundation of China(61572098, 61572102, 61562080) and the National Key Research Development Program of China(2016YFB1001103)
王健(1967-),女,教授,博士生导师,主要从事信息检索、文本挖掘和自然语言处理研究.E-mail:wangjian@dlut.edu.cn
1000- 565X(2017)03- 0076- 06
TP 391;TP 18
10.3969/j.issn.1000-565X.2017.03.011
