刺参4个功能基因的表达分析
2017-06-19顾晨雷周晓旭刘洋于海龙孔德荣宋坚仇雪梅常亚青王秀利大连海洋大学水产与生命学院辽宁大连6023大连市水产产业技术创新联合会辽宁大连6023
顾晨雷,周晓旭,刘洋,于海龙,孔德荣,宋坚,仇雪梅,常亚青,王秀利(.大连海洋大学水产与生命学院,辽宁大连6023;2.大连市水产产业技术创新联合会,辽宁大连6023)
刺参4个功能基因的表达分析
顾晨雷1、2,周晓旭1,刘洋1,于海龙1,孔德荣1,宋坚1,仇雪梅1,常亚青1,王秀利1、2
(1.大连海洋大学水产与生命学院,辽宁大连116023;2.大连市水产产业技术创新联合会,辽宁大连116023)
为研究刺参Apostichopus japonicus的生长发育,采用实时定量PCR方法,对刺参腱生蛋白基因、胶原蛋白琢2基因、整合蛋白琢V基因和整合蛋白茁L基因等4个候选功能基因不同月龄的组织表达进行了分析。结果表明:刺参12、13、14月龄时,腱生蛋白基因在刺参肠、管足、呼吸树、皮肤、体壁、体腔液细胞、疣足和纵肌等8个组织中均有不同程度的表达,其中在体腔液细胞和呼吸树组织中的表达量较高(P< 0.05);胶原蛋白琢2基因在刺参管足、呼吸树、体壁、疣足和纵肌组织中的表达量随月龄的增加而逐渐增加,其中在呼吸树组织中表达量最高,在纵肌、疣足、体壁、肠和体腔液细胞等组织中的表达量逐渐降低;整合蛋白琢V基因在纵肌组织中的表达量随月龄的增加而逐渐增加;整合蛋白茁L基因在皮肤、体壁、疣足和管足组织中的表达量较低。本研究结果为深入研究刺参这4个功能基因提供了参考。
刺参;腱生蛋白基因;胶原蛋白琢2基因;整合蛋白琢V基因;整合蛋白茁L基因;表达分析
刺参Apostichopus japonicus是中国海水养殖单种产值最高的经济种类之一。多年来,科研工作者对刺参的生物学进行了全方位的研究,包括刺参的消化生理、生态免疫、行为生态、夏眠、再生、白化、营养和健康苗种繁育等方面,为刺参养殖、繁育、病害防治等更深入地研究提供了坚实的理论基础[1]。此外,国内外许多学者利用分子生物学手段对刺参做了大量的研究工作,取得了较大的阶段性成果。王秀利等[2]、彭薇[3]、Li等[4]、Li等[5]、Du等[6]、Peng等[7]、Yang等[8]在刺参SSRs、AFLP、SNPs的开发与应用中进行了研究。Wang等[9]、丁君等[10]和孙国华等[11]对刺参微卫星标记与经济性状相关性进行了研究,并筛选出连锁位点。孙文静[12]进行了刺参生长性状的QTL分析,并采用复合区间作图法在刺参的连锁群上定位了21个与生长相关性状(体长、体宽、体质量、体壁质量和出肉率)的QTL。刘进等[13]采用流式细胞术测定刺参基因组大小为(880.20依58.68)Mb,刺参体腔细胞的单倍体基因组含量的C-值为(0.90依0.06)pg。聂鸿涛[14]利用微卫星标记构建了刺参遗传图谱。杜慧霞[15]构建了刺参遗传图谱并对刺参夏眠发育相关的转录组进行了分析。鄢荣歆等[16]、杨爱馥等[17]、王艳玲等[18]、Qiu等[19]、Zhou等[20]对刺参组织蛋白酶L基因、microRNAs、转铁蛋白等功能基因进行了克隆与表达研究。Zhao等[21]和Chen等[22]对刺参夏眠时DNA甲基转移酶、甲基化结合域基因、肠中miRNAs的差异表达进行了分析。Sun等[23]构建了刺参肠道和体壁再生时的大规模基因表达谱。还有一些学者对刺参Toll样受体、凝集素、补体、热激蛋白,以及功能酶、多糖、三萜甙等免疫相关基因的克隆、表达及相关活性物质进行了研究[24-33]。
腱生蛋白(Tenascin)、胶原蛋白(Collagen)和整合蛋白(Integrin)是动物蛋白质中的主要成分。腱生蛋白也称细胞黏合素,是由一种星形细胞合成并分泌的细胞外基质蛋白,主要功能是加强结缔组织,调节细胞形态。胶原蛋白是一种生物高分子,是动物结缔组织中的主要成分,占蛋白质总量的25%~30%,在某些生物体中胶原蛋白甚至高达80%以上[34]。整合蛋白又称整合素,是一类普遍存在于动物细胞表面,依赖于Ca2+或Mg2+的异亲型细胞黏附分子,介导细胞和细胞间以及细胞和细胞外基质间的相互识别和黏附,具有联系细胞外部与细胞内部结构的作用[35]。腱生蛋白基因、胶原蛋白基因和整合蛋白基因的表达对动物生长发育具有重要的科学意义和实际应用价值,但这3个蛋白基因在刺参生长发育阶段的表达等方面的文献不多,研究相对零散,还未见系统的研究报道。在前期工作的基础上[36-37],本研究中利用候选基因策略,对腱生蛋白基因、胶原蛋白琢2基因、整合蛋白琢V基因和整合蛋白茁L基因等4个功能候选基因进行分析,旨在为今后深入研究这4个基因的功能及其在刺参健康养殖和育种上的应用提供参考。

表1 本试验中采用的qRT-PCR引物序列Tab.1Primer sequences of qRT-PCR used in the experiment
1 材料与方法
1.1 材料
试验用刺参为大连湾养殖场的同一群体刺参,并在大连海洋大学农业部北方海水增养殖重点实验室暂养。在刺参生长至12、13和14月龄时,随机选择30头刺参,分别采集肠、管足、呼吸树、皮肤、体壁、体腔液、疣足、纵肌等组织,在液氮中保存备用。每个月龄的刺参个体间的同一种组织样品量基本相同,用每10头刺参的相同组织组成1个混合样品(pool),每个生长阶段每种组织均有3个平行的混合样品(pool)。
rTaq DNA聚合酶、dNTPs、M-MLV反转录酶、RNA酶抑制剂、PrimeScriptTMRT reagent Kit反转录试剂盒、SYBR Primix Ex TaqTM域试剂盒、EASY Dilution、RNase free dH2O等均购自大连宝生物有限公司;TransStart襃Top Green Qpcr SuperMix购自全式金生物公司;Trizol试剂购自美国Life公司;其他常规试剂取自大连海洋大学基因工程实验室。
1.2 方法
1.2.1 总RNA的提取提取刺参3个生长阶段(12、13和14月龄)时肠、管足、呼吸树、皮肤、体壁、体腔液、疣足、纵肌等组织的总RNA,对每个生长阶段每种组织的3个混合样品(pool)分别提取总RNA。按照Trizol总RNA提取试剂说明书方法进行总RNA的提取。总RNA质量检测合格后进行下一步试验。
1.2.2qRT-PCR的引物设计根据本研究的前期工作[35,37],对4个功能基因(腱生蛋白基因、胶原蛋白琢2基因、整合蛋白琢V基因和整合蛋白茁L基因),利用生物学软件Primer Premier 5.0进行引物设计。内参基因选择细胞色素B基因,其引物序列参照参考文献[38]。4个功能基因和内参基因的qRT-PCR引物序列见表1。
1.2.3 反转录使用PrimeScriptTMRT reagent Kit试剂盒进行cDNA第一链的合成,反应步骤按试剂盒说明书进行。反转录反应体系(共10 m L):5伊PrimeScriptTMBuffer 2 m L,PrimeScriptTMRT Enzyme Mix玉0.5 m L,Oligo dT Primer(50滋mol/L)0.5 m L,Random 6 mers(100滋mol/L)0.5 m L,Total RNA(100 ng/m L)5 m L,RNase free dH2O 1.5 m L。反转录反应在ABI2720 PCR仪中进行,反应条件如下:37益下反转录反应15 min;85益下灭活5 s,4益下保存。反转录完成后使用核酸分析仪测定cDNA模板浓度。
1.2.4 实时荧光定量PCR在qRT-PCR前,每一个生长阶段刺参各种组织的cDNA调整至相同的浓度。实时荧光定量采用SYBR Green玉嵌合荧光法,选择相对定量中的2-吟吟Ct法分析基因的表达水平。
1.2.4.1 实时定量PCR体系及反应条件实时定量PCR总体系为20 m L,其中包括2伊TransStart襃Top Green qPCR SuperMix 10滋L,上、下游引物(10滋mol/L)各0.4滋L,Passive Reference Dye (50伊)(optional)0.4滋L,模板cDNA 1滋L,无Rnase酶水7.8滋L。反应条件为:95益下预变性30 s;95益下变性10 s,60益下退火和延伸25 s,共进行40个循环。
1.2.4.2 标准曲线的建立采用相对定量2-吟吟Ct法,根据预试验结果选择表达量较高(高拷贝数)且均一稳定的标准品。内参基因和目的基因分别建立一条标准曲线,标准品初始浓度为50 ng/滋L, 10倍稀释,5个稀释点,每个稀释点重复3次。
1.2.4.3 模板的稀释根据预试验结果,最佳模板浓度为12.5 ng/m L。以刺参各组织总RNA反转录成的cDNA浓度为50 ng/滋L,所有待测样品需稀释4倍以达到最佳浓度,取10滋L浓度为50 ng/滋L的cDNA,加入30滋L EASY Dilution,稀释好备用。
1.2.4.4 实时定量PCR使用ABISteponePlus荧光定量PCR仪,按照TransStart襃Top Green qPCR SuperMix实时定量试剂盒说明书配制混合试剂,按照软件程序设定反应条件进行定量PCR。每个反应重复3次,每个基因分别做3个阴性对照。
1.2.4.5 实时定量数据分析实时定量数据分析采用2-吟吟Ct法,功能基因表达的相对浓度计算公式为[38-39]

其中:Ct为荧光信号达到设定域值时经历的循环数;CtA为目的基因的Ct值;CtB为内参基因的Ct值;ExA为目的基因的扩增效率;ExB为内参基因的扩增效率。具体计算过程如下:
(1)计算每个个体的吟Ct,吟Ct=CtA-CtB;
(2)计算各组刺参吟Ct平均值,选择吟Ct平均值最高的一组作为参照组;
(3)计算每个个体的吟吟Ct,公式为
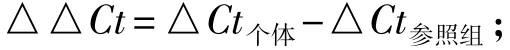
(4)计算每个个体的2-吟吟Ct值,计算结果取平均值和标准偏差。
2 结果与分析
2.1 刺参组织总RNA、内参基因和功能候选基因的标准曲线和溶解曲线
在刺参3个生长阶段中,每一种组织均按试验材料和方法中的步骤进行总RNA的提取。总RNA经琼脂糖凝胶电泳检测以及浓度和纯度测定,总RNA的数量和质量满足后续试验的需要。
以细胞色素B基因为内参基因,对所有组织的4个基因建立标准曲线和溶解曲线。标准曲线斜率(slope)为-3.339,扩增效率(Eff)为96.889%,相关系数(R2)为0.972,相关系数大于0.97,显示了良好的线性关系。溶解曲线峰值单一,未产生非特异性扩增和引物二聚体,反应结果十分理想。腱生蛋白基因、胶原蛋白琢2基因、整合蛋白琢V基因和整合蛋白茁L基因的标准曲线和溶解曲线结果均十分理想。
2.2 刺参4个功能基因在不同生长阶段及不同组织间的差异表达
腱生蛋白基因、胶原蛋白琢2基因、整合蛋白琢V基因和整合蛋白茁L基因分别在刺参12、13、14月龄的差异表达以及在不同组织中的差异表达见图1和图2。在3个月的时间点,腱生蛋白基因在肠、管足、呼吸树、皮肤、体壁、体腔液细胞、疣足和纵肌组织中均有不同程度的表达(图1-A);其中12月龄时,腱生蛋白基因于在体腔液细胞中表达量最高,且显著或极显著高于其他组织(P<0.05或P<0.01),在呼吸树组织中表达量较高(P< 0.05),在纵肌、皮肤、疣足、体壁和肠等组织中的表达量依次降低;13、14月龄时,腱生蛋白基因在呼吸树组织中表达量最高,在体腔液细胞、纵肌等组织中的表达量依次降低。胶原蛋白琢2基因在管足、呼吸树、体壁、疣足和纵肌组织中,随着刺参月龄的增加其表达量逐渐增加,在其他组织中表达量与月龄之间无明显的关系;所有组织中,胶原蛋白琢2基因的表达量在呼吸树组织中最高,且显著或极显著高于其他组织(P<0.05或P< 0.01),在纵肌、疣足、体壁、肠、体腔液细胞等组织中的表达量逐渐降低(图1-B)。12月龄时,整合蛋白琢V基因在体腔液中表达量最高,且显著或极显著高于其他组织(P<0.05或P<0.01);而13、14月龄时,整合蛋白琢V基因在肠组织中表达量最高,在体腔液细胞、呼吸树、纵肌、疣足和体壁组织中的表达量依次降低(图2-A);整合蛋白琢V基因在纵肌和疣足组织中随着月龄的增加其表达量逐渐增加,但在肠、呼吸树、管足、皮肤中,该基因在13月龄时的表达量显著高于在12和14月龄的表达量(P<0.05)。3个生长阶段,整合蛋白茁L基因在体腔液中的表达量显著高于其他组织(P<0.05),在体壁、管足、疣足中的表达量无显著性差异(P>0.05);不同生长阶段,各组织整合蛋白茁L基因表达量无一致趋势(图2-B),但在皮肤、体壁、疣足和管足组织中的表达量较低。

注:相邻、相隔小写字母之间分别表示同一生长时期不同组织之间基因表达量存在显著性差异(P<0.05)和极显著性差异(P<0.01),相邻、相隔大写字母之间分别表示同一组织不同生长时期基因表达量存在显著性差异(P<0.05)和极显著性差异(P<0.01),下同Note:Themeanswith adjacentand apart different letters in different organizations in the same growth period are significant difference(P<0.05)and very significant difference(P<0.01)in genesexpression quantity,respectively,and themeanswith adjacentand apart different capital letters in dif-ferent growth period in the same organizations are significant difference(P<0.05)and very significant difference(P<0.01)in genes expression quantity,et sequentia

图2 整合蛋白琢V基因和整合蛋白茁L基因在刺参不同生长阶段不同组织中表达Fig.2Relative expression of the integrin琢V gene and integrin茁L gene in various tissues of different month old sea cu-cumber Apostichopus japonicus
3 讨论
目前,尽管对刺参腱生蛋白、胶原蛋白和整合蛋白等基因表达方面的研究较少,但仍有部分报道。Ba等[40]克隆了腱生蛋白基因,并对腱生蛋白基因在刺参再生过程中的表达进行了分析,研究结果表明,腱生蛋白基因表达的最高水平出现在刺参去内脏后第20天的肠和呼吸树组织中,以及刺参被损伤后45 min(早期再生)时和7 d(晚期再生)时。原位杂交分析表明,腱生蛋白基因在体壁、纵肌、肠和呼吸树中均有表达,但该研究未分析腱生蛋白基因在管足、皮肤、体腔液和疣足等组织中的表达情况[40]。本研究结果表明,腱生蛋白基因在呼吸树组织中表达量较高,与Ba等[40]的研究结果一致。此外,腱生蛋白基因在体腔液、呼吸树组织中高表达,在其他组织中低表达,造成与Ba等[40]的研究结果不一致,其原因可能是经生物信息学分析,本研究中获得的腱生蛋白基因为腱生蛋白家族一员,但与Ba等[40]克隆的腱生蛋白同源性为50%,故造成表达量及在不同组织表达差别。
本研究表明,8种组织中胶原蛋白琢2基因在刺参呼吸树组织中表达量最高,在疣足组织中表达量相对较低,这与Zhou等[37]的试验结果一致。但胶原蛋白琢2基因在肠、体壁、体腔液细胞和纵肌等组织也有低表达,在管足和皮肤组织中基本不表达,而Zhou等[37]的研究结果表明,胶原蛋白琢2基因在皮肤组织中有较低的表达。
本研究中,整合蛋白琢V基因和整合蛋白茁L基因在肠、管足、呼吸树、体腔液细胞和纵肌等组织中均有不同程度的表达,但在同一种组织中不同月龄时的表达量差异较大。同时,这两个基因在皮肤和疣足组织中表达量较低,该结果与Zhou等[37]的研究结果一致。
本研究中发现,刺参4个功能基因在各组织中的12、13、14月龄时的表达情况较混乱,但大体趋势符合刺参的生物学特性。个别基因在不同组织中和不同月龄时表达情况出现的较大差异,需今后深入研究。当然,个别基因在不同组织中和不同月龄时的表达情况与预期的结果有较大出入,可能有以下原因:(1)本研究中所采用的刺参群体尽管来源于同一群体,但是由多雄和多雌的亲参繁殖后代组成的群体,可能造成群体中的个体差异较大。(2)每个月龄时采集30头刺参,可能是样本量较少造成的误差。(3)所用刺参群体如果存在近交的情况,也可能造成生长发育不规律以及生长势、适应性和抗逆性下降等衰退现象。本研究结果为今后深入研究这4个功能基因的功能及表达规律提供了一定参考。
[1]杨红生,周毅,张涛.刺参生物学——理论与实践[M].北京:科学出版社,2014.
[2]王秀利,单雪,仇雪梅,等.海参微卫星DNA的多态性[J].生物技术通报,2006(6):118-121.
[3]彭薇.仿刺参(Apostichopus japonicus)微卫星标记的开发与应用[D].青岛:中国海洋大学,2011.
[4]LiQ,Chen L,Kong L.A genetic linkagemap of the sea cucumber, Apostichopus japonicus(Selenka),based on AFLP and microsatel-litemarkers[J].Animal Genetics,2009,40(5):678-685.
[5]Li C H,Feng W D,Qiu L H,et al.Characterization of skin ulcera-tion syndrome associated microRNAs in sea cucumber Apostichopus japonicus by deep sequencing[J].Fish&Shellfish Immunology, 2012,33(2):436-441.
[6]Du Huixia,Bao Zhenmin,Hou Rui,etal.Transcriptome sequencing and characterization for the sea cucumber Apostichopus japonicus (Selenka,1867)[J].PLoSOne,2012,7(3):e33311.
[7]Peng Wei,Bao Zhenmin,Du H X,et al.Development and charac-terization of 70 novelmicrosatellite markers for the sea cucumber (Apostichopus japonicus)[J].Genetics&Molecular Research, 2012,11(1):434-439.
[8]Yang Aifu,Sun Dapeng,Liu Shikai,et al.Characterization of fif-teen SNPmarkersbymining EST in sea cucumber,Apostichopusja-ponicus[J].Journal of Genetics,2012,91(1):1-5.
[9]Wang Xiuli,Shan Xue,Qiu Xuemei,etal.Microsatellite DNA poly- morphisms and the relation with body weight in sea cucumber Apos-tichopus japonicus[J].Chinese Journal of Oceanology and Limnolo-gy,2009,27(2):331-336.
[10]丁君,李娇,常亚青.刺参微卫星分子标记与经济性状的相关性分析[C]//2010年中国水产学会学术年会论文摘要集.北京:中国水产学会,2010:251-253.
[11]孙国华,杨建敏,孙孝德,等.刺参微卫星标记与生长性状体重、体长的相关分析[J].水产学报,2011,35(4):501-508.
[12]孙文静.刺参生长相关性状QTL分析[D].青岛:中国海洋大学,2012.
[13]刘进,张晓军,苏琳,等.仿刺参基因组大小的测定[J].水产学报,2012,36(5):686-695.
[14]聂鸿涛.两种经济贝类和刺参微卫星——着丝粒作图及遗传图谱的整合[D].青岛:中国海洋大学,2012.
[15]杜慧霞.仿刺参(Apostichopus japonicus)转录组分析与遗传图谱构建[D].青岛:中国海洋大学,2013.
[16]鄢荣歆,徐赛涛,丛丽娜,等.海刺参组织蛋白酶L基因的克隆、重组表达及活性鉴定[J].大连工业大学学报,2009,28 (6):391-396.
[17]杨爱馥,周遵春,孙大鹏,等.仿刺参铁蛋白ferritin基因的序列分析及表达[J].水产学报,2010,34(6):710-717.
[18]王艳玲,李东,王秀利.海参免疫相关基因的研究进展[J].生物技术通报,2011(9):22-26.
[19]Qiu Xuemei,Li Dong,Cui Jun,et al.Molecular cloning,charac-terization and expression analysisofmelanotransferrin from the sea cucumber Apostichopus japonicus[J].Molecular Biology Reports, 2014,41(6):3781-3791.
[20]Zhou Z C,Dong Y,Sun H J,et al.Transcriptome sequencing of sea cucumber(Apostichopus japonicus)and the identification of gene-associated markers[J].Molecular Ecology Resources, 2014,14(1):127-138.
[21]Zhao Ye,Chen Muyan,Su Lin,et al.Molecular cloning and ex-pression-profile analysis of sea cucumber DNA(Cytosine-5)-methyltransferase 1 and methyl-CpG binding domain type 2/3 genes during aestivation[J].Comparative Biochemistry and Phys-iology Part B:Biochemistry and Molecular Biology,2013,165 (1):26-35.
[22]Chen Muyan,Zhang Xiumei,Liu Jianning,et al.High-throughput sequencing reveals differential expression ofmiRNAs in intestine from sea cucumber during aestivation[J].PLoS One,2013,8 (10):e76120.
[23]Sun Lina,Chen Muyan,Yang Hongsheng,et al.Large scale gene expression profiling during intestine and bodywall regeneration in the sea cucumber Apostichopus japonicus[J].Comparative Bio-chemistry and Physiology Part D:Genomics and Proteomics, 2011,6(2):195-205.
[24]Sun Hongjuan,Zhou Zunchun,Dong Ying,etal.Identification and expression analysis of two Toll-like receptor genes from sea cu-cumber(Apostichopus japonicus)[J].Fish&Shellfish Immunolo-gy,2013,34(1):147-158.
[25]Yuan Zhe,Dahms H U,Han Lulu,etal.Cloning and characteriza-tion of a trypsin-like serine protease gene,a novel regenerationrelated gene from Apostichopus japonicus[J].Gene,2012,502(1):46-52.
[26]Han Lulu,Yuan Zhe,Dahms H U,et al.Molecular cloning,char-acterization and expression analysis of a C-type lectin(AJCTL) from the sea cucumber Apostichopus japonicus[J].Immunology Letters,2012,143(2):137-145.
[27]Zhou Zunchun,Sun Dapeng,Yang Aifu,et al.Molecular charac-terization and expression analysis of a complement component3 in the sea cucumber(Apostichopus japonicus)[J].Fish&Shellfish Immunology,2011,31(4):540-547.
[28]Zhong Lei,Zhang Feng,Chang Yaqing.Gene cloning and function analysis of complement B factor-2 of Apostichopus japonicus[J]. Fish&Shellfish Immunology,2012,33(3):504-513.
[29]Wang Xiaoyu,Zhou Zunchun,Yang Aifu,et al.Molecular charac-terization and expression analysis of heat shock cognate 70 after heat stress and lipopolysaccharide challenge in sea cucumber (Apostichopus japonicus)[J].Biochemical Genetics,2013,51(5-6):443-457.
[30]Zhao Huan,Yang Hongsheng,Zhao Heling,et al.The molecular characterization and expression of heat shock protein 90(Hsp90) and 26(Hsp26)cDNAs in sea cucumber(Apostichopus japoni-cus)[J].Cell Stress and Chaperones,2011,16(5):481-493.
[31]Yang Aifu,Zhou Zunchun,Dong Ying,et al.Expression of im-mune-related genes in embryos and larvae of sea cucumber Apos-tichopus japonicus[J].Fish&Shellfish Immunology,2010,29 (5):839-845.
[32]Zhu Beiwei,Zhao Lulu,Sun Liming,et al.Purification and char-acterization of a cathepsin L-like enzyme from the body wall of the sea cucumber Stichopus japonicus[J].Bioscience,Biotechnol- ogy,and Biochemistry,2008,72(6):1430-1437.
[33]Liu Xin,Sun Zhenliang,Zhang Miansong,et al.Antioxidant and antihyperlipidemic activities of polysaccharides from sea cucumber Apostichopus japonicus[J].Carbohydrate Polymers,2012,90(4): 1664-1670.
[34]Hulmes D JS.Building collagen molecules,fibrils,and suprafi-brillar structures[J].Journal of Structural Biology,2002,137(1-2):2-10.
[35]Marsden M,Burke R D.The茁L integrin subunit is necessary for gastrulation in sea urchin embryos[J].Developmental Biology, 1998,203(1):134-148.
[36]Wang Hongdi,Liu Shikai,Cui Jun,et al.Identification and char-acterization ofmicroRNAs from longitudinalmuscle and respirato-ry tree in sea cucumber(Apostichopus japonicus)using highthroughput sequencing[J].PLoSOne,2015,10(8):e0134899.
[37]Zhou Xiaoxu,Cui Jun,Liu Shikai,et al.Comparative transcrip-tome analysis of papilla and skin in the sea cucumber,Aposticho-pus japonicas[J].Peerj,2016,4(5):e1779.DOI:10.7717/ peerj.1779.
[38]郭晓黎,仇雪梅,常亚青,等.虾夷马粪海胆CYP17基因的克隆及其表达差异[J].大连海洋大学学报,2013,28(1):1-6.
[39]陈孝红,仇雪梅,郝薇薇,等.斑马鱼CYP 11a1基因在不同性腺发育时期的表达[J].大连海洋大学学报,2015,30(1):13-17.
[40]Ba Huazhong,Yao Feng,Yang Lei,et al.Identification and ex-pression patterns of extracellularmatrix-associated genes fibropel-lin-ia and tenascin involved in regeneration of sea cucumber Apostichopus japonicus[J].Gene,2015,565(1):96-105.
Expression analysis of four functional genes in sea cucumber Apostichopus japonicas
GU Chen-lei1,2,ZHOU Xiao-xu1,LIU Yang1,YU Hai-long1,KONG De-rong1, SONG Jian1,QIU Xue-mei1,CHANG Ya-qing1,WANG Xiu-li1,2
(1.College of Fisheries and Life Science,Dalian Ocean University,Dalian 116023,China;2.Dalian Fisheries Industry Technology Innovation Associ-ation,Dalian 116023,China)
Tissue expression levels of four candidate genes including tenascin gene,collagen琢2 gene,integrin琢V gene and integrin茁L gene were investigated in differentmonth old sea cucumber Apostichopus japonicus by RTPCT.The results showed that therewere different expression levels of tenascin gene in intestines,tube foot,respir-atory tree,skin,body wall,coelomic fluid,parapodium and longitudinalmuscle of the sea cucumber at age of12, 13,and 14 months,significantly higher in coelomic fluid and respiratory tree than in other tissues(P<0.05).The expression levels of collagen琢2 gene were shown to be increased in tube foot,respiratory tree,body wall,parapo-dium and longitudinalmuscle with increase in month age,themaximal expression in respiratory tree,and gradual decline in longitudinalmuscle,parapodium,body wall,intestines and coelomic fluid.The expression levels of in-tegrin琢V gene were found to be gradually increased in longitudinalmuscle,parapodium and body wallwithmonth age increasing.There were lower expression levels of integrin茁L gene in skin,body wall,parapodium and tube foot.The findings provided foundation for further understanding of the four genes in sea cucumber.
Apostichopus japonicus;tenascin gene;collagen琢2 gene;integrin琢V gene;integrin茁L gene;expres-sion analysis
S917
A
10.16535/j.cnki.dlhyxb.2017.03.002
2095-1388(2017)03-0262-06
2016-05-30
国家自然科学基金资助项目(31572608);辽宁省高等学校优秀人才支持计划项目(LR2014022);大连市科技计划项目(2012B11NC049)
顾晨雷(1991—),男,硕士研究生。E-mail:nkss@qq.com
王秀利(1964—),男,博士,教授。E-mail:xlwang@dlou.edu.cn
