柴达木黄牛Y染色体单倍型组构成及父系起源
——基于USP9Y基因多态性的分析
2017-06-10马志杰胡双龙李瑞哲陈生梅雷初朝晁生玉
马志杰,胡双龙,李瑞哲,陈生梅,雷初朝,晁生玉
(1.青海大学畜牧兽医科学院,青海西宁 810016;2.青海省格尔木市乌图美仁乡畜牧兽医站,青海格尔木816000;3.西北农林科技大学动物科技学院,陕西杨凌 712100;4.青海省海西州动物疫病预防控制中心,青海德令哈 817099)
柴达木黄牛Y染色体单倍型组构成及父系起源
——基于USP9Y基因多态性的分析
马志杰1*,胡双龙2,李瑞哲1,陈生梅1,雷初朝3,晁生玉4
(1.青海大学畜牧兽医科学院,青海西宁 810016;2.青海省格尔木市乌图美仁乡畜牧兽医站,青海格尔木816000;3.西北农林科技大学动物科技学院,陕西杨凌 712100;4.青海省海西州动物疫病预防控制中心,青海德令哈 817099)
为从分子水平上探究柴达木黄牛的父系遗传多样性、起源及群体遗传结构,对62头柴达木黄牛公牛进行Y染色体USP9Y基因多态性分析。结果表明:柴达木黄牛USP9Y基因的PCR产物显示471 bp和552 bp 2种带型,其中552 bp带型不能被SspI酶酶切,这2种带型对应Y1和Y2 2种普通牛Y染色体单倍型组,表明柴达木黄牛含有Y1和Y2 2个父系支系,有2个普通牛父系起源。单倍型多样度为0.3369,说明柴达木黄牛具有较低的父系遗传多样性。
柴达木黄牛;Y染色体;USP9Y;多态性;单倍型组;父系起源
柴达木黄牛是我国优良的地方黄牛品种之一,主产于青海省柴达木盆地边缘的都兰县、乌兰县和格尔木市辖区等2 600~3 200 m高海拔地区。柴达木黄牛具有较强的抗逆性和适应能力,可为该地区蒙、藏少数民族提供乳、肉主要畜产品和役力,在遗传方面是一个宝贵的基因库[1]。目前,有关柴达木黄牛遗传多样性、群体遗传结构及遗传背景的研究相对较少。在生化水平上,张才骏等[2]对柴达木黄牛14个血液和乳生化遗传标记进行了系统分析,结果表明11个标记存在多态性,平均基因杂合度为0.2677,平均有效等位基因数为1.4792个。随后,张才骏等[3]探究了柴达木黄牛与我国其他黄牛品种/群体之间的遗传关系,对血红蛋白(HB)、运铁蛋白(TF)、后运铁蛋白(PTF)、后白蛋白(PA)和碱性磷酸酶(ALP)5个血液基因座进行了分析,发现柴达木黄牛与青海东部黄牛存在最近的亲缘关系,与北方牛系蒙古牛、延边牛和安西牛具有较近的亲缘关系,而与其他黄牛品种的亲缘关系较远。在分子水平上,目前尚未见对柴达木黄牛品种的父系遗传多样性、起源及群体遗传结构的相关研究报道。
动物Y染色体遵循父系遗传,单倍型完整,突变率低,不易受重组和回复突变等因素影响,是探明父系遗传多样性、起源和驯化历史等研究的理想工具[4]。近年来,众多关于家牛Y染色体SNPs标记(Y-SNPs)的研究表明[5-14],家牛由3种Y染色体单倍型组组成(即普通牛Y1和Y2单倍型组以及瘤牛Y3单倍型组)。与此同时,Bonfiglio等[10]基于Y染色体USP9Y(Y-linked Ubiquitin-specific Protease 9)基因内含子26上1个81 bp的插入和1个限制性酶切位点的结合,发展出一种不用测序而只通过PCR和酶切电泳就可以直接鉴别这3种单倍型组的方法。基于此,本研究利用USP9Y基因PCR和酶切电泳技术结合直接测序方法对柴达木黄牛进行Y染色体单倍型组及父系起源分析,以期弄清柴达木黄牛的父系遗传多样性、起源和群体遗传结构,为今后开展该品种的保种、选育策略制定和分子育种奠定理论基础。
1 材料与方法
1.1 采样及基因组DNA提取 在青海省格尔木市乌图美仁乡(34头)、都兰县宗加镇(20头)和乌兰县柯柯镇(8头)共采集62头柴达木黄牛公牛血样或耳组织带回实验室,采用基因组DNA提取试剂盒(北京艾德莱生物科技有限公司)提取基因组DNA,稀释终浓度至25~50 ng/μL,保存备用。
1.2 引物合成和PCR扩增 参照Bonfiglio等[10]报道的普通牛USP9Y基因序列合成1对引物,其中上游引物(PF):5'-AAACCCTTCAAGG TAATAAAACAAAA-3',下游引物(PR):5'-CACAGCTCCTCAAAACCAGA -3',由生工(上海)有限公司合成。
25 μL PCR体系:2×PrimeSTAR Max Premix (上海宝生物公司) 10.5 μL,上、下游引物(10 pmol/L)各0.5 μL,模板DNA(25~50 ng/μL)1 μL,超纯水12.5 μL。PCR反应程序:98℃变性10 s,57℃退火15 s,72℃延伸10 s,35个循环,后冷却至4℃保存。PCR产物经1.5%琼脂糖凝胶(含Gold View核酸染料)电泳后,凝胶成像系统分析检测。
1.3 PCR产物的电泳检测和酶切分析 先对PCR产物进行1.5%的琼脂糖凝胶电泳检测,随后根据其带型情况,对部分PCR纯化产物用SspI限制性内切酶再进行酶切分析,判定其单倍型组。其中20 μL SspI酶切体系:SspI酶(12 U/μL)1 μL,10×SspI Buffer 2 μL,PCR产物10 μL,超纯水7 μL。37℃酶切8~10 h,后对酶切结果进行电泳检测。
1.4 序列测定及比对分析 根据酶切结果,对不同单倍型组个体PCR纯化产物用3730XL型DNA测序仪(Applied Biosystems公司)进行正、反向测序,测序结果用Chromas 2.3软件进行核实和校正,得到每头柴达木黄牛公牛的USP9Y基因序列。后用BioEdit 7.0.9软件[15]进行序列比对分析,以确认是否与酶切分析结果一致,用Arlequin 3.11软件[16]进行序列变异分析并计算群体单倍型多样度,多样度用平均值±标准差表示。
2 结果与分析
2.1 柴达木黄牛USP9Y 基因扩增多态性与SspI 酶酶切分析 如图1所示,62头黄牛PCR产物均获得预期的大小片段,有471 bp和552 bp 2种PCR产物,其中13头黄牛的PCR产物为471 bp,49头黄牛的PCR产物为552 bp。依据Bonfiglio等[10]的判定标准,确定这13头黄牛(471 bp)为普通牛Y1单倍型组,剩余49头黄牛尚不能确定属于普通牛Y2单倍型组还是瘤牛Y3单倍型组,随后进行SspI酶酶切分析,结果表明这49头黄牛的PCR产物都不能被SspI酶切开,与其PCR产物带型、大小完全相同(图1),因此确定这49头黄牛均为普通牛Y2单倍型组。
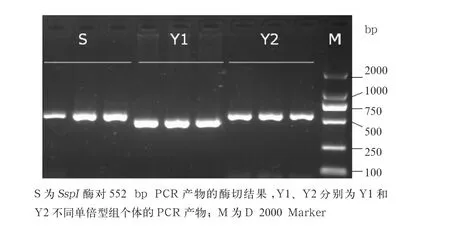
图1 柴达木黄牛USP9Y基因PCR产物多态性及酶切电泳

图2 柴达木黄牛Y1和Y2两种单倍型组序列比对分析
2.2 柴达木黄牛不同Y染色体单倍型组频率统计 由表1可知,62头黄牛中所含Y1和Y2单倍型组个体分别为13和49个,所占频率依次为0.21和0.79,表明柴达木黄牛品种由2种普通牛Y染色体单倍型组构成,以Y2单倍型组为主。

表1 柴达木黄牛单倍型组频率及单倍型多样度
2.3 柴达木黄牛不同Y染色体单倍型组序列比对分析及单倍型多样度 通过对62头柴达木黄牛公牛USP9Y基因部分序列进行测序和比对分析,发现测序结果与酶切结果完全一致,Y1和Y2单倍型组个体数分别为13头和49头,其中13头Y1单倍型组个体的序列完全相同(471 bp),剩余49头Y2单倍型组个体的序列也完全相同(552 bp)。2种单倍型组序列(GenBank登录号:KY495786~KY495787)表现为81 bp的序列插入和1个496A>C核苷酸颠换的差异(图2)。柴达木黄牛品种单倍型多样度为0.3369(表1),表明其父系遗传多样性较低。
3 讨 论
在先前对中国部分黄牛品种的父系遗传研究中,常振华等[9]基于Y-SNPs和Y-STRs标记对16个中国地方黄牛品种284头公牛进行了父系遗传多样性及起源分析,结果在16个黄牛品种中仅发现普通牛Y2和瘤牛Y3单倍型组,未发现存在普通牛Y1单倍型组,其中Y2单倍型频率在北方黄牛中占优势(98.3%),Y3单倍型频率在南方黄牛中占优势(76.1%),中原黄牛中普通牛Y2的单倍型频率(63.8%)比瘤牛Y3(36.2%)高。Li等[12-13]基于USP9Y基因多态标记以及Y-SNPs和Y-STRs标记进一步对常振华等[9]研究的16个中国地方黄牛品种302头公牛进行综合分析,结果与其研究结果一致,同时,该研究发现哈萨克、秦川、吉安3个黄牛品种除以Y2或Y3单倍型组为主外,还含有少量普通牛Y1 单倍型组个体。基于对一些西方肉牛品种的父系遗传分析[7-8,10-13](如Holstein、Aberdeen Angus等品种)和中国20世纪80年代以来的黄牛育种改良的实践[1],Li等[12-13]推测哈萨克、秦川和吉安3个黄牛品种中所含有的频率较低的Y1单倍型组可能主要来自引进的黄牛品种。随后,Yue等[14]对25头蒙古黄牛公牛进行分子遗传分析,表明蒙古黄牛由Y2单倍型组构成。在本研究中,对62头柴达木黄牛进行父系遗传分析表明酶切结果与测序结果完全一致,说明利用USP9Y基因酶切检测技术能够准确快速、经济可靠的确定黄牛品种内部的Y染色体单倍型组构成,从而揭示其群体父系遗传结构。本研究显示,柴达木黄牛由普通牛Y1和Y2单倍型组构成,以Y2单倍型组为主,提示柴达木黄牛由2个父系支系组成,有2个父系起源,这与常振华等[9]、Li等[12-13]、Yue等[14]的研究结果基本一致,说明柴达木黄牛作为中国北方黄牛品种之一,具有与中国北部、中部黄牛品种相似的父系遗传组成,即以Y2普通牛单倍型组为主。同时,与大部分中国黄牛品种相比也存在一定差异,不仅由Y2单倍型组构成,还包括一定的Y1单倍型组个体,父系遗传基础较广。柴达木黄牛在20世纪50年代后经历了秦川牛、荷斯坦牛、西门塔尔牛等品种的人工授精改良实践[1],可能由于这些牛品种雄性介导的基因渗入,导致柴达木黄牛含有与部分中国黄牛品种(如哈萨克、秦川、吉安黄牛品种)相似的Y1单倍型组[12-13],形成较宽泛的父系遗传基础和起源。
在生化水平上,张才骏等[2]对柴达木黄牛14个血液和乳生化遗传标记分析表明,多态性遗传标记基因座达78.6%,平均基因杂合度(0.2677)和有效等位基因数(1.4792个)都较高,提示该品种在生化水平上具有较高程度的多态性和较大的遗传变异;根据HB、TF、PTF、PA和ALP 5个基因座的资料,分析表明柴达木黄牛的平均基因杂合度(0.3770)和有效等位基因数(1.61)与蒙古牛(分别为0.3874和1.63)、复州牛(分别为0.3857和1.63)相似,低于绝大部分中原牛(分别为0.4262~0.4886和1.68~1.96)。而在分子水平上,Li等[12]对16个中国地方黄牛品种的父系遗传多样性分析表明,各品种单倍型多样度在0.000~0.600,平均值为0.518;其中北方黄牛、中部黄牛和南方黄牛的单倍型多样度平均值分别为0.164、0.416和0.312。在本研究中,62头柴达木黄牛单倍型多样度为0.3369,可以看出,该值低于部分国外牛品种的单倍型多样度平均值0.422[11],同时也低于Li等[12]报道的结果,但稍高于北方黄牛和南方黄牛的单倍型多样度平均值,表明柴达木黄牛单倍型多样度相对较低,这可能与柴达木黄牛在本研究父系遗传检测中不含瘤牛血统以及所处地理位置偏僻、牛群多为封闭性选育等因素相关。同时,也提示柴达木黄牛分子水平和生化水平上的遗传多样性研究结果稍有差异。柴达木黄牛和我国中部黄牛品种相比父系分子遗传多样性水平较低,可能与中部黄牛大多具有普通牛和瘤牛父系遗传血统而多样性水平较高有关[9,12-13]。当然,有关柴达木黄牛分子水平上的遗传多样性状况和种群遗传结构分析还有待通过线粒体基因组和核基因组(包括Y染色体STR标记和常染色体)遗传信息从不同角度进行综合系统研究,进而准确地揭示其遗传多样性水平和群体遗传结构,为其选育和保种提供基础材料。
[1] 国家畜禽遗传资源委员会.中国畜禽遗传资源志·牛志[M].北京:中国农业出版社, 2011:198-200.
[2] 张才骏, 王勇, 卢福山, 等. 柴达木黄牛生化遗传标记的研究[J]. 青海畜牧兽医杂志, 2001, 31(6):3-5.
[3] 张才骏, 张海峰, 晃生玉. 柴达木黄牛与我国其它品种黄牛亲缘关系的探讨[J]. 中国畜牧杂志, 2001, 37(6):9-11.
[4] Cortez D, Marin R, Toledo-Flores D, et al. Origins and functional evolution of Y chromosomes across mammals[J]. Nature, 2014, 508(7497): 488-493.
[5] Götherström A, Anderung C, Hellborg L, et al. Cattle domestication in the Near East was followed by hybridization with aurochs bulls in Europe[J]. P Roy Soc Lond B Bio, 2005, 272: 2345-2351.
[6] Bollongino R, Elsner J, Vigne J D, et al. Y-SNPs Do notindicate hybridisation between european aurochs and domestic cattle[J]. PLoS One, 2008, 3(10):e3418.
[7] Ginja C, Telod G L, Penedo M C. Y chromosome haplotype analysis in Portuguese cattle breeds using SNPs and STRs[J]. J Hered, 2009, 100(2):148-157.
[8] Pérez-Pardal L, Royo L J, Beja-Pereira A, et al. Y-specific microsatellites reveal an African subfamily in taurine (Bos taurus) cattle [J]. Anim Genet, 2010, 41(3): 232-241.
[9] 常振华. 中国黄牛Y染色体微卫星和SNPs遗传多样性与起源研究[D]. 杨凌: 西北农林科技大学, 2011.
[10] Bonfiglio S, De G A, Tesfaye K, et al. A novel USP9Y polymorphism allowing a rapid and unambiguous classification of Bos taurus Y chromosomes into haplogroups[J]. Anim Genet, 2012, 43(5):611-613.
[11] Edwards C J, Ginja C, Kantanen J, et al. Dual origins of dairy cattle farming-evidence from a comprehensive survey of European Y-chromosomal variation[J]. PLoS One, 2012, 6(1):e15922.
[12] Li R, Xie W M, Chang Z H, et al. Y chromosome diversity and paternal origin of Chinese cattle[J]. Mol Biol Rep, 2013, 40(12):6633-6636.
[13] Li R, Zhang X M, Campana M G, et al. Paternal origins of Chinese cattle[J]. Anim Genet, 2013, 44(4):446.
[14] Yue X, Li R, Liu L, et al. When and how did Bos indicus introgress into Mongolian cattle? [J]. Gene, 2014, 537(2):214-219.
[15] Hall T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/ NT [J]. Nucl Acids Symp Ser, 1999, 41(41):95-98.
[16] Excoffier L, Laval G, Schneider S. Arlequin ver. 3.11: An integrated software package for population genetics data analysis[J]. Evol Bioinform, 2005, 1: 47-50.
Y Chromosome Haplogroups and Paternal Origin of Qaidam Cattle: Based on the Polymorphism Analysis of USP9Y Gene
MA Zhi-jie1*, HU Shuang-long2, LI Rui-zhe1, CHEN Sheng-mei1, LEI Chu-zhao3, CHAO Sheng-yu4
(1. Academy of Animal Science and Veterinary Medicine, Qinghai University, Qinghai Xining 810016, China; 2. Station of Animal Science and Veterinary Medicine of Wutumeiren town in Golmud city of Qinghai province, Qinghai Golmud 816000, China; 3.College of Animal Science and Technology, Northwest A&F University, Shaanxi Yangling 712100, China; 4. Animal Epidemic Disease Prevent and Control Center in Haixi Autonomous Prefecture of Qinghai province, Qinghai Delingha 817099, China)
To explore the paternal diversity, population genetic structure and origin of Qaidam cattle, the polymorphism of Y chromosome USP9Y gene in 62 Qaidam cattle males was analyzed. The results showed that the size of PCR products of Qaidam cattle USP9Y gene was 471 bp and 552 bp respectively, and the 552 bp PCR products can’t be digested by SspI. Therefore, the above two belt types represent Y1 and Y2 cattle haplogroups, respectively, indicating that Qaidam cattle have two patrilineal branches with two paternal origins. The haplotype diversity of Qaidam cattle was 0.3369, suggesting that the Qaidam cattle have lower paternal genetic diversity.
Qaidam cattle; Y chromosome; USP9Y; Polymorphism; Haplogroups; Paternal origin
S823.2
A
10.19556/j.0258-7033.2017-06-036
2016-09-06;
2017-03-06
青海省自然科学基金(面上)项目(2017-ZJ-906);青海省“高端创新人才千人计划”项目;国家自然科学基金项目(31360267);青海省应用基础研究项目(2015-ZJ-712);国家肉牛牦牛产业技术体系(CARS-38)
马志杰(1978-),男,甘肃张家川人,博士研究生,副研究员,主要从事动物遗传资源研究,E-mail: zhijiema@126. com
* 通讯作者
