侵染甘薯的DNA病毒研究进展
2017-06-05刘起丽张建新李学成石明旺欧行奇
刘起丽, 张建新, 李学成, 石明旺, 欧行奇
(1. 河南科技学院资源与环境学院, 新乡 453003; 2. 河南师范大学水产学院, 新乡 453007; 3. 河南天方药业有限公司, 驻马店 463000)
侵染甘薯的DNA病毒研究进展
刘起丽1, 张建新2, 李学成3, 石明旺1, 欧行奇1
(1. 河南科技学院资源与环境学院, 新乡 453003; 2. 河南师范大学水产学院, 新乡 453007; 3. 河南天方药业有限公司, 驻马店 463000)
甘薯是重要的粮食作物和食品加工及工业原料。我国是世界上最大的甘薯生产国。病毒病是甘薯上的重要病害,目前世界上已报道的侵染甘薯的DNA病毒主要归属于双生病毒科Geminiviridae和花椰菜花叶病毒科Caulimoviridae。近年来,双生病毒等DNA病毒严重影响我国甘薯的产量、品质以及食品加工产业。本文简介了甘薯在我国的重要地位和种植情况;具体介绍了侵染甘薯的菜豆金色花叶病毒属Begomovirus、玉米线条病毒属Mastrevirus及杆状DNA病毒属Badnavirus的病毒特征、分子变异、分类现状和检测方法。结合甘薯生产的实际情况,提出了目前甘薯DNA病毒研究中存在的问题及思考。本文旨在为我国甘薯DNA病毒病的综合防控提供理论依据。
甘薯; DNA病毒; 菜豆金色花叶病毒属; 玉米线条病毒属; 杆状DNA病毒属
甘薯Ipomoeabatatas是世界上重要的粮食作物和食品加工及工业原料。据联合国粮农组织(FAO)统计,世界上栽培甘薯的国家一共有50多个,主要分布在亚洲、非洲的多个发展中国家,其次为拉丁美洲,欧洲的种植面积极少[1]。我国是世界上甘薯种植面积最大的国家[2],近年来随着双生病毒科菜豆金色花叶病毒属的病毒等多种DNA病毒在世界范围内多种作物上的扩展和肆虐,我国的甘薯产业也面临着多种DNA病毒的威胁,甘薯产量和品质受到严重影响。鉴于最近几年来国内外报道的侵染甘薯的DNA病毒种类不断增多、国际病毒分类委员会(International Committee on Taxonomy of Viruses,ICTV)及病毒分类权威专家关于DNA病毒分类标准的不断变化和改进,本文结合我国甘薯产业的现状和病害发生情况,对侵染甘薯的DNA病毒的种类、特征、分子变异和检测方法等进行了综述,旨在为我国甘薯DNA病毒病的综合防控提供参考和依据。
1 我国的甘薯种植现状
甘薯又称红薯、地瓜、山芋等,是旋花科Convolvulaceae甘薯属Ipomoea一年生或多年生蔓生草本植物[3],总产量居世界粮食产量的第7位[4]。中国一直是世界上最大的甘薯生产国[2]。21世纪以来中国甘薯种植总面积缓慢下降,2001年为5.507×106hm2,2010年为3.684×106hm2,年递减率约为5%,导致中国甘薯种植面积占世界总面积的比例从60.0%一直下降到45.0%[5-6]。2011年中国甘薯面积为4.6×106hm2,占世界总种植面积的1/2以上。尽管中国甘薯的种植面积缓慢下降,但单产量却逐年不断提高,甘薯产量一直保持在22.5 t/hm2,鲜薯总产量约为1.0×108t[7]。
甘薯在我国的种植范围较广,从内蒙古自治区到海南岛,自西藏自治区至浙江省都有种植,尤其在我国黄河流域和长江流域各省份,甘薯的集中区域化种植非常突出[8-9]。据统计,目前甘薯种植面积超过1.0×104hm2的省份约有16个[10-11]。依据耕作制度和气候条件的不同,我国甘薯主要分为三大主产区[12]:北方薯区(主要有河北、山东、安徽、河南和江苏共5个省份)、长江流域产区(主要有四川、湖北和重庆共3个省、直辖市)和南方薯区(主要有海南、广东和福建共3个省份)。
病毒病是甘薯上的重要病害,在世界各甘薯产区广泛存在[13]。截至2012年,世界上已报道的能够侵染甘薯的DNA病毒一共有16种[14],分别归属于双生病毒科Geminiviridae和花椰菜花叶病毒科Caulimoviridae[14]。其中双生病毒科病毒对甘薯的生长危害较重,可造成26%~63%的产量损失[15]。
2 侵染甘薯的DNA病毒种类
2.1 侵染甘薯的双生病毒科Geminiviridae病毒
双生病毒科病毒是植物病毒中数目最多的一类DNA病毒,截至2017年3月11日,经国际病毒分类委员会(ICTV)确定的已达369个种[16-17]。ICTV第十次病毒分类报告中,根据双生病毒的基因组结构特征、寄主范围大小和传播介体的不同,将双生病毒科Geminiviridae划分为9个属[16],分别为:菜豆金色花叶病毒属Begomovirus、玉米线条病毒属Mastrevirus、甜菜曲顶病毒属Curtovirus、番茄伪曲顶病毒属Topocuvirus、伊朗甜菜曲顶病毒属Becurtovirus、芜菁曲顶病毒属Turncurtovirus、画眉草条纹病毒属Eragrovirus、孔雀大戟潜隐病毒属Capulavirus和葡萄红斑病毒属Grablovirus,其中孔雀大戟潜隐病毒属Capulavirus和葡萄红斑病毒属Grablovirus是新建立的两个属[18]。已报道的侵染甘薯的双生病毒科病毒仅限于菜豆金色花叶病毒属Begomovirus和玉米线条病毒属Mastrevirus的病毒。
2.1.1 菜豆金色花叶病毒属Begomovirus
Begomovirus是双生病毒科中成员最多的属,截至2017年3月11日已确立322个正式种[17]。目前已报道的侵染甘薯的双生病毒大部分属于Begomovirus。系统发育分析发现,侵染甘薯的Begomovirus病毒分离物聚成一簇,与旧世界病毒和新世界病毒处于不同分支,且与侵染其他植物的双生病毒明显分离开来,因此又被称为“sweepoviruses”[19]。目前世界上已发现的sweepoviruses总共有11个种[17],分别为:甘薯曲叶病毒Sweetpotatoleafcurlvirus(SPLCV)、甘薯中国曲叶病毒SweetpotatoleafcurlChinavirus(SPLCCNV)、甘薯乔治亚曲叶病毒SweetpotatoleafcurlGeorgiavirus(SPLCGV)、甘薯加纳利曲叶病毒SweetpotatoleafcurlCanaryvirus(SPLCCV)、甘薯圣保罗曲叶病毒SweetpotatoleafcurlSaoPaulovirus(SPLCSPV)、甘薯南卡罗莱纳曲叶病毒SweetpotatoleafcurlSouthCarolinavirus(SPLCSCV)、甘薯乌干达曲叶病毒SweetpotatoleafcurlUgandavirus(SPLCUV)、甘薯斑驳病毒Sweetpotatomosaicvirus(SPMoV)、甘薯河南曲叶病毒SweetpotatoleafcurlHenanvirus(SPLCHnV)、甘薯四川曲叶病毒1SweetpotatoleafcurlSichuanvirus1(SPLCSiV-1)和甘薯四川曲叶病毒2SweetpotatoleafcurlSichuanvirus2(SPLCSiV-2)。
2.1.2 玉米线条病毒属Mastrevirus
双生病毒科中的玉米线条病毒属Mastrevirus病毒也能够侵染甘薯。目前已报道的仅有甘薯无症病毒1 Sweet potato symptomless virus 1(SPSMV-1)[14,20]。
2.2 侵染甘薯的花椰菜花叶病毒科Caulimoviridae病毒
截至2016年,已报道的能够侵染甘薯的花椰菜花叶病毒科Caulimoviridae的病毒有3个种[14],分别为:杆状DNA病毒属Badnavirus的Sweetpotatopakakuyvirus(SPPV),也称为 Sweet potato badnavirus A and B (SPBV-A和SPBV-B);木薯脉花叶病毒属Cavemovirus的Sweetpotatocollusivevirus,也称为Sweet potato caulimo-like virus;以及Solendovirus属的Sweetpotatoveinclearingvirus。
3 甘薯DNA病毒的基因组特征
3.1 Sweepoviruses的基因组特征及其变异
Sweepoviruses绝大多数都属于典型的旧世界病毒,即只发现了DNA-A组分,大小约为2.8 kb,其病毒正义链包含2个ORFs(编码AV1和AV2基因);病毒互补链包含4个ORFs(编码AC1~AC4基因),大多数学者的研究均未发现DNA-B组分以及伴随的卫星DNA分子的存在[21]。但值得关注的是,2013年Swapna Geetanjali等[22]在I.purpurea上发现了SPLCV的两个不同的β卫星(betasatellites):Croton yellow vein mosaic betasatellite (CroYVMβ)和Papaya leaf curl betasatellite (PaLCuβ),这是目前唯一的发现sweepoviruses的基因组伴随有β卫星的报道。2016年,Hassan等[23]发现了伴随sweepoviruses的δ卫星(deltasatellites);Gloria等[24]报道了非编码DNA satellites的存在。
目前sweepoviruses的变异研究相对较多,且主要集中于重组变异。Zhang等[15]报道了在美国发现的1个自然重组病毒:Sweet potato golden vein-associated virus,重组分析发现,该病毒极有可能是由SPLCV和SPLCGoV两个种自然重组而来,重组位点位于复制起点与AC2和AC4基因之间。Paprotk等[25]认为侵染巴西甘薯的sweepoviruses的重组位点主要位于发卡结构的结合处。Albuquerque等[26]发现巴西sweepoviruses的重组事件主要发生在IR区及AC1的中间部位。重组位点的多样性表明该类病毒的变异可能发生在不同的位点。
3.2 甘薯无症病毒1(SPSMV-1)的基因组特征
侵染甘薯的甘薯无症病毒1(SPSMV-1)的结构非常独特,与其他mastreviruses病毒相比,它有一个比同属病毒小得多的复制酶基因 (replicase gene)[14]。Mastreviruses的C1和C2蛋白质通过选择性剪接(图1,在C2白色方框所示的内含子的位置),使其具有相同的N-末端和不同的C-末端。SPSMV-1基因组比其他mastreviruses基因组小得多,且缺乏选择性剪接的C2蛋白。此外,预测在SPSMV-1基因组的大型非编码区可能存在两个茎-环结构(图1)。

图1 SPSMV-1 与其他玉米线条病毒属病毒基因组结构比较(引自Clark[14])Fig. 1 Comparison of the organization of SPSMV-1 genome with genome typical of other plant mastreviruses(cited from Clark[14])
3.3 甘薯杆状DNA病毒sweet potato badnavirus的基因组特征
2009年,侵染甘薯的杆状DNA病毒(SPBV)首次被发现。Badnaviruses病毒是一类环状双链DNA(double-stranded DNA,dsDNA)类逆转录病毒(pararetrovirus)[27]。病毒粒体杆状,大小为(25~30) nm×(60~900) nm[28]。病毒基因组大小约为7.1~8.0 kb,核酸变异很大[29-34]。典型的Badnavirus病毒包含有3个ORFs[35](图2)。侵染甘薯的杆状DNA病毒(SPBV)包含SPBV-A和SPBV-B[36]。SPBV-A和SPBV-B均含5个ORFs,前2个ORFs和最后1个ORF均编码3个小的假定蛋白(hypothetical protein);ORF3a编码运动蛋白和外壳蛋白(SPBV-A: 1 437~5 006 bp; SPBV-B: 1 486~4 998 bp);ORF3b编码天冬氨酸蛋白酶域(aspartic protease domain; SPBV-A的ORF3b编码)或天冬氨酸蛋白酶(aspartic protease, AP; SPBV-B的ORF3b编码)、逆转录酶(reverse transcriptase,RT)和RNA酶H (ribonuclease H,Rnase H)[36]。
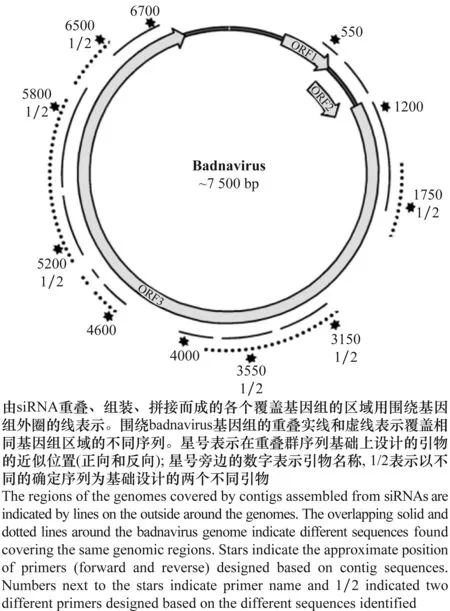
图2 杆状DNA病毒基因组结构(引自Kreuze[36])Fig.2 Viral genome structure of badnavirus(cited from Kreuze[36])
4 侵染性克隆的构建
构建侵染性克隆是研究病毒致病性的重要途径,目前侵染甘薯的DNA病毒中,仅有sweepoviruses侵染性克隆的报道,其构建原则与双生病毒的侵染性克隆构建原则一致,即需构建一个具有1.3~2.0个拷贝的正向重复的双生病毒基因组重组质粒,且该正向重复序列中必须包括2个完整的CR区[37-38]。目前国内外仅报道了两个侵染性克隆。Trenado等[37]2011年成功构建了Sweet potato leaf curl Lanzarote virus (SPLCLaV)的侵染性克隆,并研究了该侵染性克隆对不同种类植物及植物品种的致病性和症状表现。2012年,Bi等[38]构建了甘薯曲叶病毒江苏分离物(SPLCV-JS)的侵染性克隆,农杆菌介导的病毒接种试验发现,单独的SPLCV-JS侵染性克隆只能使本氏烟出现轻微症状和病毒DNA的微量积累,而将该侵染性克隆与一个异源卫星DNA分子TYLCCNV-Y10的DNAβ共同接种本氏烟后,本氏烟发病症状更加明显,病毒DNA的积累量也大大增加。侵染性克隆为这类病毒的致病性和基因功能的深入研究奠定了基础。
5 甘薯DNA病毒的分布
5.1 Sweepoviruses的分布
1985年,甘薯曲叶病在中国台湾被发现[39]。1994年,研究者从美国一种观赏甘薯上首次获得了甘薯曲叶病毒Sweetpotatoleafcurlvirus(SPLCV)[40]。目前,以色列[41]、美国[40]、日本[42]、西班牙[43]、意大利[44]、秘鲁[45]、肯尼亚[46]、韩国[47]、中国[48]和印度[49]等国家和地区均有sweepoviruses研究的报道。Sweepoviruses的多样性和基因组变异已初步被人们认知。在中国,sweepoviruses分离物仅在台湾[39]、辽宁[21,48]、江苏和浙江[38]、河北[50]、广东[51]、四川[52]和河南[53]等省份或地区有正式的报道,包括5个种:甘薯中国曲叶病毒(SPLCCNV)、甘薯曲叶病毒(SPLCV)、甘薯乔治亚曲叶病毒(SPLCGV)、甘薯河南曲叶病毒(SPLCHnV)和甘薯四川曲叶病毒1(SPLCSiV-1)。
5.2 甘薯无症病毒1(SPSMV-1)的分布
甘薯无症病毒1(SPSMV-1)已经从采自秘鲁[36]、坦桑尼亚[52]、几个中美洲和亚洲国家(CIP, unpublished)的甘薯样品上检测到,但是其检出率远远低于花椰菜花叶病毒科其他杆状DNA病毒。坦桑尼亚和秘鲁的SPSMV-1分离物的CP-MP区域相似性为100%[14],说明该病毒的CP-MP区域具有较高的保守性。国内关于甘薯无症病毒1的研究和报道非常少。2015年,Wang等[53]从采自中国14个省份的128份甘薯样品中,利用检测SPSMV-1的通用引物[52]检测到2个来自不同省份的阳性样品,所获得的甘薯无症病毒1(SPSMV-1)核酸序列与秘鲁和坦桑尼亚报道的分离物匹配核酸序列区域的相似性达到了99%~100%。这是SPSMV-1在中国甘薯上的首次报道。
6 甘薯DNA病毒的检测方法
6.1 血清学检测
目前尚未有在生产中广泛应用的检测甘薯DNA病毒的特异性抗体的报道。乔贞贞等[54]2012年在大肠杆菌中成功高效表达了甘薯曲叶病毒江苏分离物(SPLCV-JS)的CP基因;李学成等[55]2016年在大肠杆菌中高效表达了SPBV-B CP基因的部分片段,这些工作为该病毒的抗体制备和血清学检测方法的建立奠定了基础。
6.2 PCR检测
目前用于检测甘薯DNA病毒各组分的PCR引物较多。Briddon等[56]设计了BM-V/BM-C引物,可扩增到sweepoviruses的DNA-A组分的基因组近全长序列。对于卫星分子DNAβ的检测,Briddon等[57]根据SCR序列设计的通用引物Beta 01/Beta 02被研究者们普遍采用。检测甘薯无症病毒1的通用引物SPSMV-1F/SPSMV-1R(目标片段726 bp)和MastvkF/MastvsR(目标片段426 bp)主要用于扩增甘薯无症病毒1的运动蛋白和衣壳蛋白部分序列[52-53]。在甘薯杆状病毒(SPBV)的PCR检测方法中,检测SPBV-A的通用引物BadnaBKF/BadnaBsR[52]和检测SPBV-B的通用引物rt-badB-F/rt-badB-R[58]已成功应用。
6.3 siRNA深度测序
siRNA深度测序技术作为一种新的DNA测序技术,能够一次性处理大量样品,大大提高了测序效率。2009年Kreuze[36]在用siRNA深度测序时除了获得了SPFMV和SPCSV病毒序列之外,还获得了2个新的杆状DNA病毒(sweet potato badnavirus A和sweet potato badnavirus B)和1个玉米线条病毒(sweeptotato symptomless mastrevirus 1),并且发现这2个杆状DNA病毒和1个玉米线条病毒与之前报道的badnaviruses和mastreviruses分离物的核酸相似性很高[36]。在坦桑尼亚,一种新的PCR与siRNA深度测序相结合的检测技术被用来检测SPSMV-1和SPPV[52]。siRNA深度测序的成本也越来越便宜,当前制约siRNA深度测序技术的瓶颈主要是样品处理耗时及后期生物信息数据分析方面存在的问题。
7 我国甘薯DNA病毒研究存在的问题和展望
(1)一直以来,我国对甘薯病毒病的研究主要集中于甘薯羽状斑驳病毒Sweetpotatofeatherymottlevirus(SPFMV)等RNA病毒,对DNA病毒则缺乏全面系统的鉴定和研究。分子变异是致病性变异的基础,而中国甘薯DNA病毒的种类、分布、分子变异等情况一直都还不清楚,这直接导致了甘薯DNA病毒病的预警和防治工作缺乏科学依据。(2)侵染性克隆是研究病毒特性的重要途径,但目前国际上只有Trenado等[37]构建了SPLCLaV的侵染性克隆,国内仅Bi等[38]构建了SPLCV的侵染性克隆,其他种类的甘薯DNA病毒的侵染性克隆构建及生物学特性研究均尚未见报道。这部分工作亟待深入开展。(3)在检测方法上,目前许多引物在检测甘薯DNA病毒的工作中起到了重要作用,但特异性检测甘薯DNA病毒的血清学方法研究极少,这可能与近些年来国内外对甘薯病毒病的研究尚不够重视有关;多重PCR技术非常适合用来检测复合侵染现象较多的甘薯DNA病毒,但是目前已报道的多重PCR引物及方法很少;深度测序技术在甘薯DNA病毒检测中的应用的报道相对来说还比较少。制备高效的特异性抗体、多重PCR和深度测序技术均能够大大提高病毒的检测效率,在大量样品的检测工作中发挥重要作用,是值得重视的研究方向。(4)甘薯种苗的调运与管理对病毒病的防控具有一定意义。在非洲,甘薯种苗在大陆的调运是在CIP-Sweetpotato Action for Security and Health in Africa (SASHA)项目的指导下进行的,为了保证这项工作的顺利开展,肯尼亚、莫桑比克和加纳检疫中心的检测容量和相关设施都在不断改善。同时,这些检疫中心也建立了甘薯知识门户网站(http:∥sweetpotatoknowledge.org/)[14],这些好的做法都值得我们学习和借鉴。(5)在防治策略方面,目前对甘薯病毒病最为有效的防治方法就是利用茎尖分生组织培养技术培育脱毒甘薯;江苏、山东、河南等省均对多个主栽品种进行了脱毒研究并大面积示范推广,取得了显著效果[13]。在培育脱毒甘薯的基础上,大力摸索快速高效的检测方法、加强病毒病田间检测;实行甘薯种薯和薯苗严格管理审批、有序调运;做好田间防虫治虫工作、减少病毒传播的介体,这些都将为甘薯病毒病的有效防控起到积极作用。
[1] 吴雨华. 世界甘薯加工利用新趋势[J]. 食品研究与开发, 2003(10): 5-8.
[2] Tairo F, Mukasa S B, Jones R A C, et al. Unravelling the genetic diversity of the three main viruses involved in sweet potato virus disease (SPVD), and its practical implications[J]. Molecular Plant Pathology, 2005, 6(2): 199-211.
[3] 陈万祥, 杨金龙. 甘薯生产的现状及开发利用的途径[J]. 农业装备技术, 2008(4): 32-36.
[4] 刘庆昌. 甘薯在我国粮食和能源安全中的重要作用[J]. 科技导报, 2004(9): 21-22.
[5] 农业部科技教育司, 财政部教科文司. 中国农业产业技术发展报告(2009年度)[R]. 北京: 中国农业出版社, 2010.
[6] 农业部科技教育司, 财政部教科文司. 中国农业产业技术发展报告(2010年度)[R]. 北京: 中国农业出版社, 2011.
[7] 马代夫, 李强, 曹清河, 等. 中国甘薯产业及产业技术的发展与展望[J]. 江苏农业学报, 2012(5): 969-973.
[8] Zhang Liming, Wang Qingmei, Liu Qingchang, et al. Sweetpotato in China [M]∥Loebenstain G, Thottappilly G. Biology and biotechnology of sweetpotato. Netherland: Springer, 2009.
[9] Gao Feng, Gong Yifu, Zhang Pinbo. Production and employment of virus-free sweet potato in China[J]. Crop Protection, 2000, 19: 105-111.
[10]陆漱韵, 刘庆昌, 李惟基. 甘薯育种学[M]. 北京: 中国农业出版社, 1998.
[11]江苏省农业科学院, 山东省农业科学院. 中国甘薯栽培学[M]. 上海: 上海科学技术出版社, 1984.
[12]Xie Y P, Xing J Y, Li X Y, et al. Survey of sweetpotato viruses in China [J]. Acta Virologica, 2013, 57(1): 81-84.
[13]张振臣, 马淮琴, 张桂兰. 甘薯病毒病研究进展[J]. 河南农业科学, 2000(9): 19-22.
[14]Clark C A, Davis J A, Abad J A, et al. Sweetpotato viruses: 15 years of progress on understanding and managing complex diseases [J]. Plant Disease, 2012, 96: 168-185.
[15]Zhang S C, Ling K. Genetic diversity of sweet potato begomoviruses in the United States and identification of a natural recombinant betweenSweetpotatoleafcurlvirusandSweetpotatoleafcurlGeorgiavirus[J]. Archives of Virology, 2011, 156(6): 955-968.
[16]Zerbini F M, Briddon R W, Idris A, et al. ICTV virus taxonomy profiles:Geminiviridae[J]. Journal of General Virology, 2017, 98: 131-133.
[17]ICTV. ICTV-Master-Species-List-2016[EB/OL]https: ∥talk. ictvonline. org/files/master-species-lists/m/msl/6776.
[18]Varsani A, Roumagnac P, Fuchs M, et al.CapulavirusandGrablovirus: two new genera in the familyGeminiviridae[J]. Archives of Virology, 2017, doi: 10. 1007/s00705-017-3268-6.
[19]Clark C A, Valverde R A, Fuentes S, et al. Research for improved management of sweetpotato pests and diseases: cultivar decline [J]. Acta Horticulturae, 2002, 583: 103-112.
[20]ICTV.ICTV-Master-Species-List-2013 [EB/OL](2014-06-30).http:∥talk. ictvonline.org/files/ictv_documents/m/msl/4911.
[21]Luan Y S, Zhang J, Liu D M, et al. Molecular characterization ofSweetpotatoleafcurlvirusisolate from China (SPLCV-CN) and its phylogenetic relationship with other members of theGeminiviridae[J]. Virus Genes, 2007, 35(2): 379-385.
[22]Swapna Geetanjali A, Shilpi S, Mandal B. Natural association of two different betasatellites withSweetpotatoleafcurlvirusin wild morning glory (Ipomoeapurpurea) in India [J]. Virus Genes, 2013, 47(1): 184-188.
[23]Hassan I, Orílio A F, Fiallo-Olivé E, et al. Infectivity, effects on helper viruses and whitefly transmission of the deltasatellites associated with sweepoviruses (genusBegomovirus, familyGeminiviridae)[J]. Scientific Reports, 2016, 6: 30204.
[24]Lozano G, Trenado H P, Fiallo-Olivé E, et al. Characterization of non-coding DNA satellites associated with sweepoviruses (genusBegomovirus,Geminiviridae)-definition of a distinct class ofBegomovirus-associated satellites [J]. Frontiers in Microbiology, 2016, 7: 162-167.
[25]Paprotka T, Boiteux L S, Fonseca M E N, et al. Genomic diversity of sweet potato geminiviruses in a Brazilian germplasm bank [J]. Virus Research, 2010, 149(2): 224-233.
[26]Albuquerque L C, Inoue-Nagata A K, Pinheiro B, et al. Genetic diversity and recombination analysis of sweepoviruses from Brazil [J]. Virology Journal, 2012, 9: 241.
[27]King A M Q, Adams M J, Carstcns E B, et al. Virus Taxonomy-9th Report of the ICTV [R]. San Diego: Elsevier/Academic Press, 2012.
[28]Su L, Gao S, Huang Y, et al. Complete genomic sequence of Dracaena mottle virus, a distinct badnavirus [J]. Virus Genes, 2007, 35(2): 423-429.
[29]费继锋, 肖火根, 李华平, 等. 香蕉线条病毒病研究进展[J]. 病毒学报, 2001, 17(4): 381-385.
[30]Geering A D W, McMichael L A, Dietzgen R G, et al. Genetic diversity among Banana streak virus isolates from Australia[J]. Phytopathology, 2000, 90(8): 921-927.
[31]Harper G, Hart D, Moult S, et al. Banana streak virus is very diverse in Uganda [J]. Virus Research, 2004, 100(1): 51-56.
[32]Harper G, Hart D, Moult S, et al. The diversity of Banana streak virus isolates in Uganda[J]. Archives of Virology, 2005, 150(12): 2407-2420.
[33]Jaufeerally-Fakim Y, Khorugdharry A, Harper G. Genetic variants of Banana streak virus in Mauritius [J]. Virus Research, 2006, 115(1): 91-98.
[34]Muller E, Sackey S. Molecular variability analysis of five new complete Cacao swollen shoot virus genomic sequences [J]. Archives of Virology, 2005, 150(1): 53-66.
[35]Tzafrir I, Ayala-Navarrete L, Lockhart B E L, et al. The N-terminal portion of the 216-kDa polyprotein of commelina yellow mottle badnavirus is required for virus movement but not for replication [J]. Virology, 1997, 232(2): 359-368.
[36]Kreuze J F, Perez A, Untiveros M, et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: A generic method for diagnosis, discovery and sequencing of viruses [J]. Virology, 2009, 388(1): 1-7.
[37]Trenado H P, Orílio A F, Márquez-Martín B, et al. Sweepoviruses cause disease in sweet potato and relatedIpomoeaspp.: Fulfilling Koch's postulates for a divergent group in the genusBegomovirus[J]. PLoS ONE, 2011, 6(11): e27329.
[38]Bi H, Zhang P. Molecular characterization of two sweepoviruses from China and evaluation of the infectivity of cloned SPLCV-JS inNicotianabenthamiana[J]. Archives of Virology, 2012, 157(3): 441-454.
[39]Chung M L, Liao C H, Chen M J, et al. The isolation transmission and host range of sweet potato leaf curl disease agent in Taiwan [J]. Plant Protection Bulletin, 1985, 27: 333-341.
[40]Lotrakul P, Valverde R A, Clark C A, et al. Detection of a geminivirus infecting sweet potato in the United States [J]. Plant Disease, 1998, 82: 1253-1257.
[41]Cohen J, Milgram M, Antignus Y, et al. Ipomoea crinkle leaf curl caused by a whitefly-transmitted gemini-like virus[J]. Annals of Applied Biology, 1997, 131: 273-282.
[42]Onuki M, Hanada K. PCR amplification and partial nucleotide sequences of three dicot-infecting geminiviruses occurring in Japan[J]. Annals of the Phytopathological Society of Japan, 1998, 64: 116-120.
[43]Banks G K, Bedford I D, Beitia F J, et al. A novel geminivirus ofIpomoeaindica(Convolvulacae) from Southern Spain[J]. Plant Disease, 1999, 83: 486.
[44]Briddon R W, Bull S E, Bedford I D. Occurrence ofSweetpotatoleafcurlvirusin Sicily [J]. Plant Pathology, 2006, 55: 286.
[45]Fuentes S, Salazar L F. First report ofSweetpotatoleafcurlvirusin Peru [J]. Plant Disease, 2003, 87: 98.
[46]Miano D W, LaBonte D R, Clark C A, et al. First report of a begomovirus infecting sweetpotato in Kenya[J]. Plant Disease, 2006, 90: 832.
[47]Kwak H R, Kim M K, Chung M N, et al. Virus disease incidences of sweet potato in Korea [J]. The Plant Pathology Journal, 2006, 22: 239-247.
[48]Luan Y S, Zhang J, An L J. First report ofSweetpotatoleafcurlvirusin China [J]. Plant Disease, 2006, 90 (8): 1111.
[49]Prasanth G, Hegde V. Occurrence ofSweetpotatofeatherymottlevirusandSweetpotatoleafcurlGeorgiaviruson sweet potato in India [J]. Plant Disease, 2008, 92: 311.
[50]Qin Y H, Zhang Z Z, Qiao Z, et al. First report ofSweetpotatoleafcurlGeorgiaviruson sweet potato in China [J]. Plant Disease, 2013, 97(10): 1388.
[51]汤亚飞, 何自福, 韩利芳, 等. 侵染广东甘薯的甘薯曲叶病毒分子检测与鉴定[J]. 植物保护, 2013, 39(4): 25-28.
[52]Liu Qili, Zhang Zhenchen, Qi Qiao, et al. Complete genome sequence of a novel monopartite begomovirus infecting sweet potato in China [J]. Virus Genes, 2013,47(3):591-594.
[53]Liu Qili, Zhang Zhenchen, Li Jianqiang et al. Complete genome sequence of a novel monopartite begomovirus infecting sweet potato in China [J]. Archives of Virology, 2014,159(6):1537-1540.
[54]Mbanzibwa D R, Tairo F, Gwandu C, et al. First report ofSweetpotatosymptomlessvirus1 andSweetpotatovirusAin sweetpotatoes in Tanzania[J]. Plant Disease, 2011, 95(2): 224.
[55]Wang Y J, Zhang D S, Zhang Z C, et al. First Report onSweetpotatosymptomlessvirus1(genusMastrevirusfamilyGeminiviridae) in sweetpotato in China[J]. Plant Disease, 2015, 99(7): 1042.
[56]乔贞贞, 秦艳红, 乔奇, 等. 甘薯卷叶病毒江苏分离物基因组全长序列测定及其外壳蛋白基因在大肠杆菌中的表达[J]. 河南农业科学, 2012(4): 86-89.
[57]李学成, 张振臣, 乔奇, 等. 甘薯杆状DNA病毒B的分子检测及外壳蛋白CP基因的原核表达[J]. 植物保护学报, 2016,43(2): 345-346.
[58]Briddon R W, Markham P G. Universal primers for the PCR amplification of dicot-lnfecting geminiviruses [J]. Molecular Biotechnology, 1994, 1(2): 202-205.
[59]Briddon R W, Bull S E, Mansoor S, et al. Universal primers for the PCR-mediated amplification of DNAβ[J]. Molecular Biotechnology, 2002, 20(3): 315-318.
[60]Kashif M, Pietilä S, Artola K. Detection of viruses in sweetpotatoes from Honduras and Guatemala augmented by deep-sequencing of small-RNAs[J]. Plant Disease, 2012, 96(10): 1430-1437.
(责任编辑:杨明丽)
Advances in research of DNA virus infecting sweet potato
Liu Qili1, Zhang Jianxin2, Li Xuecheng3, Shi Mingwang1, Ou Xingqi1
(1. College of Resource and Environmental Science, Henan Institute of Science and Technology,Xinxiang 453003, China; 2. College of Fisheries, Henan Normal University, Xinxiang 453007, China;3. Henan Top Found Pharmaceutical Co. Ltd, Zhumadian 463000, China)
Sweet potato is an important food crops and food processing and industrial raw materials. China is the largest producer of sweet potato in the world. Virus diseases were the important diseases on sweet potato. The reported viruses infecting sweet potato mainly belonged toGeminiviridaeandCaulimoviridae. In recent years, begomoviruses and other DNA viruses became serious threats to planting industry of sweet potato in China and influenced the yield, quality of sweet potato and the food processing industry. In order to know more about the DNA viruses infecting sweet potato, the important status and planting situation of sweet potato in China were briefly introduced, and the characteristics, molecular variation, classification situation and detection methods of these viruses belonged toBegomovirus,MastrevirusandBadnaviruswere concretely described in this review. Combining with the actual situation of sweet potato planting in China, the existing problems and thinking in the research of DNA viruses infecting sweet potato were brought up. The objects of this paper were to provide the theoretical basis for the comprehensive prevention and control for sweet potato diseases infected by DNA viruses.
sweet potato; DNA virus;Begomovirus;Mastrevirus;Badnavirus
2016-06-29
2016-08-05
国家自然科学基金(31372105);国家十三五重点研发计划(2016YFD0201000);河南科技学院高层次人才科研启动项目(2015028)
S 435.31
A
10.3969/j.issn.0529-1542.2017.03.006
联系方式 E-mail: liuqili2002@163.com
