Escherichia coli str. K-12 substr. MG1655海藻糖酶Tre F的重组表达及性质研究
2017-05-19于林港宿玲恰吴敬
于林港宿玲恰吴敬,
(1. 江南大学食品科学与技术国家重点实验室,无锡 214122;2. 江南大学生物工程学院 工业生物技术教育部重点实验室,无锡 214122)
Escherichia coli str. K-12 substr. MG1655海藻糖酶Tre F的重组表达及性质研究
于林港1宿玲恰2吴敬1,2
(1. 江南大学食品科学与技术国家重点实验室,无锡 214122;2. 江南大学生物工程学院 工业生物技术教育部重点实验室,无锡 214122)
海藻糖酶(Trehalase)是一种海藻糖水解酶,能够特异性的将海藻糖分解为两分子的葡萄糖。为了将Escherichia coli str. K-12 substr. MG1655的海藻糖酶基因Tre F在E. coli BL21(DE3)中重组表达和应用,该研究通过PCR扩增获得E. coli str. K-12 substr. MG1655的海藻糖酶基因Tre F,构建了基因工程菌E. coli BL21(DE3)/pET-24a(+)-Tre F。对重组菌进行摇瓶发酵,25℃,IPTG浓度为0.4 mmol/L时,摇瓶发酵诱导24 h时得到最高酶活为107 U/mL。进一步研究了海藻糖酶的酶学性质,发现该海藻糖酶的最适pH为7.0,最适温度为50℃;此外,将该酶应用于海藻糖的水解,起始海藻糖浓度为300 g/L,初始pH 7.0、反应温度30℃,加酶量为84 U/g,反应36 h,葡萄糖转化率可达98.4%。该研究是首次将E. coli str. K-12 substr. MG1655海藻糖酶基因Tre F在E. coli BL21(DE3)宿主中重组表达的报道。
海藻糖酶;基因克隆;重组表达;酶学特性
海藻糖(Trehalose)分子是由两个吡喃葡萄糖环分子以α,α-1,1-糖苷键连接而成的一种十分稳定的非还原二糖[1]。海藻糖广泛存在于自然界的生物体内,包括细菌、真菌、昆虫、低等植物、脊椎动物,特别在真菌和昆虫中的含量非常高,是国际上最近开发的主要低聚糖之一[2-5]。虽然海藻糖的来源很广泛,但能在体内大量积累海藻糖的生物并不是很多[6]。目前正在研究和开发的海藻糖制备方法主要有微生物抽提法、发酵法、酶转化法及基因重组法[6]。然而从微生物中提取海藻糖成本高,提取源有限,很难实现大规模工业化生产;发酵法生产海藻糖存在转化率低,发酵液成分复杂,海藻糖的提取、精制困难等问题;基因重组法生产海藻糖目前研究还不完善,技术有待发展,不能广泛应用于工业生产。目前工业上常用的方法是采用麦芽糖或淀粉等为底物,利用与海藻糖合成有关的酶的作用转化成海藻糖。然而采用酶转化方法生产海藻糖时,依然会有一些问题需要解决,如在将海藻糖制备提取后仍然会产生含有葡萄糖和少量海藻糖等组分的母液,造成资源的浪费。如果能将母液中的海藻糖水解为葡萄糖,就可以实现母液的有效再利用,减少资源浪费。因此,本研究将对海藻糖的水解酶进行研究,具有重要的研究价值和应用前景。
海藻糖酶(EC:3.2.1.28)是一种海藻糖水解酶,能够特异性的将海藻糖分解为两分子的葡萄糖[7]。海藻糖酶最早是Bourquelot于1893年在黑曲霉中发现的,1895年Fischer在酿酒酵母里也发现了海藻糖酶[8]。随后,科研人员在不同的生物里不断地发现和鉴定了不同的海藻糖酶,这些生物包括细菌、酵母、真菌、昆虫、线虫、植物和脊椎动物[9-12]。微生物中海藻糖的水解酶分为两类[13]:中性海藻糖酶(NTH,由nth1 和nth2 基因编码)和酸性海藻糖酶(ATH,由ath1 基因编码)。其中酸性海藻糖酶定位于液泡,最适pH在4.5左右;而中性海藻糖酶位于细胞质,负责主要的胞内海藻糖分解,最适pH在7.0左右[14-16]。本研究以Escherichia coli str. K-12 substr. MG1655为出发菌株克隆出其中性海藻糖酶基因Tre F,并在E. coli BL21(DE3)中进行高效表达,进而对其酶学特性和应用性能进行了研究,为海藻糖酶的后续制备和应用研究奠定良好基础。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒 E. coli str. K-12 substr. MG1655、E. coli JM109、E. coli BL21(DE3)菌株及表达载体pET-24a(+)均为本实验室保藏。
1.1.2 试剂与培养基 Taq酶、T4 DNA连接酶、PrimerStarTMHS DNA聚合酶、限制性内切酶Nde I和Hind III、碱性磷酸酶(calf intestine alkaline phosphatase,CIAP)、琼脂糖和核酸分子量标准均购自大连宝生物工程有限公司;琼脂糖凝胶DNA回收试剂盒和质粒小提试剂盒均购自北京天根生化科技有限公司;蛋白电泳试剂盒和蛋白分子量标准均购自上海碧云天生物技术有限公司;Ezup柱式基因组DNA抽提试剂盒(细菌)和SanPrep柱式PCR产物纯化试剂盒、异丙基-β-D-硫代半乳糖苷(Isopropyl β-D-1-Thiogalactopyranoside,IPTG)、卡那霉素(Kanamycin,Kan)均购自上海生物工程股份有限公司;酵母粉和胰蛋白胨均购自英国Oxiod公司;所有引物均由上海睿迪生物科技有限公司合成;其他国产分析纯试剂均购自上海国药集团化学试剂有限公司。
LB液体培养基(g/L):NaCl 10.0,酵母粉5.0,胰蛋白胨10.0。LB固体培养基:添加质量体积分数为1.5%-2.0%的琼脂至LB液体培养基。LB-Kan培养基:在LB培养基中添加100.0 μg/mL的Kan。TB培养基(g/L):甘油5.0,酵母粉24.0,胰蛋白胨12.0,KH2PO42.3,K2HPO4×3H2O 16.4。TB-Kan培养基:在TB培养基中添加30.0 μg/mL的Kan。
1.2 方法
1.2.1 基因组DNA的提取 E. coli str. K-12 substr. MG1655基因组DNA的提取按照上海生工的细菌基因组抽提试剂盒说明书提取获得。
1.2.2 引物设计与基因克隆 根据GenBank中海藻糖酶的同源序列(NC_000913.3)为模板,按需要加入适宜的内切酶(Nde I和Hind III)位点,设计出一对PCR引物P1/P2:上游引物P1:5'-GGAATTCCA TATGCTCAATCAGAAAATTCAAAACC-3';(下划线处为酶切位点Nde I);下游引物P2:5'-CCCAAGCTTA TGGTTCGCCGTACAAACCAATTA-3'。(下划线处为酶切位点Hind III);以E. coli str. K-12 substr. MG1655基因组DNA为模板,以P1/P2为引物进行PCR扩增。PCR扩增体系(50 μL):ddH2O 33.5 μL,dNTP混合物4 μL,5×PS Buffer 10 μL,模板1 μL,上游引物P10.5 μL,下游引物P20.5 μL,PrimerSTARTMHS DNA聚合酶0.5 μL。PCR扩增条件为:于95℃预变性5 min,然后开始循环,首先于98℃变性10 s,然后于55℃退火5 s,最后于72℃延伸110 s,循环30次后于72℃延伸10 min,并于4℃保温。
将PCR产物进行琼脂糖(1%)凝胶电泳验证,将验证正确的PCR产物进行胶回收,胶回收后的产物在PCR仪或者恒温水浴锅中72℃反应10 min完成平末端加A,然后将加A后的产物进行DNA产物纯化,最后将纯化后产物连接至pMD19-T simple vector,并转化E. coli JM109感受态细胞,涂布LBKan固体培养基,于37℃恒温培养箱培养过夜。挑取阳性克隆至LB-Kan液体培养基,抽提质粒经限制性内切酶Nde I和Hind III双酶切,通过琼脂糖凝胶电泳验证,并将验证正确的序列送睿迪基因测序。测序正确的重组质粒命名为pMD19T-Tre F,-80℃甘油管保存。
1.2.3 E. coli重组表达质粒的构建 分别用限制性内切酶Nde I和Hind III对测序正确的pMD19T-Tre F质粒和表达载体pET-24a(+)进行双酶切,用DNA胶回收试剂盒将目的片段回收,然后用T4连接酶将目的基因和表达载体16℃连接过夜,转化E. coli JM109感受态细胞,涂布LB-Kan固体培养基,于37℃恒温培养箱培养过夜。挑取阳性克隆至LBKan液体培养基,抽提质粒经限制性内切酶Nde I和Hind III双酶切,通过琼脂糖凝胶电泳验证。将验证正确的序列送睿迪基因测序。测序正确的重组质粒命名为pET-24a(+)-Tre F,-80℃甘油管保存。将测序正确的质粒转化表达宿主E. coli BL21(DE3),-80℃甘油管保存。
1.2.4 重组菌的诱导表达和优化 将重组菌E. coli BL21(DE3)/pET-24a(+)-Tre F接种至LB-Kan液体培养基,于37℃,200 r/min培养8 h,以5%的接种量转接上述菌液至50 mL TB-Kan培养基中,于37℃,200 r/min培养至OD600约为1.0时,加入终浓度为0.4 mmol/L的IPTG,于25℃,200 r/min诱导发酵。
在摇瓶发酵的基础上优化了不同诱导剂浓度和不同的诱导温度对产酶的影响。本实验通过改变单因子的方法对诱导条件进行优化,根据以上测酶活方法获得的酶活大小作为优化参考标准,每个因素做3个平行。
1.2.5 重组菌生长曲线的测定和粗酶液的制备 为了检测重组菌随着时间的生长情况,分别在不同的诱导时间点取样1 mL发酵液,在吸光值600 nm测量菌体的OD600。取一定量发酵液离心收集得到菌体并采用去离子水洗涤3次后,当OD600低于5.0时直接用1 mL 50 mmol/L pH7.0 的K2HPO4-KH2PO4缓冲液悬浮菌体,当菌体生长至OD600大于5.0时,采用缓冲液稀释至5.0,超声破碎(破碎2 s、间歇3 s,总时间10 min)后离心10 min,破壁上清即为Tre F粗酶液,单位体积酶活根据相应体积发酵液菌体稀释倍数折算得出。
浓缩粗酶液的制备:取500 mL发酵液离心收集得到菌体并采用去离子水洗涤3次后,用100 mL 50 mmol/L pH 7.0 K2HPO4-KH2PO4缓冲液悬浮菌体,超声破碎(破碎2 s、间歇3 s,总时间30 min)后离心10 min,破壁上清即为浓缩的Tre F粗酶液。
1.2.6 酶活力测定 将1 mL适当浓度(80 g/L)的海藻糖溶液和0.9 mL的50 mmol/L,pH7.0的磷酸缓冲液充分混匀,在37℃预热10 min,加入100 μL适当稀释的酶液,反应10 min后加入3 mL 3,5-二硝基水杨酸(DNS),煮沸7 min迅速冷却,加蒸馏水定容至15 mL,在540 nm下测吸光度(以缓冲液同样操作作为空白对照)[17]。
在上述条件下,将每分钟水解1 μmol海藻糖生成2 μmol的葡萄糖所需酶量定义为一个酶活单位(1U)。
1.2.7 海藻糖酶的酶学性质 (1)温度对重组Tre F酶活力的影响:分别测定Tre F在30-70℃(间隔5℃)下的酶活,将Tre F最高酶活设定为100%。实验方法同上,每个梯度设置3个平行实验。(2)pH对重组Tre F酶活力的影响:分别测定Tre F在pH 4.5-8.0(间隔为0.5)的50 mmol/L柠檬酸-K2HPO4缓冲液中的酶活,将Tre F最高酶活设定为100%。实验方法同上,每个梯度设置3个平行实验。(3)重组Tre F稳定性:将Tre F分别置于最适pH下,温度为30℃、37℃以及50℃的恒温水浴锅中,测定不同时间的残留酶活,将未经过热处理的样品的酶活设定为100%。实验方法同上,每个时间点设置3个平行实验。
1.2.8 加酶量对葡萄糖转化率的影响 用pH值为7.0的50 mmol/L K2HPO4-KH2PO4缓冲液配制反应底物海藻糖溶液(300 g/L),分别加入12 U/g、24 U/g、36 U/g、48 U/g、60 U/g、72 U/g、84 U/g、96 U/g、108 U/g、120 U/g及132 U/g浓缩后的粗酶液,在30℃,150 r/min的水浴摇床中反应36 h。用HPLC检测反应液中海藻糖的残余量和葡萄糖的生成量,计算海藻糖转化生成葡萄糖的转化率,每个梯度做3个平行。
葡萄糖含量的测定:产物中的葡萄糖糖含量采用HPLC检测。色谱条件为:安捷伦 1200 HPLC色谱仪,色谱柱 APS-2 HYPERSIL(250 mm×4.6 mm),示差检测器为安捷伦 2410;流动相为80%(V/V)乙腈和水的混合溶液,流速为0.8 mL/min,柱温设定为40℃。葡萄糖转化率的计算:

2 结果
2.1 目的基因的克隆
以E. coli str. K-12 substr. MG1655的基因组DNA为模板,用引物P1/P2进行PCR扩增海藻糖酶基因Tre F,将PCR产物进行1%琼脂糖凝胶电泳,扩增出的DNA片段约在1 650 bp处,与预期大小相符(图1)。将目的基因与pMD19-T simple vector载体连接并转化E. coli JM109感受态细胞,提取质粒用Nde I和Hind III双酶切,琼脂糖凝胶电泳(图2)显示约为2 600 bp和1 650 bp两条带,与预期相符。测序结果显示目的片段全长1 650 bp,编码549个氨基酸,通过DNAMAN软件比对与E. coli str. K-12 substr. MG1655的Tre F基因序列(NC_000913.3)相似度达到99%,蛋白质序列相似度为100%。可以推断克隆的基因为E. coli str. K-12 substr. MG1655的海藻糖酶Tre F基因。
2.2 重组表达质粒的构建
将测序正确的目的基因用Nde I和Hind III双酶切,与同样酶切的表达载体pET-24a(+)进行连接,连接产物转化E. coli JM109,挑取单克隆提取质粒用Nde I和Hind III双酶切验证,结果如图3所示,琼脂糖凝胶电泳显示约为5 300 bp和1 650 bp的两条片段,重组表达质粒构建成功,命名为pET-24a(+)-Tre F。
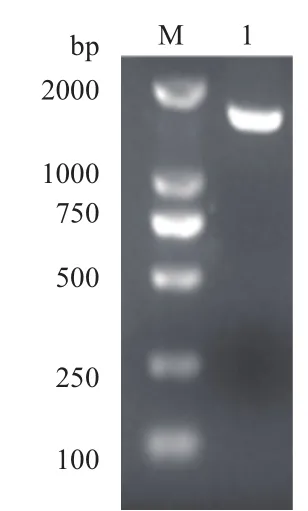
图1 PCR产物琼脂糖凝胶电泳验证
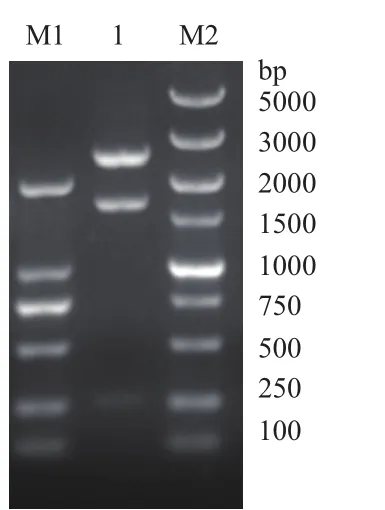
图2 pMD19T-Tre F Nde I和Hind III双酶切琼脂糖凝胶电泳验证
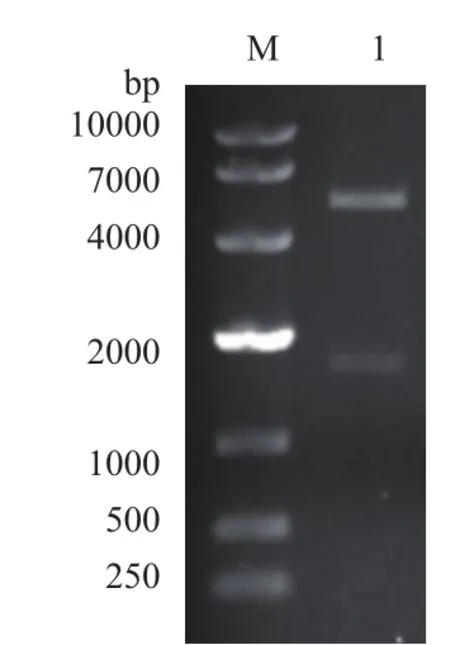
图3 pET-24a(+)-Tre F Nde I和Hind III双酶切琼脂糖凝胶电泳验证
2.3 重组酶在E. coli中的表达
将验证正确的重组质粒pET-24a(+)-Tre F转化E. coli BL21(DE3)感受态细胞,涂布LB-Kan固体平板,挑取单菌落到LB-Kan培养基中培养过夜,以5%的转接量将菌液接种到TB-Kan培养基,待OD600约为1.0时,加入终浓度为0.4 mmol/L的IPTG,于25℃,200 r/min诱导,分别在不同的时间进行取样测定重组菌的生长曲线和产酶曲线,如图4所示,发酵培养26 h时,OD600为11.9,海藻糖酶活达到最高为103 U/mL。对样品进行SDS-PAGE电泳鉴定,如图5所示,重组菌E. coli BL21(DE3)/ pET-24a(+)-Tre F成功表达了分子量约为63 kD的蛋白,与预期结果一致。
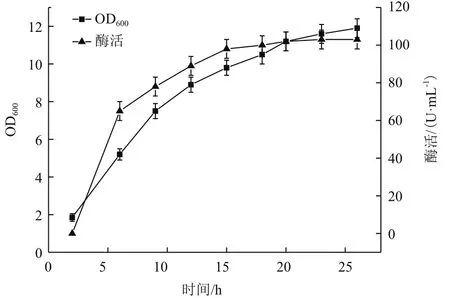
图4 重组菌E. coli BL21(DE3)/pET-24a(+)-Tre F的细胞生长情况和产酶情况
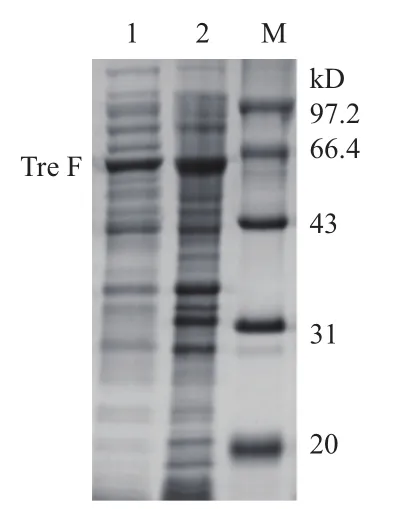
图5 重组Tre F的SDS-PAGE凝胶电泳分析
2.4 重组菌诱导条件的优化
2.4.1 IPTG最佳诱导浓度的确定 本研究选取0.1、 0.2、0.4、0.6、0.8 mmol/L等不同的IPTG浓度对重组菌进行诱导发酵。如图6所示,重组菌的OD600随着IPTG诱导浓度的增加呈现降低的趋势;而重组酶的酶活则是随着诱导剂浓度呈现先增加后下降的趋势,当IPTG的浓度低于0.4 mmol/L时,重组菌海藻糖酶的酶活力随着IPTG浓度的增加而提高,0.4 mmol/L时酶活达到最高为103 U/mL;当IPTG诱导浓度超过0.4 mmol/L时,重组菌的酶活开始下降。因此,重组菌的最佳IPTG诱导剂浓度为0.4 mmol/L。
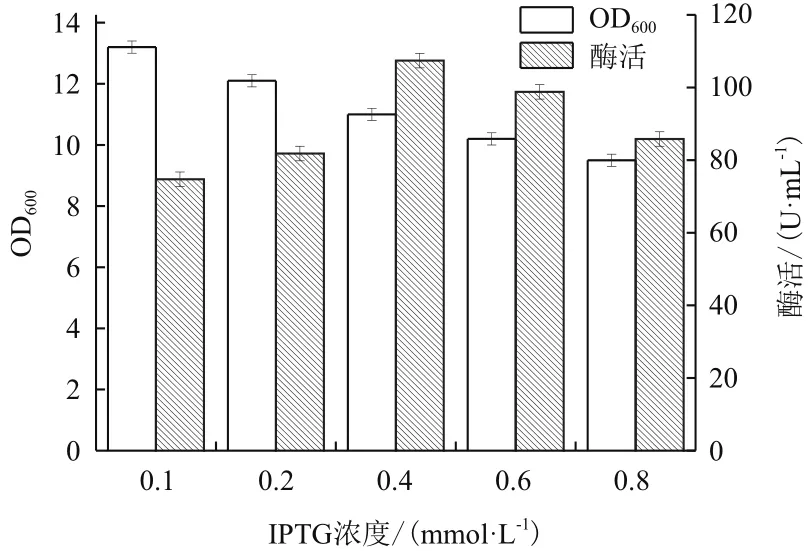
图6 IPTG浓度对重组菌的生物量和产酶情况的影响
2.4.2 最佳诱导温度的确定 温度的高低决定着微生物细胞内新陈代谢的酶催化反应,低的诱导温度可能导致重组菌生长和产酶较慢,而高的诱导温度又可能导致重组蛋白易折叠错误而以包涵体的形式从而导致酶活很低,因此需要对重组菌的诱导温度进行优化。本研究选取了3个不同的诱温度25℃、30℃和37℃,考察了诱导温度对重组菌的生长和产酶的影响,结果如图7所示。重组菌的OD600随着诱导温度的增加显示为先增加后降低,30℃时重组菌的OD600最高为12.5,25℃为11.5,37℃最低为11;而重组菌的酶活则是随着温度升高而降低,在25℃时酶活最高为107 U/mL,30℃时酶活为87.5 U/mL,当诱导温度为37℃时酶活最低仅有35 U/mL,可以看出低温有利于重组酶的表达。由于低于25℃的温度不易控制,成本较高,故本研究选取发酵产酶的最佳温度为25℃。
2.5 重组Tre F的酶学性质
2.5.1 温度对重组Tre F酶活力的影响 将酶液分别置于不同温度进行酶反应并测定酶活,以最高酶活为100%,结果如图8,海藻糖酶反应液在50-60℃时活性较高,在50℃时酶活最高,而当温度超过60℃后酶活急剧下降,由此可推断出海藻糖酶的最适反应温度为50℃。

图7 诱导温度对重组菌的生物量和产酶情况的影响
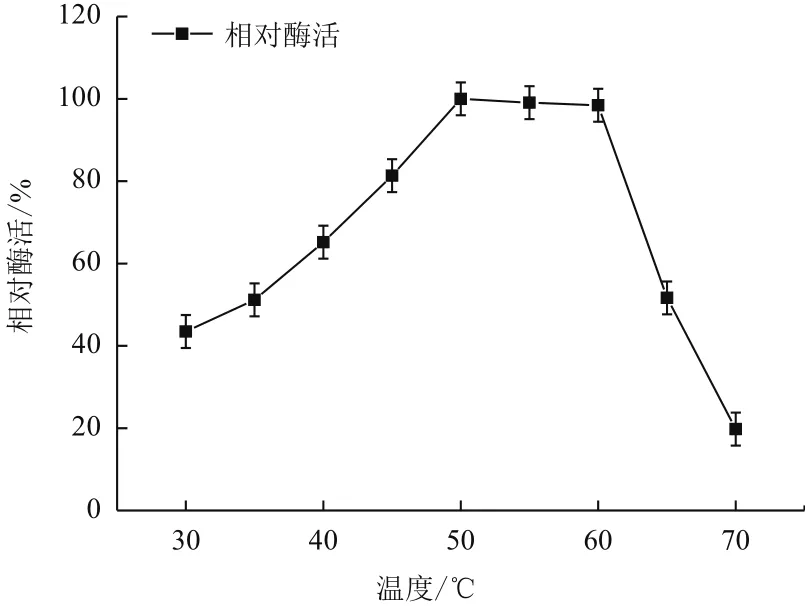
图8 反应温度对重组Tre F酶活力的影响
2.5.2 pH对重组Tre F的影响 待测样品分别置于不同pH的缓冲液中测定酶活,以最高酶活为100%,结果见图9。当pH小于7.0时海藻糖酶的酶活随着pH的增加而上升,海藻糖酶在pH 7.0处酶活最高,而当pH大于7.0时海藻糖酶的酶活随着pH的增加而逐渐下降。由此可判断海藻糖酶的最适反应pH为7.0。
2.5.3 重组Tre F的温度稳定性 将粗酶液分别置于30℃、37℃、50℃的水浴锅进行保温,测定其残留酶活。结果(图10)显示,Tre F在30℃放置24 h,酶活基本无下降,可见重组酶在30℃下具有良好的稳定性;在37℃下放置时,Tre F半衰期约为12 h,具有较好的稳定性;而在50℃下放置时,酶迅速失活,半衰期大约只有10 min,可见重组酶在较高温度条件下稳定性较差。综上所述,Tre F在30℃时具有良好的稳定性。
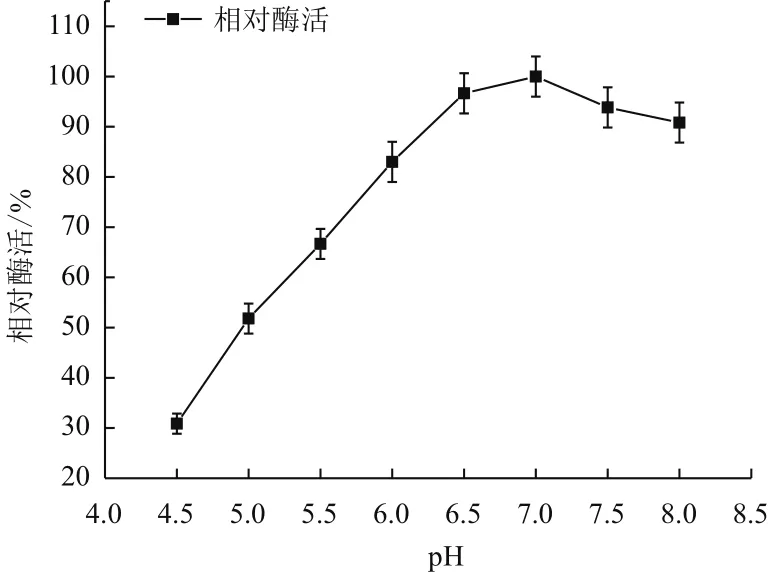
图9 pH对重组Tre F酶活力的影响
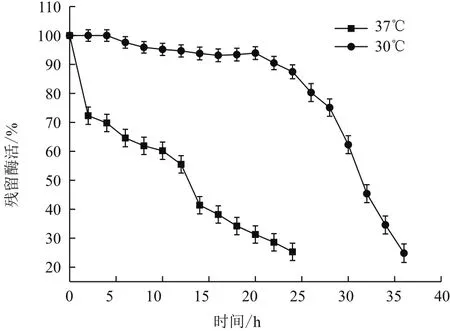
图10 重组Tre F在30℃和37℃的温度稳定性
2.6 加酶量对葡萄糖转化率的影响
由于重组Tre F在30℃的稳定性较好,保温24 h酶活基本不变,因此酶转化时选取30℃进行酶反应。葡萄糖转化率结果如图11所示,相同的反应时间内葡萄糖的转化率随着海藻糖酶的加酶量的逐渐增加而增大,当单位加酶量为24 U时转化率就能达到84.3%,当单位加酶量为36 U时转化率能达到92.2%,继续提高加酶量到单位加酶量为84 U时转化率达到最高98.4%,之后提高加酶量葡萄糖的转化率基本不变。因此,重组Tre F酶转化反应的最适条件为:单位加酶量为84 U,反应温度为30℃,pH为7.0,反应时间为36 h。

图11 加酶量对葡萄糖转化率的影响
3 讨论
目前,国内外研究较多的海藻糖酶主要有昆虫来源,如绿盲蝽(Apolygus lucorum)[18,19]、大豆蚜(Aphis glycines)[20]、棉铃虫(Helicoverpa armigera)[21]、甜菜夜蛾(Spodopteraexigua)[22]和烟粉虱(Bemisia tabaci)[23]等,真菌来源如酿酒酵母(Saccharomyces cerevisiae)等,植物来源如拟南芥(Arabidopsis thaliana)等,而对细菌来源的海藻糖酶进行研究的报道并不多。谭永安等[19]将绿盲蝽水溶性海藻糖酶ALTre-1基因在宿主E. coli BL21(DE3)中能够高效表达,纯化获得的重组蛋白具有较高的海藻糖水解活性,最适温度为55℃,最适pH为7.0,在最适温度和最适pH下纯化获得的重组蛋白酶活达到(228.59±4.62)nmol·μg-1·min-1。吕烨等[24]构建了酵母海藻糖酶缺失突变株,使突变株的海藻糖积累量和细胞密度均高于亲本,提高了其冷冻、高温、高糖和酒精耐性。郭蓓等[25]将拟南芥海藻糖酶基因在E. coli BL21(DE3)菌株中进行高效诱导表达,纯化获得的海藻糖酶蛋白在试管条件下具有较高水解活性的海藻糖酶(报道未提到酶活力情况),其最适温度为45℃。
本研究以E. coli str. K-12 substr. MG1655的基因组为模板,克隆PCR扩增出其海藻糖酶基因Tre F,选用pET-24a(+)为表达载体,构建了高效原核表达重组质粒,将重组菌转化表达宿主E. coli BL21(DE3),对重组菌进行诱导表达,并对重组菌进行诱导条件优化,最佳诱导温度为25℃,最佳IPTG诱导浓度为0.4 mmol/L。在最佳诱导条件下:海藻糖酶的酶活可达到107 U/mL。此外,通过酶学研究,发现海藻糖酶的最适反应温度为50℃,最适pH为7.0。将该酶用于海藻糖水解时,当底物海藻糖浓度为300 g/L,加酶量为每克底物84 U,初始pH 7.0,温度为30℃,150 r/min,反应36 h,葡萄糖的转化率最高为98.4%。这一过程说明,从E. coli str. K-12 substr. MG1655中克隆得到的海藻糖酶基因Tre F不但能够在E.coli BL21(DE3)中完整表达,而且得到的重组蛋白对海藻糖具有很好的水解活性。本研究是首次将E. coli str. K-12 substr. MG1655的海藻糖酶基因Tre F进行重组表达的报道,得到的重组蛋白具有较高的海藻糖水解活性(107 U/mL),在30℃具有很好的稳定性(放置24 h酶活基本无下降),将之应用于海藻糖的水解,以300g/L的海藻糖溶液为底物水解率高达98.4%。与其他来源的海藻糖酶相比,E. coli str. K-12 substr. MG1655来源的海藻糖酶基因在表达宿主E. coli BL21(DE3)重组表达是同源表达,具有表达效率高、操作简单、应用效果好等优点。
4 结论
构建了重组菌E. coli BL21(DE3)/pET-24a(+)-Tre F,对诱导温度、IPTG诱导浓度进行发酵优化,获得最佳诱导发酵条件:25℃,0.4 mmol/L IPTG,诱导24 h后,Tre F酶活达到107 U/mL。Tre F酶的最适反应pH为7.0,最适反应温度是50℃。此外,将该酶用于海藻糖水解,当底物海藻糖浓度为300 g/L,单位底物加酶量为84 U,初始pH 7.0,温度为30℃,150 r/min,反应36 h,葡萄糖的转化率达到98.4%。
[1] 马文锦, 刘树兴, 潘巨忠, 等. 海藻糖的生产制备及应用前景[J]. 现代农业科技, 2007, 24:199-201.
[2]Elbein AD, Pan YT, Pastuszak I, et al. New insights on trehalose:a multifunctional molecule[J]. Glycobiol, 2003, 13(4):17-27.
[3]Elbein AD. The metabolism of α, α-trehalose[J]. Advances in Carbohydrate Chemistry and Biochemistry, 1974, 30:227-256.
[4]Wingler A. The function of trehalose biosynthesis in plants[J].Phytochemistry, 2002, 60(5):437-440.
[5]Frison M, Parrou JL, Guillaumot D, et al. The Arabidopsis thaliana trehalase is a plasma membrane-bound enzyme with extracellular activity[J]. FEBS Letters, 2007, 581(21):4010-4016.
[6]胡宗利, 夏玉先, 陈国平, 等. 海藻糖的生产制备及其应用前景[J]. 中国生物工程杂志, 2004, 24(4):44-48.
[7]Alabran DM, Ball DH, Reese ET . Comparison of the trehalase of Trichoderma reesei with those from other sources[J]. Carbohydrate Research, 1983, 123(1):179-181.
[8]Kopp M, Müller H, Holzer H. Molecular analysis of the neutral trehalase gene from Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 1993, 268(7):4766-4774.
[9]Hill EP, Sussman AS. Purification and properties of trehalase(s)from Neurospora[J]. Archives of Biochemistry and Biophysics, 1963, 102(3):389-396.
[10]Killick KA. Alterations in trehalase solubility during development in the cellular slime mould Dictyostelium discoideum[J]. Microbiology, 1985, 131(2):273-278.
[11]Merdinger E, Lange C F, Booker BF. Isolation and identification of trehalase from Pullularia pullulans[J]. Journal of Bacteriology, 1971, 106(3):1034-1035.
[12]Retief LW, Hewitt PH. Purification and properties of trehalase from the harvester termite, trinervitermes trinervoides[J]. Insect Biochemistry, 1973, 3(12):345-351.
[13]Zähringer H, Burgert M, Holzer H, et al. Neutral trehalase Nth1p of Saccharomyces cerevisiae encoded by the NTH1 gene is a multiple stress responsive protein[J]. FEBS Letters, 1997, 412(3):615-620.
[14]Alizadeh P, Klionsky DJ. Purification and biochemical characterization of the ATH1 gene product, vacuolar acid trehalase, from Saccharomyces cerevisiae[J]. FEBS Letters, 1996, 391(3):273-278.
[15]Amaral FC, Van Dijck P, Nicoli JR, et al. Molecular cloning of the neutral trehalase gene from Kluyveromyces lactis and the distinction between neutral and acid trehalases[J]. Archives of Microbiology, 1997, 167(4):202-208.
[16]Jules M, Beltran G, François J, et al. New insights into trehalose metabolism by Saccharomyces cerevisiae:NTH2 encodes a functional cytosolic trehalase, and deletion of TPS1 reveals Ath1pdependent trehalose mobilization[J]. Applied and Environmental Microbiology, 2008, 74(3):605-614.
[17]钟国华, 胡美英, 林进添, 等. 闹羊花素-Ⅲ 对菜青虫海藻糖含量及海藻糖酶活性的影响[J]. 华中农业大学学报, 2000, 19(2):119-123.
[18]谭永安, 肖留斌, 孙洋, 等. 绿盲蝽水溶性海藻糖酶 ALTre-1基因原核表达, 纯化与酶学特性[J]. 中国农业科学, 2013, 46(17):3587-3593.
[19]谭永安, 肖留斌, 柏立新, 等. 绿盲蝽膜结合型海藻糖酶ALTre-2 基因表达, 纯化及酶学特性[J]. 棉花学报, 2015, 25(5):396-402.
[20]Bansal R, Mian MAR, Mittapalli O, et al. Molecular characterization and expression analysis of soluble trehalase gene in Aphis glycines, a migratory pest of soybean[J]. Bulletin of Entomological Research, 2013, 103(3):286-295.
[21]于彩虹, 黄莹, 林荣华, 等. 五种昆虫可溶性海藻糖酶活性比较[J]. 植物保护, 2013, 39(4):5-9.
[22]Chen J, Tang B, Chen H, et al. Different functions of the insect soluble and membrane-bound trehalase genes in chitin biosynthesis revealed by RNA interference[J]. PLoS One, 2010, 5(4):e10133.
[23]Wang J, He W, Su Y, et al. Molecularcharacterization of soluble and membrane-bound trehalases of the whitefly, Bemisia tabaci[J]. Arch Insect Biochem Physiol, 2014, 85(4):216-233.
[24]吕烨, 肖冬光, 和东芹, 等. 酵母海藻糖酶缺失突变株的构建及其耐性[J]. 微生物学报, 2008, 48(10):1301-1307.
[25]郭蓓, 胡磊, 盖颖, 等. 拟南芥海藻糖酶基因克隆及其在大肠杆菌中高效表达与功能研究[J]. 微生物学通报, 2008, 35(2):241-248.
(责任编辑 狄艳红)
Recombinant Expression and Characterization of Trehalase Tre F from Escherichia coli str. K-12 substr. MG1655
YU Lin-gang1SU Ling-qia2WU Jing1,2
(1. State Key Laboratory of Food Science and Technology,Jiangnan University,Wuxi 214122;2. School of Biotechnology and Key Laboratory of Industrial Biotechnology Ministry of Education,Jiangnan University,Wuxi 214122)
Trehalase is a trehalose hydrolase,which can distinctively catalyze trehalose into two molecules of glucose. In order to have a recombinant expression and application of the trehalase gene Tre F from Escherichia coli str. K-12 substr. MG1655 in E. coli BL21(DE3),the Tre F was amplified by PCR,and a recombinant strain E. coli BL21(DE3)/pET-24a(+)-Tre F was constructed. The highest activity reached 107 U/mL when the strain was cultured in shake flask for 24 h at induction temperature 25℃and IPTG induction concentration 0.4 mmol/L. Further study of the enzymatic properties indicated that the optimal pH and temperature was 7.0 and 50℃,respectively. The crude enzyme was used in the hydrolysis of trehalose. The reaction conditions for the conversion was as the following:initial trehalose concentration 300 g/L,initial pH 7.0,reaction temperature 30℃,enzyme concentration 84 U/g. When the reaction was performed under this specified condition,the glucose yield reached the maximum of 98.4 % at 36 h. This paper is the first report on the recombinant expression of trehalase gene Tre F of E. coli str. K-12 substr. MG1655 in E. coli BL21(DE3).
trehalase;gene cloning;recombinant expression;enzyme properties
10.13560/j.cnki.biotech.bull.1985.2017.04.023
2016-09-30
国家自然科学基金杰出青年基金(31425020),江苏省2015年度普通高校研究生实践创新计划(SJLX15_0543)
于林港,男,硕士,研究方向:工业微生物与酶技术;E-mail:13771099654@163.com
吴敬,女,博士生导师,研究方向:食品与发酵;E-mail:jingwu@jiangnan.edu.cn
