鲍亚科似鲍属鱼类的物种界定和系统发育关系
2017-05-16李玉火李明月傅萃长
曹 恺 李玉火 何 欢 李明月 傅萃长
(复旦大学生命科学院生物多样性科学研究所, 生物多样性与生态工程教育部重点实验室, 上海 200438)
鲍亚科似鲍属鱼类的物种界定和系统发育关系
曹 恺 李玉火 何 欢 李明月 傅萃长
(复旦大学生命科学院生物多样性科学研究所, 生物多样性与生态工程教育部重点实验室, 上海 200438)
亚科似鲍属鱼类的物种界定保持争议, 系统发育关系尚待解决。研究取样似鲍属鱼类所有种, 使用核基因多位点序列重建似属鱼类的系统发育关系, 运用分子的物种界定方法并结合形态特征分析厘定我国似属鱼类的分类。贝叶斯系统发育树结果表明: 桂林似鲍与平江似是单系种; 似与扁嘴似是多系种, 前者包括五个谱系A至E, 后者包括两个谱系A与B。POFAD距离分析和Structurama分析的结果表明似和扁嘴似鲍的每个谱系是独立遗传种群, BP & P分析结果强烈支持它们是不同种。*Beast物种树结果揭示: 扁嘴似鲍谱系B位于似鲍属鱼类的基部位置; 似谱系A与B是姊妹群关系, 似谱系C是扁嘴似鲍谱系A的姊妹群, 它们一起与桂林似鲍形成姊妹群关系; 似谱系D与E是姊妹群关系, 它们一起是平江似鲍的姊妹群。结合形态证据, 对我国似属鱼类分类厘定如下: 限定严格意义似鲍包括似谱系A+B; 恢复长吻似鲍(Pseudogobio longirostris Mori, 1934)给予似鲍谱系C分类名; 似谱系D与E是隐存种, 桂林似鲍与平江似鲍是有效种。
鲤科;亚科; 似鲍属; 物种界定; 系统发育
似鲍属鱼类的物种分类一直存在争议。Bănărescu & Nalbant[1]认为似鲍属鱼类包括扁嘴似鲍1种含2亚种, 即指名亚种(Pseudogobio esocinus esocinus)与似鲍亚种(P. esocinus vaillanti)。罗云林等[5]对我国的似鲍属鱼类进行了分类厘订, 认为我国似鲍属鱼类包括1种含3亚种: 似鲍指名亚种(Pseudogobio vaillanti vaillanti)、桂林似鲍(P. vaillanti guilinensis)与长吻似鲍(P. vaillanti longirostris Mori, 1934)。乐佩琦[2]认为长吻似鲍亚种是似鲍指名亚种的同物异名, 并把似鲍指名亚种与桂林似鲍都提升为种。杨秀平等[6]利用形态可量性状使用主成分分析对我国似鲍属鱼类进行了形态比较,认为桂林似鲍为似鲍的同物异名, 我国似鲍属鱼类仅似鲍单种。
基于形态特征与分子证据重建的鲍亚科鱼类系统发育关系研究[7—12]均支持似鲍属鱼类是一单系群, 但是似鲍属鱼类种间的系统发育关系并未解析清楚。夏曦中等[13]利用线粒体细胞色素b基因,对我国辽河、长江、钱塘江、珠江与海南岛的广义似鲍进行了谱系地理研究, 结果表明广义似鲍具有明显的空间遗传结构, 能分为5大谱系, 其中两个谱系分布于长江与钱塘江, 另外三个谱系各自分布于辽河、珠江与海南岛, 谱系间遗传距离达到6%—10%。基于鱼类线粒体基因的种间平均遗传距离一般为8%左右[14—16], 夏曦中等[13]的研究结果暗示我国似鲍属鱼类可能不止似鲍单种。Tominaga等[17]使用线粒体细胞色素b基因对广义扁嘴似鲍进行了初步的谱系地理研究, 结果显示广义扁嘴似鲍是多系群, 包括两大谱系: 一个谱系是日本本岛特有的, 另一个谱系嵌套于似鲍内部。Tominaga等[18]进一步加强取样使用线粒体细胞色素b基因对广义扁嘴似鲍进行了详细的谱系地理研究, 结果表明广义扁嘴似鲍能分为三大谱系A、B与C。其中, 谱系A与分布于我国北方的似鲍形成姊妹群关系; 谱系B与谱系C是姊妹群, 它们一起与分布于我国南方的似鲍形成姊妹群关系。
综上所述, 似鲍属鱼类的物种分类保持争议,系统发育关系尚待进一步解决。清晰的物种界定是准确理解生物多样性空间格局形成过程的基础。最近的研究[19—21]提倡分子证据基础的物种界定方法并结合形态证据进行物种界定。因此, 本研究取样似鲍属鱼类所有物种, 选择5个核基因位点作为分子标记, 使用系统发育技术重建似鲍属鱼类的系统发育关系, 运用分子证据基础的物种界定方法并结合形态证据进行分类厘定, 为将来以该属鱼类作为研究对象, 理解东亚地区淡水鱼类多样性空间格局形成过程奠定基础。
1 材料与方法
1.1 物种取样和分子标记
本研究共取样似鲍属鱼类4种69尾(表 1)。其中广义似鲍58尾, 来自长江、钱塘江、瓯江、灵江、闽江、韩江、鸭绿江、大洋河与辽河水系; 平江似鲍1尾, 来自珠江水系; 桂林似鲍4尾, 来自珠江水系; 广义扁嘴似鲍6尾, 来自日本琵琶湖与云出川水系。依据Tang等[11]重建的鲍亚科鱼类分子系统发育关系, 选取长蛇鲍与棒花鱼作为外类群。分子标记选择5个单拷贝核基因: 外胚层神经皮层蛋白1基因(ENC1)、错配修复蛋白MutL基因3(MLH3)、错配修复蛋白MutS基因6(MSH6)、核位点重组激活基因1(RAG1)和核糖体蛋白S7基因第一内含子(rpS7 Intron 1)。
1.2 DNA提取、PCR扩增与产物测序
从95%酒精保存的肌肉组织中采用高盐法提取基因组DNA[22]。PCR过程使用引物[23,24]见表 2,各基因PCR扩增反应的条件如下: 94℃预变性5min; 94℃变性50s, 52℃退火50s, 72℃延伸60s; 循环40次; 72℃最后延伸8min。PCR产物纯化后在ABI 3730 DNA测序仪中使用扩增引物进行测序。
1.3 序列处理与贝叶斯树的构建
使用DAMBE ver. 5.6.7软件[25]进行蛋白质编码基因的序列对位, 使用MAFFT ver. 7.22软件[26]进行非蛋白质编码基因对位。序列对位完成后手工校正并排除具有插入与缺失的区域。使用MEGA ver. 6.06软件[27]计算基因的分子特征。蛋白质编码基因分区策略按密码子进行, 非蛋白质编码基因不分区。使用jModeltest ver. 2.1.7软件[28], 依据赤池信息准则选择每个分区的最适碱基替换模型。在Mrbayes ver. 3.2.2软件[29]下使用核基因多位点联合序列构建贝叶斯系统发育树, 主要步骤如下: 基于马尔可夫链蒙特卡罗进行5000万代运算, 取样频率为1000, 保证分离频率平均标准偏差小于0.01。运行完成后舍弃前1/4的取样树, 根据剩余的3/4取样树构建50%的多数原则一致树, 并计算每个节点的贝叶斯后验概率值。
1.4 物种界定
在POFAD ver. 1.0.7软件[30]中采用标准化矩阵计算基于核基因多位点的个体之间Genpofad距离,并在SplitsTree4 ver. 4.14.2软件[31]中构建邻接网络图(简称POFAD距离分析)。在Structurama 2.0软件[32]中采用Dirichlet 过程进行种群数最适值的测试, 运算过程中将其设为随机变量并服从形状参数为0.1,尺度参数为10的伽马分布; 共运行5000万代马尔可夫链蒙特卡罗, 每1000代取样一次, 舍弃前1/4的样本; 最终获得具有最高后验概率的种群数(简称Structurama分析)。依据贝叶斯系统发育树、POFAD距离分析以及Structurama分析的结果, 设定先验物种。在BP & P 3.0软件[33,34]中验证种的成立与否。采用无指导树物种界定方法计算指定物种划分方法的后验概率分布, 按theta和tau的先验分布运行3组独立分析: (1) thetaprior=1, 10, tauprior=1, 10; (2) thetaprior=2, 2000, tauprior=2, 2000; (3) thetaprior=1, 10, tauprior=2, 2000。共运行250万代, 每5代取样一次, 舍弃前1/5的样本(简称BP & P分析)。
1.5 *BEAST物种树的构建
在*BEAST ver. 2.3.0软件[35]中构建物种树, 主要步骤包括: 选择对数正态分布的松散钟模型作为分子钟模型, 树的先验模型设定为Yule过程; 运行4个独立重复, MCMC代数设置为20000万代, 取样频率为1000; 舍弃前1/4的样本后, 在Log Combiner ver. 2.3.0软件[35]中合并每个独立重复的log文档和trees文档; 在Tracer ver. 1.5软件[36]中估算合并后的log文档参数, 保证每个参数的有效取样大小大于200; 在Tree Annotator ver. 2.3.0软件[35]中获得最大谱系置信树。
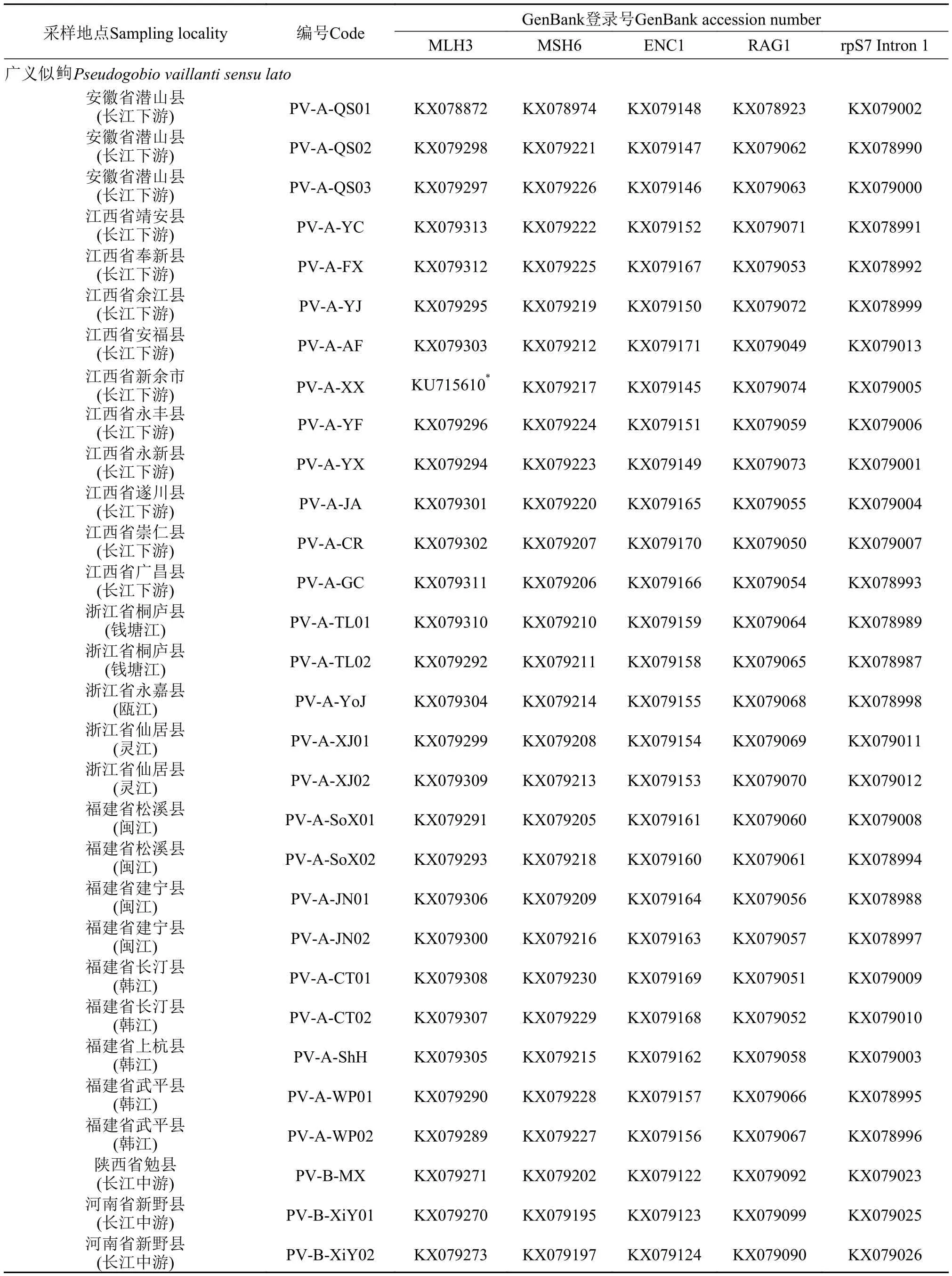
表 1 种名、采样地点、个体编号及GenBank登录号Tab. 1 Species, sampling sites, code and GenBank accession number of all individuals
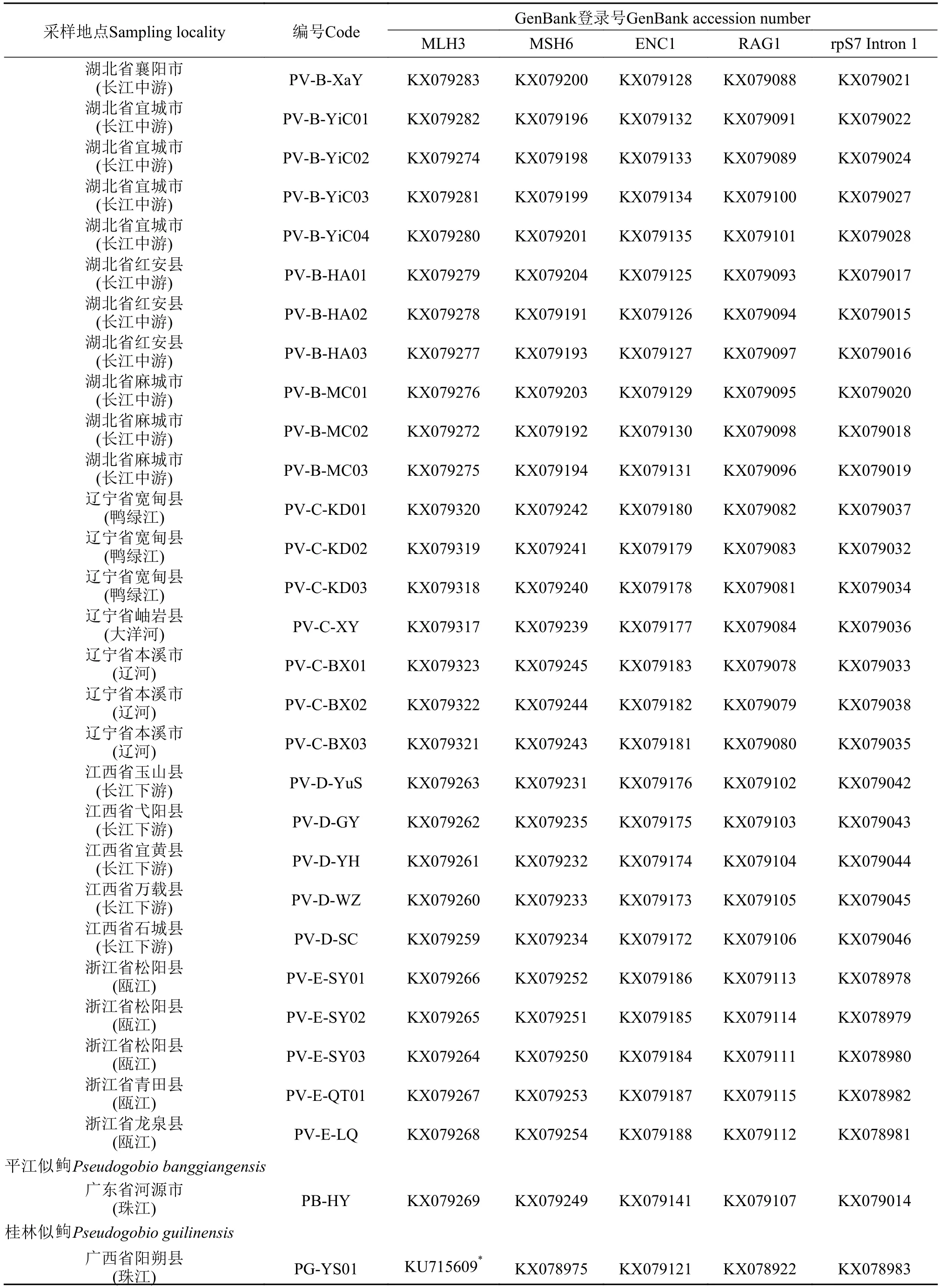
续表1
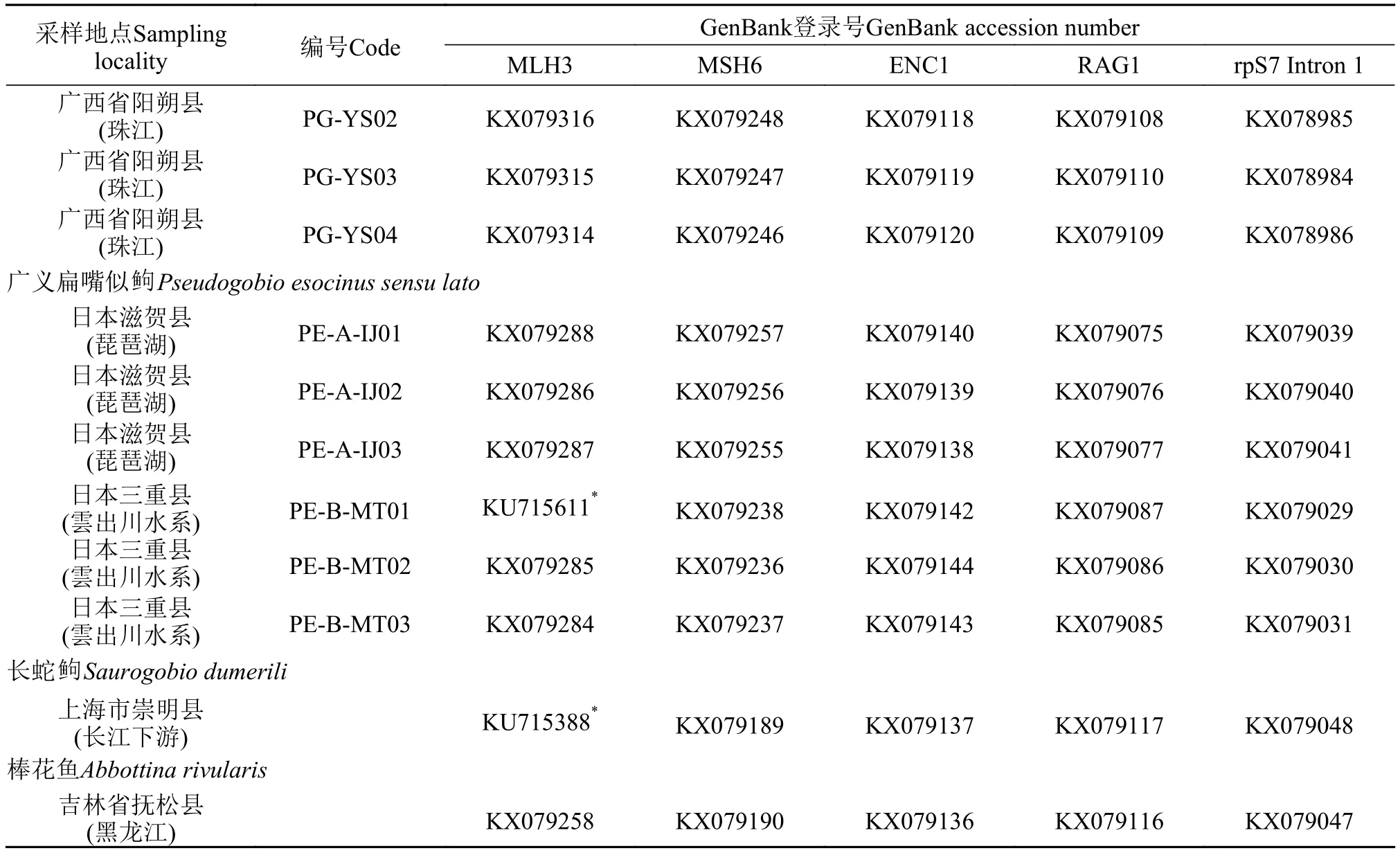
续表1
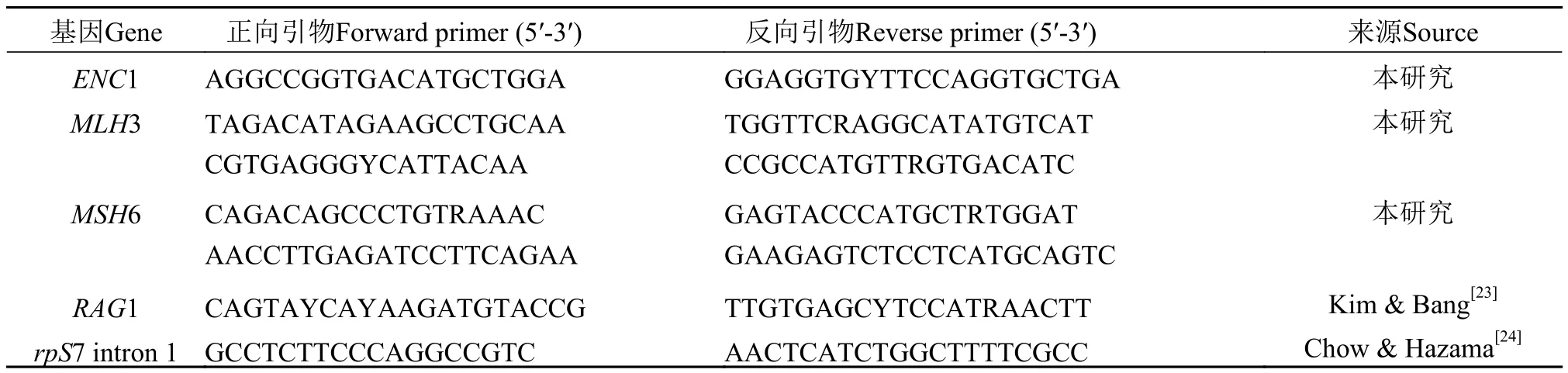
表 2 分子标记扩增使用的引物Tab. 2 Primers for the amplification of molecular markers
1.6 外部形态特征的计数与测量
统一采用鱼体背部俯视方向左侧进行计数与测量, 可数性状依据陈宜瑜[37], 可量性状依据Kottelat和Freyhof[38](图 1)。在Past ver. 1.90[39]软件下完成可量性状数据的主成分分析。为消除异速生长的影响, 可量性状数据进行对数转换并使用Burnaby方法进行校正[40,41]。
2 结果
2.1 序列特征
本研究使用的5个单拷贝核基因一起序列总长7582 bp, 变异位点数749, 简约信息位点数400。其中, ENC1序列长度为1290 bp, 变异位点数101, 简约信息位点数58; MLH3序列长度为2223 bp, 变异位点数230, 简约信息位点数115; MSH6序列长度为1992 bp, 变异位点数171, 简约信息位点数105; RAG1序列长度为1449 bp, 变异位点数88, 简约信息位点数47; rpS7 intron 1序列长度为628 bp, 变异位点数159, 简约信息位点数75。所有个体的GenBank登录号见表 1。
2.2 物种界定
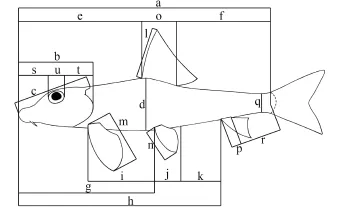
图 1 可量性状示意图(参考Kottelat & Jorg[38])Fig. 1 The diagram of morphometric characters (referring to Kottelat & Jorg[38])a. 体长; b. 头长; c. 背侧头长; d. 体高; e. 背鳍前长; f. 背鳍后长; g. 腹鳍前长; h. 臀鳍前长; i. 胸腹鳍起点间距离; j. 腹鳍肛门起点间距离; k. 肛门臀鳍起点间距离; l. 背鳍长; m. 胸鳍长; n. 腹鳍长; o. 背鳍基长; p. 臀鳍基长; q. 尾柄高; r. 尾柄长; s. 吻长; t.眼后头长; u. 眼径; v. 眼间距(图中未显示)a. Standard length (SL); b. Head length (HL); c. Dorsal head length; d. Body depth; e. Pre-dorsal length; f. Post-dorsal length; g. Pre-pelvic length; h. Pre-anal length; i. Distance between pectoraland pelvic-fin origins; j. Distance between pelvic-fin and anus origins; k. Distance between anus and anal-fin origins; l. Length of dorsal fin; m. Length of pectoral fin; n. Length of pelvic fin; o. Length of dorsal-fin base; p. Length of anal-fin base; q. Depth of caudal peduncle; r. Length of caudal peduncle; s. Snout length; t. Postorbital length; u. Eye diameter; v. Eye diameter (not shown in this figure)
基于核基因多位点联合序列重建的贝叶斯系统发育树(图 2)揭示: 广义似鲍是多系, 包括五个单系的谱系A至E; 桂林似鲍与平江似鲍都是单系; 广义扁嘴似鲍是多系, 包括两个单系的谱系A和B。POFAD距离分析(图 3)支持桂林似鲍、平江似鲍、广义似鲍谱系A至E与广义扁嘴似鲍谱系A与B的每个谱系是独立遗传种群。Structurama分析结果与POFAD距离分析结果一致。BP & P分析结果强烈支持桂林似鲍、平江似鲍、广义似鲍谱系A至E以及广义扁嘴似鲍谱系A与B的每个谱系是不同种(贝叶斯后验概率100%)。
2.3 系统发育关系
基于核基因多位点联合序列重建的贝叶斯系统发育树(图 2)与*BEAST物种树(图 4)树形基本一致。最近的鱼类分子系统发育关系研究[42—44]暗示*BEAST物种树比基于核基因多位点联合序列重建的贝叶斯系统发育树更能准确地反映系统发育关系。因此, 下面仅描述*BEAST物种树揭示的结果。
*BEAST物种树(图 4)揭示的似鲍属鱼类系统发育关系为: (1)扁嘴似鲍谱系B位于似鲍属鱼类的基部位置, 是所有其他似鲍属鱼类的姊妹群(贝叶斯后验概率100%); (2)似鲍谱系A与B是姊妹群关系, 似鲍谱系C是扁嘴似鲍谱系A的姊妹群; (3)似鲍谱系A+B与似鲍谱系C+扁嘴似鲍谱系A形成姊妹群关系(贝叶斯后验概率96%—100%), 它们一起是桂林似鲍的姊妹群(贝叶斯后验概率62%); (4)似鲍谱系D与E是姊妹群关系, 它们一起是平江似鲍的姊妹群(贝叶斯后验概率99%—100%); (5)平江似鲍+似鲍谱系D与E是桂林似鲍+扁嘴似鲍谱系A+似鲍谱系A至C的姊妹群(贝叶斯后验概率99%)。
2.4 形态特征
似鲍、桂林似鲍与平江似鲍的形态特征见表 3。可量性状的主成分分析结果(图 5)表明不能分开桂林似鲍、似鲍谱系A与B, 但能很好地分开似鲍谱系C、D与E; 桂林似鲍+似鲍谱系A与B能与似鲍谱系C至E分开。在可数性状中(表 3), 侧线鳞的数目能够很好地分开桂林似鲍(43)与似鲍谱系A至E+平江似鲍(40—42)。似鲍谱系C侧线鳞的数目(41—42)及背鳍前鳞的数目(12—13)和似鲍谱系A+B侧线鳞的数目(40—41)及背鳍前鳞的数目(10—12)也有一定差异。腹鳍分枝鳍条数与背鳍前鳞的数目能够区分平江似鲍(腹鳍分枝鳍条数8; 背鳍前鳞的数目15)和桂林似鲍+似鲍谱系A至E(腹鳍分枝鳍条数7; 背鳍前鳞的数目10—13)。似鲍谱系A与B比较、似鲍谱系D与E比较, 在可数性状上几乎没有差异。
3 讨论
3.1 我国似鲍属鱼类的分类厘定
杨秀平等[6]基于形态可量性状的主成分分析结果认为我国似鲍属鱼类仅似鲍一种。然而, 本研究基于分子的物种界定分析结果支持桂林似鲍与平江似鲍是有效种, 并认为广义似鲍谱系A至E以及广义扁嘴似鲍谱系A与B的每个谱系是不同种。由于扁嘴似鲍谱系A与B缺乏形态数据, 它们的分类有待进一步的研究再确定。下面结合形态证据就我国似鲍属鱼类的分类进行讨论。
尽管分子证据支持分布于韩江至长江的似鲍谱系A与分布于长江水系的似鲍谱系B为不同种,然而可数性状与可量性状分析结果不支持把似鲍谱系A与B划分为不同种。另外, 似鲍谱系A与B是姊妹群关系, 两者在长江水系均有分布。因此, 从形态证据、系统发育关系与地理分布三方面结合来看, 似鲍谱系A与B应属于同一物种的不同遗传谱系。基于似鲍谱系A+B分布范围包括似鲍模式地长江水系以及其形态性状符合Sauvage[45]对似鲍模式种的形态描述, 本研究限定似鲍谱系A+B为严格意义似鲍。
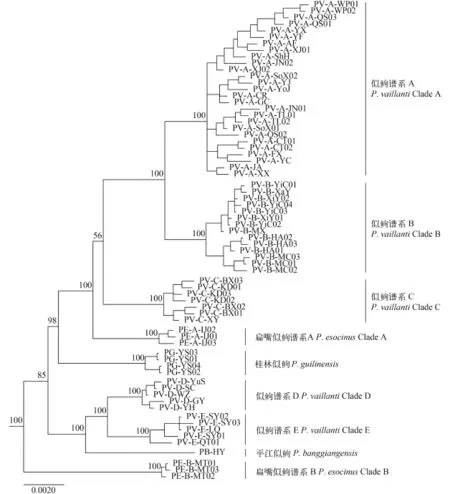
图 2 基于核基因多位点联合序列重建的似鲍属鱼类贝叶斯系统发育树Fig. 2 Bayesian tree of Pseudogobio fishes based on concatenated sequences of nuclear loci节点上方为贝叶斯后验概率, 外类群没有显示Above nodes are Bayesian posterior probabilities, and outgroup taxa are not shown
本研究结果表明分布于我国北方辽河、大洋河与鸭绿江水系的似鲍谱系C与分布于日本的扁嘴似鲍谱系A形成姊妹群关系, 它们一起是严格意义似鲍(= 似鲍谱系A+B)的姊妹群(图 4), 暗示似鲍谱系C是不同种。分子的物种界定分析结果支持似鲍谱系C作为种成立。可量性状分析结果能很好地区分似鲍谱系C与严格意义似鲍。在可数性状中, 似鲍谱系C侧线鳞的数目(41—42)及背鳍前鳞的数目(12—13)与严格意义似鲍侧线鳞的数目(40—41)及背鳍前鳞的数目(10—12)也有一定差异。依据罗云林等[5]对我国似鲍属鱼类的分类厘订, 似鲍谱系C为他们描述的长吻似鲍亚种。因此,结合分子证据、形态证据、系统发育关系与地理分布, 本研究恢复长吻似鲍(Pseudogobio longirostris Mori, 1934, 模式地辽河水系辽宁凌源)给予似鲍谱系C分类名。本研究系统发育关系结果亦表明严格意义似鲍(=似鲍谱系A+B)、长吻似鲍(=似鲍谱系C)和似鲍谱系D+E均不是姊妹群关系,暗示似鲍谱系D+E属于不同种。分子证据和形态证据进一步确认分布于长江的似鲍谱系D与分布于瓯江的似鲍谱系E各自是不同种, 并支持平江似鲍是有效种, 它们一起形成一单系群。
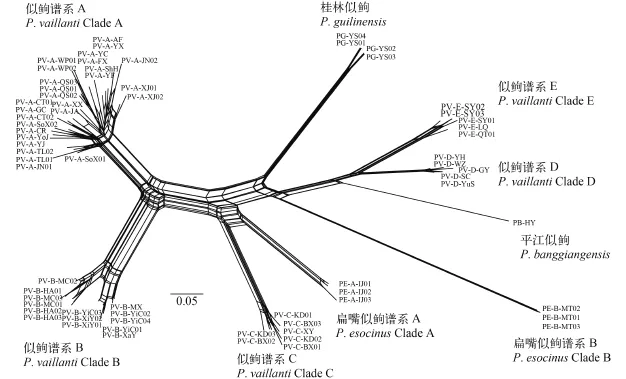
图 3 基于Genpofad距离重建的似鲍属鱼类邻接网络图Fig. 3 NeighborNet network of Pseudogobio fishes based on Genpofad distances似鲍谱系定义依据图 2Pseudogobio vaillanti Clades defined as in Fig. 2
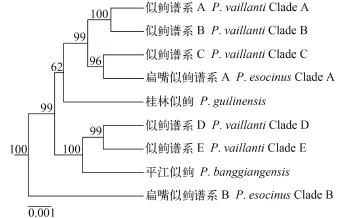
图 4 基于核基因多位点的似鲍属鱼类*BEAST物种树Fig. 4 BEAST species tree of Pseudogobio fishes based on nuclear loci节点上方为后验概率值, 外类群没有显示。似鲍谱系定义依据图 2Above nodes are posterior probabilities, and outgroup taxa are not shown. Pseudogobio vaillanti Clades defined as in Fig. 2
杨秀平等[6]基于形态可量性状的主成分分析结果认为桂林似鲍是似鲍的同物异名。本研究基于形态可量性状的主成分分析结果亦不能分开桂林似鲍和严格意义似鲍, 但它们一起能很好地与长吻似鲍、似鲍谱系D及E分开。然而, 系统发育关系结果表明桂林似鲍与严格意义似鲍+长吻似鲍+扁嘴似鲍谱系A形成姊妹群关系, 暗示桂林似鲍是有效种。分子的物种界定分析结果确认桂林似鲍是有效种。侧线鳞的数目亦能够很好地区分桂林似鲍(43)和我国分布的似鲍属鱼类其他种(40—42)。因此, 结合分子证据、形态证据与系统发育关系, 本研究支持桂林似鲍是有效种。
3.2 似鲍属鱼类的系统发育关系与生物地理过程推测
夏曦中等[13]采纳杨秀平等[6]基于形态可量性状的主成分分析结果认为我国似鲍属鱼类仅似鲍单种, 并利用线粒体细胞色素b基因开展了我国广义似鲍的谱系地理研究。随后Tominaga等[17,18]使用线粒体细胞色素b基因对分布于日本的扁嘴似鲍进行了谱系地理研究。他们的研究均发现似鲍属鱼类包括多个遗传谱系, 但都没有清晰解决似鲍属鱼类谱系间的系统发育关系。本研究利用核基因多位点数据首次清楚地解析了似鲍属鱼类的系统发育关系, 揭示平江似鲍是似鲍谱系D+E的姊妹群;桂林似鲍是严格意义似鲍(=似鲍谱系A+B)+长吻似鲍(=似鲍谱系C)+扁嘴似鲍谱系A的姊妹群; 所有谱系一起与扁嘴似鲍谱系B形成姊妹群关系。依据Tominaga等[18]的广义扁嘴似鲍谱系地理研究,扁嘴似鲍谱系A与B均为日本本岛特有。依据本研究对我国似鲍属鱼类的分类厘定, 平江似鲍与桂林似鲍分布于珠江水系, 严格意义似鲍分布于韩江至长江的水系, 长吻似鲍分布于辽河、大洋河与鸭绿江水系, 似鲍谱系D分布于长江, 似鲍谱系E分布于瓯江。结合上述系统发育关系与谱系的地理分布, 本研究提出以下生物地理过程是似鲍属鱼类物种分化的可能原因: (1)扁嘴似鲍谱系B位于系统发育树的基部位置, 暗示似鲍属鱼类可能起源于东亚北部地区, 扁嘴似鲍谱系B的物种形成可能是地理隔离造成; (2)桂林似鲍、严格意义似鲍、长吻似鲍与扁嘴似鲍谱系A一起构成一单系群, 四者地理分布不重叠, 推测四者的物种形成可能是地理隔离造成。同时, 扁嘴似鲍谱系A是长吻似鲍的姊妹群, 嵌套在似鲍属鱼类内部, 暗示扁嘴似鲍谱系A的物种形成亦可能是扩散过程造成, 即从大陆扩散到日本后的地理隔离; (3)平江似鲍、似鲍谱系D与E一起构成一单系群, 三者地理分布不重叠, 推测三者的物种形成可能是地理隔离造成; (4)平江似鲍与桂林似鲍均分布于珠江水系, 且两者不是姊妹群, 其系统位置均嵌套在似鲍属鱼类内部, 推测平江似鲍与桂林似鲍的物种形成亦可能是扩散过程造成, 即似鲍属鱼类祖先通过两次扩散进入珠江水系, 随后的地理隔离造成。
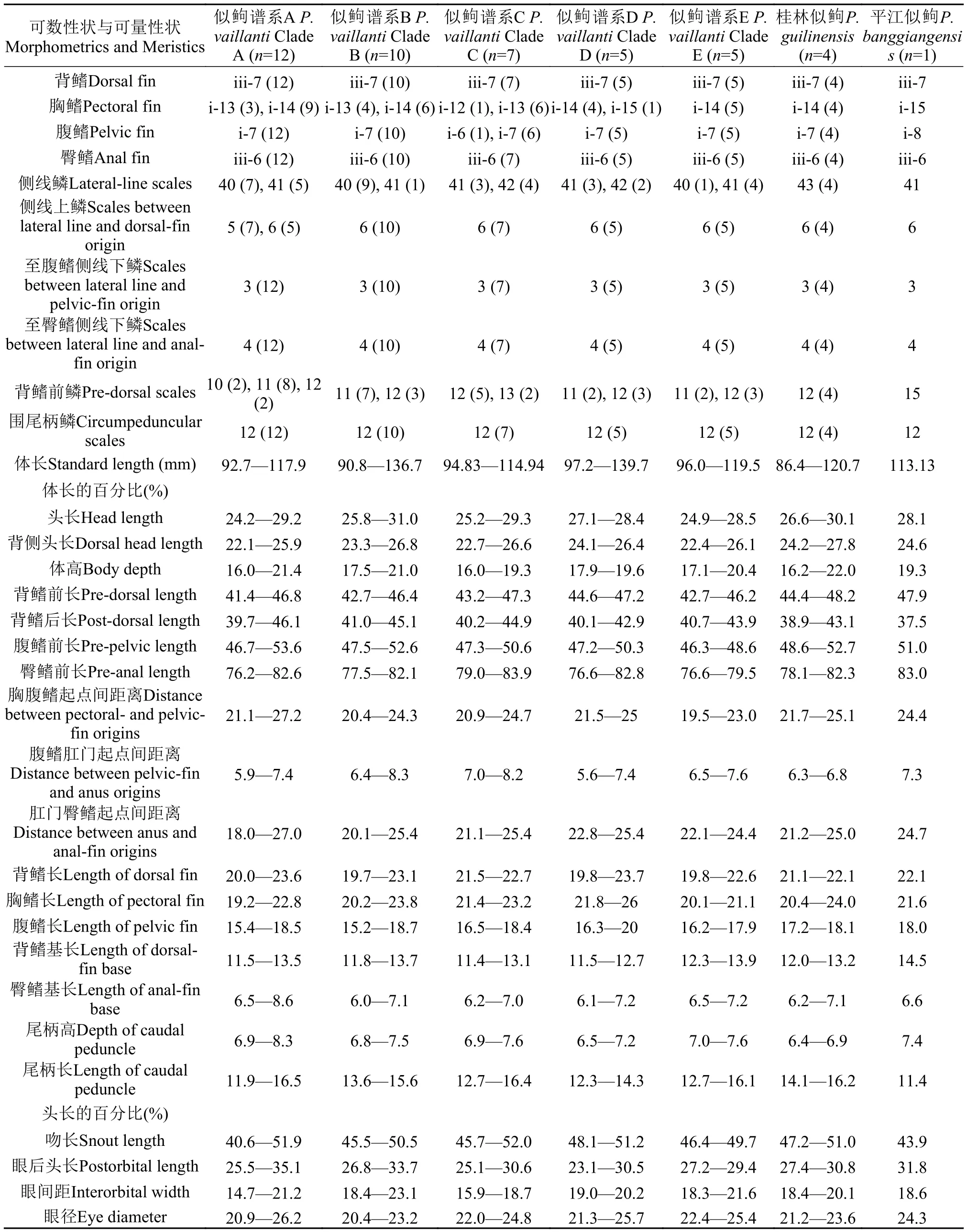
表 3 似鲍谱系A至E、桂林似鲍和平江似鲍的形态特征Tab. 3 Morphological characters of Pseudogobio vaillanti Clades A-E, Pseudogobio guilinensis and Pseudogobio banggiangensis
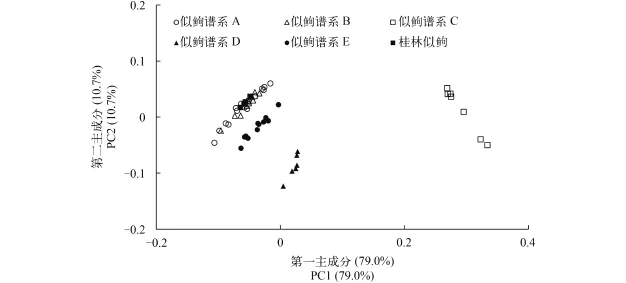
图 5 似鲍谱系A至E和桂林似鲍的第一与第二主成分得分散点图Fig. 5 Scatter plots of scores on the first and second principal components for Pseudogobio vaillanti Clades A-E and P. guilinensis
综上所述, 本研究清楚地对我国似鲍属鱼类进行了物种界定, 解析了似鲍属鱼类的系统发育关系。进一步的研究应基于动物命名法则给予似鲍谱系D与E相应的分类名称, 结合形态数据厘清广义扁嘴似鲍的分类。同时, 运用分子钟技术与祖先重建技术测试本研究提出的似鲍属鱼类物种分化的生物地理过程, 为理解东亚地区淡水鱼类多样性空间格局形成过程提供案例。
[1]Bǎnǎrescu P, Nalbant T T. Pisces, Teleostei, Cyprinidae (Gobioninae) [M]. Das Tierreich, Lieferung 93, Berlin: Walter de Guryter. 1973, 1—304
[2]Yue P Q. Pseudogobio Bleeker, 1860 [A]. In: Chen Y Y (Eds.), Fauna Sinica, Osteichthyes, Cypriniformes (II) [C]. Beijing: Science Press. 1998, 374—378 [乐佩琦. 似鲍属. 见: 陈宜瑜, 中国动物志硬骨鱼纲鲤形目(中卷).北京: 科学出版社. 1998, 374—378]
[3]Xie C X, Liu Q D. A Preliminary study on the biology of Pseudogobio vaillanti vaillanti (Sauvage) [J]. Journal of Huazhong Agricultural University, 1986, 5(1): 73—82 [谢从新, 刘齐德. 似鲍生物学的初步研究. 华中农业大学学报, 1986, 5(1): 73—82]
[4]Eschmeyer W N, Fricke R, Van der Laan R. Catalog of Fishes: Genera, Species, References, http://researcharchive.calacademy.org/research/ichthyology/catalog/fishcatmain.asp
[5]Luo Y L, Yue P Q, Chen Y Y. Pseudogobio Bleeker, 1860 [A]. In: Wu X W (Eds.), Fauna of Cyprinidae in China (II) [C]. Shanghai: Shanghai People’s Publishing House. 1977, 512—516 [罗云林, 乐佩琦, 陈宜瑜. 似鲍属. 见: 伍献文, 中国鲤科鱼类志(下卷). 上海: 上海人民出版社. 1977, 512—516]
[6]Yang X P, Zhang M Y, Liu H Z. Morphological variations and geographical differentiation of the genus Pseudogobio (Teleostei: Cyprinidae) in China [J]. Acta Hydrobiologica Sinica, 2002, 26(3): 281—285 [杨秀平,张敏莹, 刘焕章. 中国似鲍属鱼类的形态变异及地理分化研究. 水生生物学报, 2002, 26(3): 281—285]
[7]Hosoya K. Interrelationships of the Gobioninae (Cyprinidae) [A]. In: Uyeno T, Arai T, Taniuchi T, et al. (Eds.), Indo-Pacific Fish Biology: Proceeding of the Second International Conference on Indo-Pacific Fishes [C]. Tokyo: The Ichthyological Society of Japan. 1986, 484—501
[8]Yu L R, Yue P Q. Studies on phylogeny of the Pseudogobiini fishes (Cypriniformes: Cyprinidae) [J]. Acta Zootaxonomica Sinica, 1996, 21(2): 244—255 [俞利荣, 乐佩琦. 似鲍类鱼类的系统发育研究(鲤形目: 鲤科). 动物分类学报, 1996, 21(2): 244—255]
[9]Yang J Q, He S P, Freyhof J, et al. The phylogenetic relationships of the Gobioninae (Teleostei: Cyprinidae) inferred from mitochondrial cytochrome b gene sequences [J]. Hydrobiologia, 2006, 553(1): 255—266
[10]Liu H Z, Yang J Q, Tang Q Y. Estimated evolutionary tempo of East Asian gobionid fishes (Teleostei: Cyprinidae) from mitochondrial DNA sequence data [J]. Hydrobiology, 2010, 55(15): 1502—1510
[11]Tang K L, Agnew M K, Chen W J, et al. Phylogeny of the gudgeons (Teleostei: Cyprinidae: Gobioninae) [J]. Molecular Phylogenetics and Evolution, 2011, 61(1): 103—124
[12]Jiang Z G, Zhang E. Molecular evidence for taxonomic status of the gudgeon genus Huigobio Fang, 1938 (Teleostei: Cypriniformes), with a description of a new species from Guangdong Province, South China [J]. Zootaxa, 2013, 3731(1): 171—182
[13]Xia X Z, Zhang X P, Liu H Z. Molecular biological geography analysis of Pseudogobio vaillanti [J]. Progress in Natural Science, 2005, 15(9): 1064—1069 [夏曦中, 张训蒲, 刘焕章. 似鲍(Pseudogobio vaillanti)的分子生物地理学分析. 自然科学进展, 2005, 15(9): 1064—1069]
[14]Ward R D. DNA barcode divergence among species and genera of birds and fishes [J]. Molecular Ecology Resources, 2009, 9(4): 1077—1085
[15]Guo L, Li J, Wang Z S, et al. Phylogenetic relationships of noodle-fishes (Osmeriformes: Salangidae) based on four mitochondrial genes [J]. Acta Hydrobiologica Sinica, 2011, 35(3): 449—459 [郭立, 李隽, 王忠锁, et al. 基于四个线粒体基因片段的银鱼科鱼类系统发育. 水生生物学报, 2011, 35(3): 449—459]
[16]Chen H, Polgar G, Fu C Z, et al. Cryptic species and evolutionary history of Boleophthalmus pectinirostris complex along the northwestern Pacific coast [J]. Acta Hydrobiologica Sinica, 2014, 38(1): 75—86 [陈卉, Polgar G, 傅萃长, 等. 西北太平洋海岸带大弹涂鱼复合体的隐存种与进化历史. 水生生物学报, 2014, 38(1): 75—86]
[17]Tominaga K, Watanabe K, Kakioka R, et al. Two highly divergent mitochondrial DNA lineages within Pseudogobio esocinus populations in central Honshu, Japan [J]. Ichthyological Research, 2009, 56(2): 195—199
[18]Tominaga K, Nakajima J, Watanabe K. Cryptic divergence and phylogeography of the pike gudgeon Pseudogobio esocinus (Teleostei: Cyprinidae): a comprehensive case of freshwater phylogeography in Japan [J]. Ichthyological Research, 2016, 63(1): 79—93
[19]Hedin M. High-stakes species delimitation in eyeless cave spiders (Cicurina, Dictynidae, Araneae) from central Texas [J]. Molecular Ecology, 2015, 24(2): 346—361
[20]Karanovic T, Djurakic M, Eberhard S M. Cryptic species or inadequate taxonomy? implementation of 2D geometric morphometrics based on integumental organs as landmarks for delimitation and description of copepod taxa [J]. Systematic Biology, 2016, 65(2): 304—327
[21]Ojanguren-Affilastro A A, Mattoni C I, Ochoa J A, et al. Phylogeny, species delimitation and convergence in the South American bothriurid scorpion genus Brachistosternus Pocock 1893: Integrating morphology, nuclear and mitochondrial DNA [J]. Molecular Phylogenetics and Evolution, 2016, 94(2016): 159—170
[22]Miller S A, Dykes D D, Polesky H F. A simple salting out procedure for extracting DNA from human nucleated cells [J]. Nucleic Acids Research, 1988, 16(3): 12—15
[23]Kim K Y, Bang I C. Molecular phylogenetic position of Abbottina springeri (Cypriniformes, Cyprindae) based on nucleotide sequences of RAG1 gene [J]. Korean Journal of Ichthyology, 2010, 22(4): 273—278
[24]Chow S, Hazama K. Universal PCR primers for S7 ribosomal protein gene introns in fish [J]. Molecular Ecology, 1998, 7(9): 1255—1256
[25]Xia X H. DAMBE5: A comprehensive software package for data analysis in molecular biology and evolution [J]. Molecular Biology and Evolution, 2013, 30(7): 1720—1728
[26]Katoh K, Standley D M. MAFFT Multiple Sequence Alignment Software Version 7: Improvements in performance and usability [J]. Molecular Biology and Evolution, 2013, 30(4): 772—780
[27]Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0 [J]. Molecular Biology and Evolution, 2013, 30(12): 2725—2729
[28]Darriba D, Taboada G L, Doallo R, et al. jModelTest 2: more models, new heuristics and parallel computing [J]. Nature Methods, 2012, 9(9): 772
[29]Ronquist F, Teslenko M, Van der Mark P, et al. MrBayes 3.2: Efficient Bayesian phylogenetic inference and model choice across a large model space [J]. Systematic Biology, 2012, 61(3): 539—542
[30]Joly S, Bruneau A. Incorporating Allelic Variation for Reconstructing the Evolutionary History of Organisms from Multiple Genes: An Example from Rosa in North America [J]. Systematic Biology, 2006, 55(4): 623—636
[31]Huson D H, Bryant D. Application of phylogenetic networks in evolutionary studies [J]. Molecular Biology and Evolution, 2006, 23(2): 254—267
[32]Huelsenbeck J P, Andolfatto P. Inference of population structure under a Dirichlet process model [J]. Genetics, 2007, 175(4): 1787—1802
[33]Yang Z H, Rannala B. Bayesian species delimitation using multilocus sequence data [J]. Proceedings of the National Academy of Sciences, 2010, 107(20): 9264—9269
[34]Yang Z H, Rannala B. Unguided species delimitation using DNA sequence data from multiple loci [J]. MolecularBiology and Evolution, 2014, 31(12): 3125—3135
[35]Bouckaert R, Heled J, Kuhnert D, et al. BEAST 2: A software platform for bayesian evolutionary analysis [J]. PLoS Computational Biology, 2014, 10(4): 1—6
[36]Rambaut A, Drummond A. Tracer v1. 5. 2007. Available at: http://tree.bio.ed.ac.uk/software/tracer.
[37]Chen Y Y. Fauna Sinica, Osteichthyes, Cypriniformes (II) [M]. Beijing: Science Press. 1998, 16—18 [陈宜瑜. 中国动物志硬骨鱼纲鲤形目(中卷). 北京: 科学出版社. 1998, 16—18]
[38]Kottelat M, Freyhof J. Handbook of European freshwater fishes [M]. Delémont: Imprimerie du Démocrate SA. 2007, 15—16
[39]Hammer Ø, Harper D A T, Ryan P D. Past: Paleontological statistics software package for education and data analysis [J]. Palaeontologia Electronica, 2001, 4(1): 9
[40]Burnaby T P. Growth-invariant discriminant functions and generalized distances [J]. Biometrics, 1966, 22(1): 96—110
[41]Rolf F J, Bookstein F L. A comment on shearing as a method for ‘size correlation’ [J]. Systematic Zoology, 1987, 36(4): 356—367
[42]Waters J M, Rowe D L, Burridge C P. Gene trees versus species trees: reassessing life-history evolution in a freshwater fish radiation [J]. Systematic Biology, 2010, 59(5): 504—517
[43]Fu C Z, Guo L, Xia R. A multilocus phylogeny of Asian noodlefishes Salangidae (Teleostei: Osmeriformes) with a revised classification of the family [J]. Molecular Phylogenetics and Evolution, 2012, 62(3): 848—855
[44]Xia R, Durand J D, Fu C Z. Multilocus resolution of Mugilidae phylogeny (Teleostei: Mugiliformes): Implications for the family’s taxonomy [J]. Molecular Phylogenetics and Evolution, 2016, 96: 161—177
[45]Sauvage H E. Rhinogobio Vaillanti, n. sp [A]. In: Bulletin de la Societe philomathique de Paris [C]. Paris: Societe philomathique de Paris. 1878, 87
SPECIES DELIMITATION AND PHYLOGENETIC RELATIONSHIPS OF PSEUDOGOBIO FISHES IN THE SUBFAMILY GOBIONINAE
CAO Kai, LI Yu-Huo, HE Huan, LI Ming-Yue and FU Cui-Zhang
(Ministry of Education Key Laboratory for Biodiversity Science and Ecological Engineering, Institute of Biodiversity Science and School of Life Science, Fudan University, Shanghai 200438, China)
Species delimitation and phylogenetic relationships of the genus Pseudogobio fishes in the subfamily Gobioninae remain debated and unresolved. This study used molecular species delimitation methods and morphological analyses to reconstruct phylogenetic relationships of Pseudogobio fishes based on nuclear loci and the classification of this genus in China. The results of Bayesian tree showed that Pseudogobio guilinensis and P. banggiangensis were monophyletic specie, and that P. vaillanti and P. escocinus were polyphyletic species that the former included five Clades AE and the latter was comprised of two Clades A and B. The analyses of POFAD and Structurama showed that each clade of P. vaillanti and P. escocinus were independent genetic populations, and BP & P analysis strongly supported them as different species. The results of *Beast species tree revealed that P. esocinus Clade B occupied a basal position of Pseudogobio fishes; Clades A and B of P. vaillanti were the sister taxon relationship, and P. vaillanti Clade C was the sister taxon of P. esocinus Clade A, and all of them together were the sister group of P. guilinensis; Clades D and E of P. vaillanti were the sister taxon relationship, which were the sister group of P. banggiangensis. A revised classification of Pseudogobio fishes in China by molecular and morphological evidence as follows: P. vaillanti sensu stricto was limited to P. vaillanti Clades A and B; A resurrection of P. longirostris Mori, 1934 was assigned to P. vaillanti Clade C; Clades D and E of P. vaillanti were cryptic species, and P. guilinensis and P. banggiangensis were valid species, respectively.
Cyprinidae; Gobioninae; Pseudogobio; Species delimitation; Phylogeny
Q959.46
A
1000-3207(2017)03-0617-12
10.7541/2017.79
2016-04-27;
2016-08-21
国家自然科学基金(31471982)资助 [Supported by the National Natural Science Foundation of China (31471982)]
曹恺(1991—), 女, 浙江衢州人; 硕士研究生; 主要从事鱼类生态与进化研究。E-mail: 13210700119@fudan.edu.cn
傅萃长, 教授, 博士生导师; E-mail: czfu@fudan.edu.cn
