甘蓝型油菜PPR家族生物信息学分析与新疆野生油菜候选育性基因克隆
2017-05-11官春云
谭 晖,官春云
(湖南农业大学农学院/国家油料改良中心湖南分中心,长沙410128)
甘蓝型油菜PPR家族生物信息学分析与新疆野生油菜候选育性基因克隆
谭 晖,官春云*
(湖南农业大学农学院/国家油料改良中心湖南分中心,长沙410128)
通过隐马尔可夫模型从甘蓝型油菜基因组中获取1079条PPR家族蛋白序列,使用拟南芥PPR家族特征模型对其进行分类,同时进行聚类、染色体分布、亚细胞定位预测、功能注释等分析。结合不育系与恢复系分子标记筛选了定位于C09染色体的PPR基因GSBRNA2T00094406001(命名为BnPPR_C09)为新疆野生油菜潜在育性调节基因。通过分子克隆的方法从新疆野生油菜不育系1193A和恢复系1193R中分别克隆获得了长度为2514 bp的cDNA序列,序列分析显示来源于1193A的BnPPR_C09基因(BnPPR_C09b)相对源于1193R的BnPPR_C09a在+1725 bp位置存在单碱基缺失,造成移码突变,二者预测蛋白的生物信息学分析显示BnPPR_C09b蛋白的N端因为移码突变导致翻译过程在+1800位置终止,后续大量功能元件缺失,该位点的突变可能决定新疆野生油菜育性。
新疆野生油菜;生物信息学;育性;基因克隆
油菜是全球第二大油料作物,同时是我国种植面积最大,总产最高的油料作物,是我国最主要的食用油料来源,保障油菜产量对于我国粮油供给具有重要作用。细胞质雄性不育系(Cytoplasmic Male Sterility,CMS)是油菜杂种优势利用最有效、便捷的方式,对油菜的生产利用具有重要意义。油菜上较常见的胞质不育系有pol CMS,NCa CMS,Jun CMS,Hau CMS,Cam CMS,Nap CMS,Ogu CMS,Shanan2A CMS等。新疆野生油菜胞质不育系1193A(即本文中xy869与xy870)是以优良种质资源新疆野生油菜为母本,甘蓝型油菜湘油15号为父本属间杂交获得的异源胞质雄性不育株,后继续以湘油15号为轮回亲本回交多代创制而成的细胞质雄性不育系,胞质类型鉴定为新疆野油菜,与pol等有明显不同,是一种新型的胞质不育类型材料,在其姊妹系内得到恢复性稳定的恢复系1193R1[1](即本文中的xy1108、xy1109和xy1110)。
对育性恢复基因的克隆是细胞质雄性不育研究的重要内容,近年来越来越受到大家的关注。在已克隆的恢复基因中,除玉米Rf2、Rf4[2,3]和水稻Rf2、Rf17[4,5]外,其他恢复基因均属于PPR基因家族,具有PPR基因序列,这些证据对于CMS恢复基因的快速发掘与克隆具有重要作用。PPR(Pentatricopeptide repeats)是一类含有35个氨基酸重复序列的特殊蛋白家族,在植物中广泛分布,并在细胞质基因转录后加工、细胞质雄性不育相关基因的表达调控、胚胎形成和植物生长发育调控等方面发挥重要功能[6,7]。典型的PPR蛋白包含2~27个串联重复的含有35个氨基酸残基的PPR结构域或PPR相关结构域,占序列长度的三分之二左右;在其序列N端一般有长度可变、不保守的细胞器定位序列;部分序列C端含有3个可选择的非PPR结构域[8~10]。PPR基因编码蛋白具有RNA结合活性,几乎参与RNA代谢的整个过程,在细胞器RNA加工过程中发挥重要作用。
本研究基于已克隆恢复基因基本都编码PPR蛋白的特点,利用生物信息学对甘蓝型油菜PPR基因家族进行分析,结合新疆野生油菜不育系1193A已有的分子标记,筛选其候选恢复基因,并对其进行克隆和分析。
1 材料与方法
1.1 受试油菜
用于基因克隆与分析的新疆野生油菜品系(不育系xy869、xy870,恢复系xy1108、xy1109和xy1110),由国家油料改良中心湖南分中心保存并提供。
1.2 生物信息学工具
1.2.1 数据库
从BRAD数据库(http://brassicadb.org/brad/)获取甘蓝型油菜基因组数据,全部预测编码序列和全部预测蛋白数据;从Pfam[11]数据库(http://pfam.xfam.org/)获取PPR种子序列(PF01535);从TAIR数据库(http://www.arabidopsis.org/)获取拟南芥PPR蛋白家族PPR(P)、L、L2、E、E+、S和DYW 7种结构域序列。
1.2.2 本地化分析平台
基于Windows32位操作系统,搭建用于PPR基因家族的生物信息学分析平台。从NCBI网站(http://www.ncbi.nlm.nih.gov/)获取本地blast程序;从HMMER网站(http://hmmer.janelia.org/)获取HMMER3程序包;从Blast2GO网站(https://www.blast2go.com/)获取Blast2GO程序包;安装ANTHEPROT6.4、ClustalW、Fasttree等软件用于后续序列分析;安装并调试上述所有程序。
1.2.3 在线分析平台
在线工具ProtComp 9.0(http://linux1.softberry.com/)和PSORTⅡ(http://www.genscript.com/wolf-psort.html)用于亚细胞定位预测;EvolView在线工具(http://www.evolgenius.info/evolview)用于可视化树文件[12];在线工具Motifsearch(http://www.genome.jp/tools/motif/)进行蛋白功能域预测与筛选。
1.3 甘蓝型油菜PPR基因家族生物信息学分析与候选育性基因(BnPPR_C09)筛选
1.3.1 甘蓝型油菜PPR基因家族生物信息学分析
1.3.1.1 BnPPR蛋白家族筛选与鉴定
通过隐马尔可夫模型(profilehidden Markov models,profile HMMs)软件包HMMER3[13]综合分析甘蓝型油菜预测蛋白数据库,获得BnPPR蛋白家族候选序列。
通过hmmbuild程序构建PPR家族蛋白种子序列的隐马尔可夫模型矩阵(HMMER matrices);使用hmmbuild构建拟南芥7种PPR蛋白结构域序列定义的HMMER矩阵并通过hmmpress程序格式化;选择PPR家族蛋白种子序列定义的HMMER矩阵,使用hmmsearch程序检索甘蓝型油菜101 040条预测蛋白序列,定义阈值为1e-10,筛选候选BnPPR蛋白;选择拟南芥7种PPR结构域定义的HMMER矩阵,通过hmmscan程序检索候选BnPPR蛋白的PPR结构域分布,定义阈值为1e-10。PPR家族蛋白具有2~27个PPR结构域且不含其他类型结构域[5],筛选7种结构域数目在2~27之间的候选蛋白,获得BnPPR蛋白家族全部序列,对获得的BnPPR蛋白进行结构分析并分类。
1.3.1.2 多序列比对与进化树构建
通过ClustalW程序对BnPPR蛋白家族序列进行多序列比对,通过Fasttree程序[14]对比对结果分析并构建序列进化树,通过EvolView(http://www.evolgenius.info/evolview)可视化作图。
1.3.1.3 BnPPR基因的染色体定位分析
通过本地blastn程序对候选BnPPR基因序列在甘蓝型油菜基因组数据库进行比对,在甘蓝型油菜基因组中定位各候选BnPPR基因,分析甘蓝型油菜PPR基因在染色体上的分布。
1.3.1.4 PPR蛋白亚细胞定位预测
通过Softberry网站(http://linux1.softberry. com/)在线工具ProtComp 9.0[15]和WoLF PSORT(http://www.genscript.com/wolf-psort.html)在线工具[16]对甘蓝型油菜PPR蛋白进行亚细胞定位预测,统计分析两次亚细胞定位预测的结果。
1.3.1.5 PPR基因GO分析
使用Blast2GO软件[17]分析BnPPR基因家族全部成员,获得BnPPR基因家族的全部功能注释并进行统计分析。
1.3.2 候选育性基因(BnPPR_C09)筛选
尹明智[1]通过SNP芯片分析表明3个与恢复基因可能连锁的标记中有2个标记位于C09染色体上,该区域存在多个PPR蛋白基因。由于1193R之一(xy1109)系从1193A姊妹系中获得,通过杂交获得后代分离比接近3∶1,有理由推测1193A的育性为单基因控制,或者育性控制基因完全连锁,结合1193A育性基因的分子标记研究,有理由推测1193A育性控制基因可能为等位基因。提取1.3.1.3中获得的C09染色体上全部PPR基因序列与蛋白序列,并获取其blast2GO功能注释的分析结果。根据其功能注释筛选出野油胞质不育系1193A候选恢复基因。
1.4 新疆野生油菜候选育性基因BnPPR_C09 cDNA克隆
1.4.1 RNA提取与cDNA合成
取野油胞质不育系1193A材料xy869、xy870和恢复系材料xy1108、xy1109、xy1110生长过程中根、茎、叶、蕾1(2.5 mm以下小花蕾)、蕾2(2.5~3.5 mm花蕾)、蕾3(3.5 mm以上大花蕾)和花各组织放入液氮中速冻,-80℃冰箱保存。加液氮充分研磨后,使用TRIzol RNA提取试剂盒(TransGen生物技术有限公司)按照试剂盒所示方法提取总RNA,以此RNA为模板合成第1链cDNA。cDNA合成的具体方法参见Easy Script First-Strand cDNA Synthesis SuperMix试剂盒(TransGen生物技术有限公司)说明书。
1.4.2 BnPPR_C09基因的克隆
从BRAD数据库(http://brassicadb.org/brad/)获取GSBRNA2T00094406001基因(BnPPR_C09)序列。根据序列特征设计克隆引物,送擎科生物合成备用。设计的引物序列如下:
BnPPR_C09-F:5′-ATGAGTGATCAAAAAAT CAAGCGTTG-3′;
BnPPR_C09-R:5′-CTATAGCCAAACAACTC TACATGGAT-3′。
以1.3.1所述cDNA为模板进行PCR扩增。PCR体系含50 ng/μL模板1μL,10 mmol/L dNTPs 0.5μL,10μmol/L BnPPR_C09-F 0.5μL,10 μmol/L BnPPR_C09-R 0.5μL,5×反应缓冲液10 μL,TaKaRa高保真DNA聚合酶0.5μL和ddH2O 37μL。程序为预变性94℃5 min,94℃50 s,56℃45 s,72℃5 min,35个循环,72℃后延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳,用凝胶成像系统(Bio-Rad)检测和记录结果。将PCR产物回收后与pMD18-T载体(TaKaRa生物技术有限公司)连接,转化大肠杆菌感受态细胞Trans1-T1(Trans-Gen生物技术有限公司),筛选阳性克隆进行基因测序。通过BRAD数据库(http://brassicadb.org/brad/)对获得的克隆序列进行blast分析。
1.5 BnPPR_C09序列特征生物信息学分析
1.5.1 BnPPR_C09氨基酸序列特征与二级结构预测
通过ANTHEPROT 6.2软件分析BnPPR_C09蛋白序列,统计编码蛋白的氨基酸残基组分,分析编码蛋白的等电点、信号肽序列和亲疏水特征并使用Gariner模型进行二级结构预测。
1.5.2 BnPPR_C09蛋白结构域分析
使用在线工具Motifsearch(http://www.genome.jp/tools/motif/)分析BnPPR_C09蛋白序列中的特殊结构域。
1.6 甘蓝型油菜中BnPPR_C09基因时空表达分析
仔细分析1.4.2中克隆获得的BnPPR_C09序列并设计qRT-PCR引物,以actin作为内参基因,用于BnPPR_C09的时空表达分析,每样品重复3次。qRT-PCR在ABI 7500荧光定量PCR系统上运行。PCR体系包含:1μL cDNA、10μL 2×Fast-Start Universal SYBR GreenMaster with ROX、10 μmol/L正向引物和反向引物各0.5μL、8μL ddH2O。PCR程序为:95℃10 min;95℃15 s,60℃15 s,72℃32 s,35个循环。反应完成后进行95℃20 s,60℃20 s,95℃20 s,59℃20 s以绘制融解曲线,检测扩增产物的正确性和引物二聚体。
2 结果与分析
2.1 甘蓝型油菜PPR基因家族生物信息学分析
2.1.1 BnPPR蛋白家族筛选与鉴定
定义PPR家族种子序列(PF01535)的HMMER矩阵,应用hmmsearch程序检索甘蓝型油菜预测蛋白库,阈值定义为1e-10,共检索得1205条序列,排除等阈值序列后共1099条候选序列,从油菜预测蛋白序列库中提取出相应序列备用于下游分析。
以拟南芥数据库获得的PPR-P、L、L2、E、E+、S、DYW 7种结构域定义一个HMMER矩阵[18],使用hmmsearch重复检索1099条候选序列,排除其中仅含1个PPR结构域和超过27个PPR结构域的样本,共获得1079条蛋白序列。通过相同矩阵,并经由hmmscan程序检索1079条BnPPR蛋白序列,分析各序列中PPR结构域分布状况,并据此分为两个亚家族:P亚家族(PPR重复序列均为P结构)和PLS亚家族(PPR重复序列含有L,S结构域)。PLS亚家族又根据PPR-E、E+、DYW 3种结构域的分布分为PLS(不含E、E+、DYW结构域),E(含有E结构域),E+(含有E+结构域),DYW(含有DYW结构域)4个亚家族。分类统计结果如表1所示。

表1 蓝型油菜BnPPR蛋白家族分类Table 1 Classification of BnPPR protein family
由表1可见,BnPPR蛋白以PLS亚家族类型居多,共734条序列,占全部BnPPR蛋白的68%,表明BnPPR蛋白非P型结构域数目较多,分化程度较高。
甘蓝型油菜含有1079条BnPPR蛋白,是目前已知多种陆生植物中PPR家族成员最多的,接近大豆PPR蛋白数量(629)的两倍[19],这可能与甘蓝型油菜近缘亚基因组的演化有直接关联。甘蓝型油菜A、C亚基因组间有大量基因多拷贝存在,可能是BnPPR蛋白数量远多于其他陆生植物的原因。通过结构域扫描,从1079条BnPPR蛋白家族中共鉴定出9282个各类型的PPR结构域,平均每个Bn-PPR蛋白含有10.2个PPR特征结构域。
2.1.2 多序列比对与进化树构建
将1079条预测蛋白序列通过clustalW比对,使用Fasttree软件进行进化树分析,输出Newick tree格式文件,通过EvolView(http://www.evolgenius. info/evolview)进行可视化作图(图1)。通过分析Fasttree构建所得进化树文件中分枝值统计分析BnPPR各亚家族蛋白的进化关系。
BnPPR蛋白家族进化树相对较离散,保守性较差,P亚家族与PLS亚家族之间处于不同进化枝,两亚家族内均具有数目较多的离群序列,反应了BnPPR蛋白家族的高度多态性。其中GSBRNA2T 00151759001(P亚家族)、GSBRNA2T00080809001(P亚家族)和GSBRNA2T00155318001(PLS亚家族)均单独离散成一枝,但3条序列的PPR结构域均具有较完整的保守序列,显示其进化树中单独离散的原因在于其非PPR结构域。高度保守的PPR结构域为隐马尔可夫模型准确鉴定BnPPR蛋白提供了基础,聚类分析通过结合保守的PPR结构域和构成差异大的非PPR结构域能更好地将BnPPR蛋白家族进行聚类。

图1 甘蓝型油菜PPR蛋白家族进化树Fig.1 Evolutionary tree of BnPPR protein family
分析clustalW比对结果发现,BnPPR家族蛋白的保守序列均集中于PPR结构域,序列中非PPR结构域的序列构成差异很大,变异多,分化程度高,构成了BnPPR蛋白家族多态性的基础。Fasttree树文件显示,BnPPR蛋白家族的进化树节点丰富,各节点下分枝数目较多且差异较大,节点级数也较多,进化树分级次数多,充分反映了其序列构成的复杂性,这种结构复杂性可能是构成BnPPR基因家族功能多态性的分子基础。
2.1.3 BnPPR基因的染色体定位分析
通过鉴定获得的BnPPR蛋白序列号提取对应的BnPPR基因编码序列(Coding sequences,CDS),通过blastn程序检索本地甘蓝型油菜基因组数据库,将1079个BnPPR基因定位到A、C两个亚基因组19条染色体上,如表2所示,共4个BnPPR基因无明确定位信息,其中GSBRNA2T00040899001定位于A亚基因组(ChrAnn),GSBRNA2T00027616001,GSBRNA2T0086969001和GSBRNA2T00027567001定位于C亚基因组(ChrCnn)。
由表2可见,甘蓝型油菜A亚基因组含有556个BnPPR基因,C亚基因组含523个BnPPR基因,亚基因组间BnPPR基因总数大致相当,但C亚基因组中BnPPR基因密度明显低于A亚基因组,密度最高的是A06号染色体,平均每百万碱基含3.7个BnPPR基因,密度最低的是C02号染色体,每百万碱基含0.7个BnPPR基因。
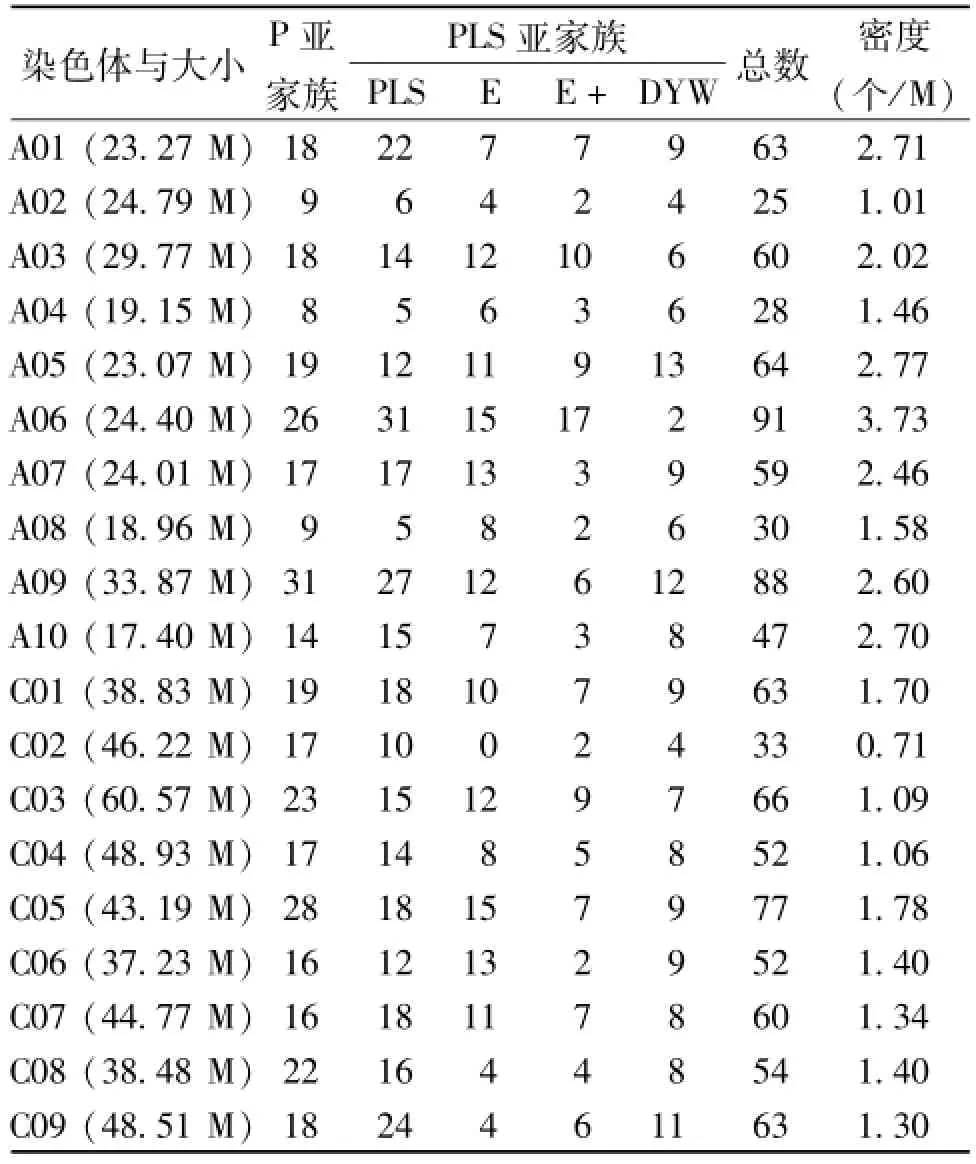
表2 甘蓝型油菜BnPPR基因的染色体分布Table 2 Chromosome destribution of BnPPR genes
2.1.4 BnPPR蛋白亚细胞定位预测
通过Softberry网站(http://linux1.softberry. com/)在线工具ProtComp 9.0[13]和WoLF PSORT(http://www.genscript.com/wolf-psort.html)在线工具[16]对甘蓝型油菜PPR蛋白进行亚细胞定位预测,统计分析两次亚细胞定位预测的结果。
通过两种不同迭代算法的在线工具进行了Bn-PPR各亚家族蛋白的亚细胞定位预测,结果如表3和表4。

表3 甘蓝型油菜PPR蛋白亚细胞定位预测(ProtComp9.0)Table 3 Subcellular localization of BnPPR proteins(Prot Comp9.0)__________
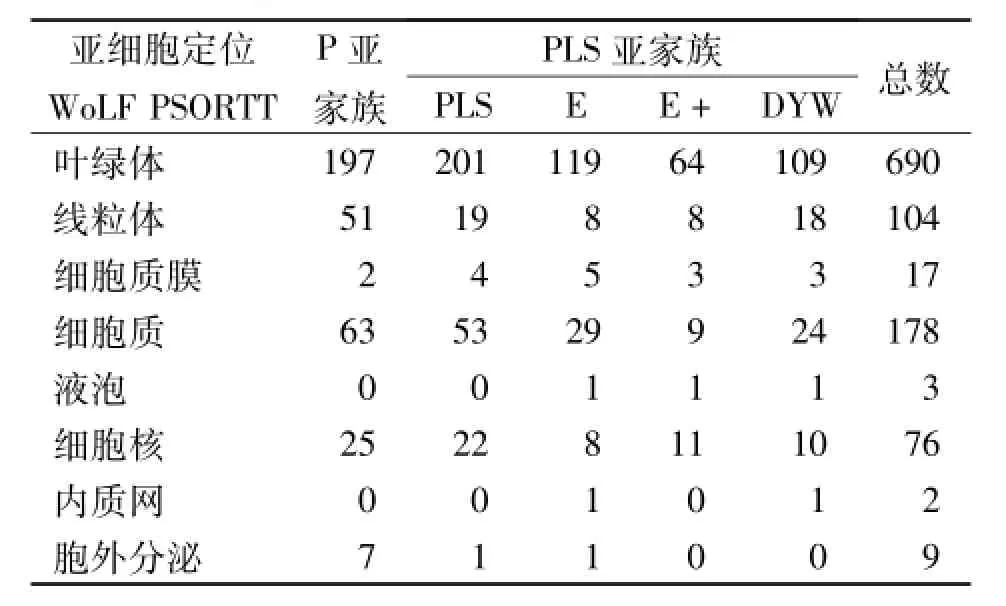
表4 甘蓝型油菜BnPPR蛋白亚细胞定位预测(W oLF PSORTT)Table 4 Subcellular localization of BnPPR proteins(W oLF PSORTT)____________
由表可见,两种算法的预测结果总体上大致相当,定位于线粒体和叶绿体的BnPPR蛋白总数基本一致(800条左右)。经仔细分析两种预测结果发现,定位于线粒体与叶绿体的全部序列是基本相同的,但分配比例有较大差异,基于ProComp9.0内核的预测结果中线粒体定位序列较多,基于PSORT内核的预测结果以叶绿体定位序列较多。各亚家族之间的亚细胞定位预测结果基本相似(定位于各亚细胞结构中BnPPR蛋白的比例基本相当),均以线粒体、叶绿体定位为主。
总体来看,两种不同算法的亚细胞定位预测结果都显示,超过75%的BnPPR蛋白定位于半自主细胞器,其次超过15%的序列定位于细胞质,极少数序列定位于细胞核或内质网等其它区域。以上结果充分显示BnPPR蛋白尽管具有极高频的分化,结构上具有较高的复杂性,但仍具有典型的定位于半自主细胞器的特点,与其他植物中PPR蛋白亚细胞定位一致。
2.1.5 PPR基因GO分析
通过blast2GO程序对BnPPR基因家族1079条CDS序列按亚家族分类进行功能注释,并对功能注释结果进行作图分析。
使用NCBI库为序列检索库,使用blastx分别对甘蓝型油菜PPR-P、PLS、E、E+和DYW 5个亚家族CDS序列进行“blasting”,使用nr数据库,设定阈值为1e-5,完成blast后通过“Gene Ontology”进行“mapping”,随后进行“annotation”,设定阈值1e-6,其它使用缺省值,最后对分子功能、生物过程与细胞组分三种不同GO注释结果进行统计分析(表5)。
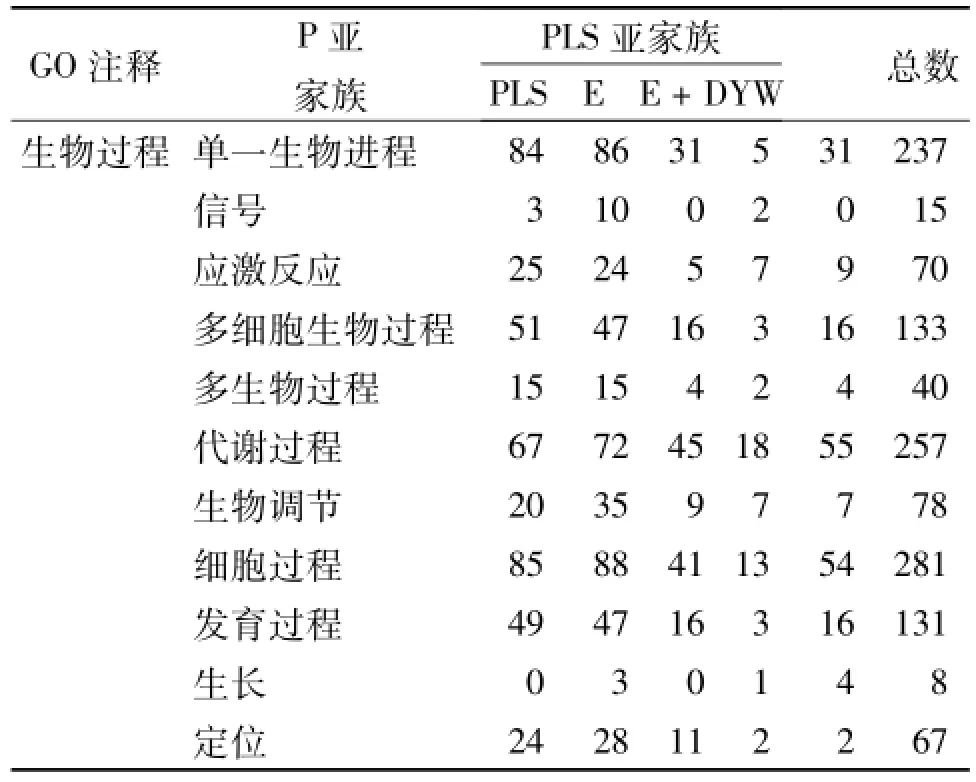
表5 BnPPR基因家族GO注释Table 5 GO annotation of BnPPR gene family

(续表5)
由表5可见,BnPPR基因家族功能注释显示BnPPR基因家族具有复杂的生物学功能,涉及到细胞组织调控、生长发育、繁殖、植物信号、植物免疫和参与细胞结构组成等多个方面。BnPPR基因家族的生物过程GO注释多态性最高,涉及单细胞、多细胞、单细胞器、多细胞器、免疫和细胞结构的组织等不同方面的多个层次,各亚家族之间没有明显差异;细胞组分GO注释相对而言比较粗放,仅注释至细胞或细胞器层面,没有进一步的功能注释,仅PLSE+亚家族GO注释为线粒体与叶绿体组分,这与其亚细胞定位预测吻合。由于GO注释是基于序列与数据库的同源比对产生,这提示PLS-E+亚家族的细胞器定位序列可能位于PPR-E+结构域区段或者与之共同出现的非PPR结构域序列;分子功能GO注释主要为催化活性与结合活性两方面,在不同的亚家族之间没有明显差异。
2.2 候选育性基因BnPPR_C09 cDNA克隆
根据甘蓝型油菜PPR基因的染色体定位与功能注释,筛选GSBRNA2T00094406001为候选育性基因,根据其序列特征设计克隆引物。分别从不育系xy869和恢复系xy1109中扩增出长度为2514 bp的cDNA片段,分别命名为BnPPR_C09b和BnPPR_C09a(图2)。

图2 BnPPR_C09 cDNA扩增结果Fig.2 cDNA am ip lification of BnPPR_C09 gene
将克隆获得的BnPPR_C09b和BnPPR_C09a连接TA克隆载体,送擎科生物测序,发现BnPPR_C09b在1695位出现3碱基替换,1725位出现单个T碱基缺失,造成后续序列移码突变。
2.3 BnPPR_C09蛋白序列分析
BnPPR_C09b和BnPPR_C09a基因分别编码长度599个和837个氨基酸残基的蛋白,分别命名为BnPPR_C09b和BnPPR_C09a。分析这2个预测的BnPPR_C09蛋白的氨基酸组成、摩尔质量和等电点于表6。
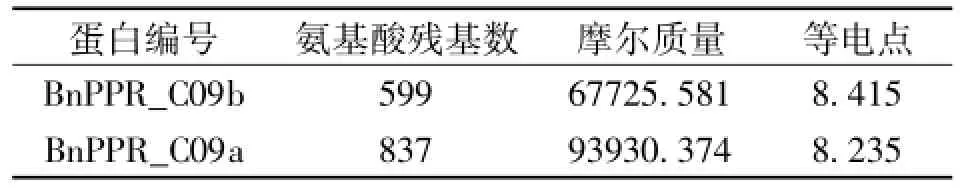
表6 BnPPR_C09蛋白信息汇总表Table 1 Summary of the deduced BnPPR_C09 proteins
分析显示BnPPR_C09b由于1725位的缺失突变导致编码蛋白提前终止,编码产生的序列长度比BnPPR_C09a少238个氨基酸残基。
通过ANTHEPROT 6.2软件预测了BnPPR_C09a\b蛋白的信号肽剪切位置(图3)和二级结构。为方便比较蛋白结构差异,将BnPPR_C09b基因1725位单碱基缺失突变后,在后续序列中间产生的终止密码移除,由此翻译一条预测蛋白命名为Bn-PPR_C09b1(图4)。

图3 BnPPR_C09蛋白信号肽预测Fig.3 Signal peptide prediction of BnPPR_C09 protein
由图3可见,在BnPPR_C09a约700氨基酸位置存在一个可能的信号肽剪切位点,而BnPPR_ C09b由于突变导致后续序列不能编码,丢失该信号肽剪切位点。
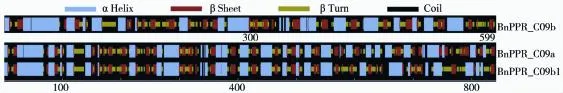
图4 BnPPR_C09蛋白二级结构Fig.4 The secondary structure of BnPPR_C09 protein
由图4可见,BnPPR_C09b1与BnPPR_C09a二级结构差异在约550氨基酸之后大量出现。二级结构差异可能会大幅影响蛋白的正确折叠,可以大胆推测BnPPR_C09a碱基移码突变之后所翻译的蛋白可能会因为结构变异而失去功能。
使用在线工具Motifsearch(http://www.genome.jp/tools/motif/)对BnPPR_C09a\b蛋白存在的保守功能结构域进行预测,结果如图5。
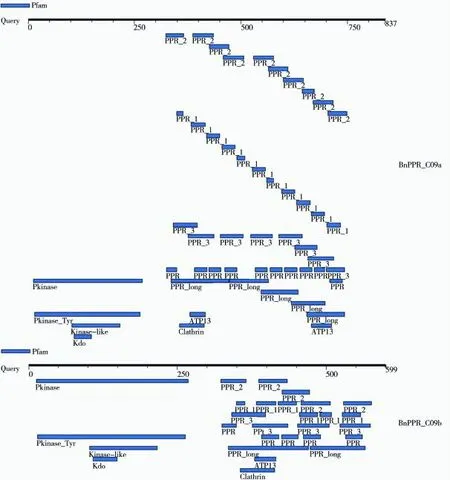
图5 BnPPR_C09功能结构域分析Fig.5 Functional domain analysis of BnPPR_C09
由图5可见,BnPPR_C09b相较BnPPR_C09a所缺失的238个氨基酸残基中包含了一个ATP13结构和多个不同类型的PPR结构元件。ATP13是线粒体ATP酶的组成亚基,其在线粒体结构形成和功能发挥中均具有重要作用[20]。由此推测BnPPR_C09b蛋白翻译提前终止造成N端序列缺失引起功能异常,造成1193A花粉败育。
2.4 甘蓝型油菜中BnPPR_C09基因时空表达分析
采用qRT-PCR法检测BnPPR_C09基因(根据5′端设计引物,BnPPR_C09a/b通用引物对)在新疆野生油菜不育系(xy869和xy870)、恢复系(xy1108、xy1109和xy1110)的根、茎、叶、蕾1、2、3和花中相对表达,结果如图6。
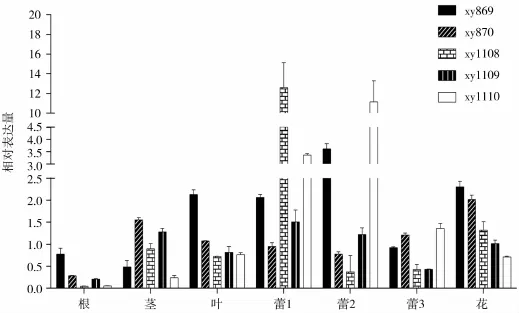
图6 BnPPR_C09表达分析Fig.6 Expression analysis of BnPPR_C09
总体来看,BnPPR_C09在不育系和恢复系各组织中均有表达,在营养器官根、茎、叶中的表达低于在生殖器官花和花蕾中的表达,其中在根中的表达量最低。不育系中BnPPR_C09在营养生长期有高于恢复系的表达,而在幼嫩花蕾中表达量则低于恢复系,最后在花中表达量高于恢复系。不育系中BnPPR_C09表达相对较稳定,而恢复系中则表现为随着生殖生长过程的推进表达量先上升后下降,在花蕾中表现为明显的高表达。
3 结论与讨论
新疆野生型油菜具有与传统pol不育系不同的不育模式,其在姊妹系中能找到稳定恢复源,表明其育性控制可能是单位点的等位基因控制。本研究发现候选育性基因BnPPR_C09在1193A与1193R中存在序列差异,不育系1193A中的BnPPR_C09在+1725位出现碱基缺失,导致蛋白翻译提前终止,可能该位点突变即1193A育性变化的原因,但尚需要进一步对BnPPR_C09在新疆野生油菜中的功能进行验证。BnPPR_C09在不育系与恢复系中表现不同的表达模式,在恢复系中呈现诱导表达,随生育期进程表现为先上升后下降的模式,不育系中则表达较稳定且在营养器官中相对高表达,这也显示了正常的BnPPR_C09蛋白可能在生殖生长期间产生功能,因此在花蕾中出现诱导高表达的特性。
生物信息学作为分子育种的辅助手段具有高通量的优势,是一种重要的辅助育种手段,值得研究和推广。本研究通过分子克隆获得了两条候选育性基因序列,是对新疆野生油菜育性研究的重要补充,但针对该基因在新疆野生油菜中的功能尚缺乏研究,需要进一步在不育系中导入正确拷贝,在恢复基因中突变或敲出正确拷贝,来进一步验证其功能。
[1] 尹明智.野油胞质雄性不育系1193A的研究[D].长沙:湖南农业大学博士学位论文,2014.
[2] Liu F,Cui XC,Horner HT,et al.Mitochondrial aldehyde dehydrogenase activity is required for male fertility in maize[J].Plant Cell,2001,13:1063-1078.
[3] Cui X,Wise RP,Schnable PS.The rf2 nuclear restorer gene ofmale-sterile T-cytoplasm maize[J].Science,1996,272:1334-1336.
[4] Fujii S,Toriyama K.Suppressed expression of RETROGRADE-REGULATED MALE STERILITY restores pollen fertility in cytoplasmic male sterile rice plants[J]. Proceedings of the National Academy of Sciences,2009,106(23):9513-9518.
[5] Itabashi E,Iwata N,Fujii S,et al.The fertility restorer gene,Rf2,for Lead Rice-type cytoplasmic male sterility of rice encodes a mitochondrial glycine-rich protein[J].The Plant Journal,2011,65(3):359-367.
[6] 何 鹏,陈海燕,俞嘉宁.PPR蛋白参与RNA编辑机制的研究进展[J].西北植物学报,2013,33(2):415-421.
[7] Wang Z,Zou Y,Li X,et al.Cytoplasmicmale sterility of rice with boro II cytoplasm is caused by a cytotoxic peptide and is restored by two related PPR motif genes via distinctmodes ofmRNA silencing[J].Plant Cell,2006,18(3):676-687.
[8] Lurin C,Andrés C,Aubourg S,et al.Genome-wide analysis of Arabidopsis pentatricopeptide repeat proteins reveals their essential role in organelle biogenesis[J].The Plant Cell,2004,16(8):2089-2103.
[9] Saha D,Prasad AM,Srinivasan R.Pentatricopeptide repeat proteins and their emerging roles in plants[J].Plant Physiology and Biochemistry,2007,45(8):521-534.
[10]Schmitz-Linneweber C,Small I.Pentatricopeptide repeat proteins:a socket set for organelle gene expression[J]. Trends in Plant Science,2008,13(12):663-670.
[11]Punta M,Coggill PC,Eberhardt RY,et al.The Pfam protein fam ilies database[J].Nucleic Acids Res,2012,40(1):290-301.
[12]Zhang H,Gao S,Lercher MJ,et al.EvolView,an online tool for visualizing,annotating and managing phylogenetic trees[J].Nucleic Acids Research,2012,40(1):569-572.
[13]Finn RD,Clements J,Eddy SR.HMMER web server:interactive sequence similarity searching[J].Nucleic Acids Res,2011,39(Web Server issue):29-37.
[14]Price MN,Dehal PS,Arkin AP.FastTree:computing large minimum evolution trees with profiles instead of a distancematrix[J].Molecular Biology and Evolution,2009,26(7):1641-1650.
[15]Hategan A,Tabus I.Protein is compressible[C].Proceedings of the 6th Nordic Signal Processing Symposium,NORSIG,2004.192-195.
[16]Horton P,Park KJ,Obayashi T,et al.WoLF PSORT:protein localization predictor[J].Nucleic Acids Research,2007,35(S2):W585-W587.
[17]Conesa A,Götz S,García-Gómez JM,etal.Blast2GO:a universal tool for annotation,visualization and analysis in functional genomics research[J].Bioinformatics,2005,21(18):3674-3676.
[18]丁安明,李 凌,屈 旭,等.番茄PPR基因家族的鉴定与生物信息学分析[J].遗传,2013,36(1):77-84.
[19]Fujii S,Small I.The evolution of RNA editing and pentatricopeptide repeat genes[J].New Phytologist,2011,191(1):37-47.
[20]Ackerman SH,Gatti DL,Gellefors P,et al.ATP13,a nuclear gene of Saccharomyces cerevisiae essential for the expression of subunit9 of themitochondrial ATPase[J]. FEBSLetters,1991,278(2):234-238.
Bioinformatics Analysis of PPR family in Brassica napus L.and Cloning of Candidate Restorer Gene of Xinjiang W ild Rapeseed
TAN Hui,GUAN Chunyun*
(College of Agronomy,Hunan Agricultural University/National Oil Crop Improvement Center in Hunan,Changsha,Hunan 410128,China)
In this study,1079 protein sequences of PPR family were obtained from the genome of Brassica napus by Hidden Markov model.Themotifs defined in the PPR family of Arabidopsis thaliana was used to classify them.The clustering,chromosome distribution,subcellular localization prediction,function Comments and so on were analyzed.Combining themolecularmarkers of the CMS line and the restorer line,the PPR gene GSBRNA2T00094406001(named BnPPR_C09)located on C09 chromosome was screened for the potential fertility regulation gene of Xinjiang wild rapeseed.A cDNA sequence with length of 2514 bp was cloned from the CMS line 1193A of Xinjiang wild rapeseed and the restorer line 1193R bymolecular cloning.Sequence analysis showed that the BnPPR_C09 gene(BnPPR_C09b)derived from 1193A had a single base deletion at+1725 bp compared with the BnPPR_C09a derived from 1193R,resulting in frameshiftmutations.The bioinformatics analysis about predicted protein showed that the frameshiftmutations caused term ination of the translation process at+1800 in the N-terminal of the BnPPR_C09b protein,and the subsequent deletion of a large number of functional components,themutationmay determine the fertility of Xinjiang wild rapeseed.
Xinjiang wild rapeseed;bioinformatics;fertility;gene clone
Q78
A
1001-5280(2017)03-0246-10 DO I:10.16848/j.cnki.issn.1001-5280.2017.03.08
2017- 03- 09
谭 晖(1992-),女,硕士研究生,Email:1132281904@qq.com。*通信作者,官春云,教授,中国工程院院士。
国家重点研发计划(2016YFD010301);湖南省省长专项(湘财农指2016114号);湖南省科技计划(2013FJ4025)。
