日本沼虾(Macrobrachiumnipponense)4个野生群体遗传多样性微卫星分析
2017-04-26武小斌穆淑梅赵玲玉康现江薛建民
武小斌,穆淑梅,赵玲玉,康现江,薛建民
(1.河北大学 生命科学学院,河北 保定 071002;2.衡水市畜牧水产局 水产技术推广站,河北 衡水 053000)
日本沼虾(Macrobrachiumnipponense)4个野生群体遗传多样性微卫星分析
武小斌1,穆淑梅1,赵玲玉1,康现江1,薛建民2
(1.河北大学 生命科学学院,河北 保定 071002;2.衡水市畜牧水产局 水产技术推广站,河北 衡水 053000)
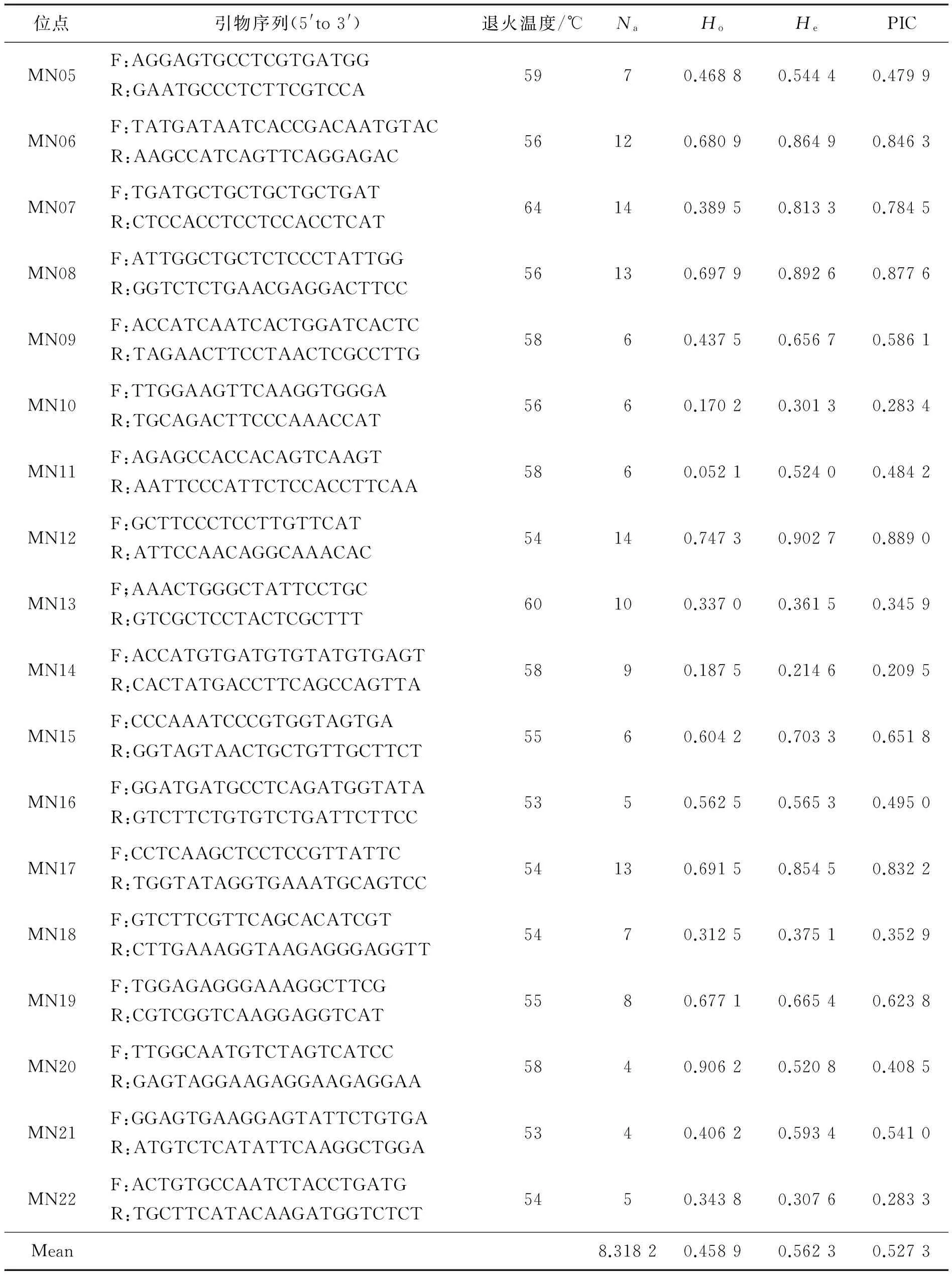
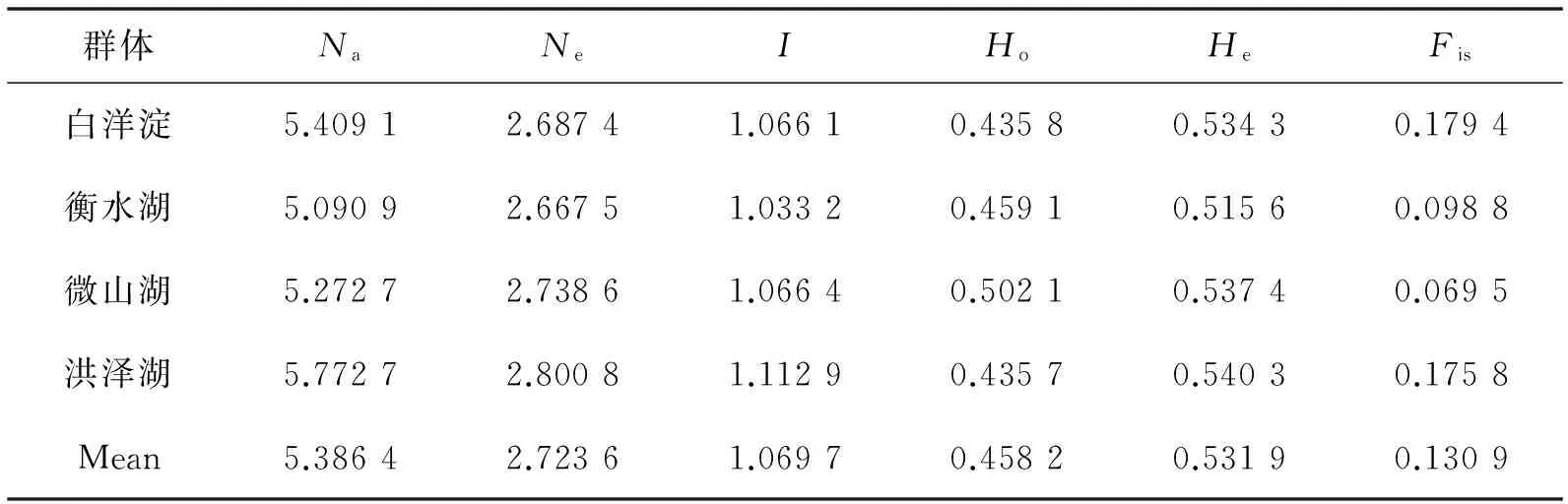
为了评价不同地区日本沼虾(Macrobrachiumnipponense)野生群体的遗传多样性和遗传分化水平,利用微卫星标记对白洋淀、衡水湖、微山湖和洪泽湖4个地区日本沼虾野生群体的遗传多样性进行分析.结果显示,22对微卫星位点都显示具足够的多态性;每个野生群体中至少有6个位点显示杂合不足,显著偏离了Hardy-Weinberg平衡;4个日本沼虾野生群体均表现出了较高的遗传多样性,其中微山湖野生群体遗传多样性水平相对较高,其他3个野生群体的遗传多样性水平则相对较低;群体间F-统计量(Fst)及分子方差分析表明,4个日本沼虾野生群体间存在中等程度的遗传差异(0.046 3 日本沼虾;微卫星;遗传多样性 日本沼虾(Macrobrachiumnipponense),俗称青虾或河虾,广泛分布于湖泊、池塘和江河中,是一种经济价值较大的淡水虾类,白洋淀、衡水湖、微山湖、洪泽湖都有日本沼虾野生资源的分布[1].因其肉质细嫩,美味可口,富含维生素、氨基酸以及Mn、Zn等多种人体必需微量元素,并有一定药用价值,深受人们喜爱并得到广泛养殖.据《中国渔业年鉴》公布,近几年全国养殖日本沼虾年产量约2×105t,年产值近100亿元,其养殖已成为我国农业增效、农民增收的重要途径之一.近年来,随着迅速推进的工业化进程,日本沼虾野生群体的栖息地环境受到人为干扰、破坏和无序开发,使得其群体结构遭到严重破坏,种质纯度降低,资源量锐减,日本沼虾的种质资源保护亟需得到重视.本文拟通过对白洋淀、衡水湖、微山湖、洪泽湖日本沼虾野生群体种质资源现状的分析研究,为日本沼虾优良种质资源保护及遗传选育研究提供数据支持. 研究物种群体的遗传结构与变化规律即群体遗传学[2],它是利用分子实验技术结合生物统计学方法研究特定群体中基因(型)频率和变化情况,从而了解物种群体内(间)的基因交流情况.分子标记作为群体遗传学研究重要工具得到越来越广泛的应用[3-5],其中微卫星标记(simple sequence repeat,SSR)具有重复性好、多态性信息丰富、数量多等优点,已经广泛应用于水产动物的遗传多样性分析、数量性状位点(quantitative trait locus,QTL)定位和功能基因发掘等研究[6-8]. 1.1 日本沼虾的获得与形态学数据测定 选取4个采样点(河北白洋淀、河北衡水湖、山东微山湖、江苏洪泽湖)获得日本沼虾野生群体样本,采样点见表1.鲜活样品采集后,解剖取肌肉保存于体积分数为95%的酒精中,-20 ℃冰箱中保存. 表1 4个日本沼虾野生群体采样数据概况 1.2 基因组DNA提取 解剖日本沼虾取肌肉组织50mg,采用海洋动物DNA提取试剂盒(康维世纪,CW2089S)提取基因组DNA.DNA质量浓度与质量通过NanoDrop2000C超微量分光光度计进行测定,根据测定结果将所有样品的DNA质量浓度调整为50ng/μL, 10g/L琼脂糖凝胶电泳检测所提取DNA的纯度及降解程度,合格DNA样品-20 ℃保存备用. 1.3 微卫星PCR反应 本研究所采用的22对微卫星引物均由本实验室自行设计开发,引物序列见表2.PCR反应体系为10μL,包括:5μL2×EsTaqMasterMix;1μLDNA模板(50ng/μL);1μL上游引物和下游引物,RNase-FreeWater补齐.采取梯度PCR方法,确定合适退火温度(表2),以减少非特异性条带.PCR反应条件:1)95 ℃变性 4min;2)94 ℃热变性 30s,各自退火温度下复性 30s,72 ℃延伸 30s,共进行35个循环;3)72 ℃延伸10min;4)PCR产物4 ℃保存.获得的PCR产物通过15g/L琼脂糖凝胶电泳进行初步检测合格后,用80g/L非变性聚丙烯酰胺凝胶电泳进行微卫星多态性检测. 1.4 统计学分析 通过QuantityOne凝胶图像分析软件分析电泳条带,根据条带位置确定基因型,利用Popgene(Version1.32)软件进行群体遗传分析,计算等位基因数目(Na)、观测杂合度(Ho)与期望杂合度(He),通过U检验进行Hardy-Weinberg平衡测试.利用PIC-CALC软件对各微卫星位点的多态信息含量(PIC)进行计算. 利用ARLEQUIN 3.1计算群体遗传分化的F-统计量(Fst)及分子方差分析(AMOVA).计算群体间Nei’s遗传距离,利用MEGA 4.1软件基于Nei’s遗传距离构建UPGMA系统进化树.釆用软件Structure 2.3.4进行群体结构分析,估算每个个体的遗传构成及其所属的亚群,并绘制群体遗传结构图. 2.1 4个日本沼虾野生群体遗传多样性 通过22对微卫星引物对4个日本沼虾野生群体进行遗传多样性分析,所有的微卫星位点都显示足够的多态性,图1为MN05引物在4个群体中的扩增结果,扩增后各个位点等位位点数及座位多态信息含量如表2. 22个引物扩增共获得183个等位位点,等位位点数在4~14,平均等位位点数8.318 2.其中引物MN07和MN12获得的等位位点数最多,均为14个;引物MN20和MN21均获得4个等位位点,等位位点数最少.观测杂合度Ho值在0.052 1~0.906 2,平均为0.458 9;期望杂合度He值在0.214 6~0.892 6,平均值为0.562 3,大于平均观测杂合度.22个等位位点的多态信息含量PIC介于0.209 5~0.889 0,平均值为0.527 3,可有效进行遗传多样性分析. 位点引物序列(5'to3')退火温度/℃NaHoHePICMN01F:CGGCGAGTGACAATGAAGR:GAAGCTCATGTTGGCTCTTAT56110.50000.47780.4581MN02F:ACAGCCATTTGTCTCAGTATCTR:TCCCAGCAGAAAGACGAG54100.41670.36400.3479MN03F:ACGAGCACCATAACAACCAR:AAATGTGATGCTGAGTGGC5780.34780.61590.5779MN04F:GGCGTTCACCTACACCACTAR:AGAAGTCCTAGATGCTGCTCTG6050.15790.25110.2420 续表2 4个日本沼虾野生群体的遗传多样性数据见表3.总体来看,各野生群体的等位基因数(Na)、有效等位基因数(Ne)、香农指数(I)、观测杂合度(Ho)和期望杂合度(He)接近.就近交系数(Fis)而言,4个日本沼虾群体间存在一定的差别,其中河北白洋淀野生群体Fis最高,山东微山湖野生群体Fis最低,4个群体Fis大小顺序依次为白洋淀>洪泽湖>衡水湖>微山湖,各群体Fis值均为正值. 对88个群体位点组合(4个种群×22个引物)进行Hardy-Weinberg平衡检验,55(62.5%)个群体位点组合符合Hardy-Weinberg平衡(P>0.05),33(37.5%)个群体位点组合则显著偏离Hardy-Weinberg平衡(P<0.05).Hardy-Weinberg平衡检验P值结果显示,白洋淀、衡水湖和洪泽湖群体显著偏离Hardy-Weinberg平衡的组合,都为9个,微山湖6个. 表3 日本沼虾4个群体的遗传多样性统计 2.2 日本沼虾群体的遗传结构 4个群体间的Nei’s遗传距离和遗传分化指数(Fst)如表4所示.洪泽湖和白洋淀野生群体间的遗传距离最远(0.167 0);白洋淀与衡水湖野生群体间遗传距离最近(0.078 6).4个群体的Fst均介于0.046 3~0.101 2,白洋淀与衡水湖野生群体间的遗传分化指数最小(0.046 3);洪泽湖和白洋淀野生群体间的遗传分化指数最大(0.101 2).UPGMA系统进化树显示(图2),白洋淀和衡水湖群体聚在一起,微山湖和洪泽湖群聚在一起.分子方差分析如表5所示,4个日本沼虾野生群体中的遗传变异93.07%存在于群体内,仅有6.93%的遗传变异来自于群体间,但各野生群体间遗传分化极显著(P<0.01). 表4 4个日本沼虾野生群体间的Nei’s遗传距离(对角线上)和Fst值(对角线下)矩阵 图2 4个地理群体遗传聚类分析结果Fig.2 UPGMA clustering tree of Macrobrachium nipponense based on Nei’ s genetic distance among the four populations 变异来源自由度平方和变异组分变异比例/%群体间375.2860.39716.93*群体内个体间92555.1460.699412.20*个体内96445.0004.635480.87*总变异1911075.4325.7319 *经过1 000次模拟检验后极显著(P<0.01). 本实验设置了100 000个burnin运算长度,预设1~10个假设K值,即群体的理论划分数目,每个K值重复运算10次,通过不同假设K值下的DeltaK值确定最适K值,图3为分别在K=2、3、4的情况下4个群体的遗传结构图.STRUCTURE软件分析结果显示,当K=2时,白洋淀群体和衡水湖群体聚为一簇,微山湖群体和洪泽湖群体聚为一簇,与遗传发育树结果相似,分为北方群体和南方群体.当K=3时,白洋淀群体和洪泽湖群体被独立集群,因此从遗传学角度可以把它们看作不同群体.当K=4时,4个群体多数个体归于各自集群. 图3 日本沼虾群体在不同假设K值下的遗传结构Fig.3 STRUCTURE genetic cluster analysis for the four populations of Macrobrachium nipponense(K=2—4) 3.1 群体遗传多样性分析 遗传多样性水平是群体种质资源评价的重要依据,而高水平的遗传多样性表明群体具有良好的生存能力和繁殖潜力[9].在本研究中,4个野生群体平均观测杂合度介于0.435 7~0.502 1,高于淮河群体(0.222~0.330)[10],低于黄河群体(0.470 5~0.573 1)[11]和太湖群体(0.750 0~0.822 2)[12].本实验所得结果与黄河、千岛湖、太湖日本沼虾种群遗传多样性研究结果类似[11-13],部分群体-位点组合显著偏离Hardy-Weinberg平衡,这种现象极有可能是随机变化和/或小样本量所引起.在本实验中,考虑到He明显高于Ho,杂合子缺失可能是偏离Hardy-Weinberg平衡最有力的证据[14].通过比较Ho和Hardy-Weinberg平衡,可以推断本实验研究的4个地区日本沼虾野生群体生长在合适的水域环境中,表明日本沼虾野生群体还保留着相当高的遗传多样性. 3.2 群体遗传结构分析 遗传分化指数(Fst)值是揭示群体间的遗传分化程度的重要参数.Fst值在0~0.05,群体的遗传分化水平较低;Fst值在0.05~0.15时,群体显示中等程度的遗传分化水平,Fst值大于0.15时,群体显示出较高的遗传分化水平[15].AMOVA结果显示,本研究中4个日本沼虾野生群体的Fst值均介于0.046 3~0.101 2,表明4个日本沼虾群体间存在中等程度的遗传差异,这与其他水域日本沼虾野生群体的微卫星遗传多样性研究结果基本吻合[11-13,16],而对中国沿海7个拟穴青蟹(Scyllaparamamosain)野生群体分析的Fst值介于0.035 5~0.081 7[17];中国和泰国6个罗氏沼虾(M.rosenbergii)养殖群体间的Fst值介于0.006 1~0.093 1[18];克氏原螯虾(Procambarusclarkia)的长江下游几个地理种群中,该值介于0.040 5~0.155 9[19],这几种重要的经济类甲壳动物野生群体的遗传分化水平均与日本沼虾野生群体相近.Brown等[20]研究表明异交物种90%以上的遗传变异来自于群体内部,本研究分子方差分析结果表明,日本沼虾野生群体之间的遗传变异仅占变异总数的6.93%,部分的遗传变异来源于群体内各个体间(12.20%),绝大部分的遗传变异来自于个体内部(80.87%),表明日本沼虾野生群体变异水平较低.根据计算出的Nei’s遗传距离对日本沼虾野生群体进行聚类分析,结果表明:地理位置接近的白洋淀和衡水湖群体聚类为一支;微山湖和洪泽湖群体聚类为一支,与实际的地理间隔相符,随着距离的增大,亲缘关系渐远.可见地理距离对于日本沼虾野生群体分化程度有较为重要的影响.同时亲缘关系与生活环境和生活习性密切相关,白洋淀和衡水湖2个群体的生活环境比较封闭,且日本沼虾本身的游泳能力有限,只做近距离游动,仅在幼苗时期随水流动或人为因素被动扩散外,与其他野生群体的基因交流较少;南方河流分布比较密集,微山湖和洪泽湖日本沼虾野生群体生活环境比较开放,存在着一定程度的基因交流. STRUCTURE软件可根据个体遗传组成进行群体遗传结构模拟分析,不受各实验群体的样本量的影响,可以作为群体遗传结构分析的重要工具[21].STRUCTURE条形图显示,当K=2时DeltaK值最大,说明存在2个亚群,白洋淀群体和大部分衡水湖群体分配到1号亚群,衡水湖的少部分个体和其余2个群体中大部分个体分配到2号亚群,与4个日本沼虾群体的聚类分析结果相符合. 通过以上分析,揭示了白洋淀、衡水湖、微山湖和洪泽湖4个地区日本沼虾野生群体的遗传多样性现状及群体遗传结构,这些结果可为日本沼虾优良种质资源保护及遗传选育研究提供基础数据支持. [1] 李新正,刘瑞玉,梁象秋.中国长臂虾总科的动物地理学特点[J].生物多样性,2003,11(5):393-406.DOI:10.3321/j.issn:1005-0094.2003.05.005.LIXZ,LIURY,LIANGXQ.ThezoogeographyofChinesePalaemonoideafauna[J].Biodiversity Science,2003,11(5):393-406.DOI:10.3321/j.issn:1005-0094.2003.05.005. [2] RITLAND K,NEWTON C,MARSHALL H D.Inheritance and population structure of the white-phased “Kermode” black bear[J].Current Biology,2001,11(18):1468-1472.DOI:10.1016/S0960-9822(01)00448-1. [3] CRUZ V M,KILIAN A,DIERIG D A.Development of DArT marker platforms and genetic diversity assessment of the U.S.collection of the new oilseed crop lesquerella and related species[J].Plos One,2013,8(5):e64062.DOI:10.1371/journal.pone.0064062. [4] HEIKRUJAM M,KUMAR J,AGRAWAL V.Genetic diversity analysis among male and female Jojoba genotypes employing gene targeted molecular markers,start codon targeted(SCoT)polymorphism and CAAT box-derived polymorphism(CBDP)markers[J].Meta Gene,2015,5:90-97.DOI:10.1016/j.mgene.2015.06.001. [5] SEYEDIMORADI H,TALEBI R.Detecting DNA polymorphism and genetic diversity in Lentil(LensculinarisMedik.)germplasm:comparison of ISSR and DAMD marker[J].Physiology and Molecular Biology of Plants,2014,20(4):495-500.DOI:10.1007/s12298-014-0253-3. [6] AN H S,NAM M M,MYEONG J I,et al.Genetic diversity and differentiation of the Korean starry flounder(Platichthysstellatus)between and within cultured stocks and wild populations inferred from microsatellite DNA analysis[J].Molecular Biology Reports,2014,41(11):7281-7292.DOI:10.1007/s11033-014-3614-7. [7] KESSUWAN K,KUBOTA S,LIU Q,et al.Detection of growth-related quantitative trait loci and high-resolution genetic linkage maps using simple sequence repeat markers in the kelp grouper(Epinephelusbruneus)[J].Marine Biotechnology(NY),2016,18:57-84.DOI:10.1007/s10126-015-9673-5. [8] SONG W T,LI Y Z,ZHAO Y W,et al.Construction of a high-density microsatellite genetic linkage map and mapping of sexual and growth-related traits in half-smooth tongue sole(Cynoglossussemilaevis)[J].Plos One,2012,7(12):e52097.DOI:10.1371/journal.pone.0052097. [9] O'CONNELL M,WRIGHT J M.Microsatellite DNA in fishes[J].Reviews in Fish Biology and Fisheries,1997,7:331-363.DOI:10.1023/A:1018443912945. [10] 姜虎成,冯建彬,丁怀宇,等.淮河安徽段日本沼虾野生群体遗传结构的微卫星分析[J].上海海洋大学学报,2012,21(2):167-175. JIANG H C ,FENG J B,DING H Y,et al.Genetic structure analysis of naturalMacrobrachiumnipponensepopulations in Anhui section of Huaihe River based on microsatellite[J].Journal of Shanghai Ocean University,2012,21(2):167-175. [11] QIAO H,LV D,JIANG S F,et al.Genetic diversity analysis of oriental river prawn,Macrobrachiumnipponense,in Yellow River using microsatellite marker[J].Genetics and Molecular Research,2013,12(4):5694-5703.DOI:10.4238/2013.November.18.18. [12] 冯建彬,吴春林,马克异,等.太湖日本沼虾野生群体遗传结构的微卫星分析[J].应用生态学报,2011,22(6):1606-1614. FENG J B,WU C L,MA K Y,et al.Genetic structure of wildMacrobrachiumnipponensepopulations in Taihu Lake based on microsatellite analysis[J].Chinese Journal of Applied Ecology,2011,22(6):1606-1614. [13] MA K Y,FENG J B,LI J L.Genetic variation based on microsatellite analysis of the oriental river prawn,Macrobrachiumnipponensefrom Qiandao Lake in China[J].Genetics and Molecular Research,2012,11(4):4235-4244.DOI:10.4238/2012.September.20.1. [14] QUAN Y C,SUN X W,LIANG L Q.Genetic polymorphism of microsatellite DNA in two populations of northern sheatfish(Silurussoldatovi)[J].Acta Genetica Sinica,2006,33(10):908-916.DOI:10.1016/S0379-4172(06)60125-X. [15] BALLOUX F,LUGON-MOULIN N.The estimation of population differentiation with microsatellite markers[Review][J].Molecular Ecology,2002,11(11):155-165.DOI:10.1046/j.0962-1083.2001.0143 6.x. [16] 马克异,冯建彬,谢楠,等.钱塘江日本沼虾野生群体遗传变异的SSR分析[J].动物学研究,2011,32(4):363-370.DOI:10.3724/SP.J.1141.2011.04363. MA K Y,FENG J B,XIE N,et al.Microsatellite analysis of genetic variation of the oriental river prawnMacrobrachiumnipponensein Qiantang River[J].Zoological Research,2011,32(4):363-370.DOI:10.3724/SP.J.1141.2011.04363. [17] 舒妙安,周宇芳,朱晓宇,等.中国沿海拟穴青蟹群体遗传多样性的微卫星分析[J].水产学报,2011,35(7):977-984 .DOI:10.3724/SP.J.1231.2011.17102. SHU M A,ZHOU Y F,ZHU X Y,et al.Microsatellite analysis on genetic diversity of seven wild populations ofScyllaparamamosainin China[J].Journal of Fisheries of China,2011,35(7):977-984.DOI:10.3724/SP.J.1231.2011.17102. [18] 孙成飞,叶星,董浚键,等.罗氏沼虾6个养殖群体遗传多样性的微卫星分析[J].南方水产科学,2015,11(2):20-26.DOI:10.3969/j.issn.2095-0780.2015.02.003. SUN C F,YE X,DONG J J,et al.Genetic diversity analysis of six cultured populations ofMacrobrachiumrosenbergiiusing microsatellite markers[J].South China Fisheries Science,2015,11(2):20-26.DOI:10.3969/j.issn.2095-0780.2015.02.003. [19] 王长忠,李忠,梁宏伟,等.长江下游地区4个克氏原螯虾群体的遗传多样性分析[J].生物多样性,2009,17(5):518-523.DOI:10.3724/SP.J.1003.2009.09017. WANG C Z,LI Z,LIANG H W,et al.Genetic diversity in fourProcambarusclarkiipopulations in the lower reaches of the Yangtze River[J].Biodiversity Science,2009,17(5):518-523.DOI:10.3724/SP.J.1003.2009.09017. [20] BROWN A H D,CLEGG M T,KAHLER A L,et al.Plant population genetics,breeding,and genetic resources[M].Sunderland :Sinauer Associates Inc.,1989:43-63. [21] 傅建军,李家乐,沈玉帮,等.草鱼野生群体遗传变异的微卫星分析[J].遗传,2013,35(2):192-201.DOI:10.3724/SP.J.1005.2013.00192. FU J J,LI J L,SHEN Y B,et al.Genetic variation analysis of wild populations of grass carp(Ctenopharyngodonidella)using microsatellite markers[J].Hereditas,2013,35(2):192-201.DOI:10.3724/SP.J.1005.2013.00192. (责任编辑:赵藏赏) Microsatellite analysis of genetic diversity in four wild populations of oriental river prawn(Macrobrachiumnipponense) WU Xiaobin1,MU Shumei1,ZHAO Lingyu1,KANG Xianjiang1,XUE Jianmin2 (1.College of Life Sciences,Hebei University,Baoding 071002,China;2. Fishery Technology Extending Station, Bureau of Animal Hasbandry and Aquiculture of Hengshui,Hengshui 053000,China) Genetic diversity and differentiation of different wild population of oriental river prawn(Macrobrachiumnipponense)in Baiyangdian Lake,Hengshui Lake,Weishan Lake and Hongze Lake,were investigated using 22 microsatellite DNA loci.The data showed that all the 22 loci were highly polymorphic.An exactP-value test indicated that most loci significantly deviated from Hardy-Weinberg in the Baiyangdian Lake,Hengshui Lake and Hongze Lake populations with the number of 9 loci,respectively;the deviations of Hardy-Weinberg were also detected in Weishan Lake populations with the number of 6 loci.All of the four wild populations showed high genetic diversities,and the genetic diversity in Weishan populations was relatively higher than the others three populations.TheFstand AMOVA analysis suggested that there was a moderate degree of genetic divergence among the four populations(0.046 3 Macrobrachiumnipponense;microsatellite;genetic diversity 10.3969/j.issn.1000-1565.2017.02.009 2016-03-25 河北省现代农业产业技术体系淡水养殖创新团队项目 武小斌(1992—),男,河北张家口人,河北大学在读硕士研究生.E-mail:hbdxwxb@163.com 康现江(1964—),男,河北邯郸人,河北大学教授,博士生导师,主要从事细胞发育分化及其调控方面的研究. E-mail:xjkang218@126.com Q347 A 1000-1565(2017)02-0161-081 材料与方法

2 结果与分析





3 讨论
