玉米大斑病菌Homeobox转录因子家族全基因组鉴定及其表达规律分析
2017-04-10赵洁赵立卿巩校东冯胜泽刘星晨郑亚男李志勇孙海月王冬雪韩建民谷守芹董金皋
赵洁,赵立卿,巩校东,冯胜泽,刘星晨,郑亚男,李志勇,孙海月,王冬雪,韩建民,谷守芹,董金皋
(1河北农业大学真菌毒素与植物分子病理学实验室,河北保定 071000;2河北省农林科学院谷子研究所,石家庄 050000)
玉米大斑病菌Homeobox转录因子家族全基因组鉴定及其表达规律分析
赵洁1,赵立卿1,巩校东1,冯胜泽1,刘星晨1,郑亚男1,李志勇2,孙海月1,王冬雪1,韩建民1,谷守芹1,董金皋1
(1河北农业大学真菌毒素与植物分子病理学实验室,河北保定 071000;2河北省农林科学院谷子研究所,石家庄 050000)
【目的】从全基因组水平上鉴定玉米大斑病菌(Setosphaeria turcica)Homeobox转录因子家族及其分布,阐明该家族的序列及进化特征,分析该家族基因在病菌不同生长发育时期的表达规律。【方法】利用生物信息学手段搜索玉米大斑病菌全基因组数据库,鉴定Homeobox转录因子家族;采用MEGA 5.0软件进行系统进化树分析;利用在线工具GSDS(gene structure display server)(http://gsds1.cbi.pku.edu.cn/index.php)绘制基因结构图;利用Clustal X 1.83软件分析Homeobox保守结构域(HOX保守结构域)的氨基酸序列特征;利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.plpage=npsa_sopma.html)对Homeobox蛋白的二级结构进行在线预测;利用实时荧光定量 PCR(qRT-PCR)技术分析Homeobox转录因子家族在病菌不同发育时期的表达模式。【结果】在玉米大斑病菌中鉴定了8个Homeobox转录因子家族成员(StHTF1-8),根据基因结构及系统进化特征将其分为4类;亚细胞定位预测分析表明,这8个蛋白全部定位在细胞核中;该家族成员均含有HOX保守结构域,其二级结构具有特征性的“螺旋-转角-螺旋”(helix-turn-helix)结构;利用qRT-PCR技术对该家族成员在菌丝、分生孢子形成、芽管形成、附着胞及侵入丝形成等5个时期的表达规律分析,发现不同基因在病菌不同发育时期具有不同的表达水平,其中StHTF1在菌丝发育、分生孢子及附着胞形成等3个时期的表达水平相对较高,StHTF3、StHTF4在分生孢子形成时期表达水平最高,StHTF6在芽管形成时期的表达水平最高,StHTF2、StHTF5、StHTF7和StHTF8在附着胞形成时期的表达水平均较高。【结论】玉米大斑病菌包括Homeobox转录因子家族包含8个成员,在进化上分为4大类,全部成员均分布在细胞核内,其编码蛋白质均含有保守的HOX结构域及“螺旋-转角-螺旋”空间结构;该基因家族成员在病菌不同发育时期呈现不同的表达规律。
玉米大斑病菌;Homeobox转录因子家族;系统进化;基因表达
0 引言
【研究意义】Homeobox转录因子家族是一类结构和功能均具有保守性的转录调控因子,该基因家族成员编码的同源异型结构域蛋白(Homeobox protein)可以与靶基因的调控序列结合而影响靶基因的转录,从而调控个体的生长发育及细胞分化等方面[1-2]。研究玉米大斑病菌(Setosphaeria turcica)基因组中Homeobox转录因子家族的分布、基因结构特征与表达模式,对进一步研究其在该病菌生长发育及致病性中的功能具有重要意义。【前人研究进展】Homeobox protein是广泛存在于真核生物中的一类转录调控因子,同源结构域含有3个α-螺旋,其中螺旋Ⅰ位于肽段的N末端,与螺旋Ⅱ之间有一个环(loop),且螺旋Ⅱ与螺旋Ⅲ之间形成“螺旋-转角-螺旋”(helix-turn-helix)DNA结合基序[3]。研究还发现,螺旋Ⅲ结构最为保守,其中的几个氨基酸残基(如玉米Kn1氨基酸序列中W48、F49、N51、R53)几乎在所有同源蛋白中都是相同的[4]。研究植物及动物中的Homeobox基因时发现,这些不同的基因可以被划分为不同的类型。在植物中可以分为14个类群,分别是HD-ZIPⅠ、HD-ZIPⅡ、HD-ZIPⅢ、HD-ZIPⅣ、WOX、PLINC、BEL、KNOX、DDT、PINTOX、PHD、LD、NDX及SAWADEE[5-6];在动物中划分为8个不同类型,依次为Antp、TALE、SIX、LIMJPOU、PRD、 CUTZF、HNF和PROS[7-8],这些不同的进化特征表明不同类型的Homeobox基因可能具有不同的功能。研究还发现,真菌中的Homeobox基因家族成员主要参与菌丝的丝状生长、分生孢子萌发、有性生殖及致病性等生物过程。如在已经完成全基因组测序的模式真菌酿酒酵母(Saccharomyces cerevisiae)中发现9个不同的Homeobox基因[1],其中参与调控病菌生物过程的基因有7个,如Yox1和Yhp1是两个起负调控作用的转录因子,它们的表达影响细胞减数分裂过程中M/G1时期的生理活动[9];另外,YHP1通过结合IME1启动子区影响减数分裂过程[10],转录抑制因子NRM1和YHP1抑制减数分裂G1时期特异基因的转录,从而影响细胞分化过程[11];在丝状真菌玉米黑粉菌(Ustilago maydis)中的Homeobox基因bW和bE与病菌的菌丝生长及致病性相关[12];在禾谷镰孢(Fusarium graminearum)中的Homeobox 基因htf1可特异调控分生孢 子的产生[13]。但在玉米大斑病菌中对Homeobox家族的组成、分子结构特征及表达规律的研究尚未见报道。【本研究切入点】玉米大斑病(Northern corn leaf blight)是由玉米大斑病菌引起的一种重要的真菌性病害,该病害在世界玉米产区普遍发生,流行年份可导致玉米产量减产达50%以上[14]。玉米大斑病菌全基因组测序的完成为从分子水平上分析病菌基因家族成员的结构特征提供了良好的平台。基于此,本文在通过搜索玉米大斑病菌基因组数据库、获得Homeobox转录因子家族序列的基础上,整体分析该家族的结构特征及其表达规律。【拟解决的关键问题】利用生物信息学手段从全基因组水平上鉴定玉米大斑病菌Homeobox转录因子家族的数目及其分布,阐明该基因家族的序列、结构及进化特征,利用real-time PCR技术分析该基因家族在病菌各个生长时期的表达特点,探索Homeobox转录因子家族在病菌不同生长发育时期的表达规律。
1 材料与方法
试验于2015—2016年在河北农业大学完成。
1.1 供试菌株
玉米大斑病菌野生型菌株01-23,由河北农业大学真菌毒素与植物分子病理学实验室保存。
1.2 主要试剂
总RNA提取试剂盒、RNA反转录试剂盒购于上海生工生物工程有限公司。
1.3 玉米大斑病菌Homeobox转录因子家族的鉴定
主要参照经典的Homeobox转录因子家族的鉴定方法进行并略有改动[4,7]。利用PFAM数据库进行在线搜索获得Homeobox基因家族的隐马尔可夫模型(HMM);通过hmmsearch程序搜索玉米大斑病菌中全基因组的蛋白序列,E-value值设定为10—50;利用在线工具SMART分析结构域;选取包含Homeobox保守结构域(HOX结构域)、保守的基序序列为候选成员。利用在线服务器ExPAS中的生物信息学软件ExPASy-ProtParam tool(http://web.expasy. org/protparam/)对玉米大斑病菌中所获得的Homeobox蛋白进行理化性质分析,如氨基酸数目、相对分子质量、不稳定系数及等电点。通过在线软件WoLF PSORT(http://www.genscript.com/psort/wolf_ psort.html)预测所有蛋白的亚细胞定位。
1.4 玉米大斑病菌Homeobox转录因子家族的结构特征及系统进化分析
通过对玉米大斑病菌全基因组数据库(JGI,http://genome.jgi-psf.org/Settu1/Settu1.home.html)的搜索得到Homeobox基因家族基因的CDS序列和全基因组序列。利用在线工具GSDS(gene structure display server)(http://gsds1.cbi.pku.edu.cn/index.php)绘制基因结构图。利用在线生物信息学软件SMART(http://smart.embl-heidelberg.de/)对上述蛋白进行Homeobox结构域分析,并利用Clustal X 1.83软件对各个Homeobox结构域的氨基酸序列进行比对。使用软件MEGA 5.0中的邻接法构建系统发育树,bootstrap值设置为1 000。
1.5 玉米大斑病菌Homeobox转录因子家族表达模式分析
菌丝的收集:将野生型菌株01-23接种于PDA培养基中,用封口膜密封培养皿,25℃黑暗培养10 d,收集菌丝体;分生孢子的收集:将野生型菌株01-23接种于LCA培养基(产孢培养基)中,25℃黑暗培养16 d,诱使菌丝产生分生孢子,然后在平皿中加入10 mL灭菌的ddH2O,用枪头轻刮培养基表面,将菌丝与分生孢子悬浮液经3层纱布过滤以除去菌丝体,收集分生孢子;芽管、附着胞及侵入丝时期的病菌材料的收集:将上述过滤后的孢悬液稀释至浓度为1×104个孢子/mL,然后在表面消毒的复印膜上滴加分生孢子悬浮液,25℃黑暗保湿条件下诱使其萌发,当诱导时间分别为4、10、24 h时,分别收集孢悬液至1.5 mL EP中,3 000 r/min离心收集芽管时期、附着胞时期及侵入丝形成时期的材料。将收集的样品保存于-80℃备用。所有试验设置3次重复。
表达模式分析:利用UNIQ-10柱式Trizol总RNA抽提试剂盒提取玉米大斑病菌野生型总RNA,按照张鑫等[15]的方法合成第一链cDNA。以18S rRNA作为内参,扩增Homeobox转录因子家族cDNA片段,引物设计见表1。分别以不同发育时期的第一链cDNA为模板,按以下条件进行real-time PCR扩增,10 μL扩增体系中含有cDNA模板1.0 μL;Mix 5.0 μL;正义引物(10 μmol·L-1)及反义引物各0.4 μL;ROX 0.2 μL;ddH2O 3 μL。上述步骤完成后,将样品加入96孔板,放于ABI Stepone plus型荧光定量PCR仪中进行反应。每个样品进行3次重复。以野生型菌株的基因转录水平作对照,采用比较Ct值法(2-ΔΔCt法)分析各个基因在野生型菌株中的相对表达量。该家族的各基因在不同发育时间的相对表达量以菌丝时期的基因转录水平为对照。
2 结果
2.1 玉米大斑病菌Homeobox转录因子家族的鉴定
利用生物信息学方法从玉米大斑病菌全基因组中共鉴定得到8个Homeobox转录因子基因,分别命名为StHTF1—StHTF8。进一步利用基因组序列及注释信息分析该基因家族的基因组定位,发现该家族成员散布在病菌基因组中,分布在6个scaffold上,并且位于负链的家族成员居多。其中,StHTF1单独定位于scaffold_2正链上,StHTF2单独定位于scaffold_6(正链)上,StHTF3单独定位于scaffold_7(正链)上,StHTF4和StHTF5定位于scaffold_11负链,且StHTF6定位于scaffold_11正链上,StHTF7和StHTF8定位于scaffold_3负链上(表2)。
利用在线软件ProtParam预测病菌中所有Homeobox蛋白的理化特性,结果发现这8个Homeobox蛋白在氨基酸数目、相对分子质量及等电点方面均不同。从该家族各基因所编码的蛋白来看,其长度在337—869 aa,且大部分长度在600 aa左右;从预测的Homeobox蛋白的等电点数据可以得知,这些蛋白大多呈酸性,其中只有2个基因编码的蛋白(StHTF4和StHTF7)呈碱性;通过预测还发现这些蛋白都属于不稳定蛋白(不稳定系数>40考虑为不稳定);通过亚细胞定位预测分析发现,这8个蛋白均定位在细胞核中(表2)。

表1 实时荧光定量PCR扩增所用引物Table 1 Primers used in qRT-PCR
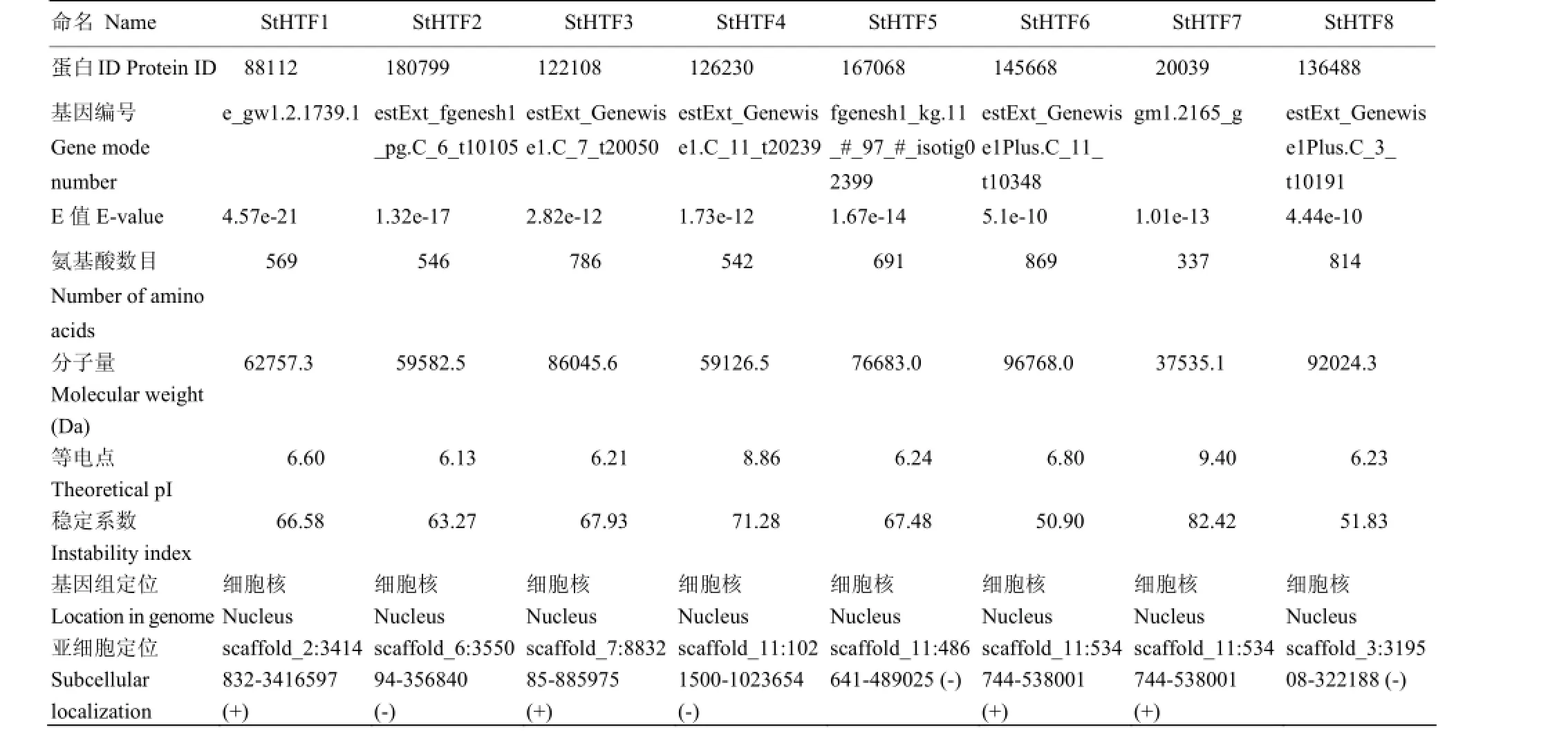
表2 玉米大斑病菌中的Homeobox转录因子家族信息Table 2 The information of Homeobox transcription factor family in S. turcica
2.2 玉米大斑病菌Homeobox转录因子家族基因结构特征及系统进化分析
通过对Homeobox转录因子家族的基因结构分析显示,所有家族成员基因均有内含子,其中StHTF1、StHTF3及StHTF5均含有1个内含子,StHTF4有3个内含子,其余基因含有2个内含子(图1)。
为了深入分析玉米大斑病菌Homeobox转录因子家族与其他物种同源基因的进化关系,对该家族进行了系统发育分析。通过与酿酒酵母、禾谷镰孢、粗糙脉胞菌(Neurospora crassa)、灰葡萄孢(Botrytis cinerea)等其他真菌物种的氨基酸序列对比,并利用MEGA 5.0中的邻接法构建系统发育树(图2)。结果发现真菌Homeobox转录因子家族可以进一步分为4类:其中在玉米大斑病菌中StHTF1、StHTF2为第Ⅰ类;StHTF3、StHTF4为第Ⅱ类;StHTF5、StHTF6、StHTF7和StHTF8为第Ⅲ类,第Ⅲ类还分为2个亚类,StHTF7属于一个亚类,其他3个蛋白都具有2—4个串联的ZnF-C2H2锌指结构聚为第Ⅳ类。进一步根据与其他物种中Homeobox转录因子同源基因的亲缘关系的远近,分别将玉米大斑病菌Homeobox转录因子家族成员命名为StHTF1—StHTF8。
2.3 玉米大斑病菌Homeobox转录因子家族保守基序分析及序列比对
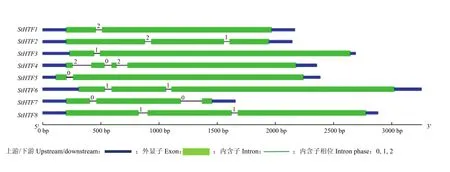
图1 玉米大斑病菌Homeobox转录因子家族基因结构特征Fig. 1 The structure characteristics of Homeobox transcription factor family in S. turcica
利用在线软件SMART和InterProScan对这8个蛋白序列进行保守结构域分析,结果发现8个基因所编码的蛋白均含有同源异型盒结构域HOX,但该结构域在各个蛋白中的位置有所不同,在StHTF1、StHTF3、StHTF4、StHTF6和StHTF8中HOX靠近N末端;在StHTF2和StHTF5中位于中部,而在StHTF7中则靠近C末端。进一步分析还发现,部分基因除了含有HOX结构域外,还有其他结构域,如StHTF5、StHTF6和StHTF8还含有2—4个串联的ZnF-C2H2锌指结构(图3-A)。
利用SMART在线软件分析病菌中HOX蛋白的氨基酸序列,并从稻瘟病菌(Magnaporthe oryzae)、禾谷镰孢、酿酒酵母、粗糙脉胞菌和黑腹果蝇(Drosophila melanogaster)的数据库中分别找到已经注释为Homeobox蛋白的HOX结构域的氨基酸序列,用ClustalX软件对这些氨基酸序列进行比对分析,再利用在线软件BoxShade Server进行多重比对序列着色标记,结果发现所比较的这些蛋白中同源域的氨基酸序列相似,且在L16、P29、W52和R57位置上均具有相同的氨基酸残基(图3-B)。
通过对蛋白质的二级结构预测分析发现,这些Homeobox蛋白在空间结构上都具有典型的螺旋结构(图3-B黑色横线)。这4个相同的保守氨基酸残基分别位于螺旋Ⅰ、loop环和螺旋Ⅲ,且这种螺旋结构与Homeobox蛋白的空间结构“螺旋-转角-螺旋”(helix-turn-helix)相符。通过比对还发现,不同物种间HOX蛋白的氨基酸残基在螺旋Ⅲ位置上具有相对多的着色区域,说明螺旋Ⅲ具有更高的保守性(图3-B)。利用SOPMA在线服务器预测Homeobox家族蛋白的二级结构,结果发现这8个蛋白的二级结构中无规则卷曲含量都比较高,如StHTF7蛋白中自由卷曲度高达64.99%,StHTF3蛋白中含量为63.99%,其次为α-螺旋,最后是β-折叠,且β-转角含量最少。
2.4 玉米大斑病菌Homeobox转录因子家族表达模式分析
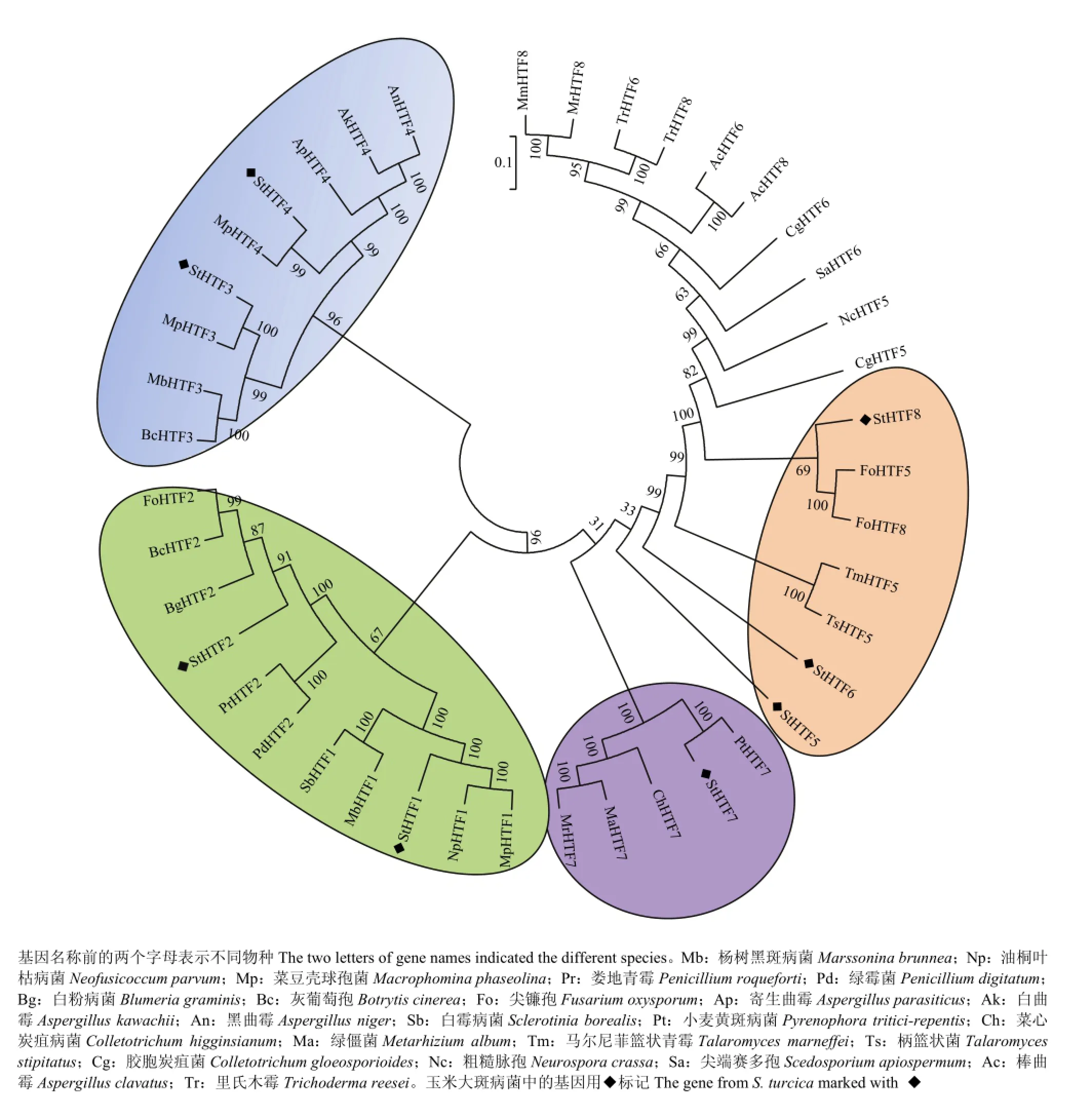
图2 玉米大斑病菌Homeobox基因家族系统发育树分析Fig. 2 The rooted phylogenetic tree of Homeobox transcription factor family in S. turcica
利用实时荧光定量PCR方法对Homeobox转录因子家族在菌丝发育、分生孢子形成、芽管萌发、附着胞及侵入丝形成等5个时期基因相对表达量进行分析。结果发现Homeobox转录因子家族中的8个基因在这5个时期均有表达,但在不同阶段基因表达水平存在显著差异(P≤0.05)。其中StHTF3、StHTF4具有相似的表达模式,在分生孢子形成时期表达量均显著高于菌丝时期,而在芽管萌发、附着胞及侵入丝形成时期表达量均显著下降;StHTF7、StHTF8也具有相似的表达模式,在分生孢子及附着胞形成时期的表达量均显著高于其他时期;StHTF5、StHTF6在芽管和附着胞时期显著高于菌丝和附着胞时期,而相较于StHTF5,StHTF6在侵入丝时期的表达量均比菌丝发育及分生孢子形成时期高40多倍,甚至比附着胞时期高2倍;StHTF2在附着胞形成时期的表达量显著高于其他时期,而在芽管形成和侵入丝时期表达量较低;StHTF1在这5个生长时期的表达量均较低,但在菌丝发育、分生孢子及附着胞形成等3个时期的表达水平相对较高(图4)。
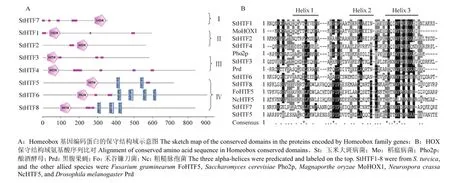
图3 玉米大斑病菌Homeobox转录因子家族保守基序分析及序列比对Fig. 3 Conservative motif analysis and sequence alignments of Homeobox transcription factor family from S. turcica
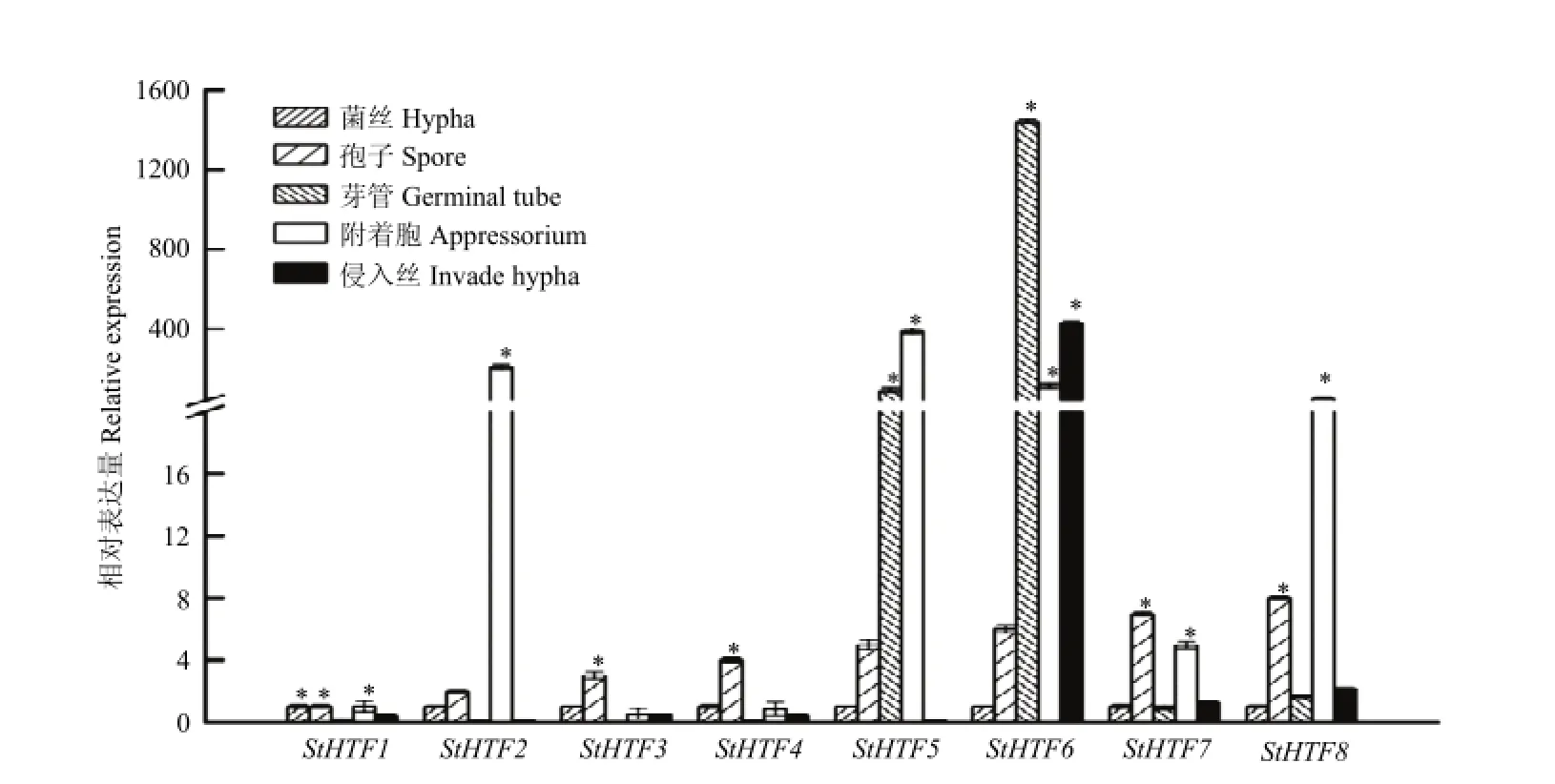
图4 Homeobox转录因子家族在玉米大斑病菌不同发育时期的表达模式Fig. 4 Expression profiles of 8 Homeobox genes assayed by qRT-PCR in S. turcica
3 讨论
随着对动物、植物及真菌基因组研究的深入,利用比较基因组学研究策略分析基因家族及基因功能已成为目前基因功能研究的热点之一。在动物、植物及真菌中有关基因家族的研究已有报道,例如大豆LEA基因家族[16]、线虫bHLH家族[17]以及酵母YRF基因家族[18]、玉米大斑病菌MAPK超家族[19]及CaMK家族[20]等,这为后续基因家族的功能分析鉴定了理论基础。
迄今为止,在某些动物[21]、植物[22]及真菌[23-24]中均鉴定了Homeobox基因家族,但其家族成员的种类和数目有所不同。在动物中发现的Homeobox基因共有16类,包括ANTP、PRD、PRD-LIKE、POU、HNF、CUT、LIM、ZF、CERS、PROS、SIX/SO、IRO、MKX、TGIF、PBC和MEIS等[25];在植物中发现的Homeobox基因主要分为11类,包括HD-ZIP、WOX、NDX、PHD、PLINC、LD、DDT、SAWADEE、PINTOX、KNOX 和BEL等[25];在模式生物酿酒酵母中鉴别出9个Homeobox基因[26];在禾谷镰孢中鉴定了12个可能的Homeobox基因[4];在稻瘟病菌中鉴定出8个Homeobox基因[27]。本研究通过对玉米大斑病菌全基因组分析,鉴定出了8个Homeobox基因。对于上述在不同生物类群及物种中Homeobox基因家族种类及数目存在较大差异的原因,可能是因为物种本身在进化中的位置不同,在越高等的物种如动物及植物中该基因家族成员数目越庞大,在较为低等的物种如酵母和丝状真菌中该家族成员数目较少,这也从另外一个角度说明该家族在调控不同物种生长发育过程中具有重要地位。但由于家族成员较多,不同家族成员间结构还存在一定差异,是否需要根据其他结构特征对该家族进行更细致的分类尚需深入探讨。
研究发现在酿酒酵母及丝状真菌中发现的Homeobox基因家族成员除含有HOX保守结构域外,还含有ZnF-C2H2锌指结构域,但其数目有所不同。其中子囊菌亚门(Ascomycete)肉座菌亚纲(Hypocreomycetidae)的真菌中包含较多数量的Homeobox-C2H2基因,比如在棉花黄萎病菌(Verticillium dahliae)和尖镰孢中均鉴定出7个Homeobox-C2H2基因[28];在粪壳菌亚纲(Sordariomycetidae)和锤舌菌纲(Leotiomycetes)中则含有较少的Homeobox-C2H2基因,如稻瘟病菌、粗糙脉胞菌、灰葡萄孢、核盘菌(Sclerotiniasclerotiorum)等都是只包含1个Homeobox-C2H2基因,而酵母中不含Homeobox-C2H2基因[28]。本研究发现,在玉米大斑病菌中存在8个Homeobox基因,其中3个基因StHTF5、StHTF6、StHTF8具有ZnF-C2H2锌指结构域。另外,在稻瘟病菌中的研究发现,含有ZnF-C2H2锌指结构域的Homeobox-C2H2基因(MGG_01730)对病菌的生长发育和致病性都没有作用[27],而本研究发现玉米大斑病菌中具有类似结构的基因具有不同的作用,其中StHTF5、StHTF8参与附着胞的形成,StHTF6对芽管形成具有重要的调控作用。由此可见,不同真菌中含有Homeobox-C2H2基因数目的不同与物种的进化水平密切相关,造成这种现象的原因可能是由于基因复制事件。从其功能分析来看,其保守的Homeobox-C2H2结构可能通过调控不同的靶基因来调控不同的代谢过程,其具体的机制尚需要深入研究。
Homeobox基因编码的Homeobox蛋白作为一类转录调控因子,在真核生物的个体发育及细胞分化的调控中起着重要作用。在动物中Homeobox基因在胚胎发育、基因转录调节、细胞分化等方面发挥重要作用[21];在植物中Homeobox基因主要是影响胚胎发育、叶的发育、细胞间的物质运输及花的演化等方面[15-22];在真菌中,酿酒酵母、柄孢霉菌(Podospora anserina)、玉米黑粉菌等中均发现Homeobox基因,但其功能存在差异,如在酿酒酵母中Homeobox基因CUP9在乳酸为唯一碳源时,能使酵母细胞在铜离子高度富集的环境中生长[23];在玉米黑粉菌中Homeobox基因参与调控菌丝的极性生长、致病性及有性世代[23-24];在柄孢霉菌中Homeobox基因抑制分生孢子的产生并影响菌丝的形态[29]。本研究发现,玉米大斑病菌中的8个Homeobox基因可能分别参与病菌的不同生长发育过程,如StHTF2、StHTF5、StHTF6、StHTF7和StHTF8均在附着胞形成时期具有较高水平的表达量,而StHTF5、StHTF6、StHTF7和StHTF8除了在附着胞形成时期具有较高的表达量外,StHTF5在芽管时期,StHTF6在芽管和侵入丝时期,StHTF3、StHTF4、StHTF7、StHTF8在分生孢子均具有较高的表达量。由此可见,在不同的丝状真菌中Homeobox基因具有不同的生物学功能,其每个成员的具体调控机制尚需要深入探讨。下一步将利用靶基因敲除、ChIP等技术分析每个基因的功能及其调控机制,以期为明确真菌中Homeobox基因家族的功能提供更多可以借鉴的数据支持。
4 结论
玉米大斑病菌包括8个Homeobox转录因子基因,共分为4类,全部成员均分布在细胞核内,所有基因的编码蛋白质均含有HOX保守结构域及“螺旋-转角-螺旋”空间结构;该基因家族成员在病菌不同发育时期呈现不同的表达规律。
[1] AI L P, SHEN A, GAO Z C, LI Z L, SUN Q L, LI Y Y, LUAN W J. Reverse genetic analysis of transcription factorOsHox9, a member of homeobox family in rice.Rice Science, 2014, 21(6): 312-317.
[2] LAPPIN T R, GRIER D G, THOMPSON A, HALLIDAY H L. HOX genes: seductive science, mysterious mechanisms.The Ulster Medical Journal, 2006, 75(1): 23-31.
[3] BHATTACHARJEE A, GHANGAL R, GARG R, JAIN M. Genomewide analysis of homeobox gene family in legumes: identification,gene duplication and expression profiling.PLoS ONE, 2015, 10(3): e0119198.
[4] 郑文辉, 赵旭, 张承康, 鲁国东, 王宗华, 王爱荣. 禾谷镰刀菌Homeobox基因结构及其演化关系分析. 热带作物学报, 2009, 30(2): 205-210.
ZHENG W H, ZHAO X, ZHANG C K, LU G D, WANG Z H, WANG A R. Gene structure and phylogenetic relationships of Homeobox transcriptional factors inFusarium graminearum. Chinese Journal of Tropical Crops, 2009, 30(2): 205-210. (in Chinese)
[5] JAIN M, TYAGI A K, KHURANA J P. Genome-wide identification, classification, evolutionary expansion and expression analyses of homeobox genes in rice.TheFEBS Journal, 2008, 275(11): 2845-2861.
[6] MUKHERJEE K, BROCCHIERI L, BÜRGLIN T R. A comprehensive classification and evolutionary analysis of plant homeobox genes.Molecular Biology and Evolution, 2009, 26(12): 2775-2794.
[7] HOLLAND P W, BOOTH H A, BRUFORD E A. Classification and nomenclature of all human homeobox genes.BMC Biology, 2007, 5(1): 47.
[8] TAKATORI N, BUTTS T, CANDIANI S, PESTARINO M, FERRIER D E, SAIGA H, HOLLAND P W. Comprehensive survey and classification of homeobox genes in the genome of amphioxus,Branchiostoma floridae.Development Genes and Evolution, 2008, 218(11/12): 579-590.
[9] PRAMILA T, MILES S, GUHATHAKURTA D, JEMIOLO D, BREEDEN L L. Conserved homeodomain proteins interact with MADS box protein Mcm1 to restrict ECB-dependent transcription to the M/G1 phase of the cell cycle.Genes & Development, 2002, 16(23): 3034-3045.
[10] KUNOH T, KANEKO Y, HARASHIMA S. YHP1 encodes a new homeoprotein that binds to the IME1 promoter inSaccharomyces cerevisiae.Yeast, 2000, 16(5): 439-449.
[11] OSTAPENKO D, SOLOMON M J. Anaphase promoting complex-dependent degradation of transcriptional repressors Nrm1 and Yhp1 inSaccharomyces cerevisiae.Molecular Biology of the Cell, 2011, 22(13): 2175-2184.
[12] BAKKEREN G, KÄMPER J, SCHIRAWSKI J. Sex in smut fungi: structure, function and evolution of mating-type complexes.Fungal Genetics and Biology, 2008, 45(Suppl.1): 15-21.
[13] 张红红, 郑文辉, 骆琼, 乔向威, 陈阿海, 余文英, 王宗华. 禾谷镰刀菌Homeobox转录因子FgHtf1敲除突变体基因差异表达分析.中国科技论文, 2015, 10(12): 1392-1397.
ZHANG H H, ZHENG W H, LUO Q, QIAO X W, CHEN A H, YU W Y, WANG Z H. Differential analysis of gene expression in the Homeobox transcription factor FgHtf1 deletion mutant ofFusarium graminearum.China Science Paper, 2015, 10(12): 1392-1397. (in Chinese)
[14] DEGEFU Y, LOHTANDER K, PAULIN L. Expression patterns and phylogenetic analysis of two xylanase genes (htxyl1andhtxyl2) fromHelminthosporium turcicum, the cause of northern leaf blight of maize.Biochimie, 2004, 86(2): 83-90.
[15] 张鑫, 曹志艳, 刘士伟, 郭丽媛, 董金皋. 玉米大斑病菌聚酮体合成酶基因StPKS功能分析. 中国农业科学, 2011, 44(8): 1603-1609.
ZHANG X, CAO Z Y, LIU S W, GUO L Y, DONG J G. Functional analysis of polyketide synthase geneStPKSinSetosphaeria turcica.Scientia Agricultura Sinica, 2011, 44(8): 1603-1609. (in Chinese)
[16] 李乐, 许红亮, 杨兴露, 李雅轩, 胡英考. 大豆LEA基因家族全基因组鉴定、分类和表达. 中国农业科学, 2011, 44(19): 3945-3954.
LI L, XU H L, YANG X L, LI Y X, HU Y K. Genome-wide identification, classification and expression analysis of LEA gene family in soybean.Scientia Agricultura Sinica, 2011, 44(19): 3945-3954. (in Chinese)
[17] 王勇, 姚勤, 陈克平. 动物bHLH转录因子家族成员及其功能. 遗传, 2010, 32(4): 307-330.
WANG Y, YAO Q, CHEN K P. Progress of studies on family members and functions of animal bHLH transcription factors.Hereditas, 2010, 32(4): 307-330. (in Chinese)
[18] 王小龙, 李宏. 酵母螺旋酶(Y RF)基因家族的同源性分析. 内蒙古大学学报(自然科学版), 2003, 34(5): 530-535.
WANG X L, LI H. Homology analysis yeast helicase gene family.Acta Scientiarum Naturalium Univrsitatis NeiMongol, 2003, 34(5): 530-535. (in Chinese)
[19] 巩校东, 张晓玉, 田兰, 王星懿, 李坡, 张盼, 王玥, 范永山, 韩建民, 谷守芹, 董金皋. 玉米大斑病菌MAPK超家族的全基因组鉴定及途径模型建立. 中国农业科学, 2014, 47(9): 1715-1724.
GONG X D, ZHANG X Y, TIAN L, WANG X Y, LI P, ZHANG P, WANG Y, FAN Y S, HAN J M, GU S Q, DONG J G. Genome-wide identification MAPK superfamily and establishment of the model of MAPK cascade pathway inSetosphaeria turcica.Scientia Agricultura Sinica, 2014, 47(9): 1715-1724. (in Chinese)
[20] 吴迪, 赵梓彤, 李志勇, 杨峥, 檀迎会, 申珅, 曹志艳, 董金皋, 田菲菲, 郝志敏. 玉米大斑病菌钙/钙调素依赖性蛋白激酶(CaMK)基因家族的鉴定与表达模式分析. 农业生物技术学报, 2015, 23(8): 1020-1030.
WU D, ZHAO Z T, LI Z Y, YANG Z, TAN Y H, SHEN S, CAO Z Y, DONG J G, TIAN F F, HAO Z M. Identification and expression pattern analysis of calcium/calmodulin-dependent protein Kinases (CaMKs) gene family inSetosphaeria turcica. Journal of Agricultural Biotechnology, 2015, 23(8): 1020-1030. (in Chinese)
[21] GUBENKO I S. Drosophila genes that encode LIM-domain containing proteins: their organization, functions, and interactions.Tsitologiia I Genetika, 2006, 40(4): 44-67.
[22] JASINSKI S, KAUR H, TATTERSALL A, TSIANTIS M. Negative regulation of KNOX expression in tomato leaves.Planta, 2007, 226(5): 1255-1263.
[23] LIU W D, XIE S Y, ZHAO X H, CHEN X, ZHENG W H, LU G D, XU J R, WANG Z H. A homeobox gene is essential for conidiogenesis of the rice blast fungusMagnaporthe oryzae.Molecular Plant-Microbe Interactions, 2010, 23(4): 366-375.
[24] CASSELTON L A, OLESNICKY N S. Molecular genetics of mating recognition in basidiomycete fungi.Microbiology and MolecularBiology Reviews, 1998, 62(1): 55-70.
[25] BÜRGLIN T R, AFFOLTER M. Homeodomain proteins: an update.Chromosoma, 2016, 125(3): 497-521.
[26] GEHRING W J, HIROMI Y. Homeotic genes and the homeobox.Annual Review of Genetics, 1986, 20: 147-173.
[27] KIM S, PARK S Y, KIM K S, RHO H S, CHI M H, CHOI J, PARK J, KONG S, PARK J, GOH J, LEE Y H. Homeobox transcription factors are required for conidiation and appressorium development in the rice blast fungusMagnaporthe oryzae.PLoS Genetics, 2009, 5(12): e1000757.
[28] XIONG D G, WANG Y L, DENG C L, HU R W, TIAN C M. Phylogenic analysis revealed an expanded C2H2-homeobox subfamily and expression profiles of C2H2zinc finger gene family inVerticillium dahliae.Gene, 2015, 562(2): 169-179.
[29] ARNAISE S, ZICKLER D, POISIER C, DEBUCHY R. Pah1: a homeobox gene involved in hyphal morphology and microconidiogenesis in the filamentous ascomycetePodospora anserine.Molecular Microbiology, 2001, 39(1): 54-64.
(责任编辑 岳梅)
Identification of Homeobox Transcription Factor Family in Genome-Wide and Expression Pattern Analysis of the Members in Setosphaeria turcica
ZHAO Jie1, ZHAO LiQing1, GONG XiaoDong1, FENG ShengZe1, LIU XingChen1, ZHENG YaNan1, LI ZhiYong2, SUN HaiYue1, WANG DongXue1, HAN JianMin1, GU ShouQin1, DONG JinGao1
(1Mycotoxin and Molecular Plant Pathology Laboratory, Agricultural University of Hebei, Baoding 071001, Hebei;2Millet Research Institute, Hebei Academy of Agriculture and Forestry, Shijiazhuang 050000)
【Objective】 The objectives of this study are to identify Homeobox transcription factor family fromSetosphaeria turcicagenome, and to predict the locations of their corresponding coding proteins in the cells, gene structure and evolutionarycharacteristics, as well as to explore their expression patterns at different growth and development stages inS. turcica. 【Method】Homeobox transcription factor family was identified based onS. turcicagenome database and bioinformatics methods. Phylogenetic tree were created using the MEGA5.0 program. Gene structure characteristics were analyzed by GSDS software, which was a gene structure display server online (http://gsds1.cbi.pku.edu.cn/index.php). Amino acid sequence characteristics of the genes in Homeobox transcription factor family were compared using software Clustal X 1.83. The secondary structures of homeodomain proteins were predicted by employing software SOMPA. Three-dimensional structures of Homeobox transcription factor family were doped out using Software I-TASSER. Homeobox genes expression patterns were detected at different developmental stages based on quantitative real-time PCR (qRT-PCR) analysis. 【Result】 A total of 8 Homeobox genes were systematically identified fromS. turcicagenome and classified into 4 groups according to the gene structure and phylogenetic tree. Subcellular localization showed that all of the predicted proteins located in the cell nuclei. The family members contained conserved Homeobox domains and typical“helix-turn-helix” secondary structures. Different gene family members took on different expression levels at different development stages inS. turcica. The expression level ofStHTF1was significantly higher at mycelium, spore and appressorium formation stages than other stages.StHTF3,StHTF4had the highest expression level at spore formation stage than other stages inS.turcica, whileStHTF6was the highest at germ tube formation stage. Moreover,StHTF2,StHTF5,StHTF7andStHTF8appeared higher expression level at appressorium formation stage than other genes. 【Conclusion】 Homeobox transcription factor family inS. turcicacontains 8 genes which are classed into 4 categories and located in the nuclei. The proteins encoded by the genes contain conserved Homeobox domain and “helix-turn-helix” spatial structure. Homeobox gene family members inS. turcicaappeared diverse expression profiles which will lay the foundation for identifying the functions of the gene family.
Setosphaeria turcica; Homeobox transcription factor family; system evolution; gene expression
10.3864/j.issn.0578-1752.2017.04.007
2016-10-24;接受日期:2016-11-16
国家自然科学基金(31271997,31371897)、大学生创新创业训练计划(201610086052,20161805)
联系方式:赵洁,E-mail:295277949@qq.com。赵立卿,E-mail:370172469@qq.com。赵洁和赵立卿为同等贡献作者。通信作者谷守芹,E-mail:gushouqin@126.com。通信作者董金皋,E-mail:dongjingao@126.com
