2011—2016年浙江省猪流行性腹泻病毒S基因与ORF3基因遗传进化分析
2017-04-08吴旺霞王一成袁秀芳徐丽华李军星
吴旺霞,王一成,吴 润,袁秀芳,徐丽华,李军星,苏 菲
(1.甘肃农业大学 动物医学院,甘肃 兰州 730000; 2.浙江省农业科学院 畜牧兽医研究所,浙江 杭州 310021)
2011—2016年浙江省猪流行性腹泻病毒S基因与ORF3基因遗传进化分析
吴旺霞1,2,王一成2,*,吴 润1,袁秀芳2,徐丽华2,李军星2,苏 菲2
(1.甘肃农业大学 动物医学院,甘肃 兰州 730000; 2.浙江省农业科学院 畜牧兽医研究所,浙江 杭州 310021)
为调查近年来浙江省猪流行性腹泻病毒流行毒株与疫苗株的S基因和ORF3基因遗传变异情况,2011—2016年,从浙江省各地区采集疑似猪流行性腹泻病料并提取其RNA,用3对特异性引物对S基因和ORF3基因进行RT-PCR扩增、克隆及测序,并对其序列变化、遗传进化情况和抗原位点进行分析。结果表明:与国内疫苗株CV777相比,12个样品株的S基因存在15个核苷酸插入和6个核苷酸缺失,导致氨基酸序列存在5个氨基酸插入(58QGVN61和l40N)和2个氨基酸缺失(163DI164),且在主要突变区S1区的2个中和表位(499-638和764-771 aa)有8处氨基酸突变;样品株的ORF3基因有7处氨基酸变异。遗传进化分析结果显示,12株分离株的S基因与亚洲主要疫苗株(国内疫苗株CV777和韩国弱毒疫苗株DR13)的同源性仅为93.3%~94.9%,而与2011—2016年中国流行株(BJ-2011和HuN)的同源性高达97.8%~99.9%,表明近年来猪流行性腹泻病毒呈现较大程度的变异,可为研制更加有效的疫苗奠定基础。
猪流行性腹泻病毒;S基因;ORF3基因;序列特点;基因进化分析
猪流行性腹泻病(Porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(Porcineepidemicdiarrheavirus,PEDV)引起的一种急性肠道传染病,病发主要特征表现为腹泻、呕吐、脱水等,且哺乳仔猪具有高发病率和高死亡率。猪流行性腹泻病于1971年首次在英国发现。中国在20世纪80年代初以来相继有报道,2010年以来,随着新毒株的出现,该病在我国的流行情况更趋严重[1]。美国自2013年5月PED首次暴发以来,已迅速蔓延至30多个州,波及4 000多个养殖场,累计死亡仔猪超过700万头。日本、加拿大、墨西哥等国也相继暴发此病,欧洲多国也开始研究防止PEDV传入的策略[2-3]。
PEDV属于冠状病毒科冠状病毒属,病毒核酸为正义单链RNA,其主要的结构蛋白包括S纤突蛋白(Spike,180~220 ku)、E蛋白(Envelope,8.8 ku)、M膜蛋白(Membrane,27~32 ku)以及N核衣壳蛋白(Nucleocapsid,55~58 ku),其中,S蛋白是纤突糖蛋白,该蛋白由1 383个氨基酸组成,由4 149个碱基编码,预测有29个潜在的糖基化位点,这些糖基化位点可能具有重要的生物学意义[4]。基于其他冠状病毒S蛋白保守基因序列,将PEDV S蛋白人为地划分为S1区(1-789位氨基酸,抗原区)和S2区(790-1383位氨基酸,膜融合区)[5],S1是变异较大的区域,也是S蛋白的主要功能区域,S2较为保守。S蛋白介导病毒与细胞的吸附和融合,并能诱导宿主产生中和抗体。ORF3是PEDV唯一的辅助基因,位于S和E基因之间,编码含224个氨基酸的蛋白,该蛋白为一种跨膜蛋白,具有离子通道活性。有研究表明,ORF3与PEDV细胞适应性和毒力有关,ORF3基因的突变可以作为衡量PEDV毒力致弱的重要标志[6],因此,ORF3是PEDV分子流行病学研究的重要方向。
病原研究的初步结果表明,目前我国从本次流行的腹泻病猪中检测到的PEDV大多数为基因2型,与泰国、菲律宾、越南等地2009—2010年所流行的毒株非常相似。序列分析表明,2010年以前PEDV毒株S基因序列与英国标准株CV777同源性很高,达到98%,2010年后,S基因序列与PEDV韩国分离株同源,与CV777的同源性仅有85%左右,存在基因的插入和缺失,PEDVS基因序列的改变可能导致了其流行规律的变化。本实验通过对浙江省近6年的猪流行性腹泻病毒S基因和ORF3基因全序列进行比较分析,着重分析其与亚洲主要疫苗株以及近6年中国流行株的序列差异以及抗原位点变化,为更好地监测猪流行性腹泻并制定有效的防控措施打下基础。
1 材料与方法
1.1 病料样品
小肠及内容物采集于浙江省不同地区猪场具有严重腹泻、呕吐、脱水和死亡等疑似感染PEDV临床表现的仔猪。
1.2 酶及化学试剂
MN病毒RNA提取试剂盒购自上海拜力生物科技有限公司;反转录试剂盒、DNA平末端加A试剂盒、T-Vector pMD18、DL 2 000 DNA Marker购自宝生物工程(大连)有限公司;DNA琼脂糖凝胶回收试剂盒、质粒小提试剂盒购自天根生化科技(北京)有限公司;Pfu DNA polymerse购自生工生物工程(上海)有限公司;大肠埃希菌Trans 10感受态细胞购自北京全式金生物技术有限公司。
1.3 实验方法
1.3.1 引物设计与合成
根据GenBank中发表的PEDVS基因和ORF3基因序列,使用Primer 6.0软件,设计出3对特异性引物(表1),其中第3对引物扩增的片段包括ORF3基因。第1对(F1/R1)和第2对(F2/R2)引物拟扩增片段长度为2 000 bp左右,第3对(F3/R3)引物拟扩增长度为1 500 bp左右。
表1 PEDVS基因和ORF3基因扩增引物
Table 1 Amplification primers of PEDVSgene andORF3 gene

引物名称Primernames序列(5'-3')Sequence(5'-3')位置Position/nt片段长度Fragmentlength/bpF1GAAGAATGGTAAGTTGCTAGTGCGTA20559~227562197R1AGCTAACAACTGTCCAGAATCAGATGF2AAGGGTGAGTTGATTACTGGCACG22503~247412238R2CTAGTAATGACACAACAAAGATGAGAACF3ACCAGATGTAATCCCAGATTACATCG24347~259421595R3GTATCCATAGAATAGCCATCTTGACAC
1.3.2 PEDV的RNA提取
取粪便或肠液,按1∶5用PBS稀释后研磨,反复冻融3次,8 000g离心10 min取上清,用MN Viral RNA isolation Kit按使用说明书提取病毒RNA,置于-70 ℃保存备用。
1.3.3S基因各片段的扩增和鉴定
按照PrimeScriptTM1st strand cDNA Synthesis kit使用说明书,以RNA为模板,Random 6 mers为引物,反转录为cDNA。PCR扩增体系(25 μL):10 × Pfu buffer 2.5 μL,dNTPs(2.5 mmoL·L-1)2 μL,反转录产物(cDNA)2 μL,上下游引物(10 μmoL·L-1)各1μL,Pfu polymerse 0.5 μL,灭菌水16 μL。反应条件为:95 ℃ 5 min;98 ℃ 10 s,56 ℃ 15 s,72 ℃ 4 min 15 s(第1对和第2对引物扩增)和3 min 30 s(第3对引物扩增),35个循环;72 ℃ 10 min。用1.5%的琼脂糖凝胶电泳对PCR产物进行鉴定。
1.3.4S基因各片段的克隆和测序
使用DNA A-Tailing Kit给PCR产物碱基序列平末端加A,将回收纯化后的PCR产物连接到pMD-18T载体后,转化至Trans 10感受态细胞。利用蓝白斑筛选,挑取白色菌落进行菌液PCR验证,每个克隆挑取3个鉴定阳性的菌液委托杭州擎科梓熙生物技术有限公司进行碱基序列测定。
1.3.5S基因和ORF3基因的测序与比对分析
利用DNAStar软件中的EditSeq程序拼接所有序列;用MegAlign软件比较分析样品毒株与参考毒株(表2)S基因和ORF3基因的核苷酸序列,用DNAStar中的Protean软件比较分析样品毒株与参考毒株氨基酸的抗原性。
2 结果与分析
表2 用于序列比对及遗传进化分析的PEDV参考毒株
Table 2 PEDV strains for sequence comparison and analysis of genetic evolution

序列编号SequenceNo.毒株Strains国家,年份Countriesandyears序列登录号AccessionNo.1CV777Belgium,1977AF3535112Br1/87Britain,1987Z254833DR13Korea,2006DQ4624044Spk1Korea,2002AF5002155KNU-0801Korea,2008GU1801426Chinju99Korea,1999AY1675857LZCChina,2006EF1859928BJ-2011China,2011JN8257129HuNChina,2011JQ462404
2.1 RT-PCR
以病料样品提取的RNA为模板,反转录得到cDNA,分别用3对引物对PEDV的S基因进行PCR扩增,经过琼脂糖凝胶电泳分析,可见到大小分别为2 000和1 500 bp的特异性扩增片段,与预期结果相符(图1)。
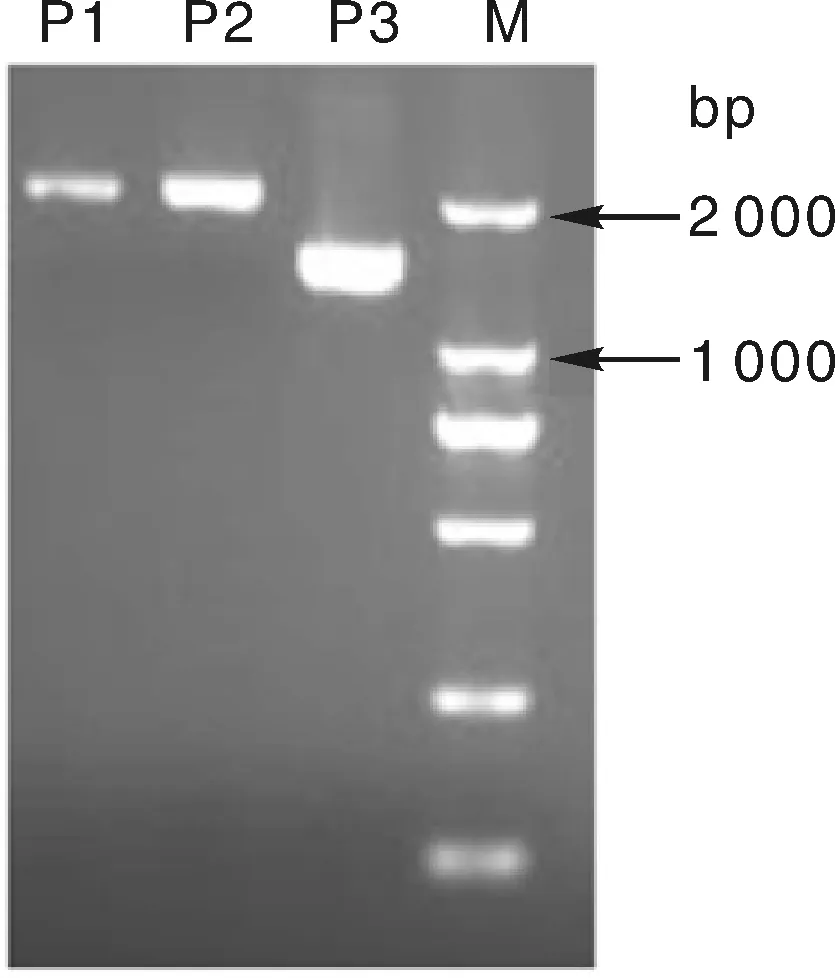
P1、P2、P3分别为第1、2、3对引物扩增PEDV S基因获得的目的条带;M,DL 2 000 DNA markerP1、P2 and P3 were the target bands of S gene amplified using 1st, 2nd, 3rd pairs of primers, respectively; M, DL 2 000 DNA marker图1 PCR扩增S基因各片段电泳结果Fig.1 Electrophoresis results of different regions of S gene amplified by PCR
2.2 pMD-18T-S阳性克隆的鉴定
如图2所示,3个片段的重组质粒均扩增出目的条带,而且目的片段大小正确,测序结果也表明片段大小分别为2 197、2 238和1 595 bp,证明S基因3个片段已成功与pMD-18T载体连接。
2.3 PEDV-S基因的同源性和遗传变异
将3段测序结果拼接出完整的PEDVS基因序列,根据样品采集时间与地点将12个样品株进行命名(表3)。结果表明,2011—2016年浙江省PEDV毒株S基因由4 161个碱基组成,均编码1 383个氨基酸。
本研究分离的2011—2016年12个样品株之间S基因的氨基酸序列同源性为97.7%~100%;12个样品株与Genbank中公布的2011—2016年中国流行株(BJ-2011和HuN)S基因的氨基酸序列同源性高达97.8%~99.9%,而与亚洲主要疫苗株(中国疫苗株CV777和韩国弱毒疫苗株DR13)的氨基酸同源性仅为93.3%~94.9%。
与CV777S基因全长(4 152 bp)相比,12个样品株的N端存在15个碱基的插入和6个碱基的缺失,分别为164~166位插入TTG,177~185位插入GGGTGTCAA,417~419位插入TAA,480~485位缺失TGGAAA。该插入和缺失导致样品株S基因的氨基酸序列中存在5个氨基酸的插入(58QGVN61和l40N)、2个氨基酸的缺失(163DI164)(图3)。
PEDVS基因编码的蛋白被人工划分为S1区(第1~789位氨基酸)和S2区(第790~1 383位氨基酸)[7]。目前,在S1区鉴定出3个诱导中和抗体产生的表位,分别为499~638[8]、748~755和764~771 aa[9]。与CV777相比,样品株的S蛋白氨基酸突变位点大多数位于S1区域,S2区域相对比较保守。样品株的信号肽区域(1~20 aa)发生了4个氨基酸位点的突变,分别为第2位(R→K)、第5位(I→T)、第10位(L→H)和第15位(P→S)。样品株的中和抗原表位COE基因在499~638 aa区域发生了8个氨基酸突变,分别是第522位(A→S)、526位(L→H)、528位(S→G)、532位(V→I)、554位(T→S)、599位(G→S)、610位(A→E)、617位(L→F)氨基酸;在748~755 aa区域没有氨基酸突变;在764~771 aa区域中的第771位氨基酸发生突变(Y→S)。另外,与CV777相比,样品株在S1区还存在较大变异,分别为第27~29位(QST→PAN),第55~57位(SMN→IGE),第68~72位(GTGIE→AGQHP),第86~87位(DS→RG),第130~131位(DN→SI),第159~162位(RDGK→SEHS),第201~203位(RRS→SGG),第247~249位(DS→EP)。除此之外,还存在大量的点突变,包括第82位(L→V)、84位(Y→H)、89位(Q→H)、120位(I→T)、138位(V→A)、157位(Y→H)、179位(A→S)、184位(H→Y)、187位(L→F)、197位(R→K)、211位(T→E)、228位(Y→S)、230位(E→Q)、303位(H→Q)、305位(M→I)、347位(L→F)、361位(I→T)、371位(E→V)、459位(S→A)、640位(I→V)、672位(I→F)、712位(N→D)、724位(N→S)、729位(N→S)。
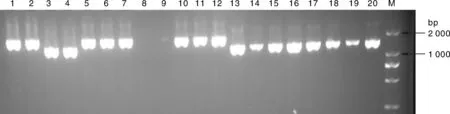
1-6,S1片段(2 197 bp);7-12,S2片段(2 238 bp);13-20,S3片段(1 595 bp);其中3、4、8、9为阴性。M,DL 2 000 DNA Marker1-6, S1 fragment (2 197 bp); 7-12, S2 fragment (2 238 bp); 13-20, S3 fragment (1 595 bp); 3, 4, 8, 9 were negtive. M, DL 2 000 DNA marker图2 阳性克隆验证Fig.2 Verification of the positive clones
表3 十二份PEDV阳性病料的来源及名称
Table 3 The sources and names of 12 PEDV positive strains
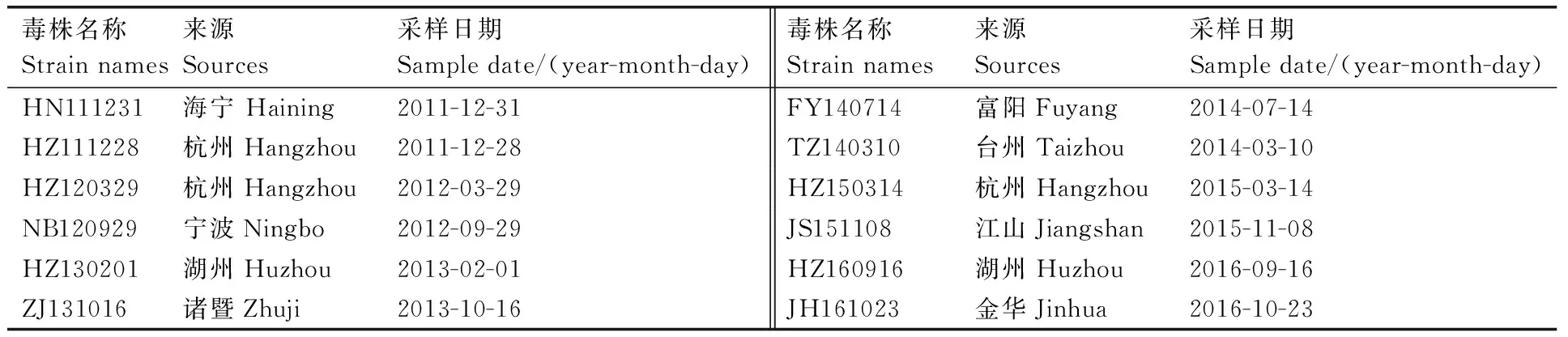
毒株名称Strainnames来源Sources采样日期Sampledate/(year-month-day)毒株名称Strainnames来源Sources采样日期Sampledate/(year-month-day)HN111231海宁Haining2011-12-31FY140714富阳Fuyang2014-07-14HZ111228杭州Hangzhou2011-12-28TZ140310台州Taizhou2014-03-10HZ120329杭州Hangzhou2012-03-29HZ150314杭州Hangzhou2015-03-14NB120929宁波Ningbo2012-09-29JS151108江山Jiangshan2015-11-08HZ130201湖州Huzhou2013-02-01HZ160916湖州Huzhou2016-09-16ZJ131016诸暨Zhuji2013-10-16JH161023金华Jinhua2016-10-23
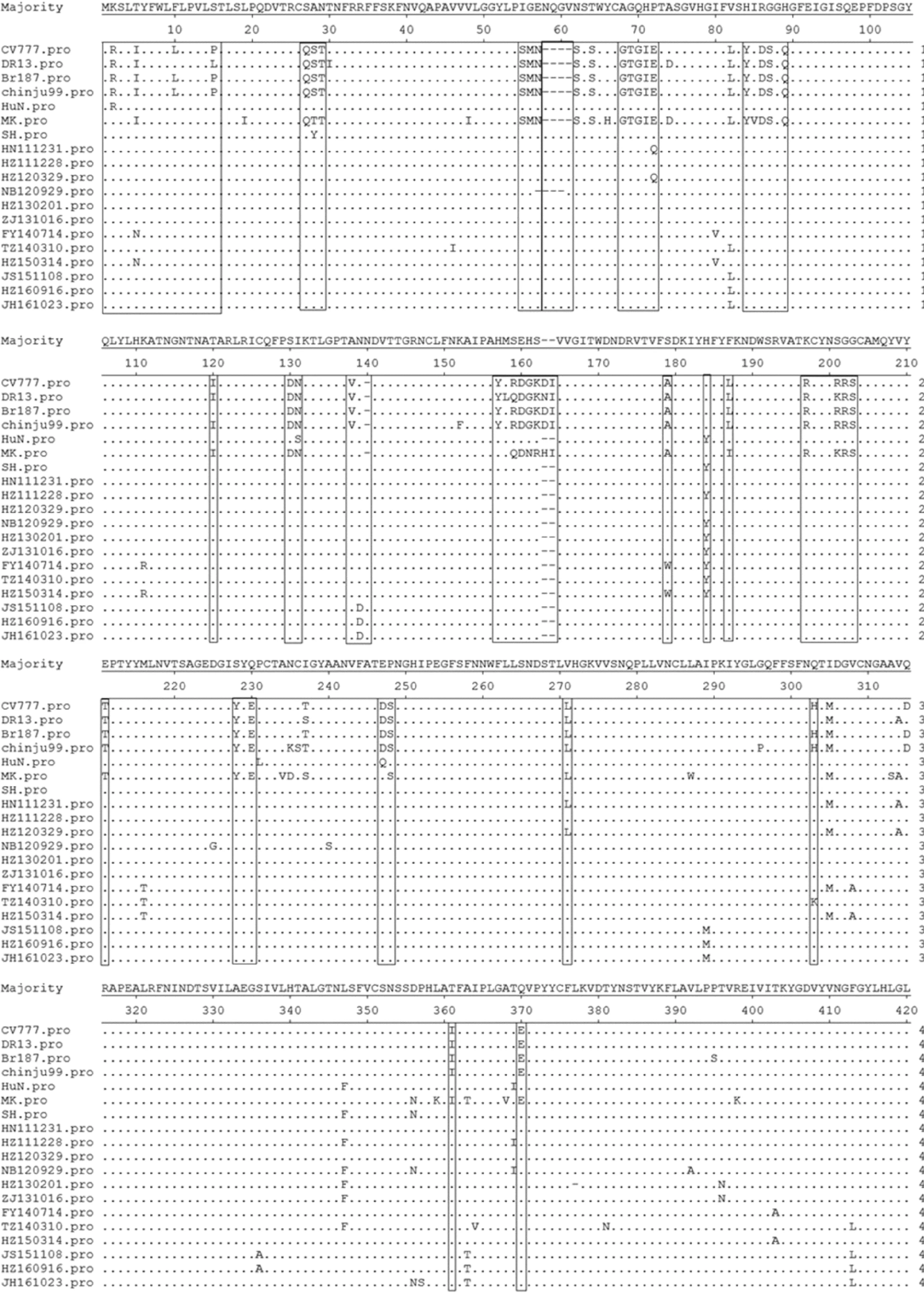
图3 2011—2016年分离毒株与参考毒株S1蛋白(1-789 aa)氨基酸序列比对Fig.3 Analysis of S1 protein (1-789 aa) amino acid sequences of the separated and referenced PEDV strains during 2011 to 2016
2.4 S蛋白的遗传进化
根据PEDV S蛋白的遗传变异分析结果,一般将PEDV分为3个群[10](图4)。本研究中的12个样品株都属于第1个群,同属该群的还有中国其他地区的流行毒株(BJ-2011和HuN);而年代较早的分离株,包括欧洲毒株(CV777和Brl-87)、韩国毒株(DR13和chinju99)和中国LZC株同属于第2个群;此外,韩国2002年分离的Spk1株和2008年分离的KNU-0801株同属于第3个群。
2.5 S蛋白抗原位点变化
12个样品株可以分为3类,选取具有代表性的2株TZ140310和JH161023,用DNAStar Protean程序,根据抗原指数(antigenic index)对疫苗株CV777、参考毒株KNU-0801、BJ-2011和选取的2个代表毒株S蛋白的抗原表位区进行分析。结果显示,抗原位点的改变主要集中在S1区。选取变异较大的1~200位氨基酸序列进行抗原表位区比较,如图5所示,与CV777相比,参考毒株和样本株S蛋白1~200位氨基酸序列的抗原表位明显不同(图5中箭头所示),表明流行毒株与疫苗株在抗原性上可能存在差异。
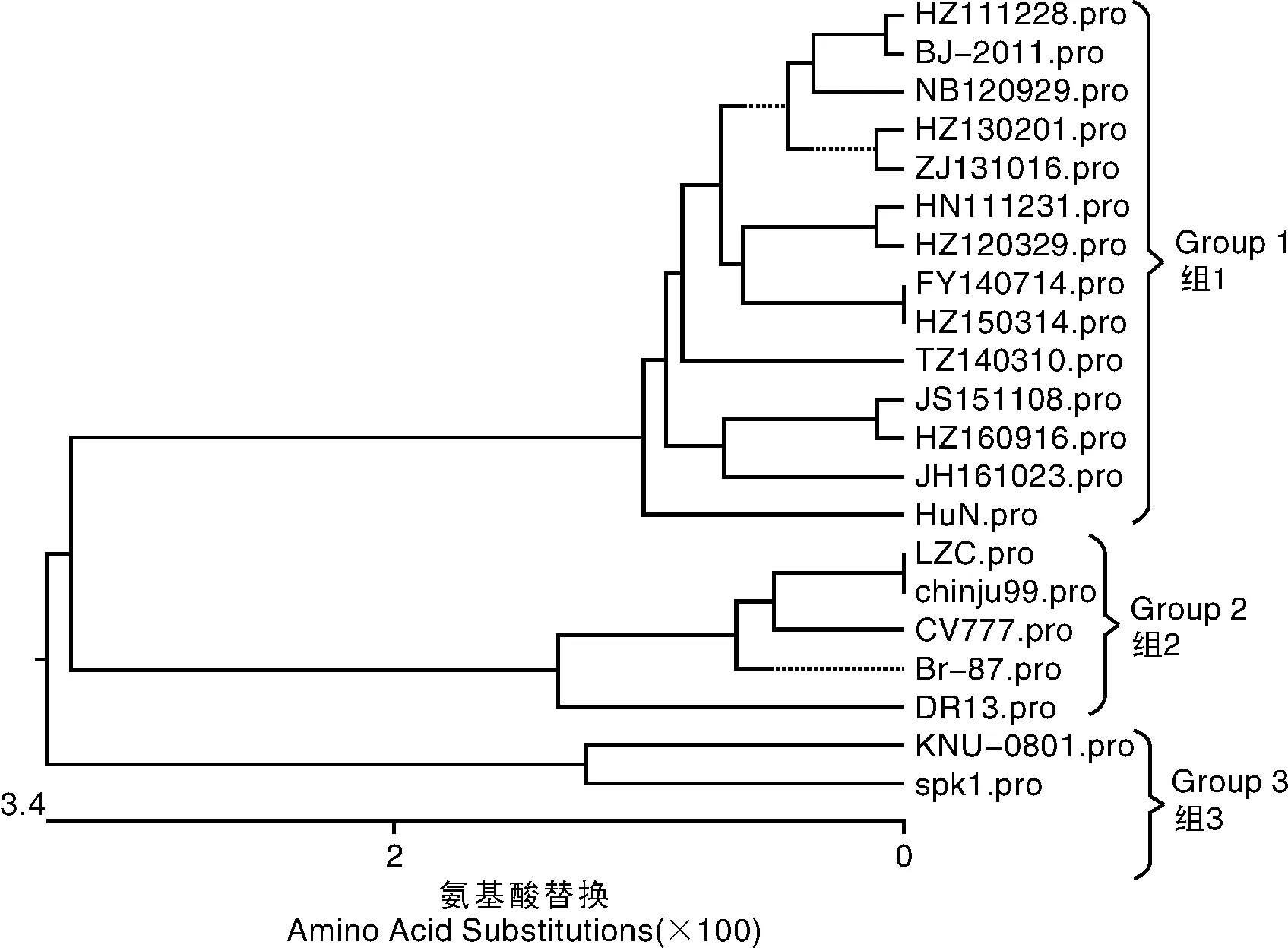
图4 PEDV S蛋白氨基酸序列的进化树Fig.4 Phylogenetic tree of amino acid sequences of PEDV S protein
2.6ORF3基因氨基酸序列同源性及变异
2011—2016年浙江省PEDV毒株ORF3基因全长675 bp,均编码225个氨基酸。将分离的12株样品株的ORF3基因编码的氨基酸序列与参考毒株进行同源性比对(图6)。结果表明:12株样品株ORF3基因彼此之间的氨基酸序列同源性为96.9%~100%,其与疫苗株CV777的同源性为95.6%~96.4%,与中国流行株BJ-2011的同源性为97.8%~99.6%,与韩国毒株chinju99和DR13的同源性为96.9%~99.6%。
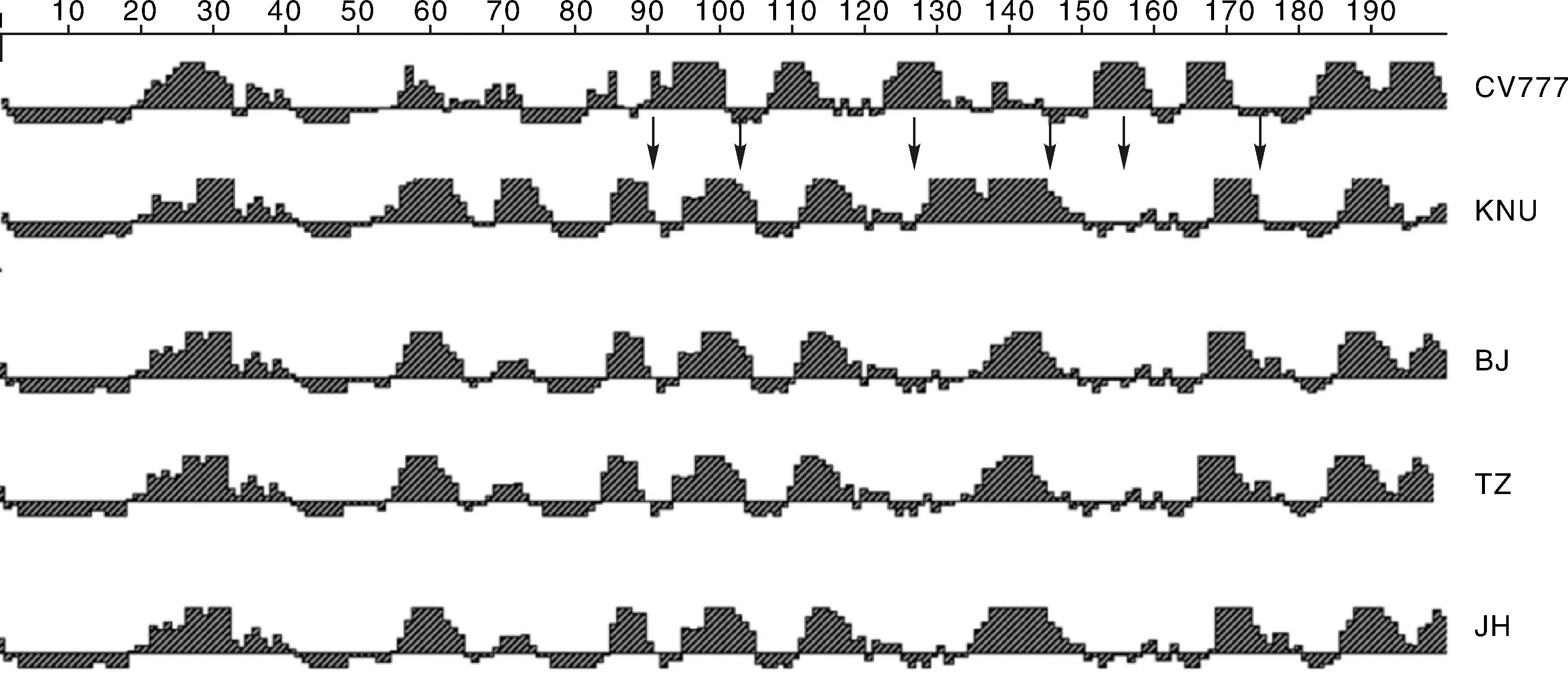
箭头所示为抗原指数不同区域Differences in antigen index were shown as arrows图5 S蛋白(1~200 aa)抗原指数预测Fig.5 Prediction of antigenic index of S protein(1~200 aa)
利用DNAStar中的MegAlign软件,对12个样品株的ORF3基因编码的氨基酸序列与参考毒株进行了比对分析。结果表明,与氨基酸序列同源性相对比较低的疫苗株CV777相比,分离的12株样品株ORF3序列存在7处氨基酸变异,包括第21位(V→A)、54位(V→I)、79位(V→I)、92位(L→F)、101位(A→T)、166位(N→S)、182(H→Q),还存在部分点突变。
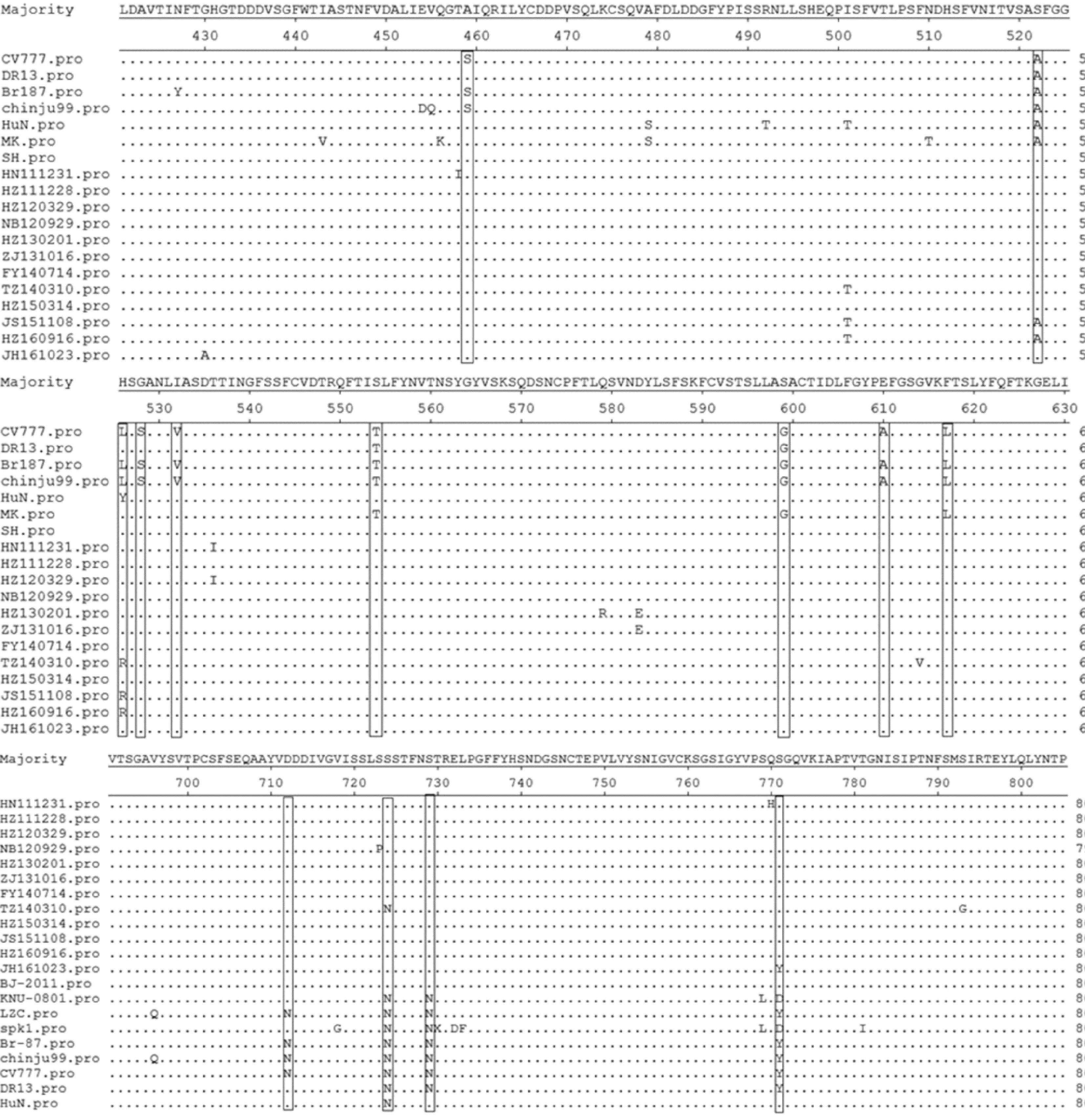
图6 ORF3基因编码的氨基酸序列变异位点分析Fig.6 Analysis of the mutation sites in the amino acid coded by ORF3 gene
3 讨论
PED是近年来危害养猪业最重要的疾病之一,严重影响了养猪业的健康发展。自2010年下半年以来,猪流行性腹泻病在中国出现新一轮的暴发流行。最早发生在广西,然后湖南、华北、华东、山东、河北等地普遍发病,呈现全国性大流行,造成哺乳仔猪的大量死亡。根据杜晓莉等[11]2010—2013年仔猪腹泻临床检测结果分析,浙江省总体PEDV检出率为69.30%,并呈现逐年上升趋势,这说明浙江省仔猪腹泻的主要病原是PEDV,因此,有必要对PEDV毒株的遗传变异情况进行调查研究。
PEDV为单股正链RNA病毒,由于RNA酶缺乏校正功能,使得RNA病毒很容易发生变异,这可能是导致PED再次爆发的主要原因。郑逢梅等[12]报道,疫苗免疫或母源抗体并不足以保证猪免受现有PEDV流行毒株的感染,原因可能有2个:一是流行毒株易发生变异,二是流行毒株毒力增强。2011年,刘孝珍等[13]通过分析PEDV的S基因序列,发现了PEDV新变异株。甘振磊等[14]研究表明,PEDV毒株只有1个血清型,目前还没有迹象表明存在不同的PEDV血清型。但作为RNA的冠状病毒,在不同的猪群环境、免疫压力和药物作用条件下极有可能发生变异。
S基因是PEDV的主要抗原决定基因,其基因结构和功能与病毒的感染性、致病性、抗体的产生和细胞结合能力密切相关。本研究中2011—2016年PEDV样品株S基因与疫苗株CV777相比,同源性较低,而且存在碱基插入、缺失和突变,而这些插入、缺失和突变最终导致样品株中出现5个氨基酸的插入(58QGVN61和140N)和2个氨基酸的缺失(163DI164);与蔡青秀等[15]的研究结果相比,变异位点增加,出现4处氨基酸位点的突变,在中和抗原表位区域,有8处氨基酸位点的突变,在其非功能区域同样存在大量的变异。S基因的遗传变异分析结果表明,浙江地区获得的阳性PEDV毒株属于同一分支,与2011年中国BJ株和HuN株亲缘关系较为密切,但它们均与1999年韩国株Chinju99、2002年韩国株Spk1、2006年中国株LZC及欧洲株(CV777和Brl/87)亲缘关系较远,这与蔡青秀等[15]的分类结果一致。抗原位点变化分析结果表明,流行毒株与疫苗株的抗原性也存在较大差异,这也可能是导致免疫失败的一个重要原因。
样品株与近年来中国其他地方流行株的同源性较高,表明S基因无明显的地域性分布,但是氨基酸的改变导致蛋白质结构和功能的改变,是否会引起流行株感染能力以及流行趋势的变化,有待进一步研究。样品株和疫苗株CV777的ORF3基因序列,虽然相对于S基因来说比较保守,但是也存在7处氨基酸变异,并且不同的流行株之间存在差异。流行毒株之间的差异说明变异的多样性,PEDV流行毒株ORF3的差异对其毒力的影响还有待深入的研究。
综上,浙江省近6年分离的样品株与中国近几年其他地区分离的毒株关系密切,推测中国现流行的PEDV可能具有相同的毒株来源;但是与疫苗株相比,在S和ORF3基因上出现了碱基的缺失、插入和突变,由此导致相关毒力及免疫原性出现差异,这很可能是传统疫苗保护效力降低的原因。因此,有必要找出不同毒株S基因的抗原位点差异性,研制出更加有效的保护机制。
[1] CHEN J F, SUN D B, WANG C B, et al. Molecular characterization and phylogenetic analysis of membrane protein genes ofporcineepidemicdiarrheavirusisolates in China[J].VirusGenes, 2008, 36(2): 355-364.
[2] JUNG K, WANG Q, SCHEUER K A, et al. Pathology of USporcineepidemicdiarrheavirusstrain PC21A in gnotobiotic pigs[J].EmergingInfectiousDiseases,2014, 20(4):668-671.
[3] WANG L, BYRUM B, ZHANG Y. New variant ofporcineepidemicdiarrheavirus, United States, 2014[J].EmergingInfectiousDiseases, 2014, 20(5): 917-919.
[4] 张强敏, 郭福生, 尹燕博, 等. 猪流行性腹泻病毒分子生物学特征[J]. 中国病毒学, 2002,17(4):381-384. ZHANG Q M, GUO F S, YIN Y B, et al. Molecular biological traits ofPorcineepidemicdiarrheavirus[J].VirologicaSinica, 2002, 17(4): 381-384. (in Chinese)
[5] SUN D B, FENG L, SHI H Y, et al. Spike protein region (aa 636789) ofporcineepidemicdiarrheavirusis essential for induction of neutralizing antibodies[J].ActaVirologica, 2007, 51(3): 149-156.
[6] 陈如敬, 吴学敏, 车勇良, 等. 猪流行性腹泻病毒FJ-11A株的分离与ORF3基因序列分析[J]. 福建农业学报, 2011, 26(6): 947-951. CHEN R J, WU X M, CHE Y L, et al. Isolation ofPorcineepidemicdiarrheavirusstrain FJ-11A in Fujian Province and sequence analysis of theORF3 gene[J].JournalofFujianAgriculturalSciences, 2011, 26(6): 947-951. (in Chinese with English abstract)
[7] NGUYEN V D, JUNZO N, MASUO S, et al. US-like isolates ofporcineepidemicdiarrheavirusfrom Japanese outbreaks between 2013 and 2014[J].SpringerPlus, 2015, 4(1): 1-10.
[8] CHANG S H, BAE J L, KANG T J, et al. Identification of the epitope region capable of inducing neutralizing antibodies against theporcineepidemicdiarrheavirus[J].MoleculesandCells, 2002, 14(2): 295.
[9] SUN D B, FENG L, SHI H Y, et a1. Identification of two novel B cell epitopes onporcineepidemicdiarrheavirusspike protein[J].VetMicrobiolVeterinaryMicrobiology, 2008, 131(1/2): 73-81.
[10] 孙东波, 冯力, 时洪艳,等. 猪流行性腹泻病毒分子生物学研究进展[J]. 动物医学进展, 2006, 27(10): 11-14. SUN D B, FENG L, SHI H Y, et a1. Progress on molecular biology ofPorcineepidemicdiarrheavirus[J].ProgressinVeterinaryMedicine, 2006, 27(10): 11-14. (in Chinese)
[11] 杜晓莉, 王一成, 吴润, 等. 2010—2013年浙江省猪流行性腹泻病毒临床检测及PEDV-S基因型分析[J]. 浙江农业学报, 2014, 26(3): 581-587. DU X L.WANG Y C, WU R, et al. Genotyping and clinical detection ofporcineepidemicdiarrheavirusduring 2010-2013 in Zhejiang Province[J].ActaAgriculturaeZhejiangensis, 2014, 26(3): 581-587. (in Chinese with English abstract)
[12] 郑逢梅, 霍金耀, 赵军, 等. 2010-2012年华中地区猪流行性腹泻病毒分子特征和遗传进化分析[J]. 病毒学报, 2013 (2): 197-205.
ZHENG F M, HUO J Y, ZHAO J, et a1. Molecular characterization and phylogenetic analysis ofporcineepidemicdiarrheavirusfield strains in central China during 2010-2012 outbreaks[J].ChineseJournalofVirology, 2013, 29(2): 197-205. (in Chinese with English abstract)
[13] 刘孝珍, 陈建飞, 时洪艳, 等. 2011年猪流行性腹泻病毒的遗传变异分析[J]. 中国预防兽医学报, 2012, 34(3): 180-183. LIU X Z, CHEN J F, SHI H Y, et a1. Genetic variation analysis ofporcineepidemicdiarrheavirusisolated in 2011[J].ChineseJournalofPreventiveVeterinaryMedicine, 2012,34 (3):180-183. (in Chinese with English abstract)
[14] 甘振磊, 汤德元, 李春燕, 等. 猪流行性腹泻流行特点及流行现状的研究[J]. 猪业科学, 2010, 27(12): 24-28. GAN Z L, TANG D Y, LI C Y, et al. Research of PEDV trendy characteristics and epidemic status[J].SwineIndustryScience, 2010, 27(12):24-28. (in Chinese)
[15] 蔡青秀, 陈晓春, 吴华伟, 等. 4株猪流行性腹泻病毒S蛋白序列特征和遗传进化分析[J]. 中国兽药杂志, 2015, 49(7): 1-7. CAI Q X, CHEN X C, WU H W, et a1. Sequence characteristics and genetic analysis of S protein of 4 porcine epidemic diarrhea strains[J].ChineseJournalofVeterinaryDrug, 2015, 49(7): 1-9. (in Chinese with English abstract)
(责任编辑 侯春晓)
Genetic analysis ofSandORF3 gene inPorciheepidemicdiarrheavirusduring 2011—2016 in Zhejiang Province
WU Wangxia1,2, WANG Yicheng2,*, WU Run1, YUAN Xiufang2, XU Lihua2, LI Junxing2, SU Fei2
(1.CollegeofVeterinaryMedicine,GansuAgriculturalUniversity,Lanzhou730070,China; 2.InstituteofAnimalHusbandryandVeterinaryScience,ZhejiangAcademyofAgriculturalSciences,Hangzhou310021,China)
In order to investigate recent genetic variations ofSandORF3 genes inPorcineepidemicdiarrheavirus(PEDV) epidemic and vaccine strains in Zhejiang Province, virus RNA was extracted from suspected PEDV samples which were collected from various areas in Zhejiang Province since 2011 to 2016, then RT-PCR, cloning and sequencing onSandORF3 genes were performed using three pairs of specific primers to analyze the sequence variation, genetic evolution and antigen sites. The results showed that as compared to vaccine strain CV777, 15 nucleotides were inserted and 6 nucleotides were absent in theSgenes of 12 epidemic strains, leading to the insertion of 5 amino acids (58QGVN61and140N) and deletion of 2 amino acids (163DI164) relatively. Moreover, in epidemic strains, totally 8 amino acid mutations were found in two neutralizing epitopes (499-638 and 764-771 aa) on the major mutation region S1; 7 amino acids were variated inORF3 gene. According to the analysis results of genetic evolution, these 12 epidemic strains shared 97.8%-99.9% homology with China epidemic strains BJ-2011 and HuN inSgene, but only 93.3%-94.9% homology with vaccine strains (CV777 and Korean attenuated strain DR13), indicating that huge variations and fast evolution had emerged in PEDV recently. This study could lay a foundation for development of effective vaccines and therapies.
Porcineepidemicdiarrheavirus;Sgene;ORF3 gene; sequence characteristics; genetic evolution analysis
10.3969/j.issn.1004-1524.2017.03.05
2016-08-17
公益技术研究农业项目(2015C32028,2016C32072)
吴旺霞(1991—),女,甘肃白银人,硕士研究生,研究方向为兽医微生物与免疫学。E-mail: 1174478344@qq.com
*通信作者,王一成,E-mail: 95711@sina.com
S858.28
A
1004-1524(2017)03-0380-09
浙江农业学报ActaAgriculturaeZhejiangensis, 2017,29(3): 380-388
http://www.zjnyxb.cn
吴旺霞, 王一成, 吴润, 等. 2011—2016年浙江省猪流行性腹泻病毒S基因与ORF3基因遗传进化分析[J]. 浙江农业学报, 2017, 29(3): 380-388.
