肠道细菌对厚壳贻贝稚贝附着的作用研究
2017-04-03彭莉华沈和定杨金龙
杨 娜, 梁 箫,, 彭莉华, 沈和定,, 杨金龙,
(1. 上海海洋大学 海洋生物科学国际联合研究中心, 上海 201306; 2. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室, 上海 201306; 3. 上海海洋大学 水产科学国家级实验教学示范中心, 上海 201306)
厚壳贻贝(Mytilus coruscus), 隶属于软体动物门(Mollusca), 瓣鳃纲(Lamellibranchia), 贻贝目(Mytilodia), 贻贝科(Mytilidae), 是我国沿海人工养殖的重要水产经济贝类[1]。与其他许多水产无脊椎动物类似, 厚壳贻贝幼虫经过浮游阶段后, 浮游末期的成熟眼点幼虫会经历一个附着的过程, 变态成为稚贝[2]。在稚贝生长为成贝过程中, 当外界生活条件不利时,稚贝就会放弃原足丝, 重新选择合适的附着基再分泌新足丝进行附着[3]。海洋环境中, 一旦把表面洁净的物体放入水体中, 海洋细菌将会很快附着在其表面进行生长繁殖, 并随后与硅藻、真菌、原生动物及无机颗粒和有机碎屑等共同形成一层微生物被膜[4-5]。研究表明微生物被膜可以不同水平地诱导或抑制水产无脊椎动物的附着过程[6-7]。近些年来, 厚壳贻贝的野生资源日益匮乏, 大规模养殖技术尚未完善, 苗种供不应求[8]。研究厚壳贻贝幼虫和稚贝附着及其生长发育则显得尤为重要。
动物体内栖息着数量大、种类多的细菌群落, 而以肠道内的细菌群落最多, 且与宿主互相依赖、彼此制约, 因此会在宿主肠道内形成一种稳定的微生态系统[9]。宿主与细菌的关系大致有3种: 致病、互利共生和同食共生[10], 通常状况下, 宿主与肠道细菌是互利共生的。研究表明肠道细菌在宿主的生长发育、免疫和营养代谢的过程中发挥重要作用。Sommer和Backhed[11]研究发现, 与正常小鼠相比, 在无菌小鼠肠道中, 肠绒毛长而细, 没有复杂的血管网, 肠隐窝较浅, 增生干细胞略少且黏液厚度较薄。在研究斑马鱼无菌肠道的发育时期时, 对比斑马鱼正常的肠道, Bates等[12]发现斑马鱼无菌肠道的上皮细胞的分化受到阻碍。Zokaeifar等[13]研究表明, 凡纳滨对虾(Litopenaeus vannamei)的肠道细菌, 如枯草芽孢杆菌(Bacillus subtilis)能够分泌某些胞外消化酶从而提高虾体肠道内的消化酶活性。另有研究阐明, 肠道细菌的种类和数量随着季节的变化而发生变化, Al-Harbi和Uddin[14]测定了夏、秋、冬3个季节罗非鱼和奥尼罗非鱼(Oreochromis niloticus × Oreochromis aureus)肠道中细菌的数目和种类, 发现不同季节的肠道细菌具有很大的差异。肠道同样作为厚壳贻贝重要的消化吸收器官之一, 肠道细菌形成的微生物被膜是否会促进厚壳贻贝稚贝的附着, 肠道中的细菌与厚壳贻贝的生长发育是否有关等问题均尚未有研究明确报道。
本实验通过从厚壳贻贝的肠道中分离并鉴定肠道细菌, 探究肠道细菌对其稚贝附着的作用, 确定肠道细菌的系统发育地位, 了解其遗传距离和细菌种属与稚贝诱导活性之间的关系, 为进一步研究肠道细菌与各个发育阶段的厚壳贻贝幼虫和稚贝之间的相互作用奠定基础, 也对人工养殖(例如稚贝中间培育)具有重要的理论指导意义。
1 材料与方法
1.1 实验材料
实验所用的野生厚壳贻贝以及其稚贝均来源于浙江省嵊泗县枸杞岛。其中稚贝壳长为(1.80 ±0.05)mm, 壳高为(1.17 ± 0.03)mm。在实验室恒温培养箱18℃条件下充气且暂养1周后进行正式实验。每天按时换水, 并投喂湛江等鞭金藻(Isochrysis zhanjiangensis)。成体厚壳贻贝壳长为(8.5 ± 0.3)cm,壳高为(4.4 ± 0.4)cm, 在实验室条件下进行暂养, 养殖期间不进行饵料投喂, 以便排空肠道, 充气培养48 h后用于正式实验。
1.2 肠道细菌的分离鉴定与系统发育分析
1.2.1肠道细菌的分离
用无菌铲刀将厚壳贻贝成体解剖, 并用无菌剪刀和镊子分离出完整肠道(整个解剖操作在冰上进行); 随后将整个肠道放入盛有50 mL灭菌的过滤海水(autoclaved filtered sea water, AFSW)的烧杯中, 用无菌剪刀将其剪碎并用无菌玻璃棒搅拌, 制成悬浮液, 涂布在 ZoBell 2216E固体培养基平板上。在黑暗、25℃条件下, 倒置培养48 h。然后在固体平板上用接种环挑取菌落进行分离, 并最终获得单株的肠道细菌, 再加入 15%的丙三醇(甘油)溶液, 放于–80℃冰箱保存。OLYMPUS相机(DIGITAL CAMERA MODEL NO.E-620)用于拍摄记录每种肠道菌株的表型。
1.2.2肠道细菌16S rDNA基因的测序
按照DNA提取试剂盒上的标准流程提取单株肠道细菌的DNA。后续PCR扩增的反应体系、程序和引物均参考杨金龙等[3]。用 1.0%的琼脂糖凝胶电泳(agarose gel electrophoresis)检测PCR产物。将PCR产物送公司进行16S rDNA基因测序。将基因序列上传至NCBI数据库, 获得细菌菌名、上传序列号等基本信息。
1.2.3比对序列和系统发育分析
得到的细菌测序序列与其近源物种的 16S rDNA基因序列使用MEGA 6.06程序进行比对。系统发育分析分别基于最大简约法(MP)、最小进化法(ME)和邻接法(NJ)进行1 000次重复构建, 物种间遗传距离使用Jukes-Cantor法。构建细菌的系统发育树, 选取大肠杆菌(Escherichia coli)(序列号AONF01000005.1)作为外群序列。
1.3 微生物被膜的形成与特性分析
1.3.1微生物被膜形成
挑取分离纯化得到的单株肠道细菌到80 mL液体的ZoBell 2216E培养基中, 黑暗条件下25℃培养24 h之后, 在3 500 r/min的条件下离心15 min, 倒掉培养液, 再用 AFSW 清洗 3次, 最后加入 50 mL AFSW均匀混合至悬浮液。在无菌培养皿(经160, 2 h℃
灭菌)中先放入无菌载玻片, 再加入一定量的菌体悬浮液, 最后加入适量的AFSW使其定容至20 mL, 从而使每株肠道细菌的初始密度分别是1×109、1×1010、1×1011和 5×1011个/L。每株细菌 4个不同的初始密度分别设置 12个平行, 空白对照组设置 9个平行,将其全部置于黑暗、18℃条件下的恒温培养箱中培养48 h, 最终形成微生物被膜。
1.3.2微生物被膜的密度计数
将培养48 h的微生物被膜在5%甲醛中固定24 h,置于 0.1%吖啶橙染液中 5 min, 再放于荧光显微镜(奥林巴斯BX51)×1 000倍下进行观察, 同时随机计数10个视野, 每株细菌的4个密度梯度分别设置3个平行, 从而确定4个初始细菌密度下微生物被膜的最终密度, 其计算公式参照杨金龙等[3]。
1.3.3微生物被膜形态及厚度分析
微生物被膜经5%甲醛溶液固定24 h, 然后在黑暗条件下, 用5 mg/L的碘化丙啶(PI)溶液染色15 min, 在蔡司激光共聚焦扫描显微镜(confocal laser scanning microscopy, CLSM)630×放大倍率下拍照。实验设置3个平行组, 每个平行组随机选取 10个视野用于成像和分析。
1.4 稚贝附着实验
玻璃培养皿经高温灭菌后, 缓慢地放入附有微生物被膜的玻片, 再加20 mL AFSW和10个稚贝, 在恒温18℃的无光照培养箱中培养6、12、24和48 h后,记录稚贝在微生物被膜上的附着率, 各株细菌的每个密度梯度分别设置 9个平行组; 对照组为无微生物被膜的载玻片。根据杨金龙等[3]计算稚贝的附着率以及评价其诱导活性的高低。
1.5 数据处理
数据的差异统计分析和相关性检验使用JMP软件(版本 10.0.0)。使用 Spearman’多元分析方法分析细菌密度与稚贝诱导活性之间的相关性, 其中, P为检验值, 在P<0.05时说明具有显著性的差异; r为相关性系数, 在r<0.5时说明低等水平显著相关, 在0.5≤r≤0.7时说明中等水平显著相关, 在 r>0.7时说明极其显著相关[3]。
2 结果
2.1 肠道细菌的测序与定名
本实验的10株肠道细菌的菌名和上传序列号等信息见表1。结果显示, 10株肠道细菌共分属9个不同的菌属, 其中Tenacibaculum sp. 4、Winogradskyella sp.1和Nonlabens sp. 1属于拟杆菌门, Bacillus sp. 6和Bacillus sp. 5属于厚壁菌门, 而其他所有5株细菌均属于变形菌门(其中Paracoccus sp. 1、Roseovarius sp.1和Ruegeria sp. 1属于α-变形菌纲, Alteromonas sp.3和Vibrio sp. 5属于γ-变形菌纲)。不同肠道细菌的表型如图1。
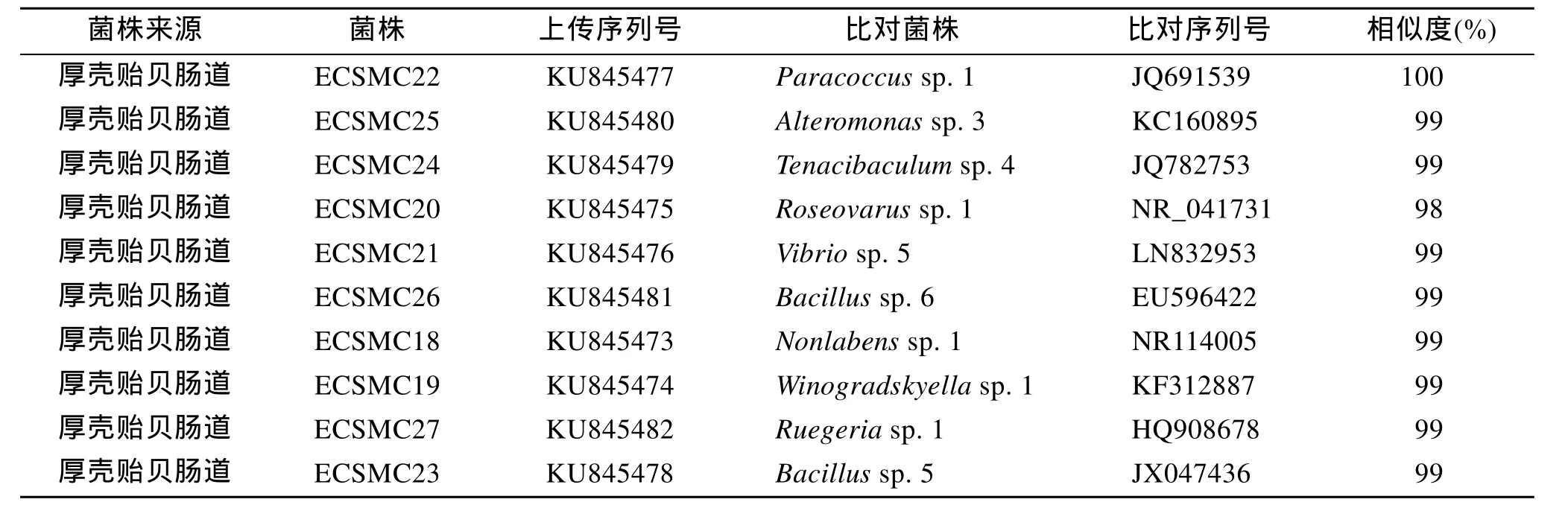
表 1 肠道细菌16S rDNA基因序列分析Tab. 1 16S rDNA gene sequence analysis of the intestinal bacterial strains
图1 不同肠道细菌的表型Fig. 1 Phenotypes of the different gut bacterial strains
2.2 不同的细菌初始密度下微生物被膜的形成
在不同的细菌初始密度条件下, 随着细菌初始密度的增加, 微生物被膜的最终密度则呈现不同程度的增加(图2)。在初始密度为1.0×109个/L和1.0×1010个/L时, Ruegeria sp. 1形成的微生物被膜的最终密度均最高, 分别为 9.1×1010个/m2和 1.4×1011个/m2,且与所有其他细菌的微生物被膜的最终密度均表现出显著性的差异(P < 0.05); Alteromonas sp. 3形成的微生物被膜的最终密度均最低, 分别为1.2×1010个/m2和 2.2×1010个/m2; 其中在初始细菌密度为 1.0×109个/L时, Alteromonas sp. 3形成的微生物被膜的最终密度与 Nonlabens sp. 1之间没有表现出显著性的差异(P > 0.05), 但与其他细菌微生物被膜的最终密度均具有显著性差异(P < 0.05); 在初始细菌密度为1.0×1010个/L时, Alteromonas sp. 3与其他细菌形成的微生物被膜的最终密度均有显著性差异(P < 0.05)。
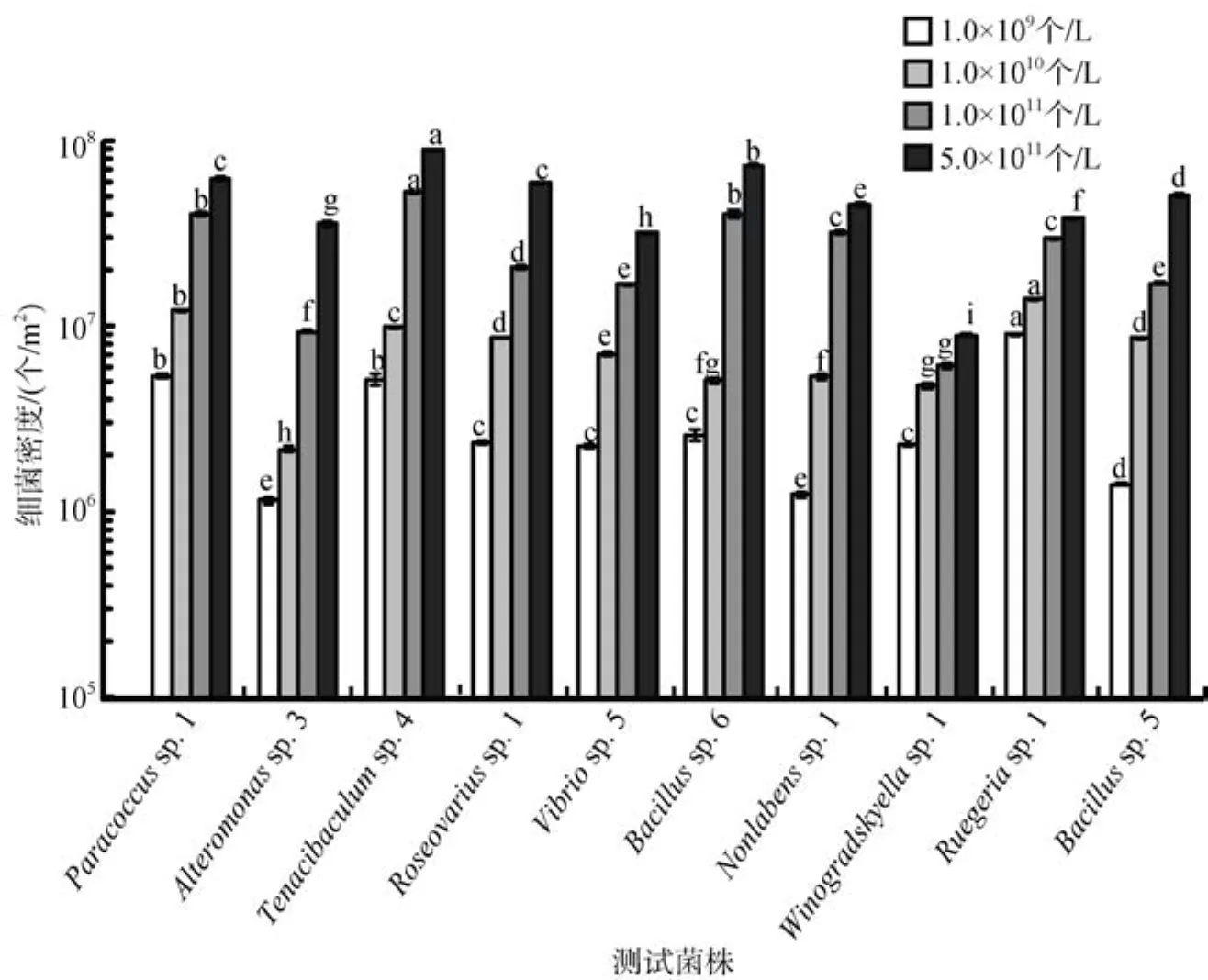
图2 不同初始细菌密度下形成微生物被膜的密度变化Fig. 2 The density of monospecific bacterial biofilms under different initial densities不同字母表示差异显著(P < 0.05)。后同
在初始细菌密度为1.0×1011个/L和5.0×1011个/L时, Tenacibaculum sp. 4形成的微生物被膜的最终密度均最高, 分别为 5.4×1011个/m2和 9.1×1011个/m2,且与其他所有细菌微生物被膜的最终密度均具有显著性的差异(P < 0.05); Winogradskyella sp. 1形成的微生物被膜的最终密度均最低, 分别为 6.2×1010个/m2和 9.1×1010个/m2, 且与其他所有细菌微生物被膜的最终密度均具有显著性的差异(P < 0.05)。结果表明,相对于其他8株细菌, Ruegeria sp. 1和Tenacibaculum sp. 4具有相对较高的微生物被膜形成能力。
2.3 肠道细菌对稚贝附着的影响
不同种属肠道细菌所形成的微生物被膜在 24 h和48 h时间段对厚壳贻贝稚贝附着的结果与12 h时的结果无明显差异, 12 h时间段的实验结果如图3所示。研究结果表明, 与空白对照组的稚贝诱导活性(14%±3%)相比, 所有试验的肠道细菌对稚贝的附着均具有不同程度的诱导活性。
Paracoccus sp. 1、Tenacibaculum sp. 4和 Bacillus sp. 6等3株细菌对稚贝的附着均具表现出中等程度的诱导作用, 其中初始细菌密度在 1.0×1010个/L时,Bacillus sp. 6表现出最高诱导活性52%; 在初始细菌密度为1.0×1011个/L时, Paracoccus sp. 1表现出最高诱导活性 61%; 而在初始细菌密度为 5.0×1011个/L时, Tenacibaculum sp. 4对稚贝附着具有最高诱导活性 53%, 其余 7株肠道细菌诱导稚贝的附着率均在50%以下, 表现出低诱导活性。
2.4 微生物被膜的不同密度对厚壳贻贝稚贝附着的诱导作用
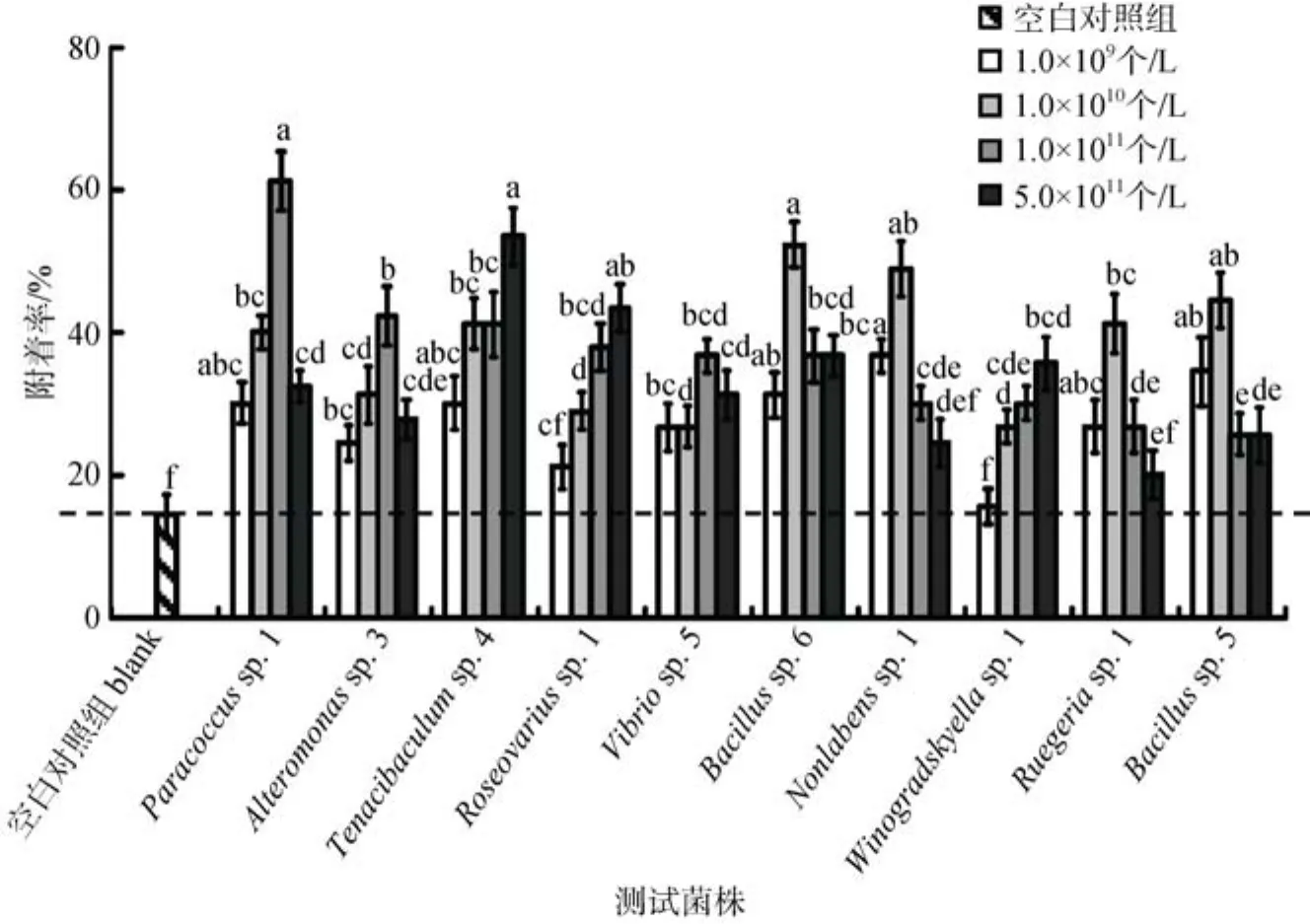
图3 不同肠道细菌对厚壳贻贝稚贝附着的诱导作用Fig. 3 Percentages of settlement of M. coruscus plantigrades on the different monospecific intestinal bacterial biofilms
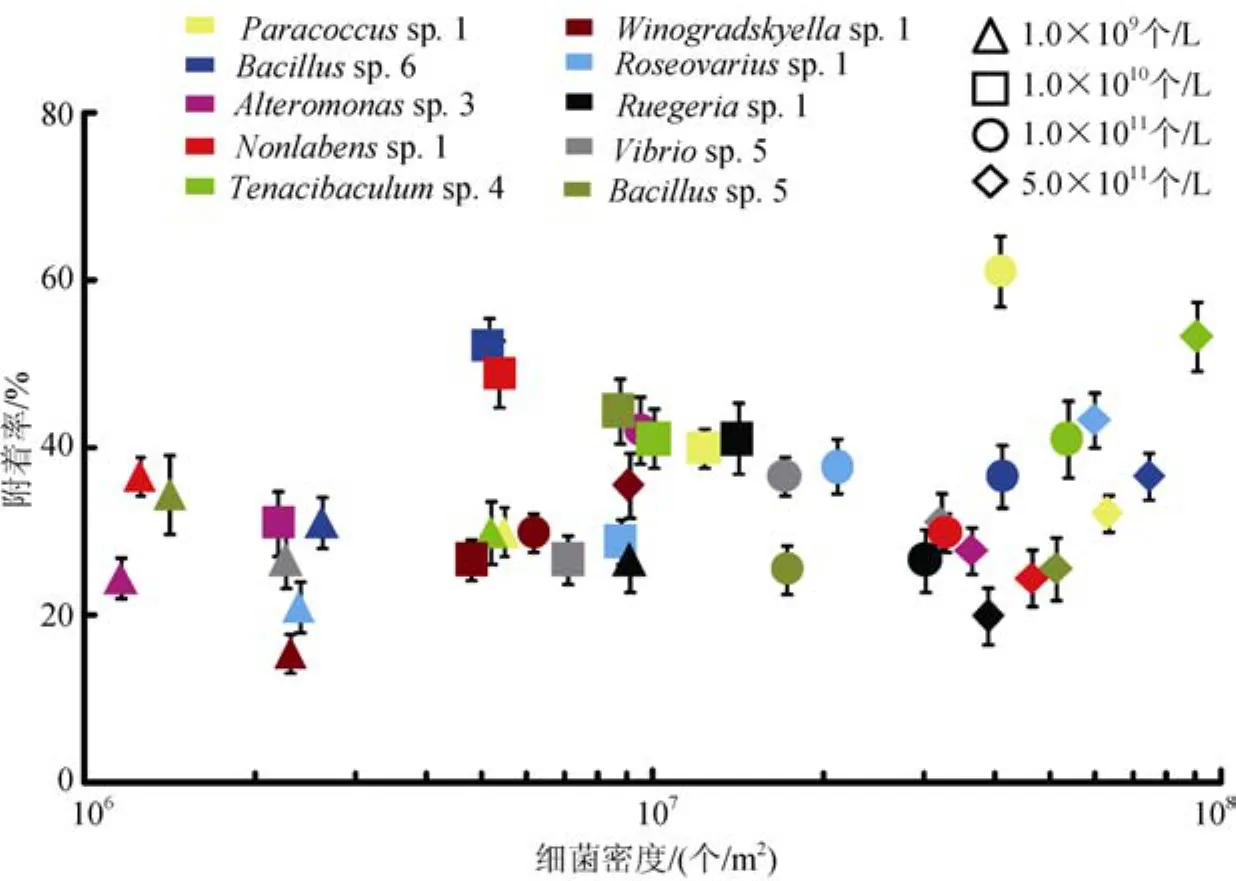
图4 单一菌株形成微生物被膜的不同密度对厚壳贻贝稚贝附着的诱导作用Fig. 4 Percentages of settlement of M. coruscus plantigrades on monospecific bacterial biofilms of varying densities
实验结果表明, 随着微生物被膜细菌密度的增多, Tenacibaculum sp. 4、Roseovarius sp. 1和Winogradskyella sp. 1等3株肠道细菌对稚贝的诱导活性逐渐升高(图4), 而其余7株肠道细菌对稚贝的诱导活性则先升高到达最大值, 然后再降低。根据相关性分析, 其中 5株肠道细菌的稚贝附着率与形成的微生物被膜密度之间具有显著相关性(P < 0.05)(表 2),其中, Tenacibaculum sp. 4、Roseovarius sp. 1和Winogradskyella sp. 1的微生物被膜密度和稚贝的诱导活性呈正显著性相关(P < 0.05), 而Nonlabens sp. 1和Bacillus sp. 5则呈负显著性相关(P < 0.05); 其余5株肠道细菌的稚贝诱导活性与形成的微生物被膜密度之间并没有表现出显著性相关(P > 0.05)。在5株显著性相关的肠道细菌中, Tenacibaculum sp. 4、Roseovarius sp. 1、Winogradskyella sp. 1和Nonlabens sp.1所形成的微生物被膜密度与稚贝的诱导活性呈中等水平显著性相关(0.5≤|r|≤0.7), 而 Bacillus sp. 5的稚贝诱导活性与微生物被膜密度之间的相关性相对较弱(|r| < 0.5)。
2.5 肠道细菌形成的微生物被膜的形态和厚度
高诱导活性和低诱导活性的两株肠道细菌的聚集状态和分布状态显著不同(图 5A)。具有高诱导活性的Paracoccus sp. 1形成的微生物被膜比低诱导活性的Bacillus sp. 5形成的微生物被膜的分布更加密集(图5A), 而且前者形成的微生物被膜膜厚(6.15 μm ±0.12 μm)显著大于后者形成的微生物被膜膜厚(3.37 μm ±0.07 μm)(P < 0.05, 图 5B)。
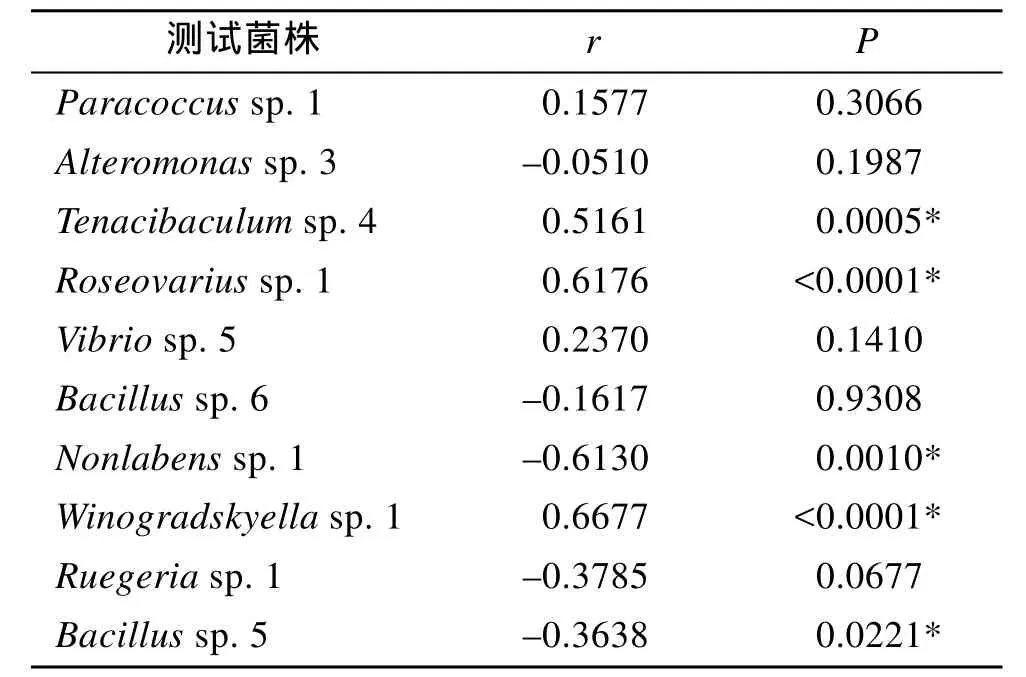
表2 微生物被膜的细菌密度与诱导活性之间的相关性分析Tab. 2 Correlation analyses between the bacterial density of biofilms and their inducing activity
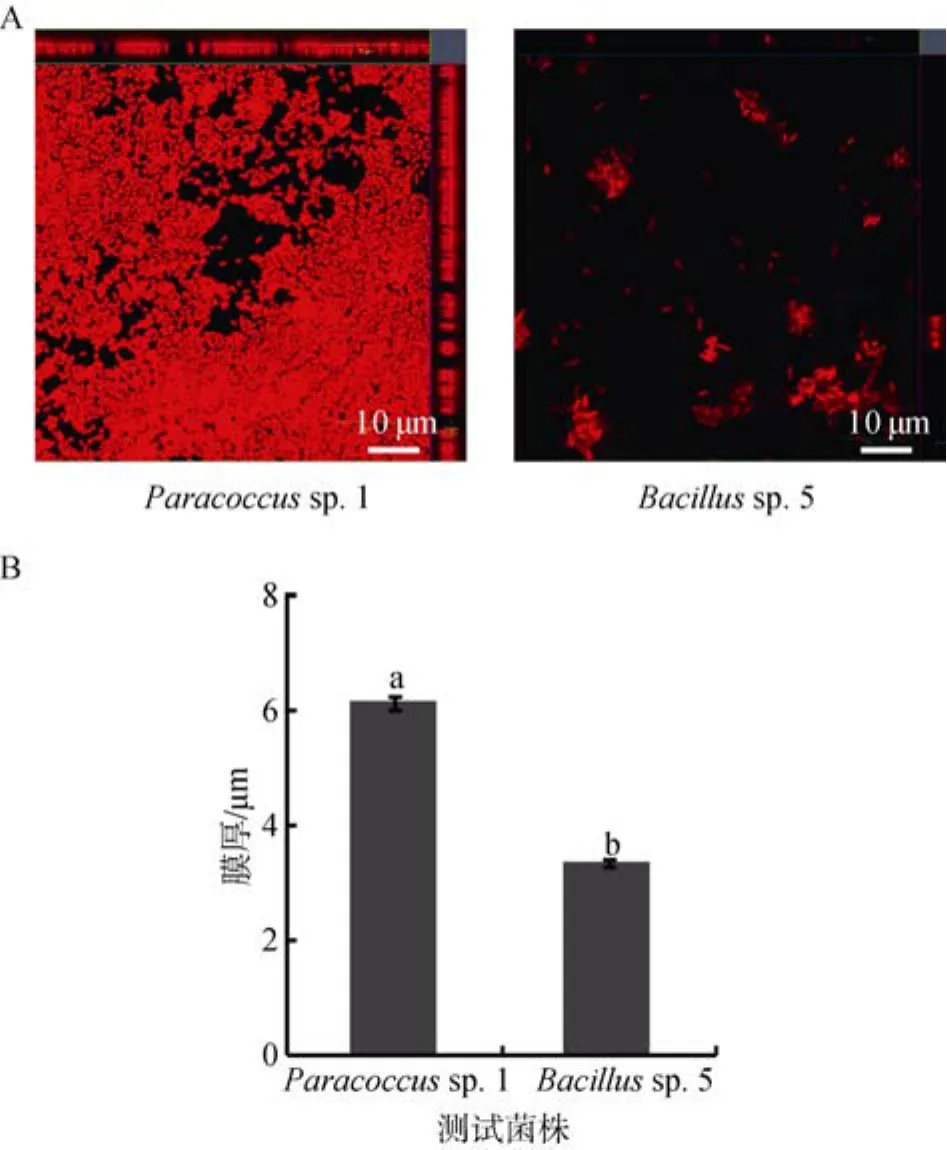
图 5 激光共聚焦扫描显微镜下Paracoccus sp. 1和Bacillus sp. 5形成的微生物被膜图像(A)及膜厚(B)Fig. 5 CLSM reveals biofilm images (A) and biofilm thickness (B) of Paracoccus sp. 1 and Bacillus sp. 5
2.6 肠道细菌的系统发育分析
采用最大简约法、最小进化法和邻接法等 3种不同的方法分析得到的系统发育分析结果基本一致,所以这里仅呈现由邻接法分析得到的系统发育树(图6)和遗传距离(表 3)。结果显示, 同属于芽孢杆菌属的Bacillus sp. 6和 Bacillus sp. 5之间的遗传距离最近, 为 0.055, 两者先聚类在一起, 但前者的诱导活性显著高于后者(P < 0.05)。而后与同属于γ-变形菌纲的两株肠道细菌Alteromonas sp. 3和Vibrio sp. 5聚为一支, 两者的诱导活性没有显著差异(P > 0.05),但形成微生物被膜的终密度具有显著性差异(P < 0.05);这一分支再与同属于 α-变形菌纲的 Paracoccus sp.1、Roseovarius sp. 1和Ruegeria sp. 1三株细菌聚为另一分支, 其中 Paracoccus sp. 1的诱导活性最高;而 Tenacibaculum sp. 4、Nonlabens sp. 1和 Winogradskyella sp. 1三株肠道细菌同属于拟杆菌门, 三者先聚为一簇, 但Tenacibaculum sp. 4的诱导活性显著高于另外两株细菌(P < 0.05); 这一簇再与前一个分支聚类, 最后与外群细菌大肠杆菌聚类。由此可见,本研究中不同肠道细菌之间的亲缘进化关系符合系统发育的规律。
3 讨论
3.1 厚壳贻贝肠道细菌的种属
单一细菌微生物被膜是海洋无脊椎动物附着过程中极其关键的化学诱导因子[15], 本研究首次证明分离自厚壳贻贝肠道的细菌所形成微生物被膜可以有效提高其稚贝的附着率。
从野生厚壳贻贝的肠道中分离纯化的肠道细菌分属于 Paracoccus、Nonlabens、Winogradskyella、Bacillus、Roseovarius、Alteromonas、Vibrio、Tenacibaculum和Ruegeria等9个菌属。在浸没于嵊泗海域由三甲氧基硅烷处理的附着基表面形成的自然微生物被膜的研究[15]中, 其分离的海洋附着细菌分属于Tenacibaculum、Staphylococcus、Vibrio、Cobetia、Pseudoalteromonas和 Bacillus等 6个菌属; 其中,Tenacibaculum、Vibrio和Bacillus等3个菌属在本研究中也同样存在。研究表明, 在浸没于自然海区的载玻片表面和野生厚壳贻贝贝壳体表形成的自然微生物被膜中, 也存在分属于Vibrio和Tenacibaculum等2个菌属的海洋细菌[16-17]。周轩等[6]在浸没于嵊泗自然海域的低湿度附着基表面的自然微生物被膜中同样分离出了Bacillus和Alteromonas 2种海洋附着细菌属。由此可见, 一些相同的细菌菌属分别存在于厚壳贻贝肠道的细菌类群中和自然微生物被膜的细菌类群中, 但同时两者也具有一定的差异。此外, 研究表明[18-22], 分属于 Paracoccus、Nonlabens、Winogradskyella、Roseovarius和Ruegeria等5个菌属的细菌也分离自不同的自然海洋环境中。根据以上结果可推测, 从野生厚壳贻贝肠道中分离纯化的10株细菌很可能来源于所处的自然海域环境。以往研究表明,肠道细菌群落源于宿主生存的外界环境[23-25], 宿主所摄食的饵料可能其肠道细菌的可能来源[24]。由此推论, 本研究中厚壳贻贝生存的外界环境在一定程度上影响了其体内肠道中的细菌类群。
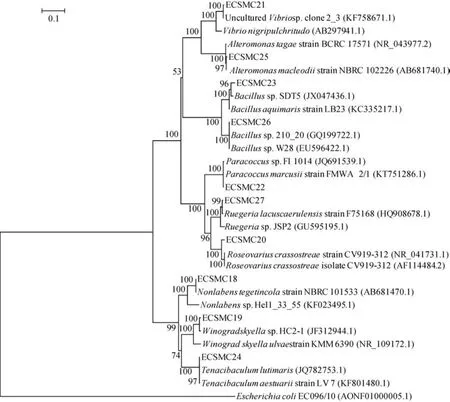
图6 细菌16S rDNA用NJ法构建的系统进化树Fig. 6 Phylogenetic tree of 16S rDNA gene sequences from bacterial isolates using the Neighbor-Joining method
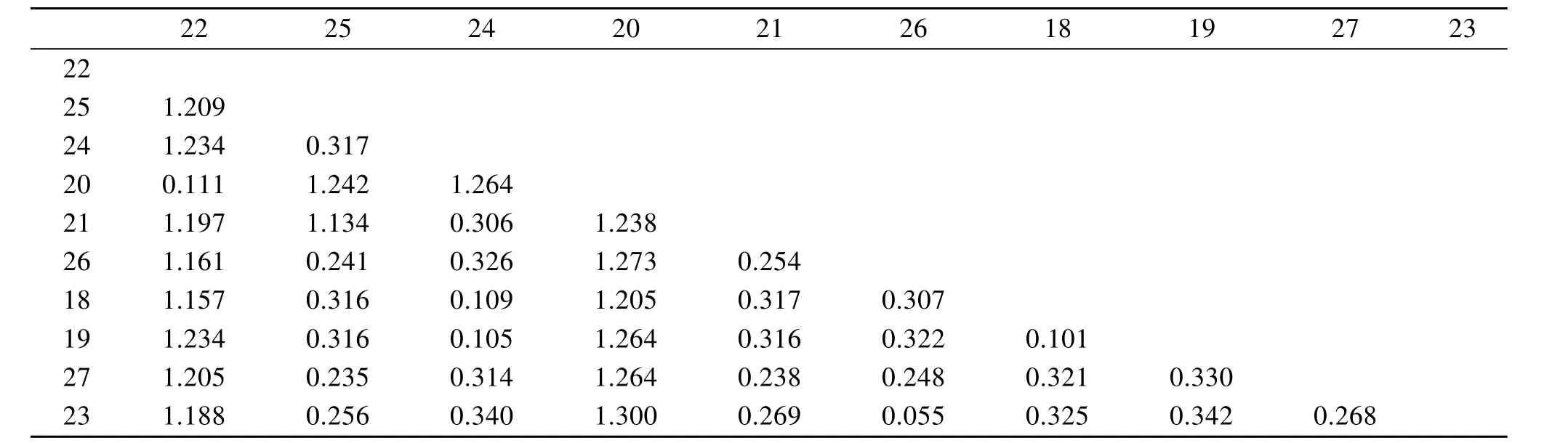
表3 所测肠道细菌的遗传距离Tab. 3 Genetic distances of the intestinal bacterial strains tested
不同种属的细菌对海洋无脊椎动物幼虫及稚贝的附着过程也是不一样的。Bao等[26]研究地中海紫贻贝(Mytilus galloprovincialis)幼虫在应对交替单胞菌Alteromonas sp. 1的附着变态反应时, 发现交替单胞菌Alteromonas sp. 1对该种幼虫的附着变态具有诱导作用, 同时其分泌的化学信号分子参与了幼虫的附着变态。本研究中, 同属于交替单胞菌属的Alteromonas sp. 3对厚壳贻贝稚贝的附着也起到了一定的诱导作用。因此推测该菌属依靠自身分泌的化学信号分子可能对不同贻贝附着都有着或高或低的诱导作用, 但作用机制需要进一步的探讨。张朝霞等[7]研究发现弧菌属的 H-4抑制了冠瘤海鞘(Styela conopus Savigny)幼虫的附着和变态, 但本研究中发现弧菌属Vibrio sp. 5却对厚壳贻贝稚贝的附着具有一定的诱导作用, 因此海洋无脊椎动物幼虫及稚贝的附着变态可能与细菌种属并无特异性关系。此外,同属于芽孢杆菌属的Bacillus sp. 5和Bacillus sp. 6这两株肠道细菌在厚壳贻贝稚贝的附着过程中具有显著不同的诱导作用, 而不同种属的Paracoccus sp.1、Tenacibaculum sp. 4和Bacillus sp. 6却均能起到中等水平的诱导作用。因此, 本研究进一步验证了细菌种属与海洋无脊椎动物幼虫及稚贝的附着变态过程没有必然的联系, 这与之前的研究结论基本相同[17]。
3.2 厚壳贻贝肠道细菌微生物被膜的形成能力
本研究结果表明, 所有测试的肠道细菌均有形成微生物被膜的能力, 且微生物被膜的最终密度受到初始细菌密度的显著影响。Huang和 Hadfield[27]研究表明, 细菌的微生物被膜终密度与初始密度具有显著相关性; 此外, Tran和Hadfield[28]的研究同样表明细菌的初始密度对微生物被膜的最终密度具有一定的影响, 这均与本研究结果一致。本研究表明,不同厚壳贻贝肠道细菌具有不同的微生物被膜形成能力, 其中Ruegeria sp. 1和Tenacibaculum sp. 4均具有较高的微生物被膜形成能力。根据 Fuqua等[29]之前的研究, 细菌分泌的某些化学信号分子或其他参与群体感应的信号物质在微生物被膜的形成和调控过程中发挥着至关重要的作用, 而本研究中特定菌株的成膜能力也可能与细菌分泌的化学信号物质具有密切的关系, 但具体的作用方式及机理仍需要进一步的深入研究。
本研究已证明, 与对照组相比所有测试肠道细菌形成的微生物被膜均对厚壳贻贝稚贝的附着具有不同程度的诱导活性。其中, 在初始细菌密度为1.0×1011个/L时, Paracoccus sp. 1的诱导活性最高, Bacillus sp. 5的诱导活性最低。很多研究[3,6,15-17]表明, 在相同的初始细菌密度下, 不同菌株对厚壳贻贝稚贝附着的诱导活性不同; 在不同的初始细菌密度下, 相同菌株对厚壳贻贝稚贝的诱导活性也不同。这可能与上述所说的菌株自身的成膜能力或细菌自身分泌的信号分子有关。
3.3 微生物被膜形成特性与贻贝附着相互关系
微生物被膜的密度与海洋无脊椎动物的附着过程具有十分密切的关系。在单一菌株形成微生物被膜的研究中, Li等[16]发现, 在所测试的10株海洋细菌中, 厚壳贻贝稚贝的附着过程与 9株细菌的微生物被膜密度具有显著的相关性。而孙俊杰等[14]研究的9株测试菌株中有7株细菌的微生物被膜密度与稚贝的附着呈显著相关。除此之外, 海洋细菌的微生物被膜密度与厚壳贻贝稚贝的诱导活性同样具有显著相关性[3]。然而, Tran和Hadfield[28]研究发现, 鹿角杯形珊瑚(Pocillopora damicornis)幼虫的附着仅与1株细菌(具有高诱导作用)的密度有显著相关性, 其余细菌密度与其幼虫的附着均没有相关性。本研究所试验的10株肠道细菌中, 只有5株形成的微生物被膜密度与厚壳贻贝稚贝的诱导活性具有显著的相关性, 说明虽然微生物被膜密度在稚贝的附着过程中具有重要的作用, 但不是适用于所有的细菌。本研究还表明, 随着细菌密度的增加, 肠道细菌对稚贝的诱导活性并非完全逐渐升高, 而是可能呈先升高达到最大值后再降低的趋势, 且最适密度取决于细菌种类。研究表明, 特定海洋细菌分泌的化学诱导信号分子在海洋无脊椎动物幼虫的附着变态过程中可能发挥着一定的作用[17,28-30]。因此, 本实验中贻贝肠道菌株所分泌的某些化学信号分子很可能对结果产生一定的影响, 但具体程度及其作用机制仍需进一步深入研究。
微生物被膜厚度、分布和聚集状态与海洋无脊椎动物的附着变态过程具有一定的相关性。根据Yang[17]等的研究发现, Tenacibaculum sp. 1形成的微生物被膜的膜厚明显大于其他细菌, 包括 Pseudoalteromonas sp. 1, 尽管这两株细菌均呈中等的诱导活性; Shewanella sp. 1的诱导活性远高于 Pseudoalteromonas sp. 4的诱导活性, 但这两株细菌形成的微生物被膜的膜厚并没有差异性, 细菌的聚集状态和分布状态也不相同。在本研究中, 高诱导活性的Paracoccus sp. 1形成的微生物被膜比低诱导活性的Bacillus sp. 5形成的微生物被膜分布更加密集, 且前者的膜厚比后者厚。由此推测, 细菌的诱导活性可能与微生物被膜膜厚没有相关性, 可能与其菌株自身有关, 例如其分泌的胞外多糖等, 因此有必要进行后续的实验分析。
综上所述, 厚壳贻贝的肠道细菌能够不同程度地诱导厚壳贻贝稚贝的附着, 说明肠道细菌在厚壳贻贝的生长发育过程中具有一定的作用。同时, 本研究成果为今后更深入地探讨肠道细菌与厚壳贻贝之间的相互作用奠定基础, 也对厚壳贻贝健康生态养殖及该过程中相关技术的提高具有一定的指导意义。
参考文献:
[1] 常亚青. 贝类增养殖学[M]. 北京: 中国农业出版社,2007.Chang Yaqing. Shellfish Culture[M]. Beijing: China Agriculture Press, 2007.
[2] 李太武. 海洋生物学[M]. 北京: 海洋出版社, 2013.Li Taiwu. Marine Biology[M]. Beijing: China Ocean Press,2013.
[3] 杨金龙, 郭行磐, 陈芋如, 等. 中湿度表面的海洋细菌对厚壳贻贝稚贝附着的影响[J]. 水产学报, 2015,39(3): 421-428.Yang Jinlong, Guo Xingpan, Chen Yuru, et al. Effects of bacterial biofilms formed on middle wettability surfaces on settlement of plantigrades of the mussel Mytilus coruscus[J]. Journal of Fisheries of China, 2015, 39(3):421- 428.
[4] Zobell C E, Allen E C. The significance of marine bacteria in the fouling of submerged surfaces[J]. Journal of Bacteriology, 1935, 29(3): 239-251.
[5] 杨金龙, 王冲, 顾忠旗, 等. 微生物膜对海洋无脊椎动物幼体附着变态的影响研究[J]. 海洋科学, 2012,36(8): 116-121.Yang Jinlong, Wang Chong, Gu Zhongqi, et al. A review on the role of marine biofilms on larval settlement and metamorphosis of marine invertebrates[J]. Marine Sciences, 2012, 36(8): 116-121.
[6] 周轩, 郭行磐, 陈芋如, 等. 低湿度表面的海洋附着细菌对厚壳贻贝附着的影响[J]. 大连海洋大学学报,2015, 30(1): 30-35.Zhou Xuan, Guo Xingpan, Chen Yuru, et al. Effects of bacterial biofilms formed on low surface wettability on the settlement of plantigrades of the mussel Mytilus coruscus[J]. Journal of Dalian Ocean University, 2015,30(1): 30-35.
[7] 张朝霞, 柯才焕, 冯丹青, 等. 海洋附着细菌对冠瘤海鞘幼体附着和变态的影响[J]. 海洋学报, 2005,27(5): 96-102.Zhang Zhaoxia, Ke Caihuan, Feng Danqing, et al. Influences of marine adhesive bacteria on settlement and metamorphosis of Styela conopus Savigny larvae[J].Acta Oceanologica Sinica, 2005, 27(5): 96-102.
[8] 常抗美, 吴剑锋. 厚壳贻贝人工繁殖技术的研究[J].南方水产科学, 2007, 3(3): 26-30.Chang Kangmei, Wu Jianfeng. Study on artificial propagation of mussel Mytilus coruscus[J]. South China Fisheries Science, 2007, 3(3): 26-30.
[9] Eckburg P B, Bik E M, Bernstein C N, et al. Diversity of the human intestinal microbial flora[J]. Science, 2005,308(5728): 1635-1638.
[10] Hooper L V, Gordon J I. Commensal host-bacterial relationships in the gut[J]. Science, 2001, 292(5519):1114-1118.
[11] Sommer F, Backhed F. The gut microbiota—masters of host development and physiology[J]. Nature Reviews Microbiology, 2013, 11(4): 227-238.
[12] Bates J M, Mittge E, Kuhlman J, et al. Distinct signals from the microbiota promote different aspects of zebrafish gut differentiation[J]. Developmental Biology,2006, 297(2): 374-386.
[13] Zokaeifar H, Balcázar J L, Saad C R, et al. Effects of Bacillus subtilis on the growth performance, digestive enzymes, immune gene expression and disease resistance of white shrimp, Litopenaeus vannamei [J]. Fish and Shellfish Immunology, 2012, 33(4) : 683-689.
[14] Al-Harbi A H, Uddin M N. Seasonal variation in the intestinal bacterial flora of hybrid tilapia Oreochromis niloticus × Oreochromis aureus cultured in earthen ponds in Saudi Arabia[J]. Aquaculture, 2004, 229(1): 37-44.
[15] 孙俊杰, 张显, 郭行磐, 等. 硅烷化表面海洋细菌对厚壳贻贝稚贝附着的影响[J]. 水产学报, 2015, 39(10):1530-1538.Sun Junjie, Zhang Xian, Guo Xingpan, et al. Effects of marinebacteria from silanizing surfaces on plantigrade settlement of the mussel Mytilus coruscus[J]. Journal of Fisheries of China, 2015, 39(10): 1530-1538.
[16] Li Y F, Guo X P, Yang J L, et al. Effects of bacterial biofilms on settlement of plantigrades of the mussel Mytilus coruscus[J]. Aquaculture, 2014, 433(6): 434-441.
[17] Yang J L, Shen P J, Liang X, et al. Larval settlement and metamorphosis of the mussel Mytilus coruscus in response to monospecific bacterial biofilms[J]. Biofouling,2013, 29(3): 247-259.
[18] Nedashkovskaya O I, Kukhlevskiy A D, Zhukova N V,et al. Winogradskyella litoriviva sp. nov. a new flavobacterium isolated from coastal seawater[J]. International Journal of Systematic & Evolutionary Microbiology, 2015, 65(10): 3652-3657.
[19] Park S, Kang C H, Yoon J H. Nonlabens arenilitoris sp.nov. a member of the family Flavobacteriaceae isolated from seashore sand[J]. Antonie Van Leeuwenhoek, 2013,103(5): 1125-1132.
[20] Vandecandelaere I, Nercessian O, Segaert E, et al.Ruegeria scottomollicae sp. nov. isolated from a marine electroactive biofilm.[J]. International Journal of Sys-tematic & Evolutionary Microbiology, 2008, 58(12):2726-2733.
[21] Pan J, Zhen Y, Jia T, et al. Roseovarius lacus, sp. nov.isolated from Yuncheng Saline Lake, China[J]. Archives of Microbiology, 2016, 199(2): 231-236.
[22] Kim K, Lee S S. Paracoccus aquimaris, sp. nov. isolated from seawater[J]. Antonie Van Leeuwenhoek, 2015,108(4): 871-877.
[23] Han S, Liu Y, Zhou Z, et al. Analysis of bacterial diversity in the intestine of grass carp (Ctenopharyngodon idellus) based on 16S rDNA gene sequences[J].Aquaculture Research, 2010, 42(1): 47-56.
[24] Cahill M M. Bacterial flora of fishes: a review[J]. Microbial Ecology, 1990, 19(1): 21-41.
[25] Wu S, Wang G, Angert E R, et al. Composition, diversity,and origin of the bacterial community in grass carp intestine[J]. Plos One, 2012, 7(2): e30440.
[26] Bao W Y, Yang J L, Satuito C G, et al. Larval metamorphosis of the mussel Mytilus galloprovincialis in response to Alteromonas sp. 1: evidence for two chemical cues?[J]. Marine Biology, 2007, 152(3): 657-666.
[27] Huang S, Hadfield M G. Composition and density of bacterial biofilms determine larval settlement of the polychaete Hydroides elegans[J]. Marine Ecology Progress, 2003, 260(1): 161-172.
[28] Tran C, Hadfield M G. Larvae of Pocillopora damicornis settle and metamorphose in response to surface-biofilm bacteria[J]. Marine Ecology Progress, 2011, 433: 85-96.
[29] Fuqua W C, Winans S C, Greenberg E P. Quorum sensing in bacteria: the LuxR-LuxI family of cell density-responsive transcriptional regulators[J]. Journal of Bacteriology, 1994, 176(2): 269-275.
[30] Chung H C, Lee O O, Huang Y L, et al. Bacterial community succession and chemical profiles of subtidal biofilms in relation to larval settlement of the polychaete Hydroides elegans[J]. ISME Journal, 2010, 4(6): 817-828.
