咖啡种质资源DNA指纹图谱的构建
2017-03-31黄丽芳董云萍王晓阳孙燕陈鹏林兴
黄丽芳 董云萍 王晓阳 孙燕 陈鹏 林兴军 闫林

摘 要 选用4份咖啡种质资源为材料,对S1-S100共100个RAPD引物进行筛选,选出多态性好的引物10个。利用这10个RAPD引物对28份咖啡资源进行扩增,共获得86条带,平均每个RAPD引物扩增出8.6条带,多态性谱带74条,多态性条带比率为86.0%。结果表明:供试咖啡种质资源遗传多样性较丰富;10个多态性好的RAPD引物中S8的鉴别效率最高,能将全部供试材料全部区分开,通过整合软件录入如所属种类、种质名称、采样地点、引物、RAPD-PCR 扩增数据以及电泳图片等相关信息,形成指纹图谱二维编码,构成咖啡种质资源的DNA指纹图谱,为咖啡种质资源品种权益保护及分子身份证构建奠定了基础。
关键词 咖啡 ;种质资源 ;RAPD ;指纹图谱
中图分类号 TS273 文献标识码 A Doi:10.12008/j.issn.1009-2196.2016.12.008
咖啡(Coffea spp.)为茜草科(Rubiaceae)咖啡属(Coffea)多年生常绿灌木或小乔木,原产非洲北部和中部的热带地区。咖啡属植物约有100多个种,通常所指的咖啡为小粒种、中粒种、大粒种和查理种。目前全世界驯化栽培的主要有小粒种(C. arabica L.)和中粒种(C. canephora Pierre),面积约占70%和30%[1-3]。小粒种咖啡为异源四倍体种,自花授粉,而其他种均为二倍体,异花授粉。实际生产中,咖啡生产者在来源或者遗传背景不甚明了的情况下进行杂交或嫁接,以及地域间的引种利用等造成咖啡品种品系良莠不齐,变异性大,产生了大量的同物异名和同名异物,导致种子、种苗市场混乱的局面,阻碍咖啡产业的健康发展。传统的咖啡品种真实性和纯度是通过形态学、化学物质含量分析或者进行杯品等方法鉴定的[4-6],耗时长、成本高、较难做到准确鉴别咖啡品种基因型。因此,对咖啡的核心种质的构建、种质的分类及真伪鉴定、品种权益保护等方面的研究显得十分必要。
DNA指纹图谱(DNA fingerprint)是鉴别不同品种、品系和不同无性系、不同基因型,以及辨别亲本与杂交后代遗传相关等方面的有力工具[7-9],RAPD标记是以PCR为基础,采用随机引物对未知序列的DNA进行检测,根据扩增的多态性来反映基因组DNA相应区域的遗传关系,具有实验操作流程简单、所需仪器设备少、快速简便,通用性好,且无须事先知道目的DNA片断序列信息等许多优点,在生物的遗传多样性检测、品系鉴定、遗传图谱构建和系统学等领域得到广泛运用[10-12]。
近年来,咖啡的分子标记研究也取得了一些进展。王晓阳等[13]从514对SSR引物中筛选出1对特异性引物进行中小粒种咖啡的鉴定。该引物在小粒种中可扩增出双带,在中粒种中扩增出单带,从扩增产物的条带数目即可进行中小粒咖啡的鉴别。黄丽芳等[14]用筛选获得的18条RAPD多态性引物对72份咖啡种质资源进行RAPD扩增,结果表明,咖啡资源在种的分类水平上存在遗传关系多样性,部分资源的分类学地位与地理来源无相关性。本文利用RAPD标记技术,对28份咖啡资源进行遗传多样性分析,并筛选出稳定的,多态性好的RAPD引物,构建DNA指纹图谱,为咖啡资源的分子身份证构建奠定基础。
1 材料与方法
1.1 供试材料
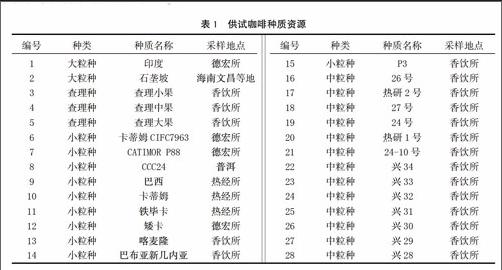
供试材料为28份咖啡种质资源,其中大粒及查理种咖啡5份(编号1~5),小粒种咖啡10份(编号6~15),中粒种咖啡13份(编号16~28)。材料采自于海南,云南省德宏热带农业科学研究所(简称德宏所),云南省农业科学院热带亚热带经济作物研究所(简称热经所),中国热带农业科学院香料饮料研究所(简称香饮所)。详见表1。
1.2 基因组DNA的提取
采用CTAB改良法进行咖啡叶片基因组DNA的提取[15]。提取的DNA经1.0%琼脂糖凝胶电泳及紫外分光光度计检测质量,稀释至20 ng/μL,置于-20℃保存备用。
1.3 引物筛选及RAPD扩增
利用4份差异较大的咖啡模板,对RAPD引物S1-S100共100条随机引物进行多态性筛选,从中选取多态性好且扩增条带清晰的对实验样品进行扩增。引物由上海生工生物工程技术股份有限公司合成。
PCR反应体系为本课题组优化过的咖啡RAPD反应体系[16]:20 μL体系中,含ddH2O 13.75 μL,10×buffer 2 μL,dNTPs(2.5 mmol/L)0.6 μL,Mg2+(25 mmol/L)1.2 μL,引物(10 μmol/L)1.2 μL,Taq酶(5 U/μL)0.25 μL,基因组DNA(20 ng/μL)1 μL。PCR扩增条件:94 ℃预变性3 min;94℃变性1 min,38℃退火1 min 45 s,72℃延伸2 min,反应40个循环;72℃再延伸7 min。扩增产物通过1.8%琼脂糖凝胶电泳观察,以DL 2 000 Marker为参照,试验重复2次。
1.4 数据分析
根据扩增引物的迁移率,利用人工方法统计读带,将清晰度高、重复性好的扩增条带赋值“1”,无条带或肉眼分辨不清的弱带赋值“0”,导入Excel 表格生成数据矩阵。通过多态性条带比率、品种最大相似系数及品种聚类区分率(Rate of distinguishing variety by cluster,RDVC)鑒别引物。采用NTSYSpc 2.1软件,根据Nei和Li相似系数计算遗传相似性GS(genetic similarity),根据非加权算术平均法(UPGMA)进行聚类分析。RDVC=(N-Ni)/N,Ni 为无法区分品种数,N为品种总数。
1.5 指纹图谱构建
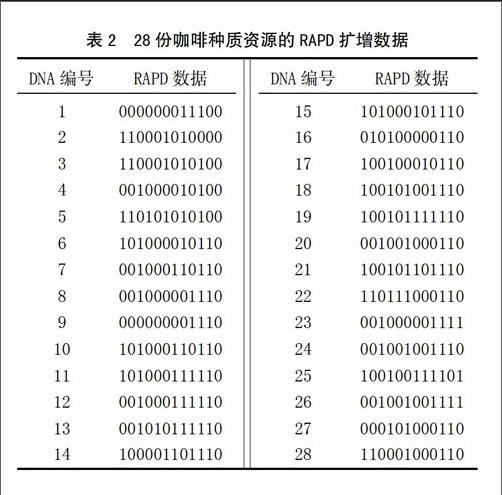
根据RAPD电泳扩增结果,从多态性比较丰富的引物中选出至少一条可将所有供试品种区分且表现优异的RAPD引物,构建咖啡品种(系)的DNA指纹图谱。咖啡的DNA指纹图谱一部分是RAPD扩增数据,由二进制代码组成,另一部分是结合草料二维码在线生成器(www.cli.im)将咖啡的种质名称、种属、采样地点、引物名称、RAPD数据代码[17],RAPD-PCR扩增数据图片等信息录入软件中,形成指纹图谱二维编码[18],可以更加快捷方便的进行实际应用。详见表2。
2 結果与分析
2.1 RAPD引物筛选与扩增片段多态性
以4份差异较大的咖啡基因组DNA为模板,对S1-S100共100个RAPD引物进行多态性筛选,筛选出10个多态性较好的引物。利用这10个引物对28份材料基因组DNA进行PCR扩增,扩增出的位点多态性数目为5~12条,产物片段大小介于350~2 000 bp ,以引物S8和S19扩增的位点数最多,10个引物共扩增出86个位点,平均每个引物扩增出8.6条谱带,其中多态性带74条,多态性条带比例为86.0%,其中S8引物多态性最为丰富。详见表3。
2.2 聚类分析
28份咖啡材料间的相似性系数变幅为0.337 8~0.905 4。其中小粒种喀麦隆(13)和中粒种热研2 号(17)的相似系数最小,为0.337 8,说明二者间的遗传距离最远,遗传差异最大。小粒种CCC24(8)和铁毕卡(11),中粒种兴32(24)和兴30(26)相似系数最大,都为0.905 4,表明它们之间的遗传距离最小,遗传差异最小。从种内遗传相似系数看,2份大粒种的相似系数为0.635 1,查理种种内最小相似系数为查理中果(4)和查理大果(5),为0.702 7,最大相似系数为查理小果(3)和查理大果(5),为0.878 4。小粒种CATIMOR P88(7)和巴布亚新几内亚(14),中粒种兴32(24)和兴31(25),它们种内最小相似系数分别为0.662 2和0.608 1。
UPGMA 聚类结果(图1)分析,该聚类图在相似性系数为0.608水平下可划分为3大类。第Ⅰ类群包括5份材料,包括2份大粒种和3份查理种,第Ⅱ类群包括全部13份中粒种材料,第Ⅲ类群包括了全部的小粒种。聚类分析结果表明,Ⅰ、Ⅱ、Ⅲ 3大类群种间关系清晰,聚类结果与经典分类结果一致。
2.3 指纹图谱的构建
根据10条引物扩增谱带的多态性,能将供试的28份咖啡材料全部区分开。但从单条RAPD引物的聚类分析显示,10条多态性较好的引物中只有引物S8能将所有供试材料一次性区分开,10条引物检测效率如表3。引物S8的多态性带数是12条,多态性百分率100%,品种最大相似性系数为0.916 7,品种聚类区分率为100%,为最有效的引物。其次为引物S11、S19,品种最大相似性系数为1,品种聚类区分率为53.57%,有13个品种无法区分开。仅用引物S8就能将所有参试品种有效区分,故选用S8的分子数据用于咖啡种质资源DNA 指纹图谱的构建。
利用草料二维码在线生成器对所有供试材料进行编码,为每份材料建立了1个DNA 指纹图谱编码,其中包含了所属种类、种质名称、采样地点、引物、RAPD-PCR扩增数据以及电泳图片,从而得到二维码结果。如图2-A为印度,包含以下信息:所属种类大粒种,种质名称印度、采样地点德宏所、引物S8、RAPD-PCR扩增数据为000000011100,以及图2-A电泳图;图2-B为巴西,包含以下信息:所属种类小粒种,种质名称为巴西,采样地点为热经所,引物S8,RAPD-PCR扩增数据为000000001110,电泳图2-B;图2-C为印度,包含信息,种类为中粒种,种质名称为兴28,采样地点为香饮所,引物S8,RAPD-PCR扩增数据为110001000110,电泳图2-C。28份咖啡资源的RAPD数据见表3。
3 讨论
任何一种分子标记在实际运用中都有各自的优缺点。RAPD 分子标记技术易受多种因素的影响,因其利用的是非特异引物,扩增结果的稳定性是相对的。但可通过多次实验摸索建立适合该物种的一套反应条件,得到令人满意的扩增结果[19-21]。
本研究所用的咖啡RAPD反应体系及程序等都经本课题组反复多次实验验证,筛选出的最优反应条件。从研究结果可看出,同一条引物在各个咖啡样品间和不同引物在相同的咖啡样品上多态性带数和扩增总带数的差异较显著,说明供试的基因组DNA多态性较丰富,也表明了RAPD标记是研究咖啡种质资源亲缘关系和构建咖啡指纹图谱的有效方法。
本研究筛选出的10条RAPD引物对28份咖啡材料进行PCR扩增,共扩增出86条带,平均每条引物扩增出8.6条带,多态性条带共74条,占总数的86%,相似性系数变幅为0.337 8~0.905 4,多样性较丰富,这与此次试验的选材也有一定关系,收集的28份咖啡样品为生产或科学研究上具有代表性的咖啡品种(品系),涵盖了咖啡属的4个种,其中小粒种和中粒种居多,大粒种和查理种因其抗逆性较好,目前在科学研究中较多用于作砧木。
为了降低成本和提高样品的检测效率,在构建DNA指纹图谱时,原则之一就是利用较少的引物分开尽量多的品种,这就意味着所用的引物多态性和稳定性要好。10条引物中,S8多态性条带12条,多态性百分率为100%,品种聚类区分率100%。因此,选择引物S8构建DNA指纹图谱。若在试验材料增加时,单条引物不能区分所有供试材料,可在引物S8的基础上增加多态性较好的引物,如S11、S19,利用引物组合区分出所有品种[22]。
DNA指纹代码构建指纹图谱的信息有限,而二维码技术可进行信息数据录入。本文首次将DNA指纹代码结合二维码技术应用在咖啡研究中,从而生成咖啡二维码,可更方便快捷地供研究人员进行对比和鉴定,为建立咖啡的网络数据库提供直观有效的数据支持。最终产生的信息也可录入到咖啡产品的物流信息中,从而形成一条完整的咖啡产品物流信息链,为咖啡产品在市场上的健康持续发展提供技术支持。
参考文献
[1] Amidou N D, Michel N, Serge H, et al. Genetic basis of species differentiation between Coffea liberica Hiem and C. canephora Pierre: Analysis of an intempecific cross[J].Genet Resour Crop Evol,2007, 54: 1 011-1 021.
[2] Aggarwal R K, Hendre P S,Varshney R K, et al. Identification, characterization and utilization of EST-derived genic microsatellite markers for genome analyses of coffee and related species[J]. Theor Appl Genet, 2007, 114: 359-372.
[3] A1-Arequi AHNA, Chliyeh M, Sghir F, et al. Diversity of arbuscular mycorrhizal fungi in the rhizosphere of Coffea Arabica in the Republic of Yemen[J]. J App1 Biosci, 2013, 64: 4 888-4 901.
[4] Carrera F, Leon-Camacho M, Pablos F, et al. Authentication of green coffee varieties according to their sterolic profile[J]. Anal Chim Acta, 1998, 370: 131-139.
[5] Martin M J, Pablos F, Gonzalez A G, et al. Fatty acid profiles as discriminant parameters for coffee varieties differentiation[J]. Talanta,2001, 54: 291-297.
[6] Jham G N, Winkler J K, Berhow M A, et al. γ-Tocopherol as a marker of Brazilian coffee(Coffea arabica L.)adulteration by corn[J]. J Agric Food Chem,2007, 55: 5 995-5 999.
[7] 薛建峰,谭美莲,严明芳,等. 我国部分蓖麻品种遗传资源SSR分析及DNA指纹图谱[J]. 中国油料作物学报,2015,37(1):048-054.
[8] 聂新辉,尤春源,李晓方,等. 新陆早棉花品种DNA 指纹图谱的构建及遗传多样性分析[J]. 作物学报,2014,40(12):2 104-2 117.
[9] 汪 斌,祁 伟,兰 涛,等. 应用ISSR分子标记绘制红麻种质资源DNA指纹图谱[J]. 作物学报,2011,37(6):1 116-1 123.
[10] 郭菊卉,田 娟,石桃雄,等. 甜荞重组自交系(RIL)亲本基因组RAPD标记的多样性分析[J]. 湖北农业科学,2015,54(2):284-288.
[11] 王红意,翟 红,王玉萍,等. 30个中国甘薯主栽品種的RAPD指纹图谱构建及遗传变异分析[J]. 分子植物育种,2009,7(5):879-884.
[12] 庞 敏,邹芳平,肖娅萍. 应用RAPD技术构建绞股蓝DNA指纹图谱[J]. 陕西师范大学学报(自然科学版),2006,34(3):87-91.
[13] 王晓阳,董云萍,黄丽芳,等. 利用SSR分子标记快速鉴别中粒种与小粒种咖啡[J].热带作物学报,2014,35(4):622-627.
[14] 黄丽芳,董云萍,王晓阳,等. 利用RAPD标记分析咖啡种质资源的遗传多样性[J]. 热带作物学报,2014,35(12):2313-2319.
[15] 黄丽芳,闫 林,董云萍,等. 咖啡叶片DNA提取方法的比较研究[J]. 热带农业科学,2011,31(12):42-45.
[16] 闫 林,黄丽芳,谭乐和,等. 咖啡ISSR与RAPD-PCR反应体系优化[J]. 热带作物学报,2012,33(5):854-859.
[17] 刘本英,孙雪梅,李友勇,等. 20个云南无性系茶树良种的DNA指纹图谱构建[J]. 热带作物学报,2011,32(4):720-727.
[18] 宋海斌,崔西波,栾非时,等. 基于SSR标记的甜瓜品种(系)DNA指纹图谱库的构建[J]. 中国农业科学,2012,45(13):2676-2689.
[19] 杨青松,熊 勇,赵 艳,等. 血满草RAPD-PCR反应体系的建立与DNA指纹图谱研究[J]. 生物技术,2014,24(5):48-51.
[20] 吕 雪. 李属种质资源的RAPD指纹图谱分析[D]. 哈尔滨:东北农业大学,2014.
[21] 熊华斌,吴渝生,程在全. 云南省3种作物品种RAPD指纹图谱的构建[J]. 种子,2004,23(2):10-13.
[22] 马瑞君,梅洪娟,庄东红,等. 不同品种(系)凤凰单丛茶DNA指纹图谱的构建[J]. 茶叶科学,2014,34(5):515-524.
