辣椒轻斑驳病毒湖南分离物的全基因序列测定及结构分析
2017-03-29刘湘宁戴良英唐前君
刘湘宁, 戴良英, 李 魏, 董 铮, 唐前君
(1. 湖南农业大学植物保护学院, 长沙 410128; 2. 湖南农业大学病毒研究所, 长沙 410128;3. 湖南省植物病虫害生物学与防控重点实验室, 长沙 410128)
辣椒轻斑驳病毒湖南分离物的全基因序列测定及结构分析
刘湘宁1,3, 戴良英1,3, 李 魏1,3, 董 铮1,3, 唐前君1,2,3*
(1. 湖南农业大学植物保护学院, 长沙 410128; 2. 湖南农业大学病毒研究所, 长沙 410128;3. 湖南省植物病虫害生物学与防控重点实验室, 长沙 410128)
采自湖南地区的辣椒轻斑驳病毒Peppermildmottlevirus(PMMoV)样品经单斑分离后,根据已经报道的PMMoV序列基因保守区设计6对简并引物,采用片段重叠法和RACE方法扩增、克隆获得一个全长为6 356 bp的湖南分离物(PMMoV-HN1,登录号:KP345899)全基因组序列,编码4个蛋白,分别为126 kD蛋白(70~3 423 nt)、183 kD蛋白(70~4 908 nt)、28 kD蛋白(4 909~5 682 nt)和17.5 kD蛋白(5 685~6 158 nt),5′-非编码区(5′-UTR)和3′-非编码区(3′-UTR)分别含有69和198个碱基,其中5′-UTR存在一个序列为m7G5′pppG的甲基化核苷酸帽子结构。一致性分析发现PMMoV-HN1与PMMoV其他分离物的核酸一致性为94%~99%,编码的氨基酸一致性为94%~99%。全基因组序列系统进化分析表明PMMoV-HN1分离物与中国首次报道的PMMoV-CN分离物亲缘关系最近。本研究是国内报道的第二例PMMoV全基因组序列。
辣椒; 辣椒轻斑驳病毒; 全基因序列; 序列分析
辣椒轻斑驳病毒Peppermildmottlevirus(PMMoV)是烟草花叶病毒属Tobamovirus成员,属于正义单链RNA病毒,最早发现于美国,后来在西班牙、德国、澳大利亚、阿根廷、加拿大、英国、巴西等国家和地区均有相关报道,曾对英国、美国的辣椒种植造成毁灭性损失[1]。PMMoV主要侵染茄科辣椒属植物,植株被侵染后表现斑驳或黄绿相间的花叶症状,果实被侵染后畸形、斑驳,可能出现凹凸斑点。PMMoV在辣椒上的种传率高达22%~29%,是温室和大棚辣椒病毒病害的重要毒源[2]。PMMoV可通过种子与染毒汁液摩擦传毒,在植物病株残体上可存活25年左右,昆虫介体不易传毒,带毒种子、感病植株和带病土壤是重要的侵染来源[3]。国内于1994年首次在新疆辣椒上发现PMMoV[4]后,陕西、山东青岛、北京及宁夏等地区相继报道了该病毒[5-7]。2006年中国首次分离测定了PMMoV的全基因组序列,命名为PMMoV中国分离物(PMMoV-CN)[8]。2013年,郑兴华等对在贵州各辣椒主产区采集的200多份辣椒病样进行血清学检测,发现贵州地区PMMoV的检出率仅次于黄瓜花叶病毒Cucumbermosaicvirus(CMV),达到22%~29%[9]。鲁宇文等[10]首次在我国辣椒田间病毒病样中检测到辣椒斑驳病毒Peppermottlevirus(PepMoV)和PMMoV复合侵染。
湖南是我国重要的辣椒产地,主要栽培品种有‘兴蔬215’、‘兴蔬16号’、‘兴蔬301’、‘兴蔬嫩辣’、‘湘研15号’、‘湘研16号’、‘博辣娇红’、‘博辣红牛’、‘博辣5号’、‘博辣6号’、‘博辣4号’、‘湘辣4号’、‘湘辣7号’等。近年来,辣椒病毒病在辣椒的种植和生产上的危害日益加重,严重影响了辣椒的产量和品质,PMMoV是造成辣椒经济损失的重要病原之一,但目前有关PMMoV的研究非常有限,同时PMMoV对湖南辣椒的侵染鲜有报道。本研究在湖南辣椒病样中检测到PMMoV,采用片段重叠法和RACE方法对湖南辣椒上PMMoV-HN1分离物的全基因序列进行测定、序列结构和一致性分析,同时发现PMMoV其他病毒属病毒存在复合侵染现象,可为PMMoV的发生规律和该分离物后续的功能研究等奠定基础。
1 材料与方法
1.1 材料
1.1.1 辣椒样本采集与保存
2013年6月随机采取叶片皱缩、畸形、扭曲、斑驳、褪绿,叶片变小、萎蔫,茎秆褪绿,植株矮化等典型病毒病危害症状的辣椒样品196份(具体辣椒品种和采集地点见表1)。经RT-PCR检测后测序确定为PMMoV侵染的辣椒样品,在苋色藜上3次单斑分离纯化,采集苋色藜单斑叶片保存于-80℃。
表1 样本来源及数量
Table 1 Sample sources and numbers
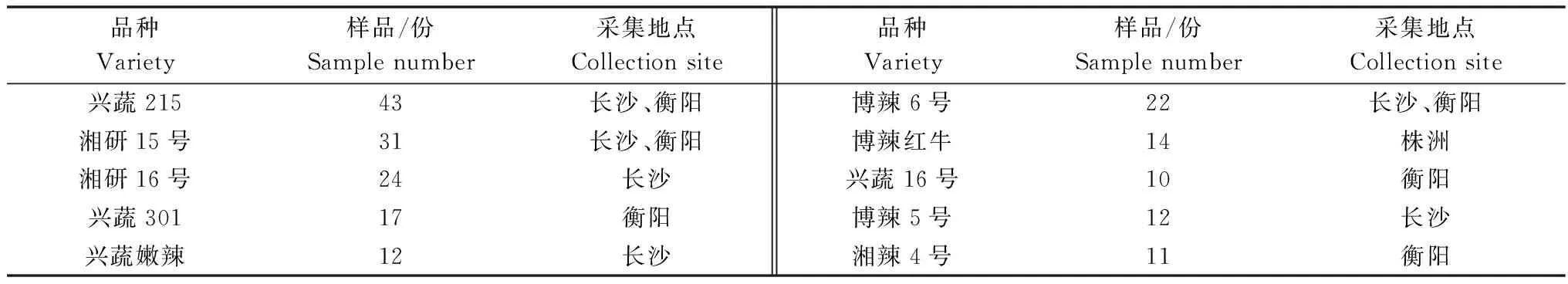
品种Variety样品/份Samplenumber采集地点Collectionsite品种Variety样品/份Samplenumber采集地点Collectionsite兴蔬21543长沙、衡阳博辣6号22长沙、衡阳湘研15号31长沙、衡阳博辣红牛14株洲湘研16号24长沙兴蔬16号10衡阳兴蔬30117衡阳博辣5号12长沙兴蔬嫩辣12长沙湘辣4号11衡阳
1.1.2 试剂
PMMoV病毒ELISA检测试剂盒购自美国Agdia公司;植物总RNA提取试剂(easyPure Plant RNA Kit)、EasyScript First-Strand cDNA Synthesis SuperMix cDNA合成试剂盒、pEASY-TI Cloning Kit、TaqDNA聚合酶和其他常规分子生物学试剂购自北京全式金生物技术有限公司;普通DNA产物纯化试剂盒购自北京天根(Tiangen)生化科技有限公司;Smarter RACE 5′/3′ Kit Race试剂盒购自宝生物(TaKaRa)大连公司。
1.2 方法
1.2.1 病叶总RNA的提取及cDNA的合成
取样品0.5 g置于研钵中,加液氮,充分研磨,按照easyPure Plant RNA Kit试剂盒说明书步骤提取植物总RNA,利用EasyScript First-Strand cDNA Synthesis SuperMix cDNA合成试剂盒将总RNA反转录成cDNA,-20℃保存。
1.2.2 辣椒样品PMMoV鉴定
参照黄粤等[6]的方法和特异性检测引物(CP基因,上游引物:ATTACTACCGCCGATGCTGAG;下游引物:TGGAGGAAAAACACTACGAG)对196份样品进行RT-PCR检测,获得的PCR产物送至生工生物工程(上海)有限公司测序,将测序结果在http:∥blast.ncbi.nlm.nih.gov/Blast.cgi上进行BLAST比对,确定鉴定结果。
1.2.3 PMMoV全基因的克隆、拼接
参照PMMoV-CN(登录号:AY859497)基因全序列,使用Premier 6.0设计出6对引物(表2),引物委托生工生物工程(上海)有限公司合成。采用片段重叠法克隆PMMoV湖南分离物基因组序列,根据RACE试剂盒说明书克隆获得PMMoV的5′端与3′序列。
表2 克隆PMMoV湖南分离物基因组的引物序列
Table 2 Primers for cloning PMMoV isolate Hunan

扩增片段Segment引物名称Primer引物序列Sequence(5'-3')引物位置Location片段长度/bpExpectedproductlengthP1P1-FCGGCGGTAAATTTTTCACAAT 1~211524P1-RAGCAGCAGTAATCTCATCC1527~1545P2P2-FCGCTTACTTACGCCAATG1310~13281570P2-RTCTGTTGATGTAAGGAGTCTG2878~2898P3P3-FGCGACCAAGGAGAATGTAA2740~2759809P3-RTGGTTACAGCATCATACTCA3549~3568P4P4-FCAGCAGACTCCTTACATCA3341~33601110P4-RCGGTAGCATAGAGGACAG4452~4470P5P5-FAAGGTTAGGCTTAGACGATT4290~43111695P5-RTCTCGGCAGTTGTAGGAT5989~6006P6P6-FGGAGTTCATCGAATCAGT5510~5528809P6-RTTCTATCTACCCGCGGTTC6320~6337
PCR产物经纯化后与T1载体连接,转入Trans1-TI感受态细胞后于含100 μg/mL氨苄青霉素的平板上培养过夜,菌液PCR筛选含重组质粒的阳性克隆,并提取质粒DNA送至上海生工生物工程有限公司测序。将测序结果在http:∥blast.ncbi.nlm.nih.gov/Blast.cgi上进行BLAST,鉴定测序的结果。利用DNAstar软件对上述6对引物扩增所得序列和RACE扩增序列进行拼接。
1.2.4 基因序列分析
采用ORF Finder(open reading frame finder)对所获得的PMMoV全基因组序列进行基因组结构分析,对PMMoV-HN1的全基因组序列进行BLAST检索,找到17个PMMoV分离物的全基因序列与7种烟草花叶病毒属病毒序列,使用DNAMAN 6.0分析25种分离物(含PMMoV-HN1)(表3)的全基因组序列一致性,利用MEGA 5.2 NJ法聚类分析法构建25种分离物(含PMMoV-HN1)全基因组序列系统进化树,使用1 000次重复的自展检验系统评价进化树拓扑结构的可靠性,其中,序列间进化距离根据Kimura-2参数遗传距离模型计算[11]。
2 结果与分析
2.1 辣椒样品PMMoV检测
对湖南10个主栽品种196份样品进行RT-PCR检测,结果发现在这些辣椒主栽品种上PMMoV检出率为9.1%~29.4%(PMMoV的检出率见表4),检测获得PCR产物(CP基因)经测序比对,其相似度为100%。
表3 本研究中涉及的病毒分离物
Table 3 Viral isolates involved in this study
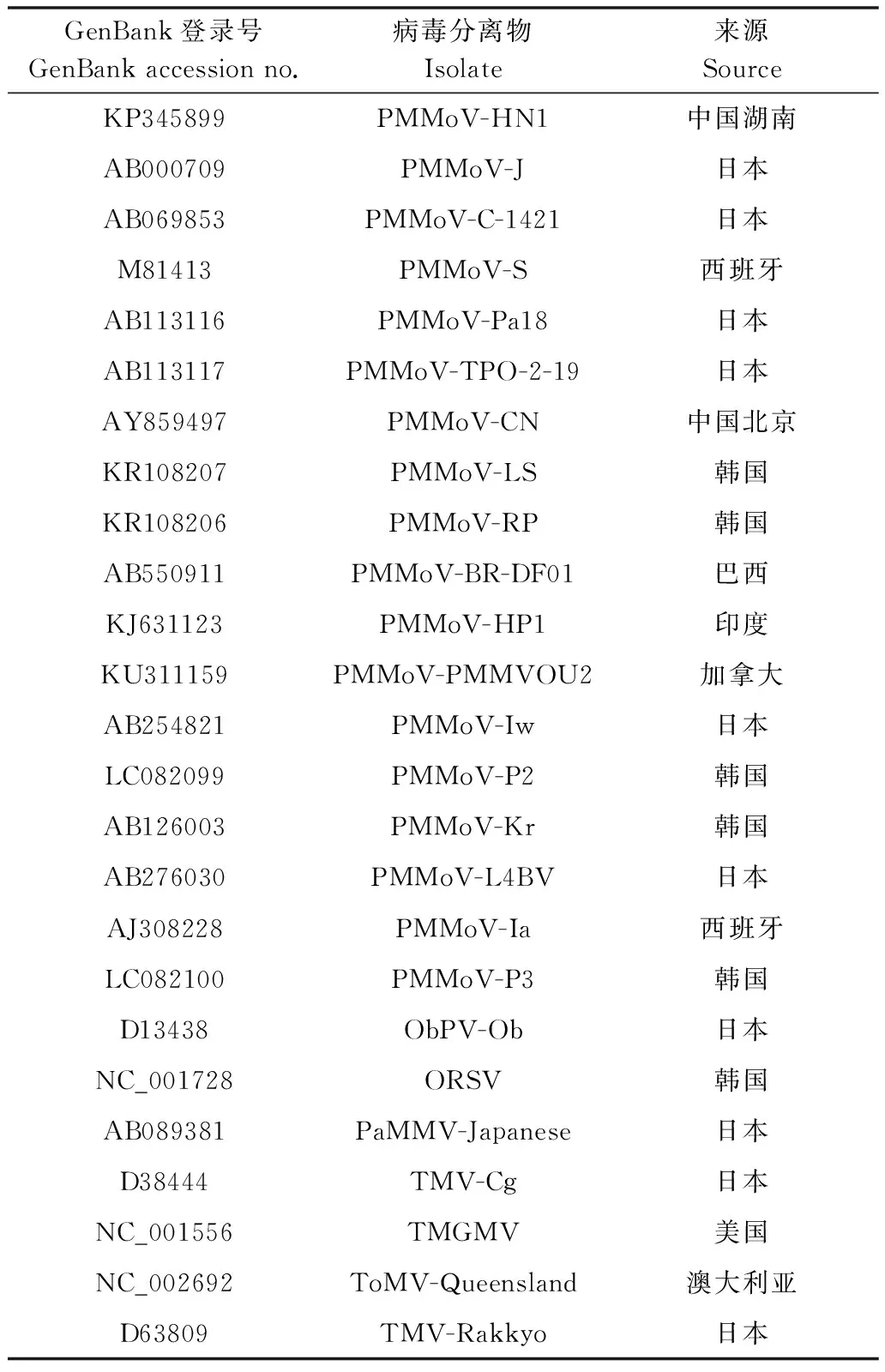
GenBank登录号GenBankaccessionno.病毒分离物Isolate来源SourceKP345899PMMoV-HN1中国湖南AB000709PMMoV-J日本AB069853PMMoV-C-1421日本M81413PMMoV-S西班牙AB113116PMMoV-Pa18日本AB113117PMMoV-TPO-2-19日本AY859497PMMoV-CN中国北京KR108207PMMoV-LS韩国KR108206PMMoV-RP韩国AB550911PMMoV-BR-DF01巴西KJ631123PMMoV-HP1印度KU311159PMMoV-PMMVOU2加拿大AB254821PMMoV-Iw日本LC082099PMMoV-P2韩国AB126003PMMoV-Kr韩国AB276030PMMoV-L4BV日本AJ308228PMMoV-Ia西班牙LC082100PMMoV-P3韩国D13438ObPV-Ob日本NC_001728ORSV韩国AB089381PaMMV-Japanese日本D38444TMV-Cg日本NC_001556TMGMV美国NC_002692ToMV-Queensland澳大利亚D63809TMV-Rakkyo日本
表4 湖南辣椒PMMoV检出率
Table 4 PMMoV detection rate in Hunan
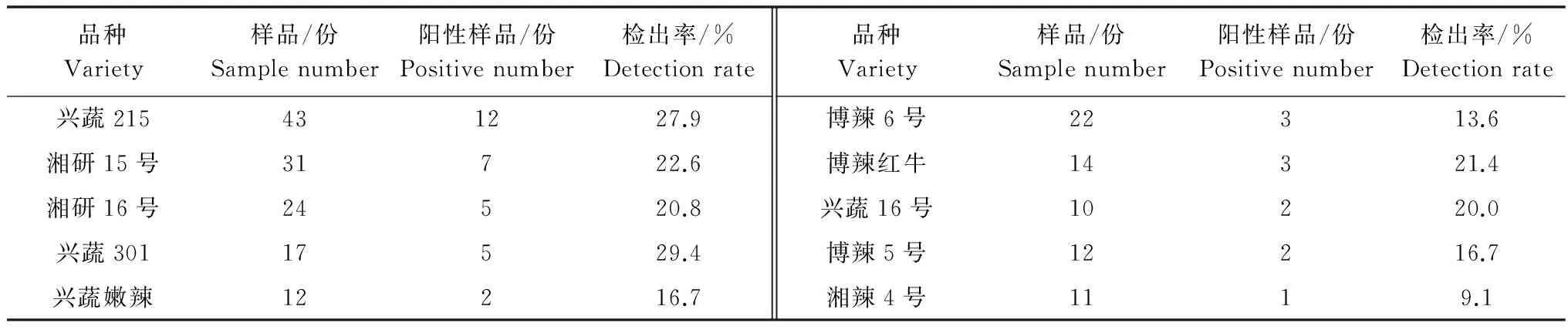
品种Variety样品/份Samplenumber阳性样品/份Positivenumber检出率/%Detectionrate品种Variety样品/份Samplenumber阳性样品/份Positivenumber检出率/%Detectionrate兴蔬215431227.9博辣6号22313.6湘研15号31722.6博辣红牛14321.4湘研16号24520.8兴蔬16号10220.0兴蔬30117529.4博辣5号12216.7兴蔬嫩辣12216.7湘辣4号1119.1
2.2 PMMoV-HN1全基因组结构分析
克隆、测序获得了PMMoV-HN1(登录号:KP345899)的全基因组序列全长6 356 bp,PMMoV-HN1的全基因组序列包含1 880个碱基A、1 777个碱基T、1 177个碱基C、1 521个碱基G,这4种碱基在PMMoV-HN1的全基因组中所占百分比依次分别为29.6%、28.0%、18.5%和23.9%。5′-非编码区(5′-UTR)和3′-非编码区(3′-UTR)分别含有69和198个碱基,其中5′-UTR存在一个序列为m7G5′pppG的甲基化核苷酸帽子结构,二级结构显示在该端形成一个发夹结构(图1)。PMMoV-HN1有4个开放阅读框架,编码4个蛋白,分别为126 kD蛋白(70~3 423 nt)、183 kD蛋白(70~4 908 nt)、28 kD蛋白(4 909~5 682 nt)和17.5 kD蛋白(5 685~6 158 nt),其中28 kD蛋白与17.5 kD蛋白由亚基因组RNA表达产生,亚基因组RNA无帽子结构。第一个开放阅读框编码的蛋白与病毒复制相关,阅读框终止于琥珀密码子UAG,同时第一个开放阅读框与第二个开放阅读框共同编码一个与病毒复制相关的蛋白,第三个开放阅读框编码的蛋白与病毒移动有关,第四个开放阅读框编码的蛋白为病毒衣壳蛋白。
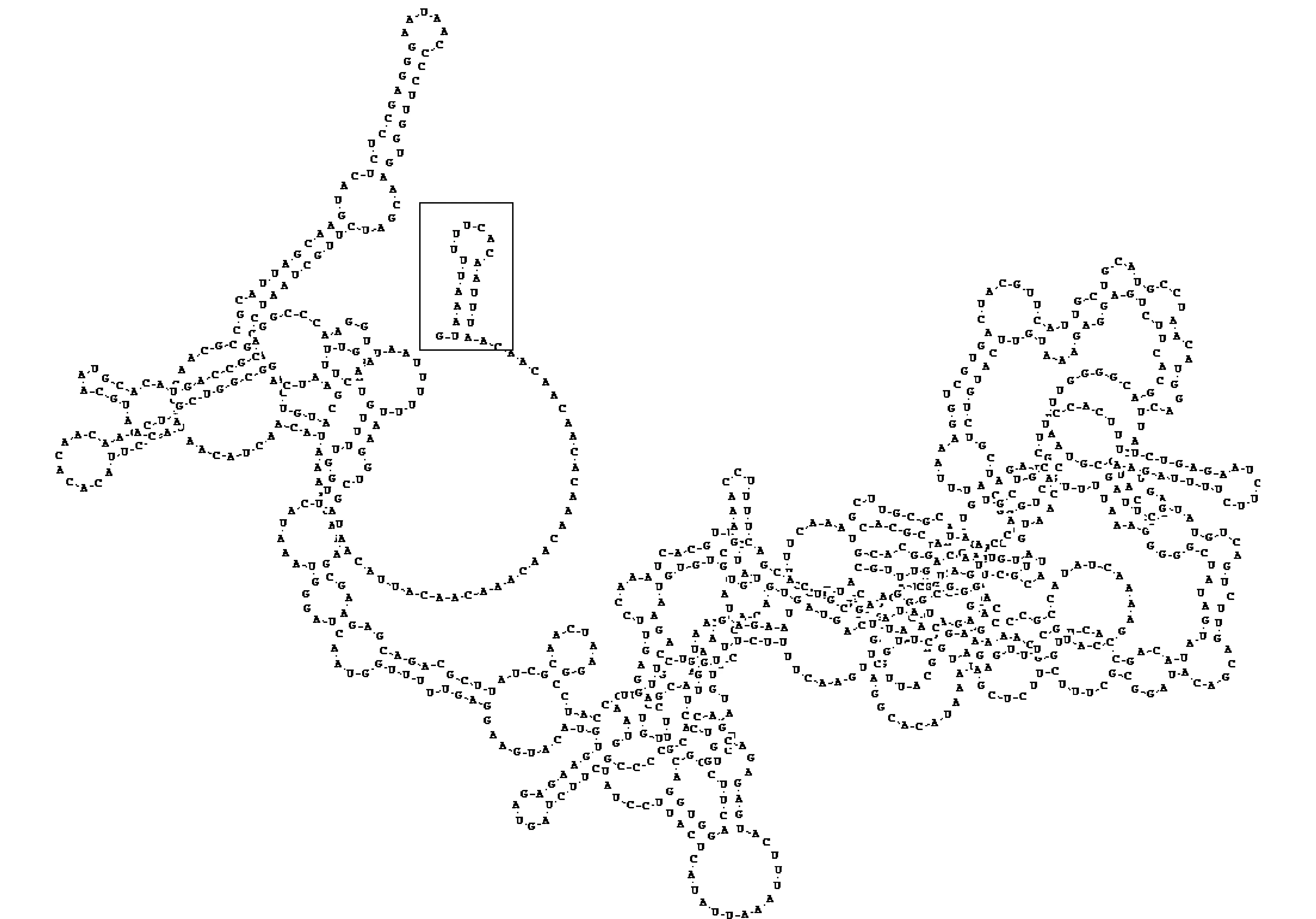
图1 PMMoV-HN1的二级结构Fig.1 Possible secondary structure of PMMoV-HN1
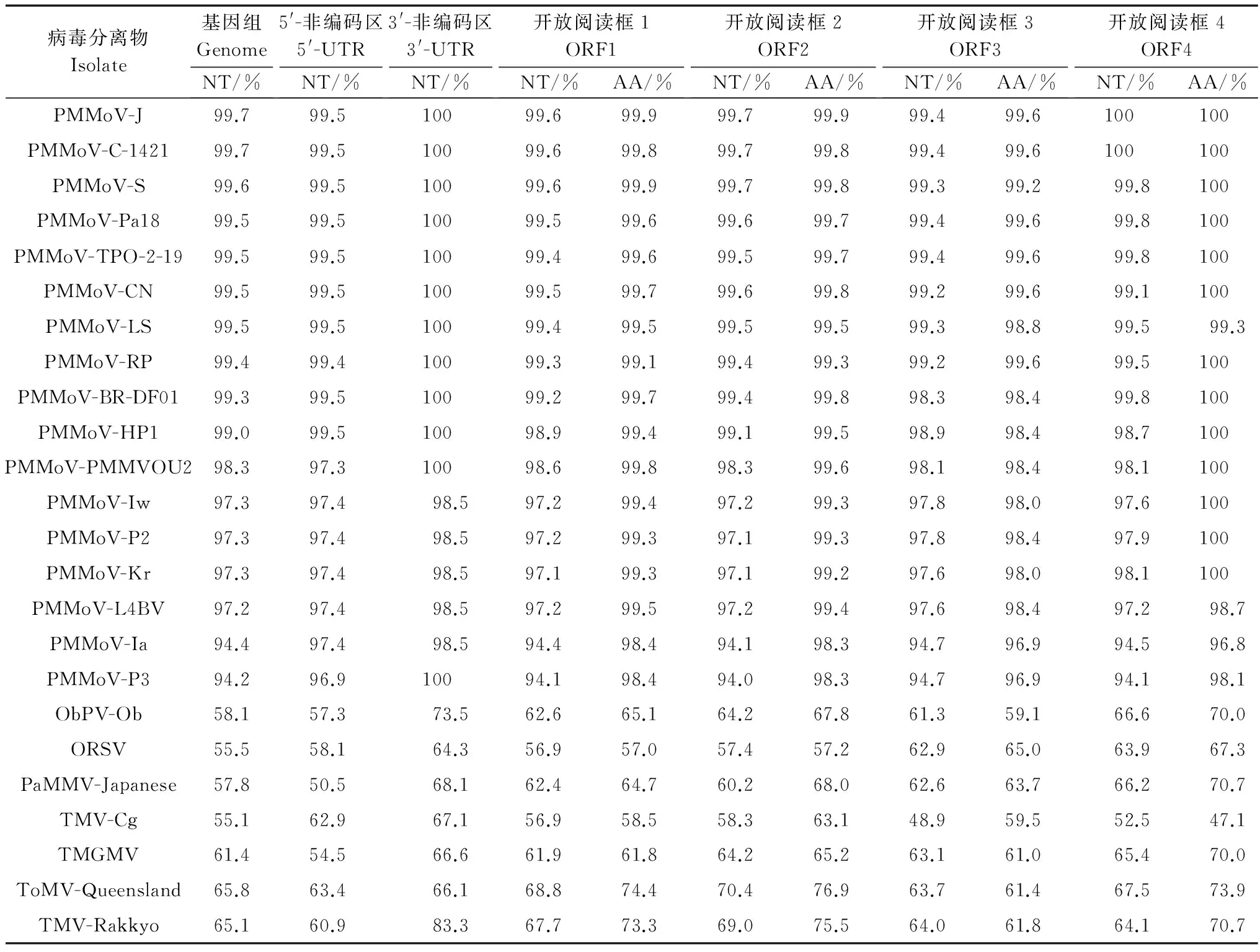
2.3 不同来源的PMMoV分离物全基因组序列分析
通过DNAMAN 6.0分析18个不同来源的PMMoV分离物及7个烟草花叶病毒属的病毒分离物的全序列基因组一致性,结果(表5)表明,PMMov-HN1与同属异种病毒的一致性仅为46.5%~65.8%,序列差异性明显;与17种PMMoV病毒分离物的一致性在94.2%~99.7%之间,其中,PMMoV-HN1与日本分离物PMMoV-J的一致性最高(99.7%),与中国分离物PMMoV-CN一致性为99.5%,与韩国分离物PMMoV-P3一致性最低(94.2%)。PMMov-HN1与17种PMMoV分离物的ORF区域氨基酸序列一致性比较结果显示,其ORF1、ORF2的一致性在98.3%~99.9%,其中与PMMoV-J仅一个氨基酸不同;ORF3的一致性在96.9%~99.6%,ORF4病毒衣壳蛋白区域除了与PMMoV-Ia的一致性为96.8%外,与其他分离物的一致性均高于98%,其中PMMoV-J、PMMoV-C-1421、PMMoV-S、PMMoV-Pa18、PMMoV-TPO-2-19、PMMoV-CN、PMMoV-RP、PMMoV-BR-DF01、PMMoV-HP1、PMMoV-PMMVOU2、PMMoV-Iw、PMMoV-P2、PMMoV-Kr编码的氨基酸序列相同。
2.4 系统进化树分析
通过MEGA5.2软件,采用NJ法构建PMMoV进化分析树显示,PMMoV分成2个大的支系群体,PMMoV-Ia与PMMoV-P3进化成一个支系,其他PMMoV分离物进化成另外一个支系,在这一支系中PMMoV-HN1(KP345899)与PMMoV-CN(AY859497)亲缘关系最近,在烟草花叶病毒属成员中,PMMoV与TMV和ToMV亲缘关系最近。
表5 PMMoV-HN1与其他来源的PMMoV分离物及同属其他病毒的一致性分析1)
Table 5 Identity analysis of PMMoV-HN1 with other PMMoV isolates and tobamovirus isolates
病毒分离物Isolate基因组GenomeNT/%5'-非编码区5'-UTRNT/%3'-非编码区3'-UTRNT/%开放阅读框1ORF1NT/%AA/%开放阅读框2ORF2NT/%AA/%开放阅读框3ORF3NT/%AA/%开放阅读框4ORF4NT/%AA/%PMMoV-J99.799.510099.699.999.799.999.499.6100100PMMoV-C-142199.799.510099.699.899.799.899.499.6100100PMMoV-S99.699.510099.699.999.799.899.399.299.8100PMMoV-Pa1899.599.510099.599.699.699.799.499.699.8100PMMoV-TPO-2-1999.599.510099.499.699.599.799.499.699.8100PMMoV-CN99.599.510099.599.799.699.899.299.699.1100PMMoV-LS99.599.510099.499.599.599.599.398.899.599.3PMMoV-RP99.499.410099.399.199.499.399.299.699.5100PMMoV-BR-DF0199.399.510099.299.799.499.898.398.499.8100PMMoV-HP199.099.510098.999.499.199.598.998.498.7100PMMoV-PMMVOU298.397.310098.699.898.399.698.198.498.1100PMMoV-Iw97.397.498.597.299.497.299.397.898.097.6100PMMoV-P297.397.498.597.299.397.199.397.898.497.9100PMMoV-Kr97.397.498.597.199.397.199.297.698.098.1100PMMoV-L4BV97.297.498.597.299.597.299.497.698.497.298.7PMMoV-Ia94.497.498.594.498.494.198.394.796.994.596.8PMMoV-P394.296.910094.198.494.098.394.796.994.198.1ObPV-Ob58.157.373.562.665.164.267.861.359.166.670.0ORSV55.558.164.356.957.057.457.262.965.063.967.3PaMMV-Japanese57.850.568.162.464.760.268.062.663.766.270.7TMV-Cg55.162.967.156.958.558.363.148.959.552.547.1TMGMV61.454.566.661.961.864.265.263.161.065.470.0ToMV-Queensland65.863.466.168.874.470.476.963.761.467.573.9TMV-Rakkyo65.160.983.367.773.369.075.564.061.864.170.7
1) NT 表示核苷酸序列的一致性;AA 表示氨基酸序列的一致性。 NT indicates the identity of the nucleotide sequence; AA indicates the identity of the amino acid sequence.

图2 不同来源的PMMoV分离物及同属其他病毒的全基因组序列系统进化树Fig.2 Phylogenetic tree of whole-genome sequences of PMMoV isolate and other tobamovirus isolates
3 讨论
PMMoV在辣椒叶片上呈现轻微的褪绿或者斑驳症状,其发病特征与烟草花叶病毒Tobaccomosaicvirus(TMV)和辣椒脉斑驳病毒Chilliveinalmottlevirus(ChiVMV)相似,但其症状严重程度随季节及环境因素变化,难以通过症状特点对病毒进行鉴定。PMMoV侵染的相关报道主要集中在华北及华中地区,最近新疆、贵州、东北、浙江等地[9-10,12-14]也相继检测出PMMoV,并提出了一些分子检测方法。然而PMMoV对湖南辣椒的侵染鲜有报道。本研究于2013年从‘兴蔬215’,‘兴蔬301’,‘湘研15号’,‘湘研16号’,‘博辣5号’,‘博辣6号’等10个湖南主要栽培品种中采集到196份疑似病毒感染的辣椒样本,进行RT-PCR检测,并测序确定PMMoV对湖南辣椒的侵染,对RT-PCR检测中扩增得到的PMMoV CP基因序列比对发现相似度为100%。随机选取PMMoV病毒侵染样本进行单斑分离后,克隆PMMoV湖南分离物基因组全序列,命名为PMMoV-HN1,并进行结构和系统进化分析,结果表明:PMMoV-HN1与中国报道的PMMoV-CN在亲缘关系上最近,表现出一定的地理相关性,与PMMoV-CN相比较,PMMoV-HN1全基因组序列的长度与PMMoV-CN序列长度完全相同,编码126 kD蛋白区域(70~3 423 nt)、编码183 kD蛋白区域(70~4 908 nt)、编码28 kD蛋白区域(4 909~5 682 nt)和编码17.5 kD蛋白区域(5 685~6 158 nt)亦在同一个位点上,而3′-UTR与PMMoV-CN完全相同[8,15],推测PMMoV-HN1与PMMoV-CN一样可以克服辣椒的L3抗性基因;同时PMMoV-HN1 CP基因序列与PMMoV-J、PMMoV-C-1421序列完全相同,而编码的氨基酸与PMMoV-J、PMMoV-C-1421、PMMoV-S、PMMoV-Pa18、PMMoV-TPO-2-19、PMMoV-RP、PMMoV-BR-DF01、PMMoV-HP1、PMMoV-PMMVOU2、PMMoV-Iw、PMMoV-P2、PMMoV-Kr所编码的氨基酸序列完全相同,进一步说明PMMoV-HN1与PMMoV-CN相同可以突破辣椒的L3抗性基因。同时通过CP基因序列和氨基酸的比对说明PMMoV的CP基因高度保守,可根据CP基因设计PMMoV特异性检测引物。二级结构显示PMMoV-HN1在5′端有一个明显的发夹结构,这个结构可能具有终止mRNA转录及保持ssRNA稳定性的作用[16],PMMoV5′-UTR基因的生物学功能有待进一步研究。检测发现侵染湖南辣椒的PMMoV CP基因序列相似度为100%,没有克隆侵染不同品种或不同地区的PMMoV的全基因,湖南地区的PMMoV病毒序列是否存在株系分化、侵染特性是否存在差异,还有待进一步研究。检测中发现PMMoV-HN1与ChiVMV和蚕豆萎蔫病毒2号Broadbeanwiltvirus2 (BBWV2)存在复合侵染现象,复合侵染的发生是否影响这些病毒对辣椒的侵染,是否导致病毒的发生规律、病毒的变异及分子进化发生变化,我们将进行进一步探索。
PMMoV影响辣椒的产量和品质,严重危害辣椒的生产。其可通过种子与染毒汁液摩擦传毒,在植物病株残体上可存活25年左右,带毒种子、感病植株和带病土壤是重要的侵染源[3]。最近的研究表明,辣椒进行工业加工后其工业产品中亦存在PMMoV[17]。同时,污水[18]与饮用水源[19]中亦存在着PMMoV。Colson等[20]从患病的人类粪便中检测到PMMoV,通过试验证明免疫反应与患者体内的PMMoV相关,同时他们将粪便中获得的PMMoV接种到寄主植物上表现出侵染症状,证明人类排泄物中的病毒RNA仍具有侵染活性,推测人类在此病毒的传播过程中可能也作为传播媒介。说明该病毒不仅可以通过种子进行传播,亦可以通过水源或人类的活动作为传播途径,从而增大了对PMMoV的监控难度。陈青等[21],郭京泽等[22]分别自我国台湾和韩国的进境辣椒种子中检测发现PMMoV。本研究检测到PMMoV-HN1,但其发生规律和传播途径还不清楚,我们正在进行相关研究。
[1] Alonso E, Garcia-Luque I, de la Cruz A, et al. Nucleotide sequence of the genomic RNA ofPeppermildmottlevirusa resistance-breaking tobamovirus in pepper [J]. Journal of General Virology, 1991, 72: 2875-2884.
[2] 张竹青. 辣椒病毒病研究[D].长沙: 湖南农业大学, 2009: 8-9.
[3] Ikegashira Y, Ohki T, Ichiki UT, et al. An immunological system for the detection ofPeppermildmottlevirusin soil from green pepper fields [J]. Plant Disease, 2004, 88(6): 650-656.
[4] 向本春, 谢浩, 崔星明, 等. 新疆辣椒轻斑驳病毒的分离鉴定[J]. 病毒学报, 1994, 10(3): 240-244.
[5] 谭根堂, 史联联, 尚惠兰, 等. 陕西线辣椒病毒病病原检测简报[J]. 辣椒杂志, 2003(4): 32-33.
[6] 黄粤, 马荣群, 岳文辉. 应用RT-PCR方法检测辣椒轻微斑驳病毒[J]. 山东农业科学, 2004(5): 56-57.
[7] 李兴红, 严红, 郭京泽, 等. 种传辣椒轻斑驳病毒病DAS-ELISA的检测[J]. 植物保护, 2005, 31(3): 66-68.
[8] Wang X, Liu F, Zhou G, et al. Detection and molecular characterization ofPeppermildmottlevirusin China [J]. Journal of Phytopathology, 2006, 154(11): 755-757.
[9] 郑兴华, 洪鲲, 杨立昌, 等. 贵州辣椒轻斑驳病毒分离物的分子鉴定[J]. 贵州农业科学, 2013(5): 30-32.
[10]鲁宇文, 彭杰军, 郑红英, 等.一种辣椒轻斑驳病毒和辣椒斑驳病毒复合侵染的线椒病毒病害分子鉴定[J]. 科技通报, 2015(9): 85-88.
[11]张卫东, 权永兵, 廖力, 等. 黄瓜绿斑驳花叶病毒广东分离物的分子鉴定[J]. 广东农业科学, 2011(20): 73-76.
[12]刘崇, 高颖, 杨洪一. 辣椒轻斑驳病毒的分子杂交检测[J]. 湖南农业大学学报(自然科学版), 2015, 41(4): 360-363.
[13]张强, 张春竹, 李克梅, 等. 辣椒轻斑驳病毒(PMMoV)新疆加工型辣椒分离物的鉴定和致病型分析[J]. 中国农学通报, 2014, 30(25): 296-302.
[14]李晓冬, 安梦楠, 王冠中, 等. 辣椒轻斑驳病毒辽宁分离物的鉴定及序列分析[J]. 沈阳农业大学学报, 2016, 47(1): 29-34.
[15]Velasco L, Janssen D, Ruiz-Garcia L, et al. The complete nucleotide sequence and development of a differential detection assay for aPeppermildmottlevirus(PMMoV) isolate that overcomes L3 resistance in pepper[J]. Journal of Virological Methods, 2002, 106(1): 135-140.
[16]魏太云, 林含新, 吴祖建, 等. 中国水稻条纹病毒两个亚种群代表性分离物全基因组核苷酸序列分析[J]. 中国农业科学, 2004, 37(6): 846-850.
[17]Peng Jiejun, Shi Bingbin, Zheng Hongying, et al. Detection of pepper mild mottle virus in pepper sauce in China [J]. Archives of Virology, 2015, 160(8): 2079-2082.
[18]Kuroda K, Nakada N, Hanamoto S, et al.Peppermildmottlevirusas an indicator and a tracer of fecal pollution in water environments: comparative evaluation with wastewater-tracer pharmaceuticals in Hanoi, Vietnam [J]. The Science of Total Environment, 2015, 506: 287-298.
[19]Haramoto E, Kitajima M, Kishida N, et al. Occurrence ofPeppermildmottlevirusin drinking water sources in Japan[J]. Applied and Environmental Microbiology, 2013, 79(23): 7413-7418.
[20]Colson P, Richet H, Desnues C, et al.Peppermildmottlevirus, a plant virus associated with specific immune responses, fever, abdominal pains, and pruritus in humans[J]. PLoS ONE, 2010, 5(4): el0041.
[21]陈青, 余芳平, 林石明, 等. 进境辣椒种子中检出辣椒轻斑驳病毒[J]. 植物检疫, 2005, 19(6): 295-296.
[22]郭京泽, 刘鹏, 崔铁军, 等. 辣椒种子中辣椒轻斑驳病毒的检测[J]. 检验检疫科学, 2007, 17(6): 32-34.
(责任编辑:田 喆)
Whole-genome sequence and structural analysis ofPeppermildmottlevirusisolate HN1
Liu Xiangning1,3, Dai Liangying1,3, Li Wei1,3, Dong Zheng1,3, Tang Qianjun1,2,3
(1.CollegeofPlantProtection,HunanAgriculturalUniversity,Changsha410128,China; 2.InstituteofVirology,HunanAgriculturalUniversity,Changsha410128,China; 3.HunanProvincialKeyLaboratoryforBiologyandControlofPlantDiseasesandInsectPests,Changsha410128,China)
Pepper samples were collected from Hunan, and identified asPeppermildmottlevirus(PMMoV). According to the previously reported gene conserved regions of PMMoV, six pairs of degenerate primers were designed. The 6 356 bp whole-genome sequence (PMMoV-HN1, accession no. KP345899) was obtained. There were four open reading frames, encoding four proteins, including 126 kD protein (70-3 423 nt), 183 kD protein (70-4 908 nt), 28 kD protein (4 909-5 682 nt) and 17.5 kD protein (5 685-6 158 nt). The 5′-non-coding region (5′-UTR) and the 3′-non-coding region (3′-UTR) contained 69 and 198 nucleotides, respectively, in which 5′-UTR had a sequence of m7G5′pppG methylation in the nucleotide cap structure. Identity analysis found that the nucleotide identity of PMMoV-HN1 and other PMMoV isolates was 94%-99%, and the identity of the encoded amino acids was 94%-99%. Phylogenetic analysis showed the closest phylogenetic relationships between PMMoV-HN1 and PMMoV-CN.
pepper; PMMoV; whole-genome sequence; sequence analysis
2016-04-21
2016-06-02
公益性行业(农业)科研专项(201303028);国家自然科学基金(31101413);湖南省烟草公司科技计划项目(13-14ZDAa04,14-16ZDAa02)
S 432.41
A
10.3969/j.issn.0529-1542.2017.02.014
* 通信作者 E-mail:tangqianjun78@163.com
