环状RNA在肝细胞癌中的差异表达研究
2017-03-29高鹏骥陈雷高杰李照王福顺冷希圣朱继业
高鹏骥,陈雷,高杰,李照,王福顺,冷希圣,朱继业
(北京大学人民医院 肝胆外科/北京市肝硬化肝癌基础研究重点实验室,北京 100044)
肝细胞癌(hepatocellular carcinoma,HCC)是常见的消化系统恶性肿瘤,严重危害患者的生命健康。尽管已经病毒性肝炎、酒精性肝病等HCC的高危因素已经明确[1-4],但是具体的发病机制尚不清楚,早期诊断的临床生物标记物也有待进一步发掘。近年来,非编码RNA领域的进展为HCC的研究带来了新的希望,研究发现长链非编码RNA和微小RNA均在HCC与癌旁组织中存在差异表达,可能与肿瘤的生物学行为密切相关[5-6]。
环状RNA(circular RNA,circRNA)是新近确认的一类特殊的非编码RNA分子,通过与疾病关联的miRNA相互作用,在疾病的发生发展过程中发挥着重要的调控作用[7-10]。文献[11-12]报道circRNA在结肠癌中存在特异性差异表达和基因调控功能,Li等[12-13]对胃癌和癌旁非肿瘤组织的研究发现,hsacirc002059在胃癌中的表达显著下调,有望成为诊断胃癌的标记物。circRNA是否在HCC中发挥作用,目前尚不明确。对其进行深入研究有可能为阐明HCC的发病机制、发现可用于早期诊断的生物学标志物取得突破。
本研究预期筛选出HCC中差异表达的circRNA,为HCC的早期诊断和治疗提供新的思路。
1 材料与方法
1.1 标本收集
3例HCC和对应的癌旁组织来源于2015年9月在北京大学人民医院接受肝癌切除手术的患者。患者的TNM分期均为I期,病灶为实性,最大径线分别为2.7、3.5、6.7cm。标本离体后10 min内取材,以生理盐水冲洗后装入冻存管投入液氮罐冷藏,随后转入-80 ℃冰箱保存至提取总RNA。HCC和癌旁组织均经病理证实。
1.2 circRNA芯片实验操作流程
总RNA提取:取100 mg标本,应用RNA抽提试剂TRIzol(Invitrogen公司)按照试剂说明分别从HCC组织和癌旁肝组织提取总RNA,应用NanoDrop ND1000进行纯度和浓度测定;RNA标记:应用RNase R(Epicentre公司)处理总RNA,去除线性RNA,富集circRNA,应用随机引物按照Arraystar Supper RNA Labeling Protocol对circRNA进行扩增并转录为荧光cRNA;芯片杂交:将荧光标记的cRNA应用RNA提取试剂盒(Qiagen公司)纯化后在circRNA芯片(8×15 K,Arraystar公司)上杂交,于分子杂交仪(Agilent公司)65 ℃孵育17 h;芯片扫描:杂交后的芯片洗片后,应用Agilent Scanner G2505C扫描。
1.3 数据采集与分析流程
原始数据提取:将芯片扫描图片导入Agilent Feature Extraction软件(版本11.0.1.1)提取原始数据;数据分析:应用R软件包对原始数据进行标准化并进行分析,两组样本间差异表达的circRNA通过变化倍数进行筛选,组间比较采用t检验。P<0.05为差异有统计学意义。
1.4 生物信息学分析
根据差异表达变化倍数筛选circRNA,应用Arraystar的miRNA目标预测软件(一款基于TargetScan[14]和miRanda[15]的软件)预测与之相互作用的miRNA,文献检索已经证实的在肿瘤发生发展中具有重要作用的miRNA,初步确定在HCC中具有重要作用的circRNA。
2 结 果
2.1 识别具有统计学意义的差异表达的circRNA
与癌旁肝组织比较,HCC组织中的circRNA表达谱发生了明显变化(图1)。图1左侧/右侧红点分别表示在HCC中表达下调/上调的满足差异倍数1.5倍以上且具有统计学意义的circRNA。

图1 circRNA火山图Figure 1 Volcano plot of circRNAs
2.2 聚类分析circRNA表达谱的差异
绘制circRNA聚类图,结果显示在HCC和癌旁组织样本间circRNA的表达谱存在差异(图2)。
2.3 HCC中差异表达circRNA的筛选
将差异表达1.5倍以上且P<0.05的circRNA确定为差异表达的circRNA。结果显示HCC组织和癌旁组织相比,1.5倍以上变化的circRNA共82条,其中上调的共21条,下调的共61条;5倍以上变化的共3条,其中上调的2条,分别为hsacirc-0043278和hsa-circ-0006220,下调的1条为hsacirc-0065214。部分差异表达的circRNA见表1。

图2 差异circRNA聚类图Figure 2 Clustering plot of the differentially expressed circRNAs
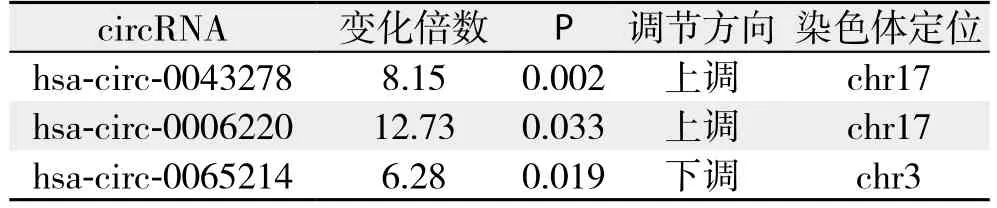
表1 部分差异表达的circRNATable 1 Part of the differentially expressed circRNAs
2.4 初步确定可能在HCC中具有重要作用的circRNA
应用Arraystar的miRNA目标预测软件对差异表达变化倍数较高的circRNA进行分析,得到可能与之相互作用的miRNA(表2)。通过文献检索发现,hsa-miR-520d-3p在胃癌和HCC组织中低表达,并与肿瘤恶性程度和化疗敏感性有关。

表2 应用Arraystar预测的可能与circRNA相互作用的miRNATable 2 The miRNAs that possiblly interacted with circRNAs predicted by Arraystar software
3 讨 论
circRNA是一类特殊的非编码RNA分子,大量存在于真核细胞,具有一定的组织、时序和疾病特异性[16-18]。circRNA又被称为miRNA海绵,含有miRNA的多个串联结合位点,通过竞争性结合miRNA来调控其表达,进而影响疾病的发生发展[19-20]。研究[21-22]显示,circRNA有望作为生物标记用于肿瘤的诊断和治疗。circRNA在HCC中的研究尚处于初级阶段,本研究结合芯片技术及生物信息学分析,希望在HCC组织中发现circRNA的差异表达,为HCC的诊断和治疗提供新的思路。
本研究中采用的circRNA芯片为Arraystar公司推出的全球首款circRNA芯片,采用特异性剪接位点探针与外切酶预处理双重保障,能够准确检测样本中circRNA的表达。借助芯片对HCC和癌旁组织提取的RNA进行分析,获得了HCC和癌旁组织中的circRNA表达谱。按照差异表达倍数≥1.5且P<0.05进行筛选,获得了82条差异表达的circRNA。其中上调的共21条,下调的共61条;5倍以上变化的共3条,其中上调的2条,下调的1条。在HCC组织中有circRNA的明显上调和下调,提示这些circRNA可能参与了HCC的发生、发展及分子调控过程。
对82条具有统计学意义的差异表达的circRNA进行分析发现,有3条circRNA差异表达倍数较高,且在3对样本中具有较好的一致性。通过生物信息学软件预测出这3条circRNA可能结合的miRNA后,通过文献检索发现,已有研究证实与hsa-circ-0043278和hsa-circ-0006220存在结合位点的hsa-miR-520d-3p在胃癌和HCC的发生发展过程中具有调控作用[23-25]。研究显示,hsa-miR-520d-3p能够抑制肿瘤的增殖和转移,hsa-miR-520d-3p水平下调将导致不良预后。因此,初步确定hsa-circ-0043278和hsa-circ-0006220可能在HCC中具有重要作用。
本研究的局限性在于仅选取了3对样本进行分析,数量相对较少,存在一定的偶然性;实验采用的芯片为Human Circlar RNA Array V1.0,有许多新近发现的circRNA未纳入;通过生物信息学预测和文献检索确定的hsa-circ-0043278和hsacirc-0006220尚未进行实验验证。下一步将增加样本量进行芯片杂交分析,对初步确定的circRNA进行qRTPCR验证,构建慢病毒载体转染HCC细胞,进行功能实验。
[1] Wanich N, Vilaichone RK, Chotivitayatarakorn P, et al. High prevalence of hepatocellular carcinoma in patients with chronic hepatitis B infection in Thailand[J]. Asian Pac J Cancer Prev, 2016,17(6):2857–2860.
[2] Joshi K, Kohli A, Manch R, et al. Alcoholic liver disease: high risk or low risk for developing hepatocellular carcinoma?[J]. Clin Liver Dis, 2016, 20(3):563–580. doi: 10.1016/j.cld.2016.02.012.
[3] Minuk GY, Bautista W, Klein J. Evidence of hepatitis B virus infection in cancer and noncancer stem cells associated with human hepatocellular carcinoma[J]. Can J Infect Dis Med Microbiol, 2016,2016:8931591. doi: 10.1155/2016/8931591.
[4] 付囡, 陈茂伟. 丙型肝炎病毒致癌机制及其防治研究进展[J]. 医学综述, 2014, 20(24):4504–4506. doi :10.3969/j.issn.1006–2084.2014.24.035.Fu N, Chan MW. Research Progress on the Carcinogenesis Mechanism of Hepatitis C Virus and Its Prevention[J].Medical Recapitulate, 2014, 20(24):4504–4506. doi :10.3969/j.issn.1006–2084.2014.24.035.
[5] 张立, 张迪, 李孝彬, 等. microRNA-616在肝癌中的表达及临床意义[J]. 中国普通外科杂志, 2016, 25(7):991–997. doi :10.3978/j.issn.1005–6947.2016.07.010.Zhang L, Zhang D, Li XB, et al. Expression of microRNA-616 in hepatocellular carcinoma and its clinical significance[J]. Chinese Journal of General Surgery, 2016, 25(7):991–997. doi :10.3978/j.issn.1005–6947.2016.07.010.
[6] 姚凯, 张光军, 黄忠明, 等. 长链非编码RNA MALAT1在肝癌中表达及功能研究[J]. 中国普通外科杂志, 2016, 25(1):90–96.doi:10.3978/j.issn.1005–6947.2016.01.014.Yao K, Zhang GJ, Huang ZM, et al. Long non-coding RNA MALAT1 expression in hepatocellular carcinoma and its action[J].Chinese Journal of General Surgery, 2016, 25(1):90–96.doi:10.3978/j.issn.1005–6947.2016.01.014.
[7] 刘彦, 周超. ciRS-7/miR-7轴调控肿瘤生长转移的研究进展[J]. 中国普通外科杂志, 2015, 24(7):1027–1031. doi: 10.3978/j.issn.1005–6947.2015.07.020.Liu Y, Zhou C. Regulative role of ciRS-7/miR-7 axis in tumor growth and metastasis: recent advances[J]. Chinese Journal of General Surgery, 2015, 24(7):1027–1031. doi: 10.3978/j.issn.1005–6947.2015.07.020.
[8] Salzman J. Circular RNA expression: Its potential regulation and function[J]. Trends Genet, 2016, 32(5):309–316. doi: 10.1016/j.tig.2016.03.002.
[9] Xuan L, Qu L, Zhou H, et al. Circular RNA: a novel biomarker for progressive laryngealcancer[J]. Am J Transl Res, 2016, 8(2):932–939.
[10] Ahmed I, Karedath T, Andrews SS, et al. Altered expression pattern of circular RNAs in primary and metastatic sites of epithelial ovarian carcinoma[J]. Oncotarget, 2016, 7(24):36366–36381. doi:10.18632/oncotarget.8917.
[11] Wang X, Zhang Y, Huang L, et al. Decreased expression of hsacirc-001988 in colorectal cancer and its clinical significances[J]. Int J Clin Exp Pathol, 2015, 8(12):16020–16025.
[12] Li P, Chen S, Chen H, et al. Using circular RNA as a novel type of biomarker in the screening of gastric cancer[J]. Clinica Chimica Acta, 2015, 444:132–136. doi: 10.1016/j.cca.2015.02.018.
[13] Li Y, Zheng Q, Bao C, et al. Circular RNA is enriched and stable in exosomes: a promising biomarker for cancer diagnosis[J]. Cell Res,2015, 25(8):981–984. doi: 10.1038/cr.2015.82.
[14] Enright AJ, John B, Gaul U, et al. MicroRNA targets in Drosophila[J]. Genome Biol, 2003, 5(1):R1.
[15] Pasquinelli AE. MicroRNAs and their targets: recognition,regulation and an emerging reciprocal relationship[J]. Nat Rev Genet, 2012, 13(4):271–282. doi: 10.1038/nrg3162.
[16] Danan M, Schwartz S, Edelheit S, et al. Transcriptome-wide discovery of circular RNAs in Archaea[J]. Nucleic Acids Res, 2012,40(7):3131–3142. doi: 10.1093/nar/gkr1009.
[17] Salzman J, Gawad C, Wang PL, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PLoS One, 2012, 7(2):e30733. doi: 10.1371/journal.pone.0030733.
[18] Cocquerelle C, Mascrez B, Hétuin D, et a1. Mis-splicing yields circular RNA molecules[J]. FASEB J, 1993, 7(1):155–160.
[19] Hentze MW, Preiss T. Circular RNAs: splicing's enigma variations[J]. EMBO J, 2013, 32(7):923–925. doi: 10.1038/emboj.2013.53.
[20] Hansen TB, Jensen TI, CIausen BH, et a1. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013,495(7441):384–388. doi: 10.1038/nature11993.
[21] Qu S, Song W, Yang X, et al. Microarray expression profile of circular RNAs in human pancreatic ductal adenocarcinoma[J].Genom Data, 2015 , 5:385–387. doi: 10.1016/j.gdata.2015.07.017.
[22] Qin ML, Liu G, Huo XS, et al. Has-circ-0001649: A circular RNA and potential novel biomarker for hepatocellular carcinoma[J].Cancer Biomarkers, 2016, 16(1):161–169. doi: 10.3233/CBM-150552.
[23] Li R, Yuan W, Mei W, et al. MicroRNA 520d-3p inhibits gastric cancer cell proliferation, migration, and invasion by downregulating EphA2 expression[J]. Mol Cell Biochem, 2014, 396(1/2):295–305.doi: 10.1007/s11010–014-2164–6.
[24] Jiang H, Dong Q, Luo X, et al. The monoclonal antibody CH12 augments 5-fluorouracil-induced growth suppression of hepatocellular carcinoma xenografts expressing epidermal growth factor receptor variant III[J]. Cancer Lett, 2014, 342(1):113–120.doi: 10.1016/j.canlet.2013.08.038.
[25] Deshpande RP, Chandra Sekhar YB, Panigrahi M, et al. SIRP Alpha Protein Downregulates in Human Astrocytoma: Presumptive Involvement of Hsa-miR-520d-5p and Hsa-miR-520d-3p[J]. Mol Neurobiol, 2016. [Epub ahead of print]
