BTB/POZ结构域蛋白7假基因1在肝细胞癌中的表达及功能的初步研究
2017-03-29陶一明胡宽汤参娥冯铁诚王志明
陶一明,胡宽,汤参娥,冯铁诚,王志明
(中南大学湘雅医院 1. 肝脏外科 2. 医学科学研究中心,湖南 长沙 410008)
BTB/POZ结构域蛋白7(BTB/POZ domaincontaining protein 7,BTBD7)基因定位染色体14q32.12,是迄今发现的一个新的癌基因,具有调控肿瘤细胞发生上皮-间质转化和肿瘤血管塑形的潜在重要功能,是肝细胞癌(HCC)新的上皮间质转化和预后标志物[1]。BTBD7的假基因(pseudogene)1、2(BTBD7P1、BTBD7P2)分别定位染色体10p13和10q25,其序列与BTBD7呈高度同源性,为高度保守的非编码序列,属于长链非编码RNA(long non-coding RNA,lncRNA)[2]。近年来研究[3-4]表明,假基因和lncRNA在肿瘤中存在异常表达,并且在肿瘤发生过程中有重要的调控作用。笔者通过实时定量RT-PCR检测HCC患者组织标本发现BTBD7P2在HCC组织中少见表达,BTBD7P1在HCC组织中表达水平显著下调,通过其表达水平与HCC临床病理特征及预后的相关性分析,BTBD7P1可能在HCC发生发展中起重要作用。通过构建BTBD7P1的慢病毒过表达载体,转染HCC细胞系Bel7404,检测转染对细胞BTBD7 mRNA、蛋白水平以及细胞增殖的影响。旨在为研究发现HCC发生发展相关新基因提供理论依据。
1 材料与方法
1.1 HCC组织标本
HCC组织样本共106例,为2009—2010年间于中南大学湘雅医院肝脏外科行手术切除的HCC患者,均已被告知并签署书面知情同意书。本课题研究内容已报中南大学湘雅医院医学伦理委员会审批备案,同意实施。HCC确诊根据2010年版世界卫生组织(WHO)病理诊断标准。
1.2 细胞系和细胞培养
HCC细胞系Bel7404购自美国保藏物中心(ATCC),生长于含体积分数为10%小牛血清、青霉素(10万U/L)和链霉素(10万U/L)的高糖DMEM培养基中,置于37 ℃、体积分数为5% CO2细胞培养箱孵育。
1.3 SYBR荧光染料法定量PCR
PCR引物碱基对序列采用美国哈佛大学引物数据库Primer Bank(http://pga.mgh. harvard.edu/primer bank)在线设计,扩增产物约100~250 bp大小,经Blast程序比对确定引物的特异性后,由大连TaKaRa生物技术公司合成。取TRIzol®Reagent(美国Thermo公司)提取新鲜HCC组织总RNA,测定浓度后合成cDNA 用于PCR扩增,用△△Ct以来计量表示该样本中目的基因的表达水平。反应体系和扩增条件参照文献。目的基因序列见表1。
1.4 慢病毒载体pLKO.1-BTBD7P1过表达质粒构建
由上海吉凯生物技术公司完成,根据BTBD7P1基因序列,合成其基因过表达序列;重组病毒包装、收集和病毒浓缩液滴度检测具体方法参照文献。得到病毒浓缩液,检测滴度约为1×109TU/mL。分装后-80 ℃冻存备用。

表1 定量PCR扩增基因序列Table 2 Oligonucleotide sequences of specific primers for quantitative real-time PCR
1.5 慢病毒质粒转染Bel7404细胞
选择Bel7404细胞为转染靶细胞[1]。分为两组:⑴ 转染LV-BTBD7P1的Bel7404细胞组(Bel7404LV-BTBD7P1);⑵ 转染空载体的Bel7404细胞(Bel7404LV-Control)作为对照组。转染条件和实验操作步骤参照文献[5]。
1.6 MTT法检测Bel7404细胞增殖抑制率
Bel7404细胞转染BTBD7P1过表达质粒和空载体后观察细胞数目,实验操作步骤参照文献[1]。每个实验重复3次取平均值。
1.7 Western blot检测BTBD7的表达
胰酶消化离心转染后的Bel7404细胞,4 ℃预冷的PBS洗涤,800 r/min,离心2次收集细胞,RIPA细胞裂解液提取细胞总蛋白,检测蛋白质浓度。电泳、转膜、封闭液实验操作步骤参照文献。BTBD7抗体工作浓度为1:1 000。Bio-Rad Scan软件计算条带的灰度值和内参α-tubulin灰度值的作为蛋白表达量。每个实验重复3次取平均值。

图1 定量PCR检测BTBD7P1与BTBD7P2的表达Figure 1 BTBD7P1 and BTBD7P2 expressions detected by real time PCR
1.8 统计学处理
2 结 果
2.1 HCC组织中BTBD7P1 mRNA表达水平下调
106例配对HCC组织中BTBD7P1 mRNA的表达水平明显低于邻近癌旁肝组织中的表达水平[(0.71±0.16) vs.( 2.14±1.44),P<0.05];HCC组织及癌旁肝组织中少见BTBD7P2 mRNA表达(图1)。
2.2 BTBD7P1表达与HCC临床病理特征的关系
根据癌组织中BTBD7P1相对表达量低于癌旁肝组织2倍为标准分为低表达和高表达。BTBD7P1低表达与肿瘤大小、卫星灶、分化程度、静脉血管侵犯、出血坏死和HCC分期有关(均P<0.05)(表2)。
2.3 BTBD7P1 mRNA表达与HCC预后的关系
本组BTBD7P1 mRNA高表达患者1、3、5年总体生存率分别为93%、74%、53%,BTBD7P1 mRNA低表达患者1、3、5年总体生存率分别为77%、44%、18%,两组间差异有统计学意义(P<0.05);BTBD7P1 mRNA高表达患者1、3、5年无瘤生存率分别为74%、57%、38%,BTBD7P1 mRNA低表达患者1、3、5年无瘤生存率分别为22%、12%、5%,两组间差异有统计学意义(P<0.05)(图2)。

表2 BTBD7P1 mRNA表达与HCC临床病理特征相的关系[n(%)]Table 2 Relations of BTBD7P1 mRNA expression with clinicopathologic characteristics of HCC [n (%)]

图2 不同BTBD7P1 mRNA表达水平患者生存情况比较 A:总体生存曲线;B:无瘤生存曲线Figure 2 Comparison of the survival conditions between patients with different BTBD7P1 mRNA expression levels A: Overall survival curves; B: Tumor-free survival curves
2.4 过表达BTBD7P1抑制HCC细胞增殖
活细胞计数检测Bel7404细胞增殖的变化(图3)。转染LV-BTBD7P1的Bel7404细胞组(Bel7404LV-BTBD7P1)与对照组细胞(Bel7404LV-Control)比较,细胞数明显减少(P<0.05)。
2.5 BTBD7P1抑制HCC细胞的BTBD7 mRNA表达
通过RT-PCR方法检测Bel7404LV-BTBD7P1和Bel7404LV-Control细胞中BTBD7 mRNA表达水平,结果显示,Bel7404LV-BTBD7P1细胞中BTBD7 mRNA表达水平较Bel7404LV-Control细胞明显下调(P<0.05)(图4)。通过Western blot检测两组细胞中BTBD7蛋白表达变化,结果显示,两组细胞BTBD7蛋白表达无统计学差异(P>0.05)(图5)。

图3 MTT检测结果Figure 3 Results of MTT assay

图4 BTBD7 mRNA表达检测Figure 4 Measurement of BTBD7 mRNA expression
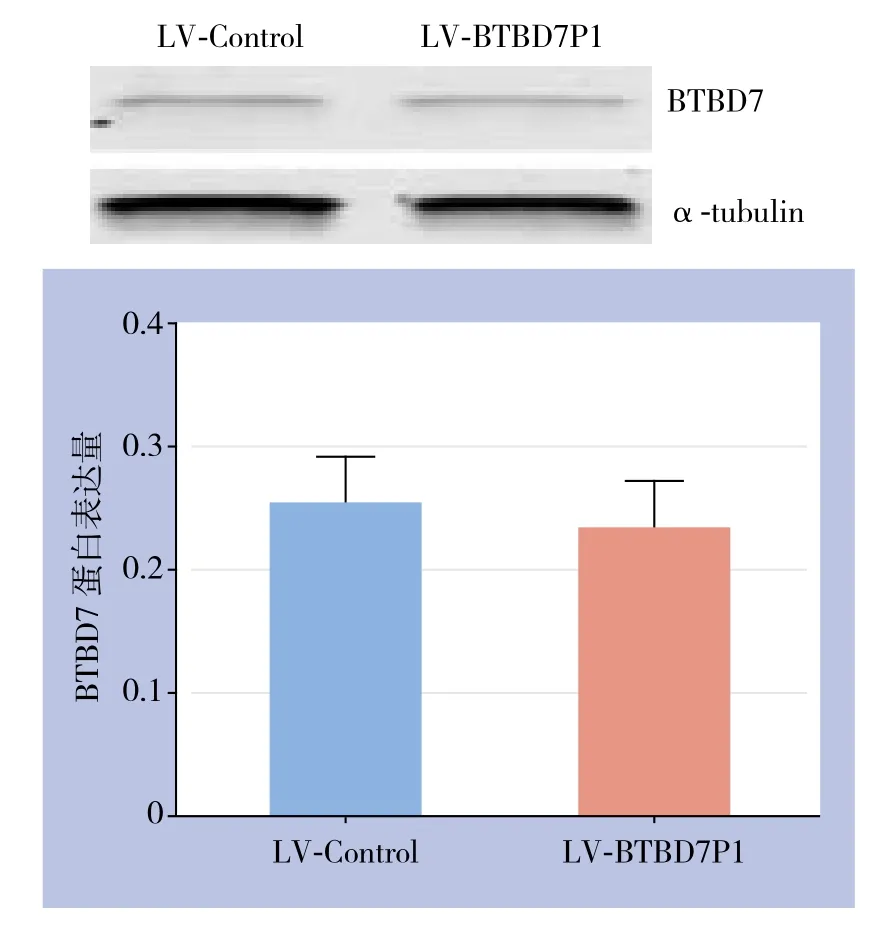
图5 BTBD7蛋白表达检测Figure 5 Measurement of BTBD7 protein expression
3 讨 论
HCC是我国常见恶性肿瘤之一。进一步探索研究新基因的功能与HCC发生发展的关系,对揭示HCC发生、发展的精确分子机制、设计合理的治疗药物及判断预后,进一步提高我国HCC的治疗水平具有重要意义。
BTBD7基因是近年来新发现的一种上皮间质转化调控关键基因,定位于人类染色体14q32.12区域,约1 233 bp大小,其结构中包括BTB/POZ结构域,CAAT增强蛋白β(C/EBP-β),GATA和AP-1转录子位点[6]。BTBD7首次被发现是因其可引导上皮细胞发生“分支形态”(branching morphogenesis)改变过程中发挥了重要作用[7]。已有的研究[8]显示,BTBD7基因也称FUP1,最先是从肝脏细胞中克隆发现被鉴定,其在肝脏组织表达有相对特异性,并被证实在HCC细胞系中表达上调。笔者[1]前期研究中证实:BTBD7表达诱导HCC细胞发生上皮间质转化表型,并激活下游RhoC-ROCK2-FAK通路信号,促使HCC细胞分泌较多的基质金属蛋白酶2/9(MMP-2/9)。目前调控BTBD7表达研究鲜见报道。
新近研究[9]表明,假基因异常表达可能在肿瘤发生中起重要作用。部分假基因对其功能基因有调节作用[10]。而且特异性假基因表达可作为肿瘤的预测因子,诸如AOC4P[11]、OCT4[12]、INTS6P1[13]等。目前关于BTBD7假基因(BTBD7P1和BTBD7P2)在HCC及其他恶性肿瘤发生发展关系未见报道。本研究结果显示,在HCC患者组织中的BTBD7P1表达水平显著低于癌旁组织,临床病理相关性分析发现BTBD7P1低表达水平与HCC的侵袭转移及预后密切相关,提示BTBD7P1的表达水平与HCC的发生可能存在一定关系。目前BTBD7P1在HCC表达下调的原因尚不清楚,可能涉及缺氧微环境的调节[14]或表观遗传调控机制[15]。
本研究发现BTBD7P1可抑制亲本基因BTBD7 mRNA的表达,但对BTBD7蛋白表达无影响,提示BTBD7P1可能扮演了内源性小RNA(esiRNA)[16]或内源性竞争环状RNA(ceRNA)重要角色[17]。BTBD7P1具有与亲本功能基因BTBD7编码的蛋白质一样的功能,即抑制HCC细胞生长。说明BTBD7P1不需要通过BTBD7蛋白发挥作用,但具体调控机理尚有待进一步探索。已有研究证据表明,假基因可通过竞争性地结合miRNA调控功能基因的表达,从而抑制或者促进肿瘤的发生发展[18]。提示上述作用分子机制可能是BTBD7 mRNA及BTBD7P1转录产物竞争某同一种或几种microRNA,激活不同信号通路发挥功能得以实现,其中哪些是起决定性因素的microRNA调节或者可能参与的信号途径值得进一步深入研究[19-20]。
[1] Tao YM, Huang JL, Zeng S, et al. BTB/POZ domain containing protein 7: Epithelial mesenchymal transition promoter and prognostic biomarker of hepatocellular carcinoma[J]. Hepatology,2013, 57(6):2326–2337. doi: 10.1002/hep.26268.
[2] Han L, Yuan Y, Zheng S, et al. The Pan-Cancer analysis of pseudogene expression reveals biologically and clinically relevant tumour subtypes[J]. Nat Commun, 2014, 5:3963. doi: 10.1038/ncomms4963.
[3] Poliseno L, Marranci A, Pandolfi PP. Pseudogenes in human cancer[J]. Front Med (Lausanne), 2015, 2:68. doi: 10.3389/fmed.2015.00068.
[4] Dickson I. Hepatocellular carcinoma: A role for lncRNA in liver cancer[J]. Nat Rev Gastroenterol Hepatol, 2016, 13(3):122–123.doi: 10.1038/nrgastro.2016.21.
[5] Tao Y, Hu K, Tan F, et al. SH3-domain binding protein 1 in the tumor microenvironment promotes hepatocellular carcinoma metastasis through WAVE2 pathway[J]. Oncotarget, 2016,7(14):18356–18370. doi: 10.18632/oncotarget.7786.
[6] Xu L, Wei Y, Reboul J, et al. BTB proteins are substrate-specific adaptors in an SCF-like modular ubiquitin ligase containing CUL-3[J]. Nature, 2003, 425(6955):316–321.
[7] David R. Development: BTBD7 branches out[J]. Nat Rev Mol Cell Biol, 2010, 11(9):605. doi: 10.1038/nrm2963.
[8] Pan W, Zhang Q, Xi QS, et al. FUP1, a gene associated with hepatocellular carcinoma, stimulates NIH3T3 cell proliferation and tumor formation in nude mice[J]. Biochem Biophys Res Commun,2001, 286(5):1033–1038.
[9] Kalyana-Sundaram S, Kumar-Sinha C, Shankar S, et al. Expressed pseudogenes in the transcriptional landscape of human cancers[J].Cell, 2012, 149(7):1622–1634. doi: 10.1016/j.cell.2012.04.041.
[10] Chen X, Zhu H, Wu X, et al. Downregulated pseudogene CTNNAP1 promote tumor growth in human cancer by downregulating its cognate gene CTNNA1 expression[J]. Oncotarget, 2016,7(34):55518–55528. doi: 10.18632/oncotarget.10833.
[11] Wang TH, Lin YS, Chen Y, et al. Long non-coding RNA AOC4P suppresses hepatocellular carcinoma metastasis by enhancing vimentin degradation and inhibiting epithelial-mesenchymal transition[J]. Oncotarget, 2015, 6(27):23342–23357.
[12] Wang L, Guo ZY, Zhang R, et al. Pseudogene OCT4-pg4 functions as a natural micro RNA sponge to regulate OCT4 expression by competing for miR-145 in hepatocellular carcinoma[J].Carcinogenesis, 2013, 34(8):1773–1781. doi: 10.1093/carcin/bgt139.
[13] Peng H, Ishida M, Li L, et al. Pseudogene INTS6P1 regulates its cognate gene INTS6 through competitive binding of miR-17–5p in hepatocellular carcinoma[J]. Oncotarget, 2015, 6(8):5666–5677.
[14] Wang TH, Yu CC, Lin YS, et al. Long noncoding RNA CPS1-IT1 suppresses the metastasis of hepatocellular carcinoma by regulating HIF-1α activity and inhibiting epithelial-mesenchymal transition[J]. Oncotarget, 2016, 7(28):43588–43603. doi: 10.18632/oncotarget.9635.
[15] Scarola M, Comisso E, Pascolo R, et al. Epigenetic silencing of Oct4 by a complex containing SUV39H1 and Oct4 pseudogene lncRNA [J]. Nat Commun, 2015, 6:7631. doi: 10.1038/ncomms8631.
[16] Chan WL, Chang JG. Pseudogene-derived endogenous siRNAs and their function[J]. Methods Mol Biol, 2014, 1167:227–239. doi:10.1007/978–1-4939–0835-6_15.
[17] Tay Y, Rinn J, Pandolfi PP. The multilayered complexity of ceRNA crosstalk and competition[J]. Nature, 2014, 505(7483): 344–352.doi: 10.1038/nature12986.
[18] Rutnam ZJ, Du WW, Yang W, et al. The pseudogene TUSC2P promotes TUSC2 function by binding multiple microRNAs[J]. Nat Commun, 2014, 5:2914. doi: 10.1038/ncomms3914.
[19] Schmitt AM, Chang HY. Long Noncoding RNAs in Cancer Pathways[J]. Cancer cell, 2016, 29(4):452–463. doi: 10.1016/j.ccell.2016.03.010.
[20] Shi X, Nie F, Wang Z, et al. Pseudogene-expressed RNAs: a new frontier in cancers[J]. Tumor Biology, 2016, 37(2):1471–1478. doi:10.1007/s13277–015-4482-z.
