乌拉尔图小麦全基因组NBS-SSR位点分析及标记开发
2017-03-28乔麟轶畅志坚张晓军刘静朱艳詹海仙郭慧娟李欣
乔麟轶,畅志坚,张晓军,刘静,朱艳,詹海仙,郭慧娟,李欣*
(1.山西大学 生命科学学院, 山西 太原 030006; 2.山西省农业科学院 作物科学研究所 / 作物遗传与分子改良山西省重点实验室,山西 太原0300311; 3.山西大学 生物工程学院, 山西 太原 030006)
乌拉尔图小麦全基因组NBS-SSR位点分析及标记开发
乔麟轶1,2,畅志坚2,张晓军2,刘静3,朱艳3,詹海仙2,郭慧娟2,李欣2*
(1.山西大学 生命科学学院, 山西 太原 030006; 2.山西省农业科学院 作物科学研究所 / 作物遗传与分子改良山西省重点实验室,山西 太原0300311; 3.山西大学 生物工程学院, 山西 太原 030006)
[目的]本研究旨在诊断乌拉尔图小麦NBS家族所在基因组序列的SSR位点,并为抗源的发掘和利用开发分子标记。[方法]利用NBS隐马模型文件检索数据库获得目标序列,通过软件诊断序列SSR位点并开发标记,利用乌拉尔图小麦抗白粉病材料UR207和感病材料UR203进行标记验证。[结果]本研究从乌拉尔图小麦全基因组中分离出463个NBS基因,分为N、CN、NL和CNL共4类。从NBS所在scaffold序列上发现4 292个SSR位点,以二碱基重复位点最多,占78.1%。随机开发了42个分布于各染色体臂的TuNBS-SSR标记,有12个标记在UR207和UR203间存在多态性。[结论]本研究从乌拉尔图小麦全基因组中诊断出4 292个NBS-SSR位点,开发的12个标记在抗感材料间有多态性,可用于乌拉尔图小麦材料抗病性鉴定和抗病基因筛选。
乌拉尔图小麦; NBS类基因; SSR标记; 抗病
植物在生长过程中不断面临着真菌、细菌、病毒等病原体的侵袭,逐渐形成了一系列复杂的防御机制,尤其是植物抗病蛋白对病原体的特异性识别。目前从植物中分离出约100多个抗病蛋白,其中80%以上都属于NBS (Nucleotide binding site) 家族。
NBS家族由约320个氨基酸组成,其结构域中高度保守的部分通过与ATP或GTP作用使蛋白构象发生变化,进而参与抗病信号的传导[1]。NBS蛋白的C端通常由数量不等的LRR (leucine rich repeats) 结构域组成,其N端往往连接一个TIR (toll and interleukin-1 receptor) 结构或者是一个CC (coiled-coil) 结构域。TIR在对病原物的识别以及各自免疫系统的信号传导中起重要作用,含有这类结构的TIR-NBS基因有N[2]、RPP1[3]等;而CC结构可通过其异亮氨酸的作用,促进蛋白质之间的特异相互作用,这类CC-NBS基因包括Pm3b[4]、Lr10[5]等。
乌拉尔图小麦(Triticumurartu, 2n=14, AA)为野生一粒小麦,是四倍体和六倍体小麦A染色体组的祖先种,具有优质、抗逆等优异性状,因经历过基因扩张,比其他二级基因源的种属以及近缘种属含有更多的抗病基因[6],对其NBS家族进行分离和研究对于普通小麦抗性改良具有重要意义。乌拉尔图小麦测序草图的公布[7],使得从全基因水平揭示重要基因家族的功能成为可能。本研究采用生物信息学方法从乌拉尔图小麦全基因组中分离了NBS家族序列,对NBS所在基因组序列上的SSR位点进行了诊断,并开发部分分子标记在2个乌拉尔图小麦的白粉病抗感材料间进行了验证。本研究结果为挖掘和利用乌拉尔图小麦中的抗病基因资源及功能研究提供参考。
1 材料与方法
1.1 植物材料
苗期抗性鉴定材料为乌拉尔图小麦UR207和UR203,由中国农业科学院作物科学研究所贾继增研究员惠赠。抗性鉴定所用白粉病菌株为 E09,由中国农业科学院植物保护研究所段霞瑜研究员惠赠。
1.2 苗期鉴定
将UR207、UR203和小麦对照材料台长29各20粒分别播于纸杯内,待植株生长至一叶一心期时,用毛笔将白粉菌菌株E09的分生孢子轻扫至小麦叶片上,之后转入人工培养箱,设温度15~20 ℃,光照时间 12~14 h。在7~10 d后当感病对照台长29充分发病时, 采用0~4级标准调查材料第1片叶的反应型[8]。
1.3 序列的分离和检测
从GIGA数据库(http://gigadb.org/)中下载乌拉尔图全基因组数据建立本地BLAST数据库,利用NBS隐马模型文件 (Pfam登录号:PF00931)检索本地数据库。利用SMART工具对检索结果进行结构域的检查。
1.4 SSR位点诊断和引物设计
利用SSRhurnter软件查找NBS所在scaffold序列的SSR位点。查找SSR的设定为:每重复单元碱基数为2~5,重复次数≥5。利用Primer5软件对SSR位点设计引物,由生工生物工程(上海)有限公司合成。
1.5 标记检测
采用 SDS法[9]提取基因组总DNA。PCR总体系为15 μL,含1.5 μL 10×buffer (TaKaRa)、0.2 mmol·L-1dNTP、1 U Taq酶(TaKaRa)、0.25 μmol·L-1引物和100 ng模板DNA。反应扩增程序为94 ℃变性5 min;94 ℃变性45 s,58 ℃复性45 s,72 ℃延伸1 min,36个循环;72 ℃延伸10 min。扩增产物用8%非变性聚丙烯酰胺凝胶电泳,硝酸银染色后观察照相。
2 结果与分析
2.1 乌拉尔图小麦NBS序列的分布及分类
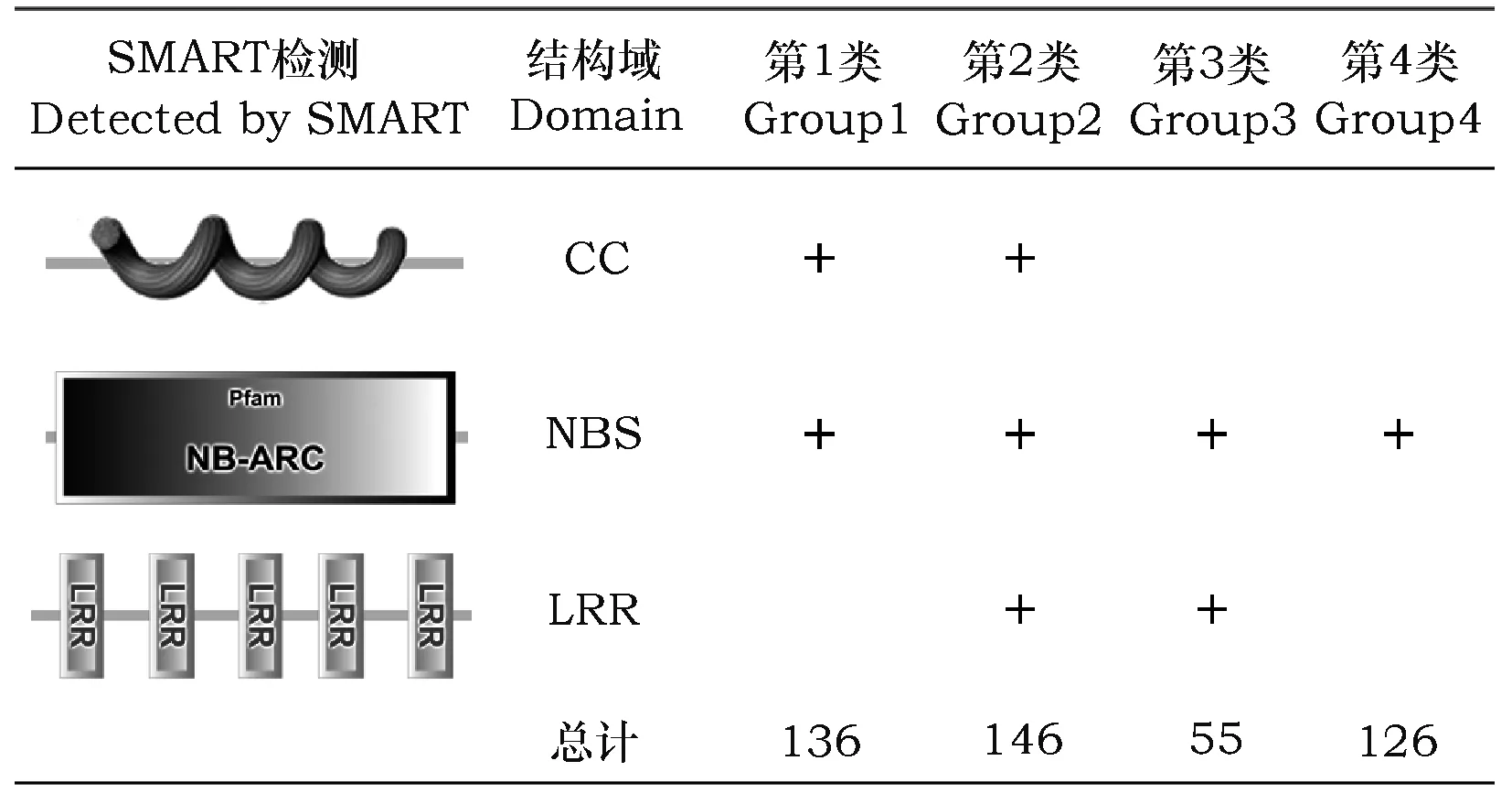
从本地乌拉尔图小麦数据库中筛选出463条完整蛋白序列,根据染色体位置信息将其定位于乌拉尔图小麦的1A~7A染色体上,分布数目依次为81、77、55、69、45、39和97,其中7A染色体上分布的NBS序列最多, 6A染色体上分布最少。检索出的463条蛋白序列均包含NBS保守结构域,根据其C端和N端是否包含其他结构域,将NBS序列分为CC-NBS-LRR、NBS-LRR、CC-NBS和NBS共4类(表1)。
2.2 TuNBS-SSR位点诊断及标记开发
查找TuNBS所在scaffold序列的SSR位点,在365条scaffold序列上共检测到4 292个SSR位点,分布于1A~7A染色体,分布比例依次为15.6%、16.6%、9.4%、18.9%、9.2%、10.1%和20.2% (图1A)。其中二碱基重复位点最多,占78.1%;五碱基重复位点最少,仅9个 (图1B)。
表1 乌拉尔图小麦TuNBS序列的结构分类
Table 1 Structural classification of TuNBS protein sequences
在每条染色体的长短臂上分别随机取3个位点,根据这些SSR位点的侧翼序列设计引物,共开发TuNBS-SSR标记42对(表2)。
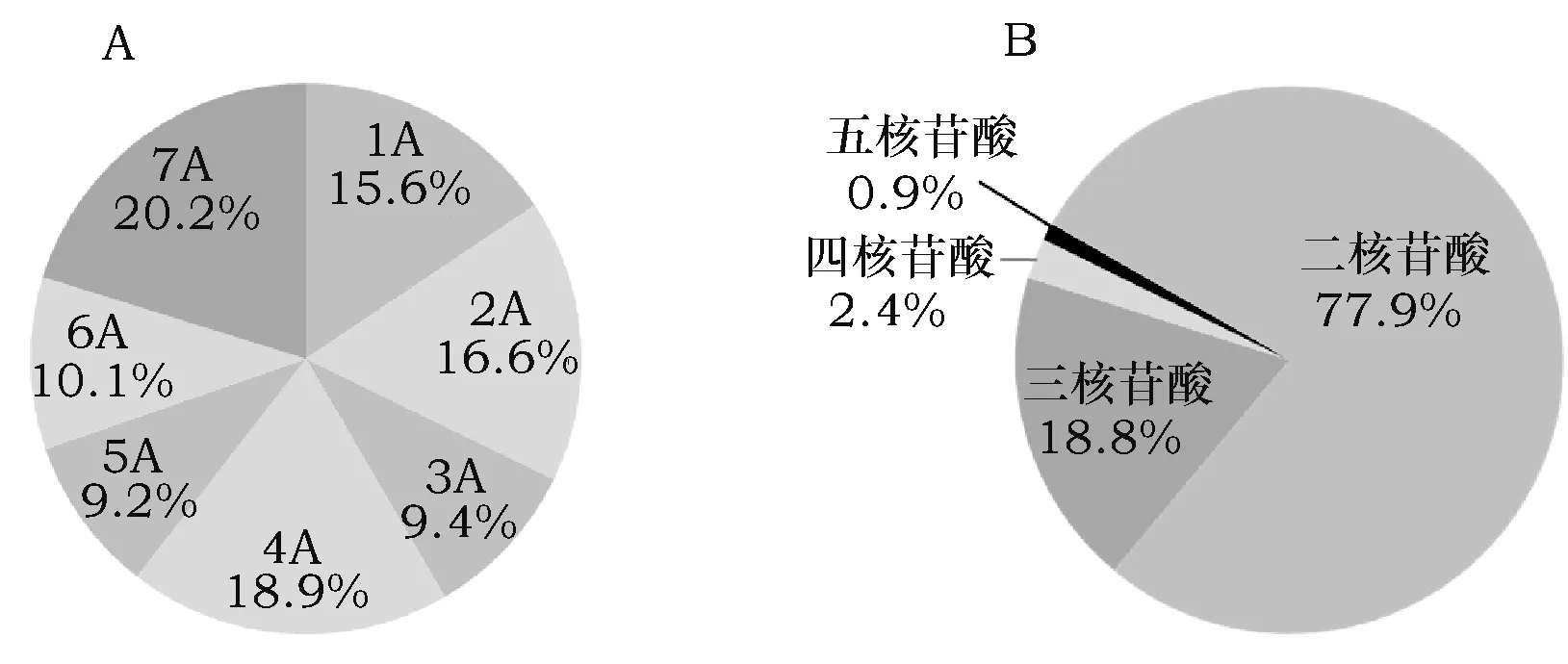
图1 乌拉尔图小麦全基因组NBS-SSR位点诊断Fig.1 The genome-wide diagnosis of NBS-related SSR loci in T. urartu
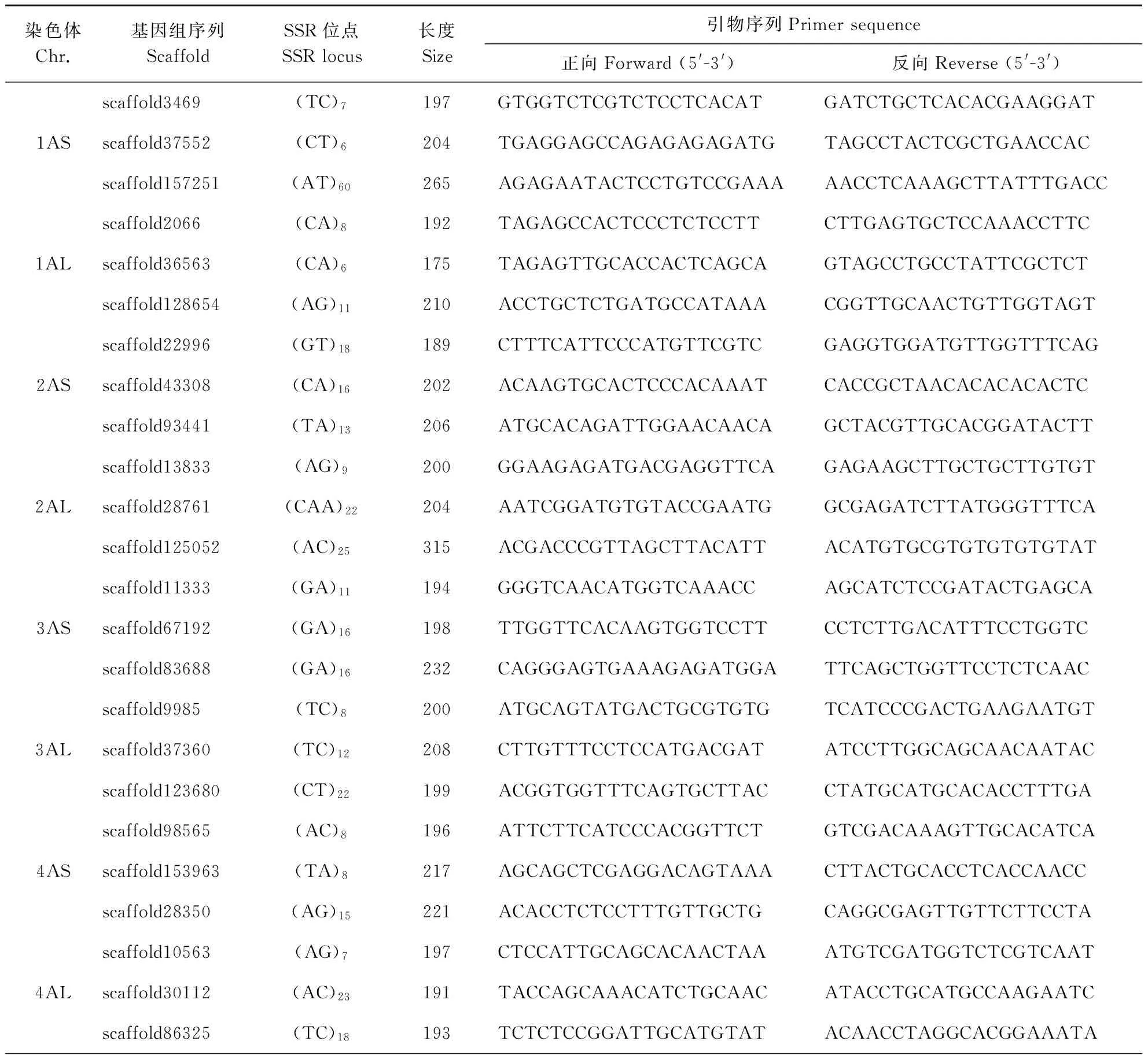
表2 42对乌拉尔图小麦NBS-SSR标记
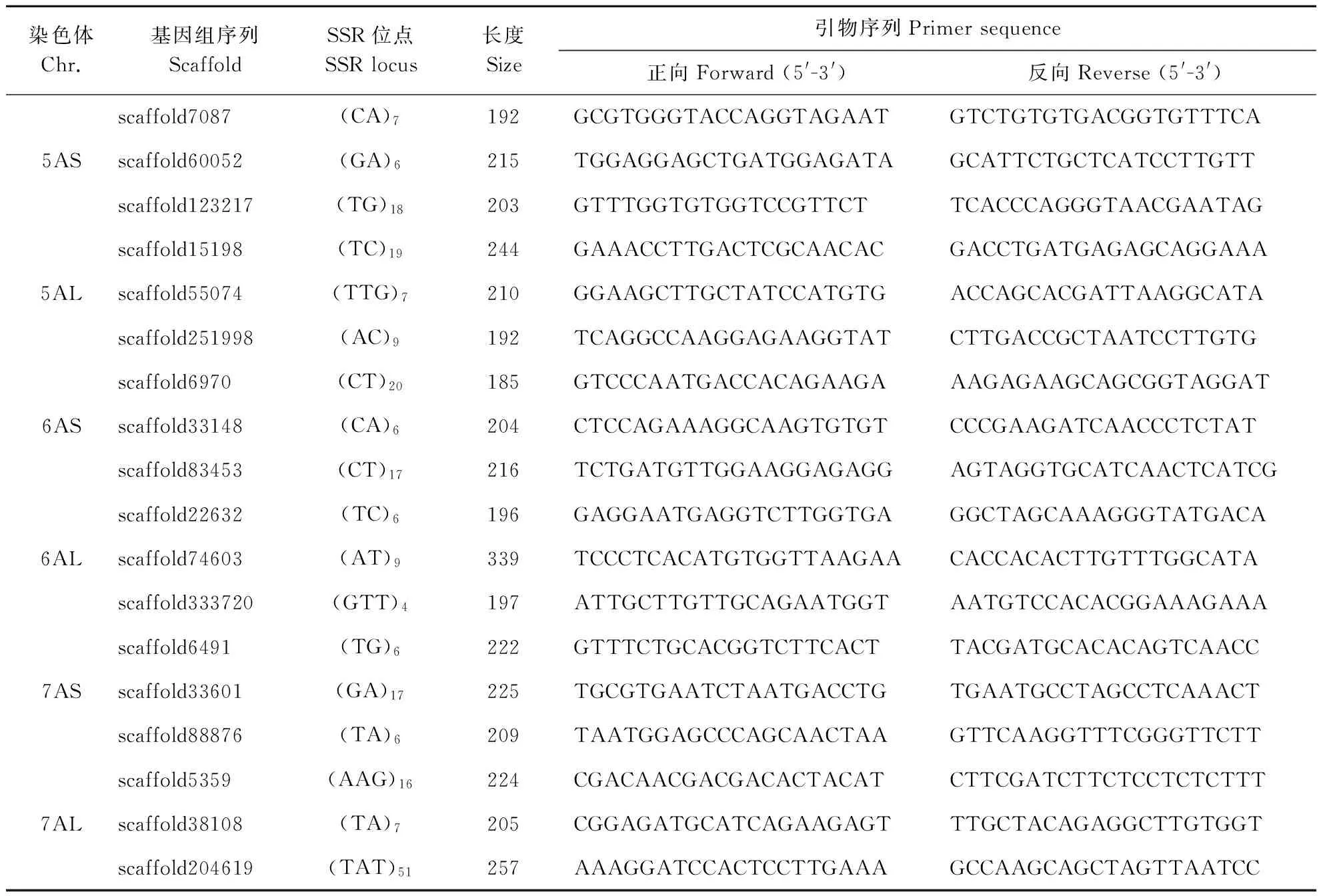
表2(续)
2.3 TuNBS-SSR标记验证
在对照材料台长29发病级别达4级时,鉴定乌拉尔图小麦UR207和UR203的抗性。结果显示UR207对白粉菌小种E09表现为近免疫(IT=0),UR203表现为感病(IT=4)。将开发的42个TuNBS-SSR标记扩增UR207和UR203的DNA,有12个标记在群体间表现出多态性 (图2)。
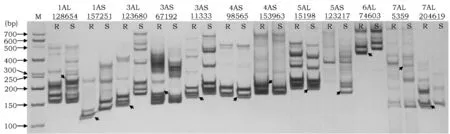
图2 TuNBS-SSR位点验证Fig.2 Diagnosis of NBS-related SSR loci in T. urartu 注:图中箭头所指为差异条带.Note: The arrows in the figure refer to differential electrophoretic bands.
3 讨论
随着测序技术的不断提高,许多植物全基因组中的NBS基因数目及种类已确定。如拟南芥149个[10],高粱211个[11],大豆319个[12]、大麦420个[13],水稻535个[14]等。本研究从乌拉尔图小麦中筛选出463个NBS基因。与大麦NBS基因数目最为接近,与水稻次之,这可能是由于乌拉尔图小麦与大麦和水稻的亲缘关系较其他物种更近,三者基因组经历了较为相似的进化过程,最终形成了成员数目相当的NBS家族。另外已有研究表明植物界TIR-NBS起源较早,但在陆生植物中TIR结构开始缺失,进化至单子叶植物中大幅度减少,因此目前大部分单子叶植物的NBS蛋白不包含TIR结构[15]。本研究从乌拉尔图小麦基因组中分离的均为non-TIR-NBS蛋白,这与高粱、大麦等单子叶植物结果一致。
本研究分离的全基因组TuNBS为乌拉尔图小麦抗病基因的筛选构建了一个候选序列库,也为小麦抗病育种中广泛发掘外源抗病基因提供了参考。我们还从TuNBS所在的基因组序列上诊断出4 292个SSR位点,并随机合成42个标记对抗感材料进行扩增,其中有12个标记表现出多态性。因此我们预计可开发出1 000余个具有丰富多态性的NBS-SSR标记,这些标记可用于乌拉尔图小麦基因组或小麦A基因组的分子图谱构建和抗病基因定位研究中。另外,本研究开发的每个SSR标记均与TuNBS序列位于同一条scaffold序列上,可作为抗病基因的共分离标记使用,适合用于抗病育种过程中的分子标记辅助选择。
4 结论
从乌拉尔图小麦基因组中分离出463条NBS序列,包含N、CN、NL和CNL共4类结构,从NBS所在基因组序列中诊断出4 292个NBS-SSR位点,随机开发了42个分布于各染色体的标记,其中有12个标记在抗感材料间有多态性,可用于乌拉尔图小麦材料抗病性鉴定和抗病基因筛选。
[1]Porter B W, Paidi M, Ming R, et al. Genome-wide analysis ofCaricapapayareveals a smallNBSresistance gene family[J]. Mol Genet Genomics,2009,281(6):609-626.
[2]Whitham S, Dinesh-Kumar S P, Choi D, et al. The product of the tobacco mosaic virus resistance geneN: similarty to toll and the interleukin-1 receptor[J]. Cell,1994,78(6):1101-1015.
[3]Botella M A, Parker J E, Frost L N, et al. Three genes of theArabidopsisRPP1 complex resistance locus recognize distinctPeronosporaparasitica avirulence determinants[J]. Plant Cell,1998,10(11):1847-1860.
[4]Yahiaoui N, Srichumpa P, Dudler R, et al. Genome analysis at different ploidy levels allows cloning of the powdery mildew resistance genePm3bfrom hexaploid wheat[J]. Plant J,2004,37(4):528-538.
[5]Sela H, Spiridon L N, Petrescu A J, et al. Ancient diversity of splicing motifs and protein surfaces in the wild emmer wheat (Triticumdicoccoides)LR10 coiled coil (CC) and leucine-rich repeat (LRR) domains[J]. Mol Plant Pathol,2012,13(3):276-287.
[6]Lee Y K, Bekes F, Gupta R, et al. The low molecular weight glutenin subunit proteins of primitive wheat variation in A genome species[J]. Theor Appl Genet,1999,98(1):119-125.
[7]Ling H Q, Zhao S, Liu D, et al. Draft genome of the wheat A-genome progenitorTriticumurartu[J]. Nature,2013,496(7443):87-90.
[8]盛宝钦. 用反应型记载小麦苗期白粉病[J].植物保护,1988,14(1):49.
[9]Sharp P J, Kreis M, Shewry P R, et al. Location of β-amylase sequence in wheat and its relatives[J]. Theor Appl Genet,1988,75(2):289-290.
[10]Meyers B C, Kozik A, Griego A, et al. Genome-wide analysis of NBS-LRR encoding genes inArabidopsis[J]. Plant Cell,2003,15(4):809-834.
[11]Paterson A H, Bowers J E, Bruggmann R, et al. TheSorghumbicolorgenome and the diversifcation of grasses[J]. Nature,2009,457(7229):551-556.
[12]Kang Y J, Kim K H, Shim S, et al. Genome-wide mapping of NBS-LRR genes and their association with disease resistance in soybean[J]. BMC Plant Biol,2012,12(1):139.
[13]Gu L J, Si W N, Zhao L N, et al. Dynamic evolution of NBS-LRR genes in bread wheat and its progenitors[J]. Mol Genet Genomic,2015,290(2):727-738.
[14]Shang J J, Tao Y, Chen X W, et al. Identification of a new rice blast resistance gene,pid3, by genome wide comparison of paired nucleotide-binding site leucine-rich repeat genes and their pseudogene alleles between the two sequenced rice genomes[J]. Genetics, 2009,182(4):1303-1311.
[15]Li J, Ding J, Zhang W, et al. Unique evolutionary pattern of numbers of gramineous NBS-LRR genes[J]. Mol Genet Genomics,2010,283(5):427-438.
(编辑:邢国芳)
Genome-wide analysis and marker development of NBS-SSR inTriticumurartu
Qiao Linyi1,2, Chang Zhijian2, Zhang Xiaojun2, Liu Jing3, Zhu Yan3, Zhan Haixian2, Guo Huijuan2, Li Xin2*
(1.CollegeofLifeScience,ShanxiUniversity,Taiyuan030006,China; 2.InstituteofCropScience,ShanxiAcademyofAgriculturalSciences/ShanxiKeyLaboratoryofCropGeneticsandMolecularImprovement,Taiyuan030031,China; 3.CollegeofBiologicalEngineering,ShanxiUniversity,Taiyuan030006,China)
[Objective]The aim of this study was to diagnose the SSR loci on genome sequences of the NBS family inTriticumurartuand to develop molecular markers for the exploitation and utilization of this resistance resources. [Method]In this paper, the target sequences were getted by searching the database using NBS HMM, and molecular markers were developed based on the SSR loci diagnosed by software, then the markers were verificated by twoT.urartumaterials which resistant(UR207) and susceptible(UR203) to theBlumeriagraministritici, respectively. [Results]463 NBS genes, which can be divided into four categories containing N, CN, NL and CNL, were separated from theT.urartugenome. 4 292 SSR loci were found on the scaffold sequences with NBS and of which the dinucleotide repeat loci is most, accounting for 78.1%. 42 TuNBS-SSR markers distributing in the each chromosome arm were developed randomly and 12 markers showed polymorphism between UR207 and UR203.[Conclusion]4 292 NBS-SSR loci were diagnosed fromT.urartugenome and 12 markers exhibited polymorphism between resistant and susceptible materials. The results can be used for the identification of disease resistance and the screening of resistance genes ofT.urartu.
Turartu, NBS-encoding genes, SSR markers, Disease resistance
2016-08-25
2016-12-07
乔麟轶 (1988-),男(汉),山西晋中人,博士研究生,研究方向:小麦抗病
*通信作者:李欣,副研究员,Tel:18635101299, E-mail:saaslixin@126.com
国家自然科学青年基金项目(31601303), 山西省青年基金项目(2015021145), 山西省农业攻关项目(20150311001-1), 山西省农科院攻关项目(15YGG01, YGG1602)
S435.121
A
1671-8151(2017)03-0153-05
