基于三种分子标记的外伶仃岛石珊瑚系统进化关系研究
2017-03-26肖宝华廖宝林杨小东谢子强
肖宝华, 廖宝林, 杨小东, 谢子强
基于三种分子标记的外伶仃岛石珊瑚系统进化关系研究
肖宝华1, 2, 廖宝林2, 杨小东2, 谢子强3
(1. 广东海洋大学, 广东 湛江 524025; 2. 广东海洋大学 深圳研究院, 广东 深圳 518108; 3. 深圳市碧海蓝天海洋科技有限公司, 广东 深圳 518108)
以广东省珠海市外伶仃岛地区常见的4属5种石珊瑚为研究对象, 对线粒体COI、16S rRNA和ITS三种基因片段数据进行分析, 并计算了种间遗传距离。序列分析结果表明5种石珊瑚的COI、16S rRNA基因片段碱基AT含量大于GC含量, 而ITS序列碱基AT含量小于GC含量; 5种石珊瑚种间的三种基因平均遗传距离分别为0.043、0.134和0.763, 所得遗传距离结果存在一定差异。将三种基因串联起来并利用邻位连接法(NJ)、最大似然法(ML)和最小进化法(ME)分别构建了分子系统树。在系统发育树中, 科一级的系统进化关系与形态学分类一致, 但蜂巢珊瑚科内的分子系统分类结果与形态学分类阐述的遗传进化关系略有差别, 说明石珊瑚传统分类可能受骨骼可塑性的影响。研究结果为外伶仃岛石珊瑚的亲缘关系研究提供相关基础数据, 并为该地区石珊瑚资源保护奠定理论基础。
石珊瑚; COI; 16S rRNA; ITS; 分子系统进化
石珊瑚分子系统进化研究始于20世纪90年代, 1996年Romano等[1]在世界上首次使用分子标记(线粒体16S rRNA和细胞核28S rRNA)对石珊瑚系统进化关系进行研究, 研究结果表明现有石珊瑚目起源于两个分支类群(坚实型类群和复合型类群)。目前, 应用于石珊瑚系统进化的分子标记主要有核糖体18S rRNA、5.8S rRNA、内转录间隔(Internal transcribed spacers, ITS)和线粒体的12S rRNA基因、16S rRNA基因和细胞色素b(cytochromeb, cyt b)基因、细胞色素c氧化酶亚基I基因(COI)等[2]。经过近20年的研究发展, 石珊瑚的分子系统进化研究已经对石珊瑚较高阶元的系统分类学产生了极大的影响, 颠覆了以往通过形态学对石珊瑚系统进化的认识[3]。
鹿角珊瑚科的石珊瑚品种是目前国内外利用分子标记研究系统进化关系最多一类珊瑚。Odorico等[4]利用ITS基因对5种鹿角珊瑚科品种的系统关系进行了初步的探讨, 为鹿角珊瑚科分子系统研究打下了坚实的理论基础。Fukami等[5]利用线粒体的细胞色素b和ATP6两种基因片段对鹿角珊瑚科4个属15个品种进行系统进化分析, 分子系统进化树显示15种珊瑚可分为三个大的珊瑚类群。Forsman等[6]利用16S rRNA作为分子标记对鹿角珊瑚科的蔷薇珊瑚属()多种蔷薇珊瑚的种间遗传关系进行分析, 构建了蔷薇珊瑚属的系统进化树。蜂巢珊瑚科的石珊瑚作为石珊瑚种类第二丰富的种类, 使用分子标记进行系统进化研究也在国内外积累了大量的报道。Huang等[7]基于线粒体COI和一个非编码区序列对新加坡附近海域的41种蜂巢珊瑚科珊瑚系统进化关系进行研究, Arrigoni等[8]采用COI作为分子标记对印度洋海域的蜂巢珊瑚科的珊瑚品种进行系统进化分析。这些学者都发现蜂巢珊瑚科的分子系统进化结果与形态学系统进化结果存在一定的差异, 再次证明石珊瑚中普遍存在并系现象。相比较国外对石珊瑚分子系统进化的报道, 国内有关石珊瑚分子系统进化的研究报道仍较少, 仅有海南[9-10]、广东徐闻[11-13]等少数地方的石珊瑚系统进化有过相关的报道。
外伶仃岛位于万山群岛中部东北端, 珠江口东侧。目前, 有关外伶仃岛附近海域石珊瑚的研究知之甚少, 仅有黄晖等[15]在2008年开展万山群岛的石珊瑚群落分布研究时对外伶仃岛海域石珊瑚情况进行了初步的调查, 调查结果显示多孔同星珊瑚()和腐蚀刺柄珊瑚()为外伶仃岛的优势种。其他有关外伶仃岛石珊瑚的研究还未见报道。
本研究采用线粒体COI、16S rRNA基因以及核糖体ITS基因的部分序列片段作为分子标记, 对外伶仃岛海域的5种石珊瑚的系统进化关系进行研究, 为外伶仃岛石珊瑚的亲缘关系研究提供相关基础数据, 并为该地区石珊瑚资源保护奠定理论基础。
1 材料与方法
1.1 样品采集和形态学鉴定
本研究的石珊瑚样品采自广东省珠海市外伶仃岛附近海域, 形态学鉴定依照邹仁林[16]的《中国动物志·腔肠动物门·珊瑚虫纲·石珊瑚目·石珊瑚》和Veron[17]的六射珊瑚骨骼形态特征分类系统。经形态学鉴定采集的样品可分为3科4属5种, 样品形态学鉴定结果见表1。截取一小段断枝保存于无水乙醇中以备后续DNA提取实验使用。
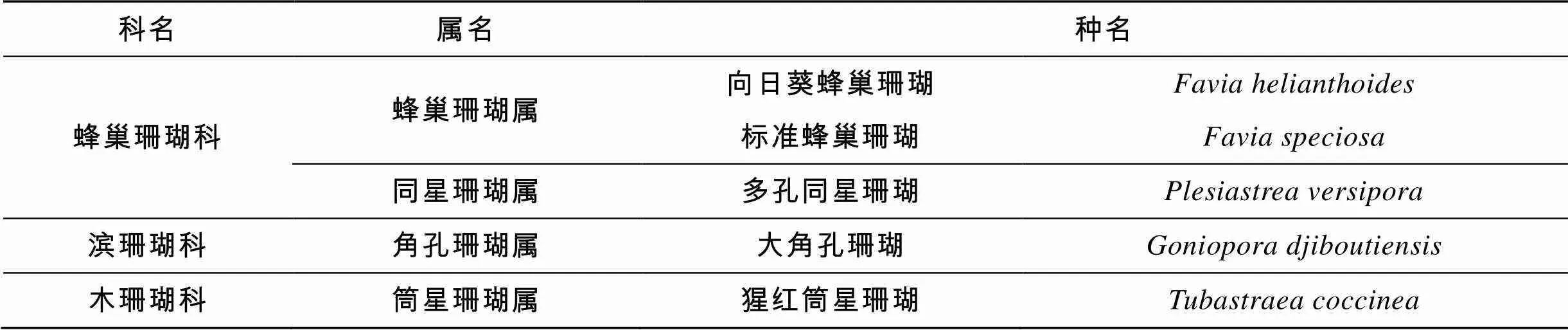
表1 珊瑚样品
1.2 基因组总DNA的提取
使用北京天根生物有限公司生产的海洋动物组织基因组提取试剂盒提取珊瑚样品的DNA, 所提取的DNA置于4℃保存备用。
1.3 引物设计合成
COI基因的PCR引物参考李晓娜等[11]的研究报道, 16S rRNA基因的PCR引物参考Forsman等[6]的研究报道, ITS基因的PCR引物参考Odorico等[4]的研究报道。引物由上海生工生物技术服务有限公司合成, 引物信息见表2。
1.4 PCR扩增和DNA测序
PCR反应体系反应总体积25 µL: 10×Buffer 2.5 µL, dNTP 2 µL, 上下游引物各1 µL, , Taq DNA聚合酶1U及DNA模板2 µL; 其余用去离子水补至25 µL。PCR反应条件: 95℃预变性5min, 95℃变性45 , 50℃退火45 s, 72℃延伸1 min 30 s, 共35个循环, 最后72℃延伸10 min。扩增产物在1.0%的琼脂糖凝胶中检测, 凝胶成像仪拍照记录。选取扩增条带单一、浓度较高的PCR产物送至上海生工生物技术公司进行测序, 获取各基因片段的序列信息。

表2 COI、16S rRNA、ITS基因的PCR引物
1.5 序列分析及系统进化树的构建
测序所得序列用DNASTAR中的Editseq软件进行编辑、剪切、校正, 再将校正好的序列与NCBI的数据库进行比对, 确认样品的分子结果。采用MEGA6.0软件进行三种基因序列的串联与分析, 统计出三种线粒体基因组序列长度、碱基组成、简约信息位点等相关信息。同时根据简约信息位点, 计算出种间遗传距离。本研究三种基因的系统进化树是使用MEGA6.0软件基于Kimu-ra双参数模型(Kimura 2-parameter, K2P), 只计算颠换(Transversional substitutions only), 采用邻位连接法(Neighbor-Joinning, NJ)、最大似然法(Minimum-Likelihood, ML)和最小进化法(Minimum-Evolution, ME)构建出来的。三种系统进化树中节点的自举置信度水平由自引导值(Bootstrapvalue) 估计, 重复次数为1 000次。
2 结果与分析
2.1 三种基因序列及碱基组成分析
本研究的5个物种的COI、16S rRNA基因以及ITS基因部分序列长度分别为464~465bp, 470~485bp, 964~988bp。从5种石珊瑚的COI、16S rRNA基因以及ITS基因部分序列的碱基组成比例来看, 两种线粒体基因(COI、16S rRNA)片段碱基中的A、T、C、G含量相差较大, 序列碱基AT含量大于GC含量, 与目前已知的无脊椎动物mtDNA序列中AT含量大于GC含量的情况相同。而ITS基因序列片段碱基中的A、T、C、G含量相差不大, 除大角孔珊瑚外(), 其余物种的碱基AT含量小于GC含量。三种片段序列基因都处于高度饱和状态。碱基组成比例见表3。
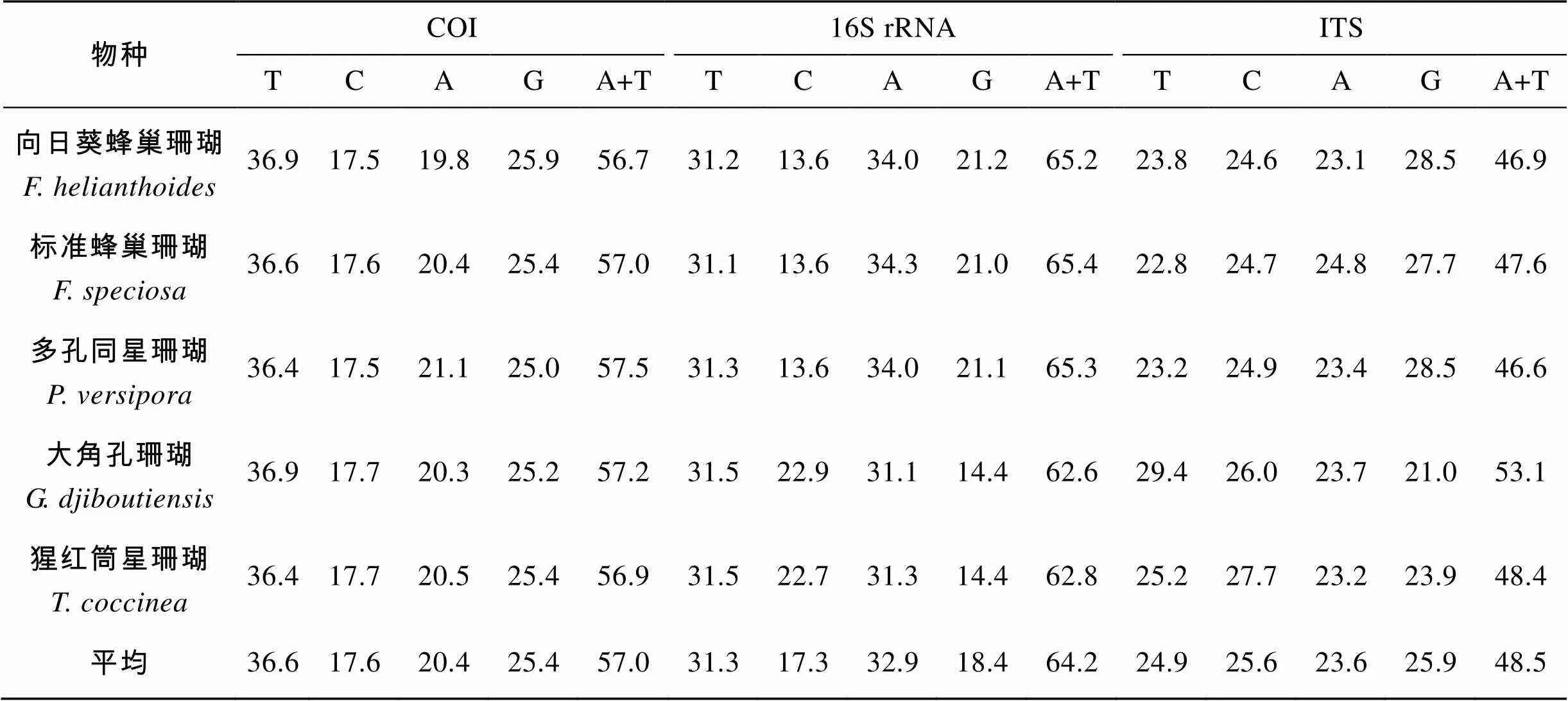
表3 5种石珊瑚COI、16S rRNA以及ITS基因片段的碱基组成比例
2.2 5种石珊瑚种间遗传距离
通过MEGA6.0计算5种石珊瑚的种间遗传距离, 得到COI、16S rRNA基因以及ITS序列间的平均距离为0.043、0.134和0.763。在COI序列中, 标准蜂巢珊瑚和大角孔珊瑚的种间遗传距离最大为0.093, 而标准蜂巢珊瑚和猩红筒星珊瑚的遗传距离最小, 为0.004。在16S rRNA基因序列中, 猩红筒星珊瑚和多孔同星珊瑚以及向日葵蜂巢珊瑚的种间遗传距离最大, 都为0.224, 而标准蜂巢珊瑚和多孔同星珊瑚的遗传距离最小, 为0.002。在ITS序列中, 大角孔珊瑚和多孔同星珊瑚的种间遗传距离最大, 为1.224, 而多孔同星珊瑚和向日葵蜂巢珊瑚的遗传距离最小, 为0.028。三种序列得出的种间遗传距离结果存在一定差异, 种间遗传距离见表4、表5和表6。
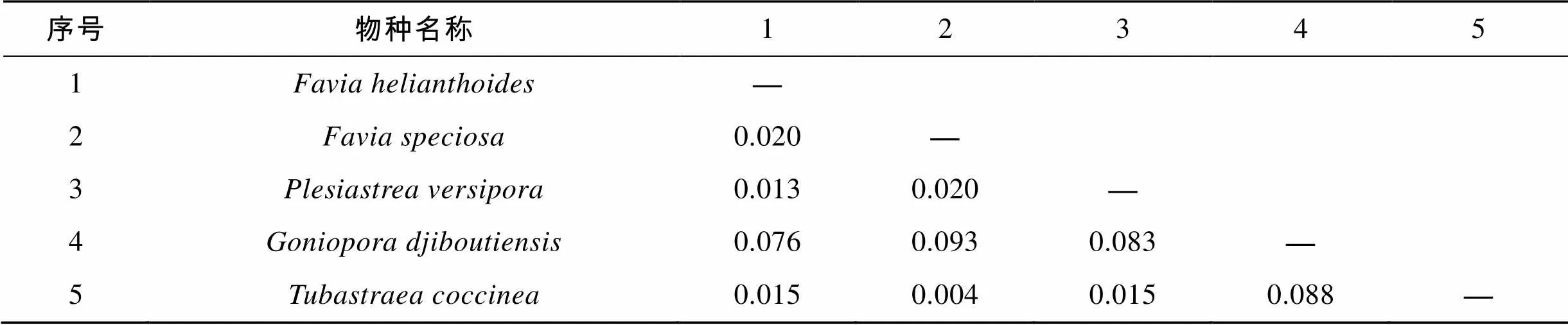
表4 基于COI的5种石珊瑚种间遗传距离
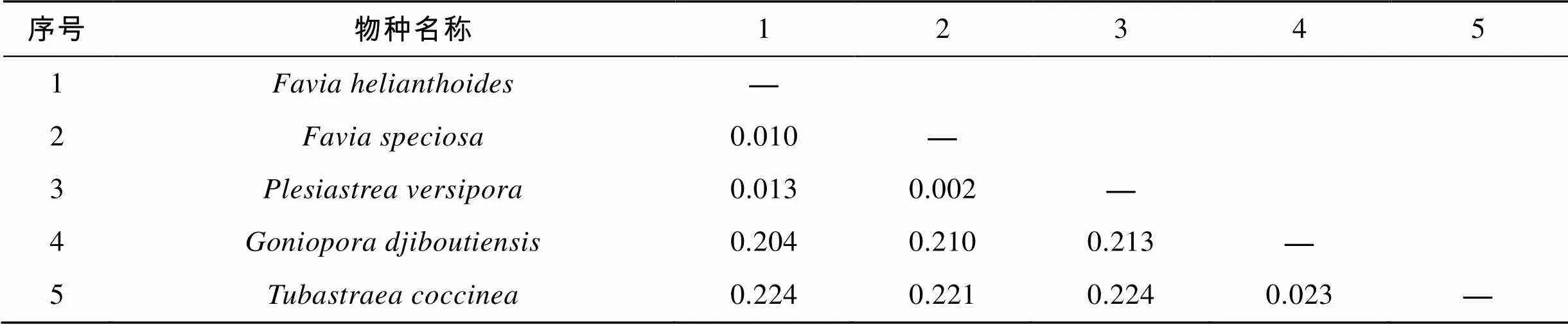
表5 基于16S rRNA基因的5种石珊瑚种间遗传距离
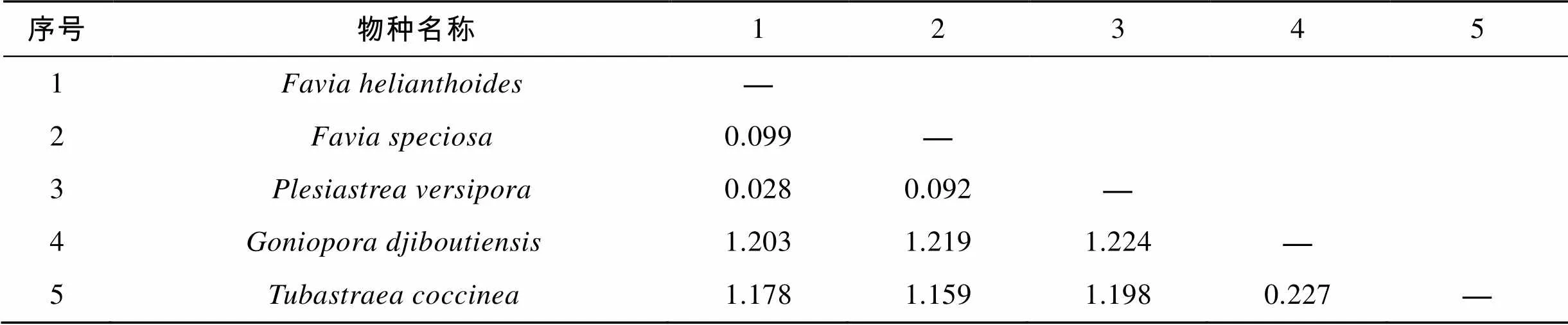
表6 基于ITS的5种石珊瑚种间遗传距离
2.3 基于三种基因序列对5种石珊瑚的系统进化分析
使用MEGA6.0软件将每种石珊瑚的COI、16S rRNA基因以及ITS三种基因片段序列串联起来, 再采用邻位连接法(Neighbor-Joinning, NJ)、最大似然法(Minimum-Likelihood, ML)和最小进化法(Minimum-Evolution, ME)构建5种石珊瑚的分子系统树, 结果显示3种方法构建的系统进化树拓扑结构完全一致, 蜂巢珊瑚科、滨珊瑚科和木珊瑚科的珊瑚品种各聚为一枝, 但在蜂巢珊瑚科中, 向日葵蜂巢珊瑚与多孔同星珊瑚先聚为一支然后再与标准蜂巢珊瑚再聚为一支, 这与传统形态学科属分类结果存在一定差异。三种基因构建的系统进化树见图1、图2和图3。

图1 利用三种基因序列数据构建的分子系统进化NJ树

图2 利用三种基因序列数据构建的分子系统进化ML树

图3 利用三种基因序列数据构建的分子系统进化ME树
3 讨论
3.1 5种石珊瑚COI、16S rRNA基因和ITS基因序列特点
线粒体细胞色素c氧化酶亚基I基因(COI)是动物线粒体DNA的重链上参与细胞色素氧化反应的关键基因。COI基因拥有长度适宜、进化速率慢及富含系统发育信号等特点, 常被当作动物界物种鉴定的DNA条形码工具[18]。近年来, 很多研究表明在许多低等动物的分子系统进化研究中COI基因具有更为缓慢的进化速率, 特别是石珊瑚。造成这一现象的原因可能是由于石珊瑚采用无性生殖方式使珊瑚虫个体可以存活上百年[19]。Hellberg[19]在使用COI基因对橙杯珊瑚()和猩红筒星珊瑚系统进化的研究中发现其较慢的进化速率导致其较低的种内分化。然而在石珊瑚种间关系的研究中, COI基因作为主要的分子标记得到了广泛的应用[20-22]。本研究的5种石珊瑚COI序列长度变化不大, 遵循真核生物线粒体基因AT偏向性的规律, 是本研究三种基因中种间序列变化程度最小的基因。
线粒体16S rRNA基因相对于其他石珊瑚的线粒体基因进化速率较慢, 且较为保守, 常用于种以上分子分类的标记[23]。本研究的5种石珊瑚16S rRNA基因序列长度变化较大, 遵循真核生物线粒体基因AT偏向性的规律。国外学者研究发现, 16S rRNA基因在石珊瑚属以上的分类阶元中种间片段长度具有较大的差异, 而属内差异片段长度差异不大[23-24]。本研究的结果也较为符合这种情况, 两种复合型珊瑚(大角孔珊瑚和猩红筒星珊瑚)16S rRNA基因具有较为相似的碱基组成, 而与3种蜂巢珊瑚科珊瑚的16S rRNA基因序列碱基组成则相差较大。从序列的碱基组成、序列变化和遗传距离上看, 16S rRNA基因比较适宜作为科、属之间的分子分类标记。
内转录间隔序列(ITS)是细胞核中核糖体18S r RNA和5.8S r RNA之间的一段非转录区序列, 由于所受的选择压力较小、DNA碱基替换速率较快, 加上较大的拷贝数、适中的长度和快速的同步化等特点, 常被用于低等真核生物属水平的种间关系研究[7]。而在石珊瑚目的鹿角珊瑚、扁脑珊瑚、沙珊瑚、滨珊瑚等类群研究中, 国内外都有相关ITS标记的报道[3, 25]。本研究的5种石珊瑚ITS序列长度变化是本研究三种基因中变化最大的基因。序列碱基组成情况上与16S rRNA基因结果较为一致, 同时由于其较高的碱基变化率, 对蜂巢珊瑚科内不同种石珊瑚遗传分化情况是三种基因中最为显著一种。从序列的碱基组成、序列变化和遗传距离上看, ITS基因比较适宜作为属间的分子分类标记。
3.2 石珊瑚系统进化分析
早在20世纪90年代, Romano和Palumbi[1]在应用16S rRNA基因对34种石珊瑚的系统进化关系进行研究的过程中, 发现全球范围内现存的石珊瑚可划分为两大分支类群, 并将其命名为坚实型(Robust)和复合型(Complex)。结合已有的形态学研究, Romano认为坚实型珊瑚骨骼钙化程度较高, 具有坚实的骨骼结构, 珊瑚外观主要以碟片状和团块状为主, 其代表种类包括蜂巢珊瑚科、石芝珊瑚科、褶叶珊瑚科等。复合型珊瑚骨骼钙化程度较低, 具有较为复杂多变的生长形态, 其代表种类包括鹿角珊瑚科、菌珊瑚科、木珊瑚科、滨珊瑚科等。经过近20年的石珊瑚分子系统学研究发展, 国内外的许多学者积累的分子数据都极力地支持了这一学说。Fukami等[26]使用多种分子标记对17科75属127种石珊瑚进行了系统进化分析, 通过构建的系统进化树将127种石珊瑚细分成21个类群, 其中9个类群属于复合型珊瑚, 12个类群属于坚实型珊瑚。Kitahara等[22]对现有324种石珊瑚COI基因的系统进化关系进行了探讨, 引入外源种进行分析后, 将坚实型分支的位置放在了复合型分支辐射范围之内。相对来说, 复合型珊瑚类群的分子系统进化关系与传统分类学的系统关系较为一致。而坚实型珊瑚类群的分子系统进化关系则与传统分类学存在很多不一致的地方, 部分科属之间关系较为混乱。其中最为混乱的第17个分类群被称为“Bigmessidae”, 里面包含了4个石珊瑚科: 蜂巢珊瑚科、裸肋珊瑚科、梳状珊瑚科、脑珊瑚科。这个类群在分子系统进化关系上存在相互并系现象, 不同科属之间的物种重新聚为新的分支[27]。蜂巢珊瑚科作为坚实型珊瑚的代表, 同时也是最为多源的系统进化种类, 在不同的分子系统进化研究中都分散在7种坚实型珊瑚分类群中, 在系统进化树上与裸肋珊瑚科、褶叶珊瑚科、梳状珊瑚科、铁星珊瑚科等珊瑚种类交杂在一起, 成为并系情况最为复杂的一类珊瑚。这些分子证据与传统形态学系统进化存在许多不协调和不统一的地方, 暗示着传统形态学可能受珊瑚骨骼生长变异性限制, 对石珊瑚的系统进化关系分析中存在错误, 尤其是坚实型珊瑚类型。
本研究采用邻位连接法(NJ)、最大似然法(ML)和最小进化法(ME)对5种石珊瑚三种基因片段的串联序列构建了系统发育树, 从科一级单元来看, 串联基因构建的系统进化关系符合Romano的两大分支类群学说。蜂巢珊瑚科的珊瑚品种属于坚实型珊瑚类群分支、而滨珊瑚科和木珊瑚科品种属于复合型珊瑚类群分支。同时, 蜂巢珊瑚科科内多源系统进化的现象也在本研究体现出来, 传统形态学分类中同属的向日葵蜂巢珊瑚和标准蜂巢珊瑚并没有聚为一支, 反而向日葵蜂巢珊瑚与不同属的多孔同星珊瑚聚率先聚为一支。
近年来, 许多学者通过显微扫描技术对石珊瑚骨骼的精细结构进行观察, 结合石珊瑚分子系统进化数据, 综合全面的分析其系统进化关系, 对Veron传统形态学研究结果进行了校正和补充[28-29]。很多精细的石珊瑚骨骼数据正逐渐改变我们对石珊瑚传统分类的看法, 同时也对分子系统进化研究结果进行了有力的佐证。在当今的石珊瑚系统进化研究中, 无论是传统的形态学方法还是利用分子技术手段, 两者都必须相互利用和补充。
[1] Romano S L, Palumbi S R. Evolution of scleractinian corals inferred from molecular systematics[J]. Science, 1996, 271(5249): 640.
[2] 尤丰, 黄晖, 董志军, 等. 造礁石珊瑚的分子系统学研究进展[J]. 海洋通报, 2010, 29(4): 459-465. You Feng, Huang Hui, Dong Zhijun, et al. Progress in taxonomy and genetic diversity of Scleratinia corals[J]. Marine Science Bulletin, 2010, 29(4): 459-465.
[3] Budd A F, Romano S L, Smith N D, et al. Rethinking the phylogeny of scleractinian corals: a review of morphological and molecular data[J]. Integrative and Comparative Biology, 2010, 50(3): 411-427.
[4] Odorico D M, Miller D J.Variation in the ribosomal internal transcribed spacers and 5.8S r DNA among five species of(Cnidaria; Sclerac-tinia): Patterns of variation consistent with reticulate evolution[J]. Molecular Biology and Evolution, 1997, 14: 465-473.
[5] Fukami H, Omori M, Hatta M. Phylogenetic relationships in the coral family Acroporidae, reassessed by inference from mitochondrial genes[J]. Zoological science, 2000, 17(5): 689-696.
[6] Forsman Z H, Concepcion G T, Haverkort R D, et al. Genetic and morphological characterization of a coral species of concern montipora dilatata in Kaneohe Bay, Oahu, Hawaii[J]. Pacific Islands Region Species of Concern, 2008: 1-17.
[7] Huang D, Meier R, Todd P A, et al. More evidence for pervasive paraphyly in scleractinian corals: Systematic study of Southeast Asian Faviidae (Cnidaria; Scleractinia) based on molecular and morphological data[J]. Molecular Phylogenetics and Evolution, 2009, 50(1): 102-116.
[8] Arrigoni R, Stefani F, Pichon M, et al. Molecular phylogeny of the robust clade (Faviidae, Mussidae, Merulinidae, and Pectiniidae): an Indian Ocean perspective[J]. Molecular Phylogenetics and Evolution, 2012, 65(1): 183-193.
[9] 齐文同, 柯叶艳, 顾红雅. 石珊瑚演化的18S rDNA分子生物学和形态学研究[J]. 北京大学学报(自然科学版), 2008, 44(2): 171-176. Qi Wentong, Ke Yeyan, Gu Hongya. Molecular systematics of the 18S rDNA within the scleractinian corals[J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2008, 44(2): 171-176.
[10] 程希婷. 石珊瑚的 DNA 条形码研究及其系统发育分析[D]. 海南: 海南大学, 2012. Cheng Xiting. DNA barcoding and molecular phylogeny of the scleractinia[D].Hainan: Hainan University, 2012.
[11] 李晓娜, 刘丽, 刘楚吾. 运用线粒体COI基因分析8种石珊瑚系统发育关系[J]. 渔业科学进展, 2012, 33(4): 83-88. Li Xiaona, Liu Li, Liu Chuwu. Phylogenetic relationship in 8 species of Scleractinian coral based on COI sequences[J]. Progress in Fishery Sciences, 2012, 33(4): 83-88.
[12] 刘丽, 李晓娜, 陈育盛, 等. 基于线粒体基因的石珊瑚分子系统学研究[J]. 海洋与湖沼, 2012, 43(4): 814-820. Liu Li, Li Xiaona, Chen Yusheng, et al. Molecular systematics study of scleractinian based on mitchondrial genes[J]. Oceanologia and Limnologia Sinica, 2012, 43(4): 814-820.
[13] 刘丽, 陈育盛, 李晓娜, 等. 基于线粒体Cytb基因的10种石珊瑚的系统发育关系[J]. 广东海洋大学学报, 2011, 31(1): 6-11. Liu Li, Chen Yusheng, Li Xiaona, et al. Phylogenetic Relationships in 10 Species of Scleractinian Coral Based on Cyt b Sequences[J]. Journal of Guangdong Ocean University, 2011, 31(1): 6-11.
[14] 廖伟群, 张加恭, 黄慧萍. 万山群岛的旅游资源及其开发利用研究[J]. 热带地理, 2000, 20(2): 134-138, 143.Liao Weiqun, Zhang Jiagong, Huang Huiping. Development of the tourism resources of the wanshan islands[J]. Tropical Geography, 2000, 20(2): 134-138, 143.
[15] 黄晖, 尤丰, 练健生, 等. 珠江口万山群岛海域造礁石珊瑚群落分布与保护[J]. 海洋通报, 2012, 31(2): 189-197. Huang Hui, You Feng, Lian Jianshen, et al. Status and conservation strategies of the scleractinian coral community in the Wanshan Islands at Pearl River Estuary[J]. Marine Science Bulletin, 2012, 31(2): 189-197.
[16] 邹仁林. 中国动物志—造礁石珊瑚[M]. 北京: 科学出版杜, 2001. Zou Renlin. Fauna Sinica: Coelenterata: Anthozoa, Scleractinia, Hermatypic coral[M]. Beijing: Science Press, 2001.
[17] Veron J E N. Corals in space and time: the biogeography and evolution of the Scleractinia[M]. Ithaca, New York: Comstock/Cornell, 1995.
[18] Yamauchi M M, Miya M U, Nishida M. Complete mitochondrial DNA sequence of the swimming crab, Portunus trituberculatus (Crustacea: Decapoda: Brachyura)[J]. Gene, 2003, 311: 129-135.
[19] Hellberg M E. No variation and low synonymous substitution rates in coral mtDNA despite high nuclear variation[J]. BMC evolutionary biology, 2006, 6(1): 24.
[20] Arrigoni R, Kitano Y F, Stolarski J, et al. A phylogeny reconstruction of the Dendrophylliidae (Cnidaria, Scleractinia) based on molecular and micromorphological criteria, and its ecological implications[J]. Zoologica Scripta, 2014, 43(6): 661-688.
[21] Gittenberger A, Reijnen B T, Hoeksema B W. A molecularly based phylogeny reconstruction of mushroom corals (Scleractinia: Fungiidae) with taxonomic consequences and evolutionary implications for life history traits[J]. Contributions to Zoology, 2011, 80(2): 107-132.
[22] Kitahara M V, Cairns S D, Stolarski J, et al. A comprehensive phylogenetic analysis of the Scleractinia (Cnidaria, Anthozoa) based on mitochondrial CO1 sequence data[J]. PloS One, 2010, 5(7): e11490.
[23] Fukami H, Knowlton N. Analysis of complete mitochondrial DNA sequences of three members of thecoral species complex (Cnidaria, Anthozoa, Scleractinia)[J]. Coral Reefs, 2005, 24(3): 410-417.
[24] Romano S L, Cairns S D. Molecular phylogenetic hypotheses for the evolution of scleractinian corals[J]. Bulletin of Marine Science, 2000, 67(3): 1043-1068.
[25] 李文娟, 刘楚吾, 刘丽, 等. 利用ITS基因研究徐闻滨珊瑚的系统发生关系[J]. 海洋科学, 2012, 36(7): 56-60. Li Wenjuan, Liu Chuwu, Liu Li, et al. Phylogeny analysis of Porites link in Xuwen using its gene[J]. Marine Sciences, 2012, 36(7): 56-60.
[26] Fukami H, Chen C A, Budd A F, et al. Mitochondrial and nuclear genes suggest that stony corals are monophyletic but most families of stony corals are not (Order Scleractinia, Class Anthozoa, Phylum Cnidaria)[J]. PloS one, 2008, 3(9): e3222.
[27] Huang D, Licuanan W Y, Baird A H, et al. Cleaning up the'Bigmessidae': Molecular phylogeny of scleractinian corals from Faviidae, Merulinidae, Pectiniidae and Trachyphylliidae[J]. BMC Evolutionary Biology, 2011, 11(1): 1.
[28] Stolarski J, Kitahara M V, Miller D J, et al. The ancient evolutionary origins of Scleractinia revealed by azooxanthellate corals[J]. BMC Evolutionary Biology, 2011, 11(1): 316.
[29] Huang D, Benzoni F, Fukami H, et al. Taxonomic classification of the reef coral families Merulinidae, Montastraeidae, and Diploastraeidae (Cnidaria: Anthozoa: Scleractinia)[J]. Zoological Journal of the Linnean Society, 2014, 171(2): 277-355.
(本文编辑: 梁德海)
Study of phylogenetic relationships of scleractinian from Wailingding Island based on three standard molecular markers
XIAO Bao-hua1, 2, LIAO Bao-lin2, YANG Xiao-dong2, XIE Zi-qiang3
(1. Guangdong Ocean University, Zhanjiang 524025, China; 2. Guangdong Ocean University, Shenzhen Institute, Shenzhen 518108, China; 3. Shenzhen Bihai Blue Sky Marine Technology Co, LTD, Shenzhen 518108, China)
The mitochondrial cytochrome oxidase subunit I (CO I), 16S rRNA, and nuclear ITS genes have been routinely used as standard molecular markers. A total of five species pertaining to four genera of scleractinian, collected from Wailingding Island, Zhuhai City, Guangdong Province, were analyzed by partial sequences, and the genetic distances of these three genes were calculated. The results indicated that the average AT contents of CO I and 16S rRNA gene fragments were obviously higher than the GC contents. However, the average AT content of ITS fragments was obviously less than the GC content. The average genetic distances of the three genes were 0.043, 0.134, and 0.763 from the five species, respectively. The neighbor-joining, maximum-likelihood, and minimum-evolution trees of combined partial sequences of these three genes revealed that the phylogenetic relationships were consistent with the morphological classification in the families. However, the phylogenetic relationships were slightly different from the morphological classification in Faviidae. This result suggested that morphological classification may be limited by coral skeleton plasticity. These findings provide basic data for studying the genetic relationship of Wailingding Island corals and a theoretical basis for resource protection of stone corals in this area.
Scleractinian; COI; 16S RNA; ITS; Molecular phylogenetics
Dec. 22, 2016
肖宝华(1978-), 男, 福建长乐人, 高级工程师, 硕士, 主要从事海洋生态学, 特别是珊瑚生态系统方面研究, 电话: 0759- 2396216, E-mail: gdouxxhpaper@126.com
Q75
A
1000-3096(2017)09-0001-08
10.11759/hykx20160414001
2016-12-12;
2017-02-22
广东省渔港建设和渔业产业发展专项(A201708D06); 大鹏新区产业发展专项(KY20160107)
Guangdong Ocean and Fishery Administration Project, No. A201708D06; Dapeng New District Industry Development Fund, No. KY20160107]
