一个新杉木纤维素合酶ClCesA基因的克隆与植物表达载体构建
2017-03-08魏晓骁王士亚陈潇潇汪凤林曹光球曹世江
魏晓骁, 王士亚, 陈潇潇, 汪凤林, 曹光球, 曹世江
(1.福建农林大学林学院;2.国家林业局杉木工程技术研究中心,福建 福州 350002)
一个新杉木纤维素合酶ClCesA基因的克隆与植物表达载体构建
魏晓骁1,2, 王士亚1,2, 陈潇潇1,2, 汪凤林1,2, 曹光球1,2, 曹世江1,2
(1.福建农林大学林学院;2.国家林业局杉木工程技术研究中心,福建 福州 350002)
为探索杉木木材形成的分子机制,以杉木嫩叶总RNA反转录的cDNA为模板,运用RT-PCR技术克隆出一个新杉木纤维素合酶基因ClCesA.该基因的开放阅读框(ORF)为2 955 bp,共编码984个氨基酸.通过TMHMM Server v.2.0和MEGA 5.1软件预测ClCesA蛋白的跨膜结构和功能,发现该蛋白N端含有锌指结构,共有8个跨膜结构,平均跨膜区域18个氨基酸残基,并具有保守的DDDQXXLRW结构域.系统进化树分析结果表明,ClCesA可能与细胞次生壁形成有关.荧光定量PCR分析表明,ClCseA基因在杉木的不同器官(根、茎、叶)中均有表达,但在杉木根中的表达含量最高,茎中次之,叶中最低.随后,为后续该基因功能验证提供基础,将ClCesA克隆到含有GFP标签和潮霉素抗性的pGWB506载体,构建植物过表达载体pGWB506-35S-ClCesA-GFP.
杉木; 纤维素合酶; 基因克隆; 表达分析; 载体构建
纤维素作为地球上最丰富的可再生资源,是木材次生木质部细胞壁的主要组成成分之一,约占木材干重的40%,是决定木材品质的重要因子之一.为此揭示植物纤维素生物合成机制对木材材质改良、木材定向培育都具有十分重要的意义.纤维素合酶(CesA)是纤维素生物合成的关键酶之一[1,2].研究表明,CesA基因通过在转录水平上对纤维素合成进行调控,对植物细胞壁发育起到重要作用[3-5].美国密西根大学在2000年初次从欧洲颤杨(Populustremuloides)中克隆得到林木的纤维素合酶基因PtrCesA1[6],并且研究发现该基因主要在植物茎中表达,表达高峰期主要出现在次生壁形成期间.此后,在毛果杨(Populustrichocarpa)[7]、大青杨(Populusussuriensis)[8]、松树(Pinusradiata)[9]、苎麻(Boehmerianivea)[10]、桉树(Eucalyptus)[11]、马尾松(Pinusmassoniana)[12]、杉木(Cunninghamialanceolata)[13]、毛竹(Phyllostachysedulis)[14]等木本植物中都陆续克隆分离出CesA基因,其中毛果杨中鉴定出18个CesA基因[7],杂交杨(Populustremula(L)×P.tremuloides)中鉴定出10个CesA基因,绿竹(Bambusabambusaoldhami)中鉴定出8个CesA基因[14].这些基因共同构成了林木的纤维素合酶基因家族.对ClCesA基因功能的研究结果表明,PtrCesA1、PtrCesA2、PtrCesA3这3个基因可能组成同一个纤维素合成酶复合体,共同完成杨树次生壁的合成[15];而PtrCesA4、PtrCesA5、PtrCesA7这3个基因可能与初生壁的形成有关[16].在毛果杨中,PtCesA4、PtCesA5、PtCesA7、PtCesA8、PtCesA17和PtCesA18在木质部特异高表达[17].Song et al[18]通过研究杨树的纤维素合酶发现,杨树的木质部细胞膜上至少具有2种纤维素合酶复合体,复合体Ⅰ由CesA4、CesA7、CesA8、CesA17和CesA18组成,参与细胞次生壁的合成;复合体Ⅱ由CesA3、CesA10、CesA11、CesA13、CesA15和CesA16组成,参与细胞初生壁和次生壁的合成.
杉木作为中国南方重要的速生用材树种之一,用途非常广泛.近几年,与杉木材性相关的分子机制研究取得一定进展[19,20],而有关杉木的纤维素生物合成分子机制的研究报道甚少.彭沙沙等[21]克隆了杉木纤维素合成酶类似蛋白ClCslD1;而庞景等[13]克隆了杉木纤维素合成酶基因CesA,通过将本研究克隆出的基因ClCseA与庞景等克隆的CesA基因的DNA和蛋白序列进行比较,发现2个基因在核苷酸的编码区有11处位点存在差异,分别为773、821、1 249、1 537、1 545、1 554、2 166、2 436、2 580、2 823、2 856位点;在氨基酸的编码区序列也发现3处位点存在差异,分别为258、274、513位点.由于杉木基因组测序未完成,关于杉木纤维素合成酶基因家族的数量、各个基因的确切功能,以及各基因间的相互作用关系均不清楚.虽然克隆出的2个基因存在相似性,但也存在差异,有可能为2个不同的基因.关于基因的功能尚需进一步验证.探索杉木中纤维素合酶基因家族的功能,特别是相关次生细胞壁形成的纤维素合酶基因,对于利用现代分子生物学手段改良杉木木材品质具有重要意义.本研究以杉木三代种子园种子发育植株为材料,利用RT-PCR技术分离克隆出1个新的杉木ClCesA基因,长度为2 955 bp,编码984个氨基酸;同时进行了生物信息学分析以及不同器官表达情况的研究;采用Invitrogen公司的gateway特异性位点重组技术将ClCesA基因构建到植物表达载体中,以期为进一步的功能解析提供依据,同时也为杉木材性的改良提供重要的基因资源.
1 材料与方法
1.1 材料收集与RNA提取
试验材料由国家林业局杉木工程技术研究中心提供,为杉木种子发育而成的杉木幼苗.取长势好的杉木幼嫩叶片组织0.2 g,按照TIANGEN公司的总RNA提取试剂盒操作说明书提取杉木总RNA,通过1%(质量分数)的琼脂糖凝胶电泳和超微量分光光度计Nanodrop分别检测样品的RNA质量及浓度.
1.2ClCesA基因克隆与序列分析
利用NCBI网上的GenBank数据库查询到已知杉木的DNA序列,采用gateway技术设计特异引物(表1).取检测合格的杉木总RNA,利用TIANScript cDNA第一链合成试剂盒进行第1条链cDNA的合成.合成的cDNA稀释10倍后进行PCR反应,具体反应体系见表2.反应程序为:95 ℃预变性3 min;94 ℃变性1 min,53 ℃退火3 min,72 ℃延伸3 min,共30循环;最后72 ℃延伸10 min.扩增后的产物经1%(质量分数)的琼脂糖凝胶电泳检测后,通过BP连接到pDONR207载体上,热激转化到大肠杆菌DH-5α感受态细胞,进行Gen抗性筛选.选取单菌落做菌落PCR,将检测为阳性的菌落进行液体LB过夜培养,使用天根质粒试剂盒提取质粒,并送铂尚生物公司进行全长测序.利用实时荧光定量PCR仪研究ClCesA基因在不同组织的表达情况.qPCR扩增试剂为SYBR Premix Ex Taq,采用的荧光染料为SYBR GreenⅠ,以杉木EF1α基因作为内参基因.在进行qCR分析前,先使用普通PCR检测体系的反应条件,如退火温度、模板量等,并将PCR产物进行测序,以保证qPCR结果的准确性.

表1 基因克隆及q-PCR反应中所用引物序列Table 1 Primers used in gene cloning and q-PCR reaction
1.3 植物表达载体的构建

表2 PCR反应体系Table 2 Reaction System of PCR
使用Gateway BP反应试剂盒,将回收带有attB位点的ClCesA基因PCR产物与入门载体pDONR207在室温下进行BP反应1 h.反应体系为:PCR产物0.6 μL,入门载体0.4 μL,BP酶0.25 μL.随后将反应产物全部转化到E.coliTrans 5α感受态细胞,涂布于含Gen(50 mg·L-1)的LB平板;次日随机挑选阳性克隆摇菌并提取质粒,经PCR检测验证正确后进行测序,即可获得入门克隆载体pDONR207-ClCesA.参考Gateway LR反应试剂盒的操作说明书,将入门载体与目的载体pGWB506进行LR反应,室温25 ℃,反应后转化大肠杆菌DH5α感受态细胞,并涂布于LB平板(含50 mg·L-1潮霉素),37 ℃培养过夜.次日挑单菌落,摇菌后用试剂盒提取质粒,经PCR检测载体构建的正确性,并进行测序验证.
1. 4 纤维素合酶基因的序列分析
在DNAMAN V6软件上设计引物;通过Vecter NTI软件进行序列比对;在TMHMM Server v.2.0软件上进行蛋白质结构分析;运用MEGA 5.1软件通过邻接法构建不同植物的基因进化树;采用Microsoft Excel 2007软件对基因表达进行作图.
2 结果与分析
2.1 杉木总RNA提取
杉木叶片总RNA经1%(质量分数)琼脂糖凝胶电泳,结果见图1.从图1可以看出,RNA条带完整,28 S rRNA和18 S rRNA 2条主带明显,28 S rRNA的亮度约为18 S rRNA的2倍,且无DNA污染.采用超微量分光光度计进行浓度测定和质量分析、检测,结果显示,样品总RNA浓度为231 μg·μL-1,D260 nm/D280 nm=1.86,其浓度与质量均满足反转录的要求,可用于后续试验.最后,将总RNA样品置于-80 ℃冰箱保存.
2.2 扩增产物的碱基序列测定与分析
取上述总RNA,采用天根公司的逆转录酶试剂盒获得cDNA,通过RT-PCR反应得到1个约3 000 bp的产物(图2),将该产物连接到pDONR207载体且转化DH-5α,挑取阳性克隆测序.测序结果显示该序列与CesA基因的同源性很高,并含有保守结构域.其氨基酸序列及结构如图3所示.
2.3 蛋白质跨膜结构的预测
从图4可以看出,新的ClCesA蛋白具有8个跨膜区,分别为175~197、204~223、760~782、794~816、831~853、878~900、910~932、945~964,最大跨膜区域为22个氨基酸残基,最小跨膜区域为12氨基酸残基,平均跨膜区域为18个氨基酸残基.
2.4 基因序列系统发生树
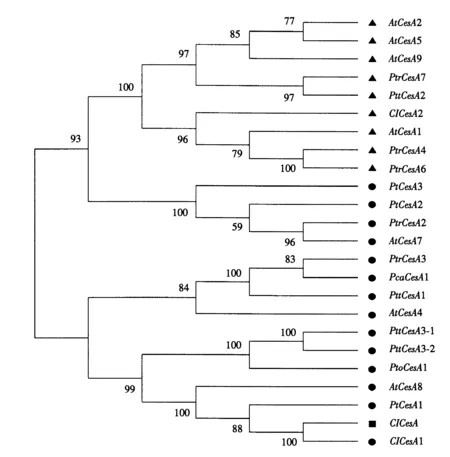
本研究运用MEGA 5.1软件构建系统进化树,将克隆到的杉木ClCesA基因编码区与拟南芥(Arabidopsisthaliana, At)、颤杨(P.tremuloides, Ptr)、欧洲山杨×颤杨(Populustremula×P.tremuloides, Ptt)、火炬松(PinustaedaL., Pt)、银灰杨(Populuscanescens(Ait.) Smith, Pca)、毛白杨(PopulustomentosaCarrière, Pto)的初生壁和次生壁形成的CesA基因进行系统进化树分析.初生壁有关基因有AtCesA1(O48946)、AtCesA2(O48947)、AtCesA5(Q8L778)、AtCesA9(Q9SJ22)、PtrCesA7(AAO25581)、PttCesA2(AAT09895)、PtrCesA4(AAO25536)、PtrCesA6(AAP40636).次生壁有关基因有PtCesA1(AAX18647)、PtCesA2(AAX18648、PtCesA3(AAX18649)、PtrCesA2(AAM26299)、PtrCesA7(AAO25581)、PtrCesA3(AAQ08987)、PcaCesA1(AAC78476)、PttCesA1(AAT09894)、PttCesA3-1(AAT09896)、PttCesA3-2(AAT09897)、AtCesA4(Q84JA6)、AtCesA8(Q8LPK5)、PtoCesA1(AAY21910).从图5可以看出ClCesA分布在次生壁较密集的分支,且与杉木ClCesA1在一个小分支,说明两者的亲缘关系较近,可能执行类似的功能,由此推测ClCesA基因可能与次生壁合成有关.

图1 杉木幼苗总RNA琼脂糖凝胶电泳Fig.1 Agarose gel of total RNA extracted from seedling of Chinese fir
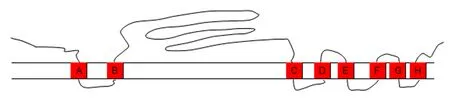
图4 杉木ClCesA蛋白氨基酸序列跨膜区域预测Fig.4 Prediction on trasmembrane domains of amino acids of ClCesA
2.5ClCesA基因在不同组织的表达
杉木纤维素合酶基因在不同器官表达情况的分析结果(图6)显示,杉木的根、茎、叶3个器官均有纤维素合酶基因表达,而且相对表达量在杉木根中最高,茎中次之,叶中最低.因此,可以推测ClCesA基因与杉木木材生长发育的调控有着密切关系.
圆圈代表与次生细胞壁有关的基因;三角形代表与初生细胞壁有关的基因.

图6 不同器官中ClCesA基因的相对表达量Fig.6 Relative expressions of ClCesA gene in different organs
2.6 植物过表达载体的构建
将检测正确的入门载体pDONR207-ClCesA和目的载体pGWB506进行LR反应,获得植物表达载体pGWB506-35S-ClCesA-GFP,载体构建策略如图7所示.使用特异性的载体引物和基因特异性引物做菌落PCR,扩增出1 000 bp的片段(图8),表明LR反应成功.重组质粒的测序结果显示,ClCesA并未产生突变,并且和原序列相同,说明本试验的植物表达载体连接正确.
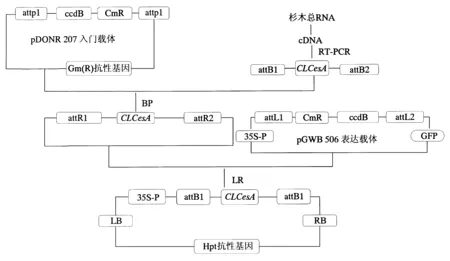
图7 植物表达载体pGWB-35S-ClCesA-GFP的构建策略Fig.7 Construction strategy of vector pGWB-35S-ClCesA-GFP
3 讨论
图8 菌落PCR反应Fig.8 Colony PCR reaction
通过序列比对与分析,Pear et al[22]首先从棉花中克隆得到与细菌CelA基因同源的纤维素合酶基因GhCesA1、2.通过基因组学分析研究,Richmond et al[23]得到了拟南芥基因组中的10个CesA基因的蛋白质序列结构特点.Suzuki et al[17]分析了杨树基因组的18个CesA基因的结构特点.目前为止,研究发现CesA基因编码的蛋白质有着相类似的结构,共有8个跨膜结构域, N端有2个, C端有6个.1个锌指结构域在N端,可能和蛋白质之间的相互作用有关.CesA蛋白都含有较典型的结构如DDDQxxRW(天冬氨酸、天冬氨酸、天冬氨酸、谷氨酞胺-x-x精氨酸—色氨酸)[24].通过对CesA基因功能的研究,已陆续鉴定出与初生壁和次生壁形成相关的基因.在欧洲颤杨中,PtrCesA1、PtrCesA2、PtrCesA3可能形成一个纤维素合成酶复合体,共同完成杨树次生壁的合成[15,24],而PtrcesA4、PtrCesA5、PtrCesA7可能与初生壁的形成有关[16].本研究克隆到一个新杉木ClCesA基因,通过系统进化树,该基因与聚类在一个进化分支并在根中表达量最高,推测该基因可能参与植物次生细胞壁的形成.而庞景等克隆到的2个CesA基因,ClcesAl与PtrCesAl的同源性最高;ClCesA2与ZmCesA2聚类在一起,两者在茎中高度表达.本研究中所用材料为培养30 d左右的杉木种子萌发而成的幼苗,而庞景所用的杉木材料为1年生杉木幼苗,且纤维素合酶CesA在植物不同发育阶段、不同器官中的表达量不同,所以这可能是造成ClCesA在杉木器官中表达量不同的原因.植物中除了CesA基因外,还有类纤维素合酶(cellulose synthase-like, CSL)基因家族参与纤维素的生物合成.拟南芥CSL蛋白有9个家族,分别为CSL(A/B/C/D/E/F/G/H/J)[25];在基因序列上,CSL基因与CesA基因具有部分同源,主要参与各种β-聚糖链的合成[23,25].彭莎莎等[21]也从杉木中克隆到了CSL基因,并分析其序列结构特点.
[1] KUMAR M, TURNER S. Plant cellulose synthesis: CESA proteins crossing kingdoms[J]. Phytochemistry, 2015,112:91-99.
[2] KAUR S, DHUGGA K, GILl K. Novel structural and functional motifs in cellulose synthase (CesA) genes of bread wheat (TriticumaestivumL.)[J]. PLoS ONE, 2016,11(1):e0147046.
[3] DESPREZ T, JURANIEC M, CROWELLE E F. Organization of cellulose synthase complexes involved in primary cell wall synthesis inArabidopsisthaliana[J]. Proceedings of the National Academy of Sciences, 2007,104(39):15 572-15 577.
[4] TAYLOR N G. Cellulose biosynthesis and deposition in higher plants[J]. New Phytologist, 2008,178(2):239-252.
[5] TANAKA K, MURATA K, YAMAZAKI M. Three distinct rice cellulose synthase catalytic subunit genes required for cellulose synthesis in the secondary wall[J]. Plant Physiology, 2003,133(1):73-83.
[6] WU L, JOSHI C P, CHIANG V L. A xylem-specific cellulose synthase gene from aspen (Populustremuloides) is responsive to mechanical stress[J]. The Plant Journal, 2000,22(6):495-502.
[7] DJERBIJ S, LINDSKOG M, ARVESTAD L. The genome sequence of black cottonwood (Populustrichocarpa) reveals 18 conserved cellulose synthase (CesA) genes[J]. Planta, 2005,221(5):739-746.
[8] 许雷,刘一星,方连玉.大青杨纤维素合成酶PuCesA6基因cDNA的克隆及序列分析[J].西南林业大学学报,2012,32(5):26-32.
[9] KRAUSKOPF E, HARRIS P J, PUTTERILL J. The cellulose synthase genePrCESA10 is involved in cellulose biosynthesis in developing tracheids of the gymnospermPinusradiata[J]. Gene, 2005,350(2):107-116.
[10] 刘昱翔,陈建荣,彭彦,等.苎麻纤维素合成酶基因BnCesA4 cDNA序列的克隆与表达分析[J].作物研究,2014,28(5):472-478.
[11] RANIK M, MYBURG A A. Six new cellulose synthase genes fromEucalyptusare associated with primary and secondary cell wall biosynthesis[J]. Tree Physiology, 2006,26(5):545-556.
[12] 阮维程,潘婷,季孔庶.马尾松纤维素合成酶基因PmCesA1的克隆及其分析[J].分子植物育种,2015,13(4):861-870.
[13] 庞景,童再康,黄华宏,等.杉木纤维素合成酶基因CesA的克隆及表达分析[J].浙江农林大学学报,2015,32(1):40-46.
[14] 邓小波,胡尚连,曹颖,等.绿竹与毛竹纤维素合成酶(CesA)的生物信息学分析[J].福建林学院学报,2011,31(1):84-90.
[15] SAMUGA A, JOSHI C P. Cloning and characterization of cellulose synthase-like gene,PtrCSLD2 from developing xylem of aspen trees[J]. Physiologia Plantarum, 2004,120(4):631-641.
[16] KALLURI U C, JOSHI C P. Differential expression patterns of two cellulose synthase genes are associated with primary and secondary cell wall development in aspen trees[J]. Planta, 2004,220(1):47-55.
[17] SUZUKI S, LI L, SUN Y H. The cellulose synthase gene superfamily and biochemical functions of xylem-specific cellulose synthase-like genes inpopulustrichocarpa[J]. Plant Physiology, 2006,142(3):1 233-1 245.
[18] SONG D, SHEN J, LI L. Characterization of cellulose synthase complexes inPopulusxylem differentiation[J]. New Phytologist, 2010,187(3):777-790.
[19] 蒋向辉,佘朝文,许栋,等.杉木CCoAOMT基因部分cDNA克隆与序列分析[J].中南林业科技大学学报,2009,29(6):24-28.
[20] 吕运舟,郑佳,陈金慧,等.参与杉木次生壁合成调控的转录因子ClMYB4的克隆及在大肠杆菌中表达[J].分子植物育种,2012,10(5):512-519.
[21] 彭沙沙,童再康,黄华宏,等.杉木纤维素合成酶类似蛋白基因ClCslD1的克隆及其生物信息学分析[J].浙江农林大学学报,2012,29(1):1-6.
[22] PEAR JR, KAWAGOE Y, SCHRECKENGOET W E. Higher plants contain homologs of the bacterialcelAgenes encoding the catalytic subunit of cellulose synthase[J]. Proceedings of the National Academy of Sciences, 1996,93(22):12 637-12 642.
[23] RICHMOND T A, SOMERVILLE C R. The cellulose synthase superfamily[J]. Plant Physiology, 2000,124(2):495-498.
[24] SAMUGA A, JOSHI CP. A new cellulose synthase gene (PtrCesA2) from aspen xylem is orthologous toArabidopsisAtCesA7 (irx3) gene associated with secondary cell wall synthesis[J]. Gene, 2002,296(1):37-44.
[25] LIEPMAN A H, CAVALIER D M. The cellulose synthase-like A and cellulose synthase-like C families: recent advances and future perspectives[J]. Front Plant Sci, 2012,3(109):1-7.
(责任编辑:叶济蓉)
Cloning of a newClCesAgene ofCunninghamialanceolataand plant expression vector construction
WEI Xiaoxiao1,2, WANG Shiya1,2, CHEN Xiaoxiao1,2, WANG Fenglin1,2, CAO Guangqiu1,2, CAO Shijiang1,2
(1.College of Forestry, Fujian Agriculture and Forestry University; 2.Chinese Fir Engineering, Technology Research Center under State Forestry Administration, Fuzhou, Fujian 350002, China)
In order to explore the molecular mechanism of Chinese fir wood formation, total RNA was extracted from Chinese fir new leaves and cDNA was synthesized. A newClCesAwas cloned using RT-PCR. The length of open reading frame ofClCesAis 2 955 bp, with 984 amino acids. Based on TMHMM Server v. 2.0 and MEGA5.1 software,ClCesAproteins consist of 8 across-membrane structures, with average 18 amino acid residues and zinc finger in N end. Also, a conservative DDDQXXLRW structure domain was found inClCesAprotein. Phylogenetic tree analysis showed thatClCesAmay be associated with secondary cell wall formation. The fluorescent quantitative PCR analysis showed thatClCesAcan be expressed in organs of root, stem and leaf in Chinese fir, with the highest expression in the root and the least in leaf. Lastly, a plant overexpression vector pGWB506-35s-ClCesA-GFP was successfully constructed from pGWB506 vector which contains GFP tag and hygromycin resistance gene by Gateway technology.
Cunninghamialanceolata; cellulose synthase; gene cloning; expression analysis; vctor construction
2016-07-03
2016-09-20
福建农林大学校重点建设项目(6112C039B);福建农林大学林学院青年基金项目(k13xjj02a).
魏晓骁(1990-),女,硕士研究生.研究方向:森林培育理论与技术.Email:695194330@qq.com.通讯作者曹世江(1984-),男,讲师,博士.研究方向:树木遗传育种教学与科研.Email:379612064@qq.com.
Q785; S791.27
A
1671-5470(2017)01-0066-07
10.13323/j.cnki.j.fafu(nat.sci.).2017.01.011
